激光显微切割技术富集肿瘤细胞在基因突变检测中的意义
2014-04-20董周寰吕亚莉汪进良朱凤伟石怀银
董周寰,吕亚莉,汪进良,钟 梅,朱凤伟,王 琼,石怀银
解放军总医院 病理科,北京 100853
激光显微切割技术富集肿瘤细胞在基因突变检测中的意义
董周寰,吕亚莉,汪进良,钟 梅,朱凤伟,王 琼,石怀银
解放军总医院 病理科,北京 100853
目的探讨激光显微切割技术富集肿瘤细胞对检测非小细胞肺癌(non-small cell lung cancer,NSCLC)表皮生长因子受体(epidermal growth factor receptor,EGFR)基因突变检测的影响以及其临床病理学意义。方法采用激光显微切割技术富集49例NSCLC患者的石蜡包埋样本的肿瘤细胞,采用PCR技术对EGFR基因第18、19、20、21号外显子片段进行扩增后直接测序,分析其结果并比较其与常规肿瘤组织筛选后进行EGFR基因突变检测结果的异同。结果49例NSCLC样本中21例(42.9%)存在EGFR基因突变,包括第19号外显子13例,第21号外显子8例,没有发现两者同时突变的样本。分析外显子突变情况发现,利用激光显微切割技术富集的样本检测突变率42.9%(21/49),高于利用常规筛选后的突变率34.7%(17/49),两种方法的差异有统计学意义(P = 0.045 5<0.05)。且比较两种方法,可以发现前者检测得到的突变峰峰高明显高于后者。结论利用激光显微切割技术富集肿瘤细胞能有效去除间质细胞对肿瘤细胞的基因组干扰,更能体现肿瘤细胞基因组状态,有效提高EGFR基因突变检测检出率,有利于患者靶向治疗的临床筛选。
激光显微切割;非小细胞肺癌;表皮生长因子;突变
基因突变检测表皮生长因子受体(epidermal growth factor receptor,EGFR)突变阳性的非小细胞肺癌(non-small cell lung cancer,NSCLC)患者,酪氨酸激酶抑制剂(tyrosine kinase inhibitor,TKI)类药物对其有效率>70%而野生型<5%,使用此类靶向药物治疗能有效延长患者的生存期[1]。目前常用的基因突变检测方法如直接测序法、基于Realtime PCR的检测法等[2]。直接测序法可以检测所有的突变,但灵敏度不高;基于Real-time PCR基础上的检测方法灵敏度高,但只能检测已知突变[3]。目前临床送检组织样本60%为穿刺样本,样本中存在含量很高的间质细胞、癌旁坏死细胞,使用常规肿瘤组织筛选(划块)很难去除。20世纪90年代中期发展起来的激光捕获显微切割技术(laser capture microdissection,LCM)能够快速准确地从复杂组织中分离均一的样品组分,可在显微镜下准确捕捉肿瘤细胞,利用激光对捕捉区域进行切割,在不破坏细胞结构、不改变组织的分子学特性的前提下富集复合组织中的肿瘤细胞,取得纯净的肿瘤细胞基因组进行突变检测,基本剔除间质及坏死细胞基因组对肿瘤细胞基因组的污染和稀释,消除因实验选用标本所造成的偏倚[4]。本实验旨在比较常规肿瘤组织筛选方法与激光显微切割技术筛选对穿刺样本检测结果的影响,进一步探索和建立更加快速准确的基因突变检测方法,为进一步使用和规范该技术提供参考依据。
材料和方法
1 材料 收集本院2012年3 - 7月经病理确诊为NSCLC的穿刺组织石蜡包埋样本49例,其中女性30例,男性19例,年龄46 ~ 79岁,中位年龄58岁,全部为腺癌样本。同批次样本在本次实验前均已利用常规方法提取样本基因组检测EGFR基因突变并得到结果。
2 方法 切片:每例标本切取覆膜切片4张,厚度6 μm。切片前,切片台用二甲苯和酒精擦拭,切片采用一次性切片刀,并用酒精清洁。切片时采用1个刀锋对1个组织块的方法,防止不同病例组织样本间的交叉污染。染色:采用HE染色,并且对后续的扩增测序反应基本无影响。富集肿瘤细胞:高年资病理科医生在显微镜下对组织切片进行甄别,用仪器圈出所需肿瘤细胞,利用激光切割(图1)进行收集。DNA提取:运用Qiagen公司的DNA提取纯化试剂盒(QIAamp DNA FFPE Tissue Kit)从石蜡组织中提取基因组DNA(具体操作步骤见试剂盒说明书),将提取的基因组DNA用紫外分光光度仪测定样本纯度和浓度,A260/ A280为1.7 ~ 2.0,A260/A230>1.7。PCR扩增:对所提取的样本DNA的EGFR基因(18、19、20、21号外显子)进行扩增。引物序列为:18号外显子上游引物:5’-CAAATGAGCTGGCAAGTGCCGT GTC-3’,下游引物:5’-GAGTTTCCCAAACACTC AGTGAAAC-3’;19号外显子上游引物:5’-GCAAT ATCAGCCTTAGGTGCGGCTC-3’,下游引物:5’-CATAGAAAGTGAACATTTAGGATGTG-3’;20号外显子上游引物:5’-CCATGAGTACGTATTTTGAAA CTC-3’,下游引物:5’-CATATCCCCATGGCAAA CTCTTGC-3’;21号外显子上游引物:5’-CTAA CGTTCGCCAGCCATAAGTCC-3’,下游引物:5’-G CTGCGAGCTCACCCAGAATGTCTGG-3’。序列测定:对PCR扩增产物进行割胶纯化,用ABI 3730XL测序仪进行双向测序,得到序列峰图后,利用Chromas软件进行基因序列的突变分析。
3 统计学分析 采用SPSS16.0软件进行统计分析,两个样本率之间差异比较用χ2检验,P<0.05为差异有统计学意义。
结 果
1 基因检测总体情况 利用激光显微切割技术富集肿瘤细胞提取基因组DNA,采用PCR技术扩增及直接测序法检测的49例样本中,总突变率为42.9%(21/49),集中于19号外显子以及21号外显子,突变率分别为26.5%(13/49)和16.3%(8/49),21号外显子为L858R密码子点突变,19号外显子为第746 ~ 753位密码子碱基缺失。未发现两者同时突变的样本。

图 1 显微激光切割收集样本前后对比 A: 激光显微切割前样本;B: 激光显微切割后样本Fig. 1 Samples before laser microdissection (A) and after laser microdissection (B)
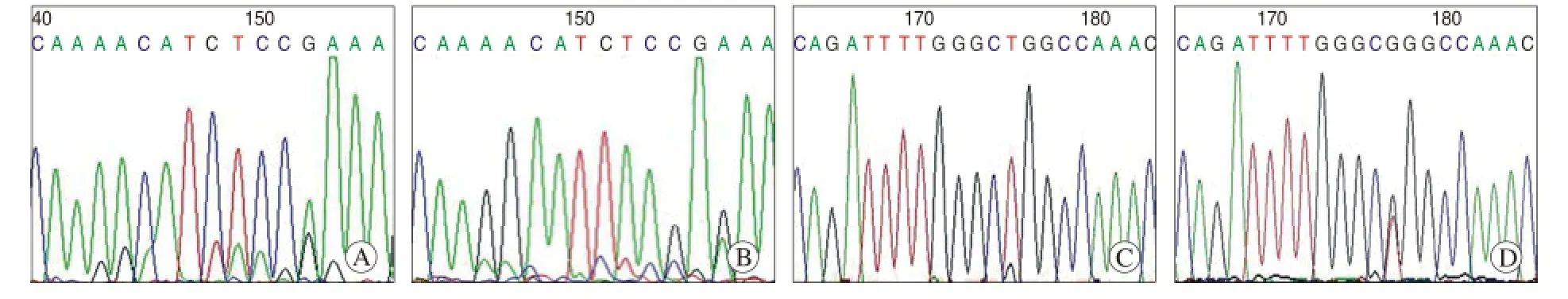
图 2 直接测序法检测样本序列峰图 A:常规方法收集样本后直接测序法测序,19外显子峰图; B:激光显微切割收集样本后直接测序法测序,19外显子峰图; C:常规方法收集样本后直接测序法测序,21外显子峰图; D:激光显微切割收集样本后直接测序法测序,21外显子峰图Fig. 2 Direct sequencing showing sequence peak in samples A: Direct sequencing showing exon 19 mutation peak in conventional sample; B: Direct sequencing showing exon 19 mutation peak after laser microdissection; C: Direct sequencing showing exon 21 mutation peak in conventional sample; D: Direct sequencing showing exon 21 mutation peak after laser microdissection
2 激光显微切割与常规划块收集样本检测突变比较 利用激光显微切割技术富集肿瘤细胞的突变率42.9%(21/49)高于同批次利用常规肿瘤组织筛选样本的突变率34.7%(17/49),两种方法的差异有统计学意义(P = 0.045 5<0.05),一致性检验有统计学意义,Kappa = 0.829 3,一致率为91.8%。且前者得到的突变峰峰高明显高于后者;在常规肿瘤组织筛选样本的测序峰图中无法判断是否存在突变峰,但在激光显微切割技术收集样本的测序峰图中,可以明确EGFR基因21号外显子发生了点突变;常规肿瘤组织筛选样本的测序峰图主峰为EGFR基因19号外显子正常序列峰图,而激光显微切割技术收集的测序峰图的主峰却为EGFR基因19号外显子突变序列峰图(图2)。
讨 论
EGFR基因的酪氨酸激酶编码区突变,是NSCLC治疗使用吉非替尼等TKI类靶向药物的重要依据和预测指标[5]。最常见的突变位点包括19号外显子碱基缺失导致受体ATP结合囊的角度的改变,以及21号外显子的L858R突变导致的A-loop稳定性提高,从而显著增强癌细胞对吉非替尼以及厄洛替尼的敏感性[6-7]。
目前,临床送检EGFR样本60%为穿刺样本,文献表明,活检和穿刺标本的EGFR突变率比手术切除标本的突变率低[8-9]。研究显示随着样本中肿瘤细胞含量的减少,EGFR突变检出率有逐渐下降的趋势。分析原因可能是因为检测技术的灵敏度不高,以及取材局限,不能完全剔除间质及坏死细胞对肿瘤细胞基因组的干扰,客观上稀释了突变细胞的含量,导致测序结果假阴性。
国外文献报道,利用激光显微切割技术进行肿瘤相关研究,用于分离肿瘤细胞和间质细胞,进行癌细胞的基因表达和突变分析[10-12]。利用激光显微切割技术,可将不同成分、不同数量的间质细胞去除,获得常规的病理取材很难取得的纯净的肿瘤细胞的DNA、RNA或者蛋白质。
实验表明,利用激光显微切割技术收集样本检测出突变率42.9%(21/49)明显高于同批次样本常规肿瘤组织筛选后检出的突变率34.7%(17/49)(P = 0.045 5<0.05)。证实了样本中的非肿瘤细胞的基因组对检测存在干扰。而无论使用直接测序法还是基于优化Real-time PCR技术的检测方法均有局限性,或敏感性低,或不能检测出非常见突变位点和未知突变类型[8,13]。利用激光显微切割技术富集肿瘤细胞联合直接测序的方法,在不破坏细胞结构、不改变组织的分子学特性的前提下富集复合组织中的肿瘤细胞,取得相对纯净的基因组进行突变检测,基本剔除间质及坏死细胞基因组对肿瘤细胞基因组污染和稀释。本实验证明,使用激光显微切割收集的肿瘤细胞,具有明显的优势,能够显示肿瘤细胞基因组突变的实际情况,有效提高EGFR检测的阳性检出率。
根据本实验获得的结果,我们发现利用激光显微切割技术可以从选材精确富集肿瘤细胞,使检测结果显示为肿瘤细胞基因组的实际情况,精确定量肿瘤细胞的基因突变以及表达情况。临床医师根据检测结果选择治疗方案时,可使患者能够最大限度地获益,值得在临床病理科推广。
1 Paez JG, Jänne PA, Lee JC, et al. EGFR mutations in lung Cancer:correlation with clinical response to gefitinib therapy[J]. Science,2004, 304(5676): 1497-1500.
2 Angulo B, Conde E, Suárez-Gauthier A, et al. A comparison of EGFR mutation testing methods in lung carcinoma: direct sequencing, real-time PCR and immunohistochemistry[J]. PLoS One, 2012, 7(8): e43842.
3 Molina-Vila MA, Bertran-Alamillo J, Reguart N, et al. A sensitive method for detecting EGFR mutations in non-small cell lung Cancer samples with few tumor cells[J]. J Thorac Oncol, 2008, 3(11):1224-1235.
4 Emmert-Buck MR, Bonner RF, Smith PD, et al. Laser capture microdissection[J]. Science, 1996, 274(5289):998-1001.
5 Sholl LM, Xiao Y, Joshi V, et al. EGFR mutation is a better predictor of response to tyrosine kinase inhibitors in non-small cell lung carcinoma than FISH, CISH, and immunohistochemistry[J]. Am J Clin Pathol, 2010, 133(6): 922-934.
6 董强刚,黄进肃,黄建,等.肺癌靶向治疗研究进展与我国肺癌的EGFR基因突变概况[J].肿瘤,2005,25(6):625-628.
7 D’angelo SP, Pietanza MC, Johnson ML, et al. Incidence of EGFR exon 19 deletions and L858R in tumor specimens from men and cigarette smokers with lung adenocarcinomas[J]. J Clin Oncol,2011, 29(15): 2066-2070.
8 冯勤,李向红,陈钊,等.非小细胞肺癌表皮生长因子受体基因突变检测及其与临床病理特征的相关性[J] .中华病理学杂志,2011,40(10):660-663.
9 孙孟红,杨飞,沈磊,等.非小细胞肺癌中表皮生长因子受体基因突变直接测序分析及其与临床病理特征的相关性[J].中华病理学杂志,2011,40(10):655-659.
10 Toruner GA, Ulger C, Alkan M, et al. Association between gene expression profile and tumor invasion in oral squamous cell carcinoma[J]. Cancer Genet Cytogenet, 2004, 154(1): 27-35.
11 Patel V, Leethanakul C, Gutkind JS. New approaches to the understanding of the molecular basis of oral Cancer[J]. Crit Rev Oral Biol Med, 2001, 12(1): 55-63.
12 Kumar S, Fend F, Quintanilla-Martinez L, et al. Epstein-Barr viruspositive primary gastrointestinal Hodgkin’s disease: association with inflammatory bowel disease and immunosuppression[J]. Am J Surg Pathol, 2000, 24(1): 66-73.
13 Pao W, Ladanyi M. Epidermal growth factor receptor mutation testing in lung Cancer: searching for the ideal method[J]. Clin Cancer Res, 2007, 13(17): 4954-4955.
Significance of laser microdissection-enriched tumor cells in detection of gene mutation
DONG Zhou-huan, LYU Ya-li, WANG Jin-liang, ZHONG Mei, ZHU Feng-wei, WANG Qiong, SHI Huai-yin
Department of Pathology, Chinese PLA General Hospital, Beijing 100853, China
Corresponding author: SHI Huai-yin. Email: shihuaiyin@sina.com
ObjectiveTo study the effect of laser microdissection-enriched tumor cells on detection of epidermal growth factor receptor (EGFR) gene mutation in non-small cell lung cancer (NSCLC) and its clinicopathological significance.MethodsParaffinembedded tumor cells from 49 NSCLC patients were enriched by laser microdissection. EGFR gene fragments from exon 18 to 21 were amplif i ed by PCR and sequenced. The EGFR gene mutations in NSCLC were compared with those in other cancers.ResultsOf the 49 NSCLC samples, 21 (42.9%) had EGFR gene mutations including exon 19 mutation in 13 and exon 21 mutation in 8. However, both exon 19 and exon 21 mutations were not detected. The analysis of exon mutations showed that the mutation rate was higher in laser microdissection-enriched samples than in conventional tumor tissue samples (42.9% vs 34.7%, P<0.05). The mutation peak was significantly higher in the former than in the latter.ConclusionLaser microdissection-enriched tumor cells can effectively prevent the intervention of tumor cell genome against interstitial cells, ref l ect the status of tumor cells genome, effectively improve the detection rate of EGFR gene mutations, thus contributing to the clinical screening of target therapy for NSCLC patients.
laser microdissection; non-small cell lung cancer; epidermal growth factor; mutation
R 34-33
A
2095-5227(2014)06-0614-03
10.3969/j.issn.2095-5227.2014.06.027
2014-03-07 10:56
http://www.cnki.net/kcms/detail/11.3275.R.20140307.1056.001.html
2014-01-07
国家自然科学基金项目(81202965)
Supported by the National Natural Science Foundation of China(81202965)
董周寰,男,技师。Email: dzh_8743@163.com
石怀银,男,硕士,主任医师。Email: shihuaiyin@sina. com
