新改良PCR/LDR/毛细管电泳技术产前诊断胎儿β-地中海贫血的可行性研究
2014-04-01廖茜易萍郑英如俞丽丽钟小林黄寅虎韩磊朱玉娟李力
廖茜,易萍,郑英如,俞丽丽,钟小林,黄寅虎,韩磊,朱玉娟,李力
传统获取胎儿遗传信息进行的产前诊断技术为有创性操作[1-4],对胎儿及母体均存在潜在的损伤,以致其临床推广及应用受到一定程度的限制[5]。自Lo等[6]发现母体循环中存在游离胎儿DNA(cell-free fetal nucleic acids,cffDNA)后,利用孕妇外周血中的cffDNA进行无创产前诊断(noninvasive prenatal diagnosis,NIPD)取得了飞跃性的进展。但胎儿DNA在孕妇血浆总DNA中所占比例甚微,其检测受到较大限制。点突变检测是目前DNA分子诊断的主要研究内容,但其技术要求高,尤其是低丰度DNA检测技术要求更甚,故需探寻一种高灵敏度和高特异性的方法。
我们在前期实验中以雌激素受体α的单核苷酸多态性(single nucleotide polymorphism,SNP)位点(c.454-397T>C)、β-地中海贫血常见的2种单核苷酸突变[IVS-Ⅱ-654(C→T)、CD17(A→T)]作为研究对象,利用聚合酶链反应(PCR)/连接酶检测反应(LDR)结合毛细管电泳技术对血浆胎儿DNA实验模型进行检测,发现该技术对低丰度基因突变的检测灵敏度可达到1:10 000,初步证明其可满足母体血浆胎儿DNA点突变检测的要求[7]。但该方法需纯化PCR产物,然后将纯化的产物用于下一步的LDR反应,纯化过程中可能出现污染,影响实验结果。因此,我们在前期实验基础上进行了改良,拟直接将PCR产物应用于LDR检测,以达到简化步骤、减少污染的目的。本研究以β-地中海贫血CD17(A→T)突变作为研究对象,探索LDR技术结合毛细管电泳检测母体血浆中胎儿父系DNA点突变的可行性,以期为胎儿单基因遗传性疾病的无创产前诊断寻求一种新的方法。
1 材料与方法
1.1 材料
1.1.1 血液标本 β-地中海贫血外周血标本:由大坪医院检验科提供,EDTA抗凝。正常人及正常孕妇外周血标本:经本人同意并签署知情同意书后抽取,EDTA抗凝。
1.1.2 主要试剂及来源 普通寡核苷酸引物、LDR寡核苷酸引物由大连宝生物(TaKaRa)公司合成;荧光定量PCR相关引物及探针由上海生工合成;PCR试剂盒购自大连宝生物(TaKaRa)公司;DNA连接酶购自美国NEB公司;全血基因组DNA提取试剂盒购于Qiagen公司。
1.2 全血DNA提取 采用全血基因组DNA提取试剂盒提取全血DNA,操作步骤按说明书进行,置-20℃冰箱内备用。PCR引物:上游5'-ACTAGCAACCTCAAACAGACACCA-3',下游5'-TCAGTGCCTATCAGAAACCCAAG-3'[7]。
1.3 PCR扩增及产物电泳 扩增体系包括PCR master mix 25μl,正、反向引物(10μmol/μl)各2μl,突变型外周血DNA 20pg,正常人外周血DNA(分别为100、50、10、1ng),ddH2O补足至50μl;另设阴性对照组,只加入正常人外周血DNA。反应条件:95℃ 5min;94℃ 30s、57℃ 30s、72℃ 30s,循环45次;72℃ 5min。PCR扩增产物行1.5%琼脂糖凝胶电泳(120V,30min),凝胶成像扫描系统下观察结果并进行图像分析。
1.4 LDR反应 LDR上游引物序列为5'-GCCGTTACTGCCCTGTGGGGCT-3',5'端6-FAM标记;下游引物序列为5'-AGGTGAACGTGGATGAAGTTGGT GGTG-3',5'端磷酸化修饰、3'端C3标记[7]。PCR产物置于95℃环境中15min,缓慢冷却至4℃备用。反应体系为:10×Buffer 2μl,LDR上、下游引物(5μmol)各0.1μl,DNA连接酶8U,PCR产物5μl,ddH2O补足至20μl。95℃变性2min;94℃ 30s、65℃ 1min,循环20次;95℃ 10min。连接产物由重庆医科大学感染分子生物中心进行检测。(1)纯化:①取8μl LDR产物加入PCR管中,加入1μl 3mol/L醋酸钠(pH5.2)和1μl 0.125mol/L EDTA,混匀后,加入20μl无水乙醇混匀,室温放置15min;②3000g离心30min,倒置PCR管,去除上清;③加入30μl 70%乙醇,轻微震荡后,1600g离心15min,倒置PCR管,去除上清;④重复步骤③;⑤在真空浓缩仪中,干燥沉淀。(2)变性:加入10μl Hi-Di甲酰胺和0.1μl GeneScanTM-500Liz®分子量内标,95℃孵育5min,冰上放置5min。(3)上机:对PCR板相应位置的样品进行编号,在室温下,于POP-6胶(ABI)、1×电泳缓冲液(ABI)中电泳,应用CEQ8000型遗传分析仪,ABI片段分析软件GeneMapper V3.5对结果进行分析。
1.5 孕妇血浆胎儿基因的检测 血浆制备:采孕妇新鲜外周血5ml,EDTA-K2抗凝,1600g离心10min,取上清,16 000g再次离心10min,取上清,-20℃保存备用。血浆DNA的提取:采用QIAamp Blood DNA midi kit提取孕妇血浆DNA,60μl洗脱液溶解过滤,将回收液再次加入吸附柱中过滤,最终所得DNA -20℃保存备用。限制性酶切:每25μl DNA加20U限制性内切酶Hinp1 Ⅰ和20U Hha Ⅰ (NEB,USA),37℃水浴16h,65℃水浴20min。实时定量PCR:以RASSF1A为靶点检测胎儿基因,扩增体系为缓冲液(含Mg2+)5μl,dNTP 4μl,TaqMan探针100μmol,Hot Start Version Taq(TaKaRa)300μmol,上、下游引物(10μmol)各1.4μl,酶切后DNA 20μl,ddH2O补足至50μl,离心混匀。50℃ 2min,95℃ 10min;95℃ 15s、62℃ 30s、72℃30s,循环50次。以β-actin基因作为内参照[8]。
1.6 孕妇血浆模型的建立及检测 正常孕妇血浆DNA中加入20pg CD17(A→T)突变杂合子外周血DNA后进行扩增,扩增产物电泳及LDR方法同前所述,以正常孕妇血浆DNA作为阴性对照。
2 结 果
2.1 测序结果 利用AB片段反向引物测序,CD17(A→T)突变的AB片段测序结果显示,第35位碱基(方框内)处有2个峰(T峰、A峰),说明此处为CD17(A→T)突变的杂合子(图1A)。利用CD片段反向引物测序,无CD17(A→T)突变的正常CD片段的测序结果显示,第41位碱基(方框内)处只有一个峰(A峰),无G峰,说明此处无CD17(A→T)突变(图1B)。

图1 CD17(A→T)突变的AB片段及正常AB片段测序结果(反向引物测序)Fig.1 Sequencing of CD17 (A→T) mutation of AB fragment (A) and normal AB fragment (B) (reverse primer sequence)
2.2 LDR检测 以含有CD17(A→T)突变的PCR扩增产物作为LDR模板,采用8U DNA连接酶进行连接反应,采用CEQ8000型遗传分析仪分析LDR产物。结果显示,LDR检测灵敏度可达1:5000,产物峰的面积随着正常人外周血DNA浓度的增加而减少,至1:10 000处不能与杂峰区分,且无CD17(A→T)突变扩增产物的阴性对照未检测到LDR产物(图2)。
2.3 孕妇血浆胎儿基因检测 将酶切前后的孕妇血浆DNA进行实时定量PCR检测(图3),可见RASSF1A基因酶切前Ct值为30.33,酶切后Ct值为32.61,以β-actin基因作为对照,其酶切前Ct值为30.33,酶切后未检测出荧光。高甲基化RASSF1A可代表胎儿游离DNA,せ SSF1A基因可作为阳性质控。
2.4 孕妇血浆模型LDR产物检测结果 正常孕妇血浆DNA中加入20pg CD17(A→T)突变杂合子外周血DNA扩增产物,LDR产物峰可明显与杂峰区分(图4)。

图2 LDR产物分析图Fig.2 Analysis of LDR productsA-D represent the LDR products with the different concentrations of normal peripheral blood DNA mixed with 20pg CD 17 (A→T) mutant hybrid DNA as template for PCR amplification, corresponding sensitivities are 1:100, 1:1000, 1:5000, 1:10000; E and F show the negative control.They are the electrophoresis results by genetic analyzer CEQ8000, abscissa indicates the length of fragment, ordinate indicates the intensity of fluorescence, arrows show connection product
3 讨 论
Lo等[5]研究表明,母体血浆中胎儿DNA浓度占血浆总DNA浓度的0.4%~11.9%,检测过程中母体等位DNA片段可干扰胎儿DNA片段的检测,增加胎儿基因突变检测的难度及误诊机会[9]。近几年比较热门的无创产前诊断检测技术包括大规模平行基因组测序、应用鸟枪测序法通过计算父母的单倍体基因来测量胎儿的基因组[10]等,但上述方法往往因成本高、操作复杂、周期长、对外周血样本要求高等问题而难以广泛开展。近期还有研究使用高分辨熔解分析(HRM)方法,用于父源性已知基因点突变的检测,但其在妊娠早期不能完全与母体血浆DNA区分[11]。此外,反向斑点杂交也是目前的一种有效方法,可用于临床检测β-地中海贫血的基因突变,但对于β-地中海贫血的点突变仍有一定误诊率。有研究证明,单基因突变位点周围的单核苷酸多态性位点可直接影响检测结果的准确性,故应根据多态性位点设计特异性的寡核苷酸探针[12]。

图3 孕妇血浆DNA酶切前后的实时荧光定量分析曲线图Fig.3 Real-time fluorescence quantitative analysis curves of pregnant women plasma DNA before and after enzyme digestion2, 3 represent the RASSF1A gene in real-time fluorescence quantitative analysis before and after enzyme digestion. 1, 4 represent the β-actin gene in real-time fluorescence quantitative analysis before and after enzyme digestion
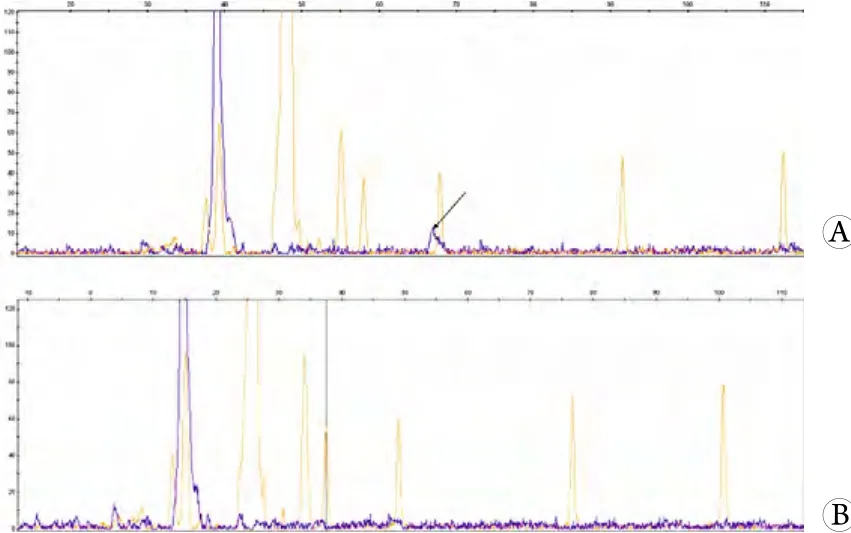
图4 孕妇血浆模型LDR产物检测分析图Fig.4 LDR products of pregnant women plasma modelA represent the LDR product which is the normal maternal plasma DNA mixed with 20pg CD 17 (A→T) heterozygous mutant DNA PCR amplification product as a template; B is a negative control. It is the electrophoresis results by genetic analyzer CEQ8000; Abscissa indicates the length of fragment, ordinate indicates fl uorescence intensity
本研究将PCR和LDR技术相结合,建立了一种可针对孕妇外周血DNA中的父源性β-地中海贫血单基因点突变进行检测的方法。将低浓度突变型外周血基因组DNA混入不同浓度梯度的正常人外周血基因组DNA,扩增后进行LDR反应,采用CEQ8000型遗传分析仪进行检测,结果发现PCR/LDR结合毛细管电泳技术可检测出50ng正常基因组DNA中含有的20pg β-地中海贫血CD17(C→T)杂合突变LDR产物,且产物峰面积随着正常模板浓度增加而降低,其基因突变检测的灵敏度可达1:5000。因此,将PCR产物直接用于LDR检测的灵敏度完全能够满足孕妇血浆中胎儿DNA的检测,从而可以简化步骤,减少纯化过程中导致污染的机会。而2006年发现的位于3号染色体的RASSF1A基因在胎儿DNA中表现为高甲基化,在母血细胞表现为低甲基化,通过这种不同甲基化形式的差异可检测出孕妇外周血浆DNA中的胎儿源性DNA[7,13]。故用甲基化敏感的限制性内切酶Hinp1 Ⅰ和Hha Ⅰ切掉非甲基化的母源性DNA,余下的即为胎儿源性DNA,将其作为下一步临床病例验证的阳性质控可降低假阴性率。在正常孕妇外周血血浆DNA中加入20pg β-地中海贫血CD17(C→T)杂合突变扩增产物,采用本研究建立的方法,LDR产物峰可明显与杂峰区分。
综上所述,PCR/LDR/毛细管电泳技术用于检测低丰度基因点突变具有很高的灵敏度,有望用于母体血浆中胎儿DNA点突变的检测,且该方法与目前常用的无创产前诊断技术相比具有操作简便、价格低廉、仅需一个工作日即可完成等优势。该方法理论上也可用于其他单基因点突变疾病的检测,但尚有待临床验证。
[1] Wang FY, Ma LX, Li CM, et al. Detecting aneuploidies of fetus by multiplex ligation-dependent probe amplification[J]. J Zhengzhou Univ (Med Sci), 2012, 47(5): 632-635. [王凤羽, 马林先, 李聪敏, 等. 多重连接探针扩增技术检测胎儿非整倍体异常[J]. 郑州大学学报(医学版), 2012, 47(5): 632-635.]
[2] Zhang HX, Ren CC, Liang YH, et al. The chromosome Karyotype analysis of chorionic tissue in early pregnancy of aged gravida[J]. Tianjin Med J, 2010, 38(7):622-624. [张海霞, 任晨春, 梁玥宏, 等. 高龄孕妇孕早期绒毛组织染色体核型分析[J]. 天津医药, 2010, 38(7): 622-624.]
[3] Wu QH, Shi HR, Lu N, et al. Genetic diagnosis of haemophilia B pedigrees and prenatal diagnosis of high-risk fetus by DNA sequencing[J]. J Zhengzhou Univ (Med Sci), 2012, 47(5): 639-642. [吴庆华, 史惠蓉, 鲁宁, 等. 血友病B 家系的基因测序与高危胎儿的产前诊断[J]. 郑州大学学报(医学版), 2012,47(5): 639-642.]
[4] Jiang SF, Gao ZY, Lu YP, et al. Clinical application of fluorescence in situ hybridization for prenatal diagnosis[J]. Med J Chin PLA,2012, 37(7): 711-714. [姜淑芳, 高志英, 卢彦平, 等. 荧光染色体原位杂交技术在产前诊断中的应用[J]. 解放军医学杂志,2012, 37(7): 711-714.]
[5] Li L, Yi P, Liao X, et al. Prenatal diagnosis and confusion[J].Chin Fam Plann Obstet, 2013, 5(4): 5-7. [李力, 易萍, 廖茜, 等.产前诊断及其困惑[J]. 中国计划生育和妇产科, 2013, 5(4):5-7.]
[6] Lo YM, Tein M S, Lau TK, et al. Quantitative analysis of fetal DNA in maternal plasma and serum: implications for noninvasive prenatal diagnosis[J]. Am J Hum Genet, 1998, 62(4): 768-775.
[7] Yi P, Chen Z, Yu L, et al. Prenatal detection of beta-thalassemia CD17 (A-->T) mutation by polymerase chain reaction/ligase detection reaction/capillary electrophoresis for fetal DNA in maternal plasma--a case report[J]. Fetal Diagn Ther, 2010,27(1): 25-31.
[8] Chan KC, Ding C, Gerovassili A, et al. Hypermethylated RASSF1A in maternal plasma: a universal fetal DNA marker that improves the reliability of noninvasive prenatal diagnosis[J].Clin Chem, 2006, 52(12): 2211-2218.
[9] Ding C, Chiu RW, Lau TK, et al. MS analysis of singlenucleotide differences in circulating nucleic acids: application to noninvasive prenatal diagnosis[J]. Proc Natl Acad Sci USA,2004, 101(29): 10762-10767.
[10] Fan HC, Gu W, Wang J, et al. Non-invasive prenatal measurement of the fetal genome[J]. Nature, 2012, 487(7407): 320-324.
[11] Hill M, Finning K, Martin P, et al. Non-invasive prenatal determination of fetal sex: translating research into clinical practice[J]. Clin Genet, 2011, 80(1): 68-75.
[12] Yenilmez ED, Tuli A, Evrüke IC. Noninvasive prenatal diagnosis experience in the Çukurova Region of Southern Turkey: detecting paternal mutations of sickle cell anemia and β-thalassemia in cell-free fetal DNA using high-resolution melting analysis[J]. Prenat Diagn, 2013, 33(11): 1054-1062.
[13] Bellido ML, Radpour R, Lapaire O, et al. MALDI-TOF mass array analysis of RASSF1A and SERPINB5 methylation patterns in human placenta and plasma[J]. Biol Reprod, 2010, 82(4):745-750.
