MiR-21靶基因预测和生物信息学分析
2014-03-25麦秀英
麦秀英, 徐 柳, 李 萍
(西南交通大学 生命科学与工程学院,成都610031)
微小RNA(microRNA,miRNA)是一类内生的非编码调控单链小分子RNA,长度约22~25个核苷酸,其通过与靶基因mRNA的3′非翻译区(3′untranslated region, 3′UTR)的不完全性互补配对,引起转录抑制或者下调,从而参与调节基因表达、生长发育和维持机体正常生理功能[1]。miRNA-21是肿瘤细胞中表达量上调最普遍的microRNA,其大多数靶基因都是抑癌基因[2]。通过功能抑制发现,miR-21几乎牵涉致癌的每个领域:促进细胞的增殖,浸润和转移,基因组不稳定和突变,炎症,复制永生化,代谢异常,血管生成,逃避细胞凋亡,免疫破坏和生长抑制[3]。因此研究miRNA在肿瘤中作用机制的关键是要准确找到其靶基因,并了解miRNA与其靶基因之间的相互作用。本研究运用生物信息学方法来预测miR-21的靶基因,并对其靶基因进行功能富集分析和信号通路富集分析,为深入研究miR-21在癌细胞中的调控机制做理论分析。
微小RNA(microRNA,miRNA)是近年来生物领域中最重要的发现之一,其调控体内多种重要的细胞进程包括细胞分化、增殖、凋亡和肿瘤的形成[3]。其中MiR-21是研究较多的miRNA,研究结果表明其多与肿瘤的发生有关,包括食管癌、乳腺癌、胶质瘤、肝癌及结肠癌等。结肠癌的发生及发展与miR-21及其靶基因有着密切的关系,敲低结肠癌HT-29细胞中miR-21会增强结肠癌细胞对放化疗的敏感性[4];在活性氧(ROS)/AMPK/mTOR/AP1/4E-BP1通路中,抑制糖醛还原酶(AR)的表达,会促使miR-21的表达水平下调,使其靶基因PDCD4的表达水平增加,从而阻止生长因子诱导的结肠癌的生长[5];而结肠癌中过表达miR-21会降低PDCD4蛋白量,刺激癌细胞入侵,内渗和转移[6]。这些均表明结肠癌的发生与miR-21的表达量之间有密切关系。因此通过生物信息学方法预测miR-21的靶基因及其靶基因可能参与的信号通路对研究miR-21在结肠癌发生中的功能及作用机制具有重要的生物学意义,也为寻找结肠癌的治疗靶点提供方向。
研究表明miR-21在多种癌症中均表达上调,但其靶基因多数只在一种癌症中被验证,只有少数几个靶基因同时在几种癌症中被发现,如Cdc25A、TGFβR2和Sprouty均只在结肠癌中证实为miR-21的靶基因,在其他癌症中是否为miR-21的靶基因还有待进一步验证;而PDCD4在结肠癌、乳腺癌和恶性胶质瘤中均证实为miR-21的靶基因,PTEN在结肠癌、肝癌和食管癌中均为miR-21的靶基因。因此,在线工具预测的miR-21的靶基因和其他癌症或疾病中miR-21的靶基因,是否为结肠癌中miR-21的靶基因还有待进一步实验验证。
1 材料与方法
1.1 材料
应用miRBase(http://www.mirbase.org/)、miRecords(http://mirecords.biolead.org/)[7]、TargetScan6.2(http://www.targetscan.org/)[8-11]、Pictar(http://pictar.mdc-berlin.de/)[12]、miRanda(http://www.microrna.org/)[13]、BINGO软件和DAVID(靶基因 的靶基因进行预测和分析。
1.2 方法
搜索miRBase查找不同哺乳动物的miR-21的成熟序列;
使用miRecords、TargetScan6.2、Pictar和miRanda 4种不同在线工具对miR-21潜在的靶基因进行预测,取预测结果的交集。4种工具预测靶基因的算法不同,所得预测结果均是其算法的最优结果,故取4种软件预测结果的交集提高预测可信度;
查找已有文献报道的miR-21的靶基因和miRrecords中已获实验验证的miR-21的靶基因;
使用cytoscape的BINGO插件和DAVID数据库对所有miR-21的靶基因用基因本体(Gene Ontology,GO)进行功能富集分类,BINGO用超几何分布算法计算出P值,DAVID 用Fisher Exact TEST计算P值,均以P<0.05为显著性阈值,得到具有统计意义的高频率注释和基因集合在GO类别上的分布信息;
用DAVID数据库中的KEGG对miR-21的所有靶基因进行信号通路富集分析。
2 结果与分析
2.1 miR-21的保守性
通过搜索miRBase发现hsa-mir-21位于染色体上17q23.2,又分别检索人、鼠、家犬和猩猩等7个物种的miR-21的成熟序列,比对结果发现7个物种的miR-21序列相似度极高,表明miR-21在不同物种间具有高度的保守性(见表1)。

表1 不同物种的miR-21的成熟序列
2.2 miR-21靶基因的预测
用TargetScan、PicTar、MiRanda和miRecords在线工具预测miR-21靶基因,分别得到164个、175个、3451个和2712个靶基因,取预测结果的交集,得到26个miR-21的靶基因(见表2)。

表2 4种在线工具预测mir-21靶基因的交集
查找文献得到在不同疾病中已证实的miR-21的靶基因42个(见表3),其中Buscaglia等综述了经实验验证的miR-21的靶基因[14],miR-21的大多数靶基因都与癌症的发生及发展有关。
在miRecords中,已证实的miR-21的靶基因有39个(见表4),其中18个靶基因是源自对乳腺癌细胞进行基因芯片和qRT-PCR的分析结果:抑制乳腺癌细胞中miR-21的表达,这18个靶基因均表达上调;而通过序列分析发现,其中BMPR2、IL6R、SOCS5、GLCCI1、APAF1、SLC16A10、SGK3、RP2、CDK6和PDCD4这10个基因与miR-21的序列至少有7-mer的种子匹配, BTG2 和 SESN1与miR-21有6-mer的种子匹配,而CDKN1A、FAS、FAM3C、HIPK3、PRRG4和ACTA2虽在抑制miR-21表达后其表达上调但与miR-21无种子匹配[15],这些与miR-21无匹配的靶基因可能为其直接靶基因也可能为其间接靶基因需要进一步地实验验证。
将预测的靶基因,查找文献获得的靶基因和miRecords中已验证的靶基因汇总,得到无重复的靶基因95个,作为进行下一步分析的基因集合。

表3 查找文献得到mir-21靶基因
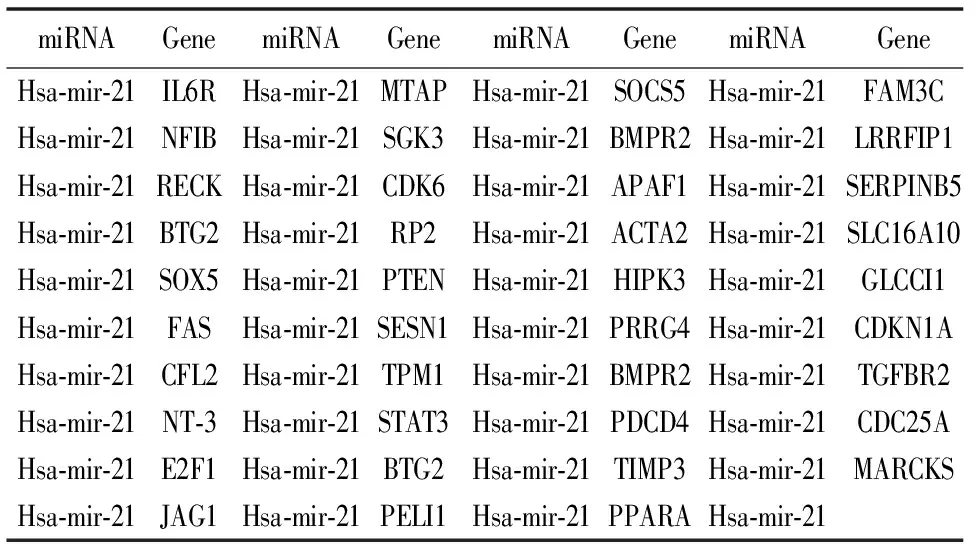
表4 miRecords中验证的has-mir-21的靶基因
2.3 mir-21靶基因的GO分析
为从基因层面认识miR-21的靶基因,对集合中的95个基因进行GO分类富集分析和分类层次网络的图形化显示。GO注释描述得到77个基因的GO生物学过程注释信息,将这77个基因投到GO的生物学过程上,根据P值选取结果,P值越小结果的可信度越高。结果(见图1和图2)发现miR-21的靶基因主要富集于生物进程、细胞进程、新陈代谢、发育、细胞增殖和细胞凋亡等生物过程。图柱越高表示参与此生物过程的基因越多,柱上方的数字为参与生物进程的基因数。

图1 mir-21靶基因在BINGO中的GO分析部分结果(生物进程)

图2 miR-21靶基因GO分类的部分层次网络Fig 2 The Hierarchical Network of GO classification节点代表一种生物进程,节点大小表示参与此进程的基因数量,颜色深浅表示P值大小,颜色越深P值越小,白色节点只起连接两个生物进程的作用,无实际研究意义。
为使基因分类富集分析结果更准确,再对集合中的95个基因用DAVID进行GO分类富集分析。GO注释描述得到44个基因的GO生物学过程注释信息,将这44个基因投到GO生物学过程,根据P值选取结果。结果(见图3)表明mir-21靶基因主要富集于调控转录、新陈代谢、基因表达和凋亡等生物进程(P<0.05,图3)。图柱越高表示参与此生物过程的基因越多,柱上方的数字为参与生物进程的基因数。
BINGO和DAVID进行的GO分类富集分析,结果表明mir-21的靶基因多富集于调控转录、新陈代谢、基因表达、细胞增殖和凋亡等生物进程,这些生物进程的异常均可能引起癌症的发生。
2.4 mir-21靶基因集合的KEGG通路分析
利用已有的生物通路对汇总的95个靶基因进行生物通路富集分析,KEGG通路富集分析得到44个基因的信号通路富集。预测结果(见图4)显示,在通路数据库KEGG中miR-21的靶基因显著富集于各种癌症(小细胞肺癌、胰腺癌、黑色素瘤、胶质瘤和结肠癌等)疾病通路中及p53 信号通路,Jak-STAT信号通路、MAPK信号通路和细胞周期等信号通路。MiR-21的靶基因集合显著富集在几个癌症通路和与癌症有关的信号通路。

图3 mir-21靶基因DAVID的GO分析(生物进程)部分结果
3 讨论
对mir-21的靶基因进行功能富集分析和信号通路富集分析,发现靶基因主要富集于癌症中通路,如P53信号通路,Jak-STAT信号通路,MAPK信号通路及与癌症相关的信号通路,并参与调控细胞周期、细胞增殖、分化和凋亡等生物进程。众所周知,P53、Jak-STAT、MAPK信号通路与肿瘤的发生有密切关系[16-17],P53信号通路已被证实与肿瘤的发生有关,但其与miR-21之间的相互作用却很少研究,有报道称抑制miR-21表达将有益于对p53缺陷的肿瘤细胞进行凋亡诱导的癌症疗法[18];STAT3是jak-STAT信号通路的重要转导子和激活子,其是癌症和白血病的著名预后因素之一[19],已有研究表明STAT3为miR-21的靶基因,因此研究结肠癌中调控jak-STAT信号通路的基因十分必要;MAPK信号通路主要指ERK、JNK和P38途径,其参与调控多种生理过程,在恶性人胚胎肺成纤维细胞(HELF)中,miR-21及其靶基因PDCD4和Spry1均是JNK/c-jun和ERK/NF-кB的调节子,表明miR-21对MAPK通路起重要的调节作用[20]。
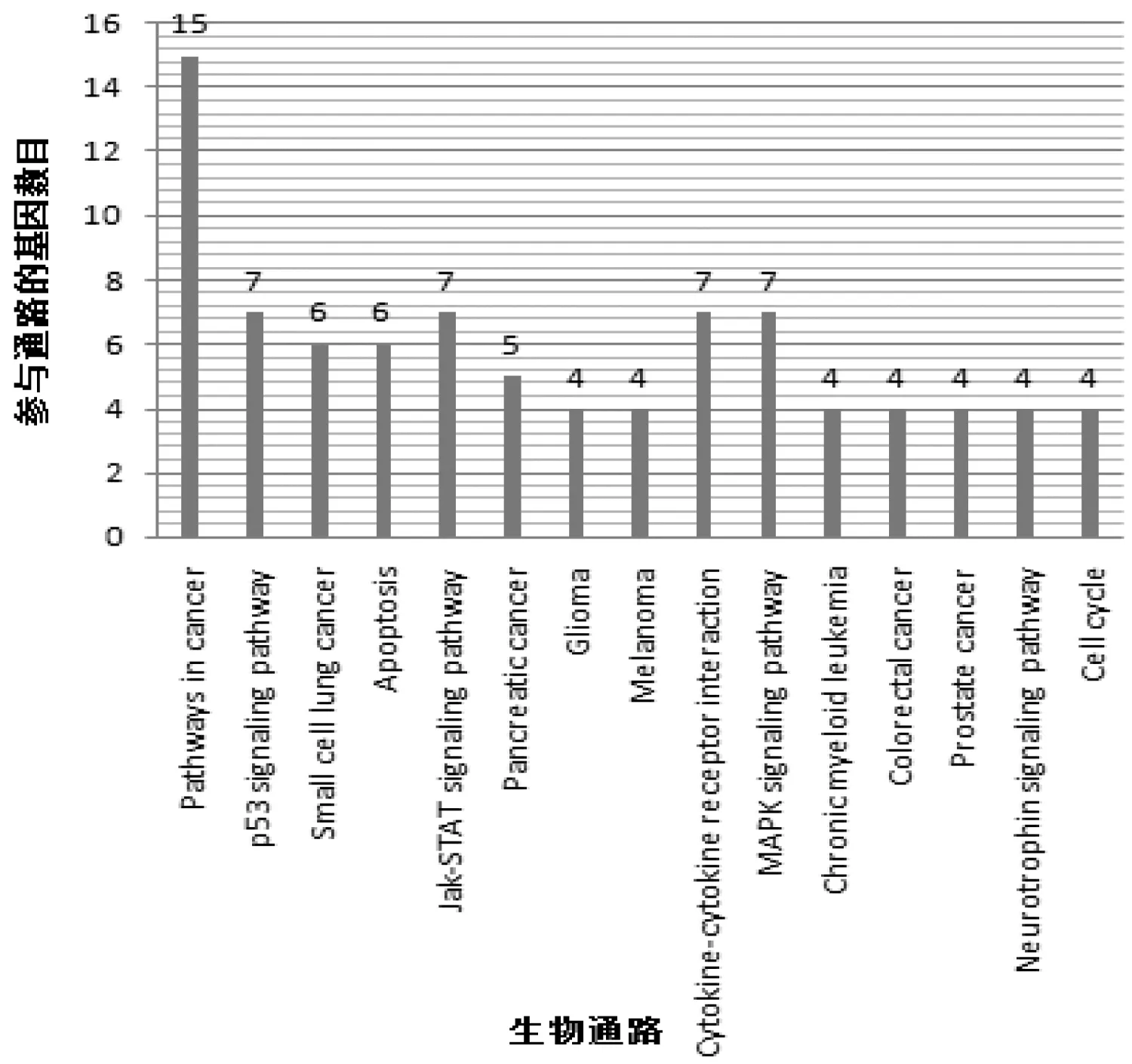
图4 mir-21靶基因KEGG通路富集分析
通过对miR-21靶基因进行生物进程分析,可以从调控网络层面挖掘miR-21的调控机制,看到基因层面看不到的信息,从而在调控层面更全面地了解miR-21的生物学功能,对了解miR-21对结肠癌的调控机制奠定基础。但所预测的miR-21靶基因及文献中查找到的靶基因在结肠癌细胞中是否为miR-21的直接靶基因或间接靶基因需要进一步实验验证。
通过生物信息学方法预测mir-21靶基因并结合部分验证的靶基因,对mir-21靶基因进行功能富集分析和信号通路富集分析,发现其靶基因多数参与癌症发生的相关信号通路,并具有调控细胞增殖、分化、凋亡及代谢的功能。由此为进一步通过RT-PCR、荧光素酶报告基因,MTT、Western blotting等实验验证mir-21在结肠癌细胞中的靶基因及靶基因的作用机制提供了一定的理论依据。
参考文献:
[1]Bushati N, Cohen S M. microRNA functions[J]. Annu Rev Cell Dev Biol, 2007, 23: 175-205.
[2]Özgün A, Karagoz B, Bilgi O, et al. MicroRNA-21 as an indicator of aggressive phenotype in breast cancer[J]. Onkologie, 2013, 36(3): 115-118.
[3]Wang Z, Han J, Cui Y, et al. miRNA-21 inhibition enhances RANTES and IP-10 release in MCF-7 via PIAS3 and STAT3 signalling and causes increased lymphocyte migration[J]. Biochemical and Biophysical Research Communications, 2013, 439(3): 384-389.
[4]Deng J, Lei W, Fu J C, et al. Targeting miR-21 enhances the sensitivity of human colon cancer HT-29 cells to chemoradiotherapy in vitro[J]. Biochemical and Biophysical Research Communications, 2014, 443(3): 789-795.
[5]Saxena A, Shoeb M, Ramana K V, et al. Aldose reductase inhibition suppresses colon cancer cell viability by modulating microRNA-21 mediated programmed cell death 4 (PDCD4) expression[J]. European Journal of Cancer, 2013, 49(15): 3311-3319.
[6]Asangani I A, Rasheed S A K, Nikolova D A, et al. MicroRNA-21 (miR-21) post-transcriptionally downregulates tumor suppressor Pdcd4 and stimulates invasion, intravasation and metastasis in colorectal cancer[J]. Oncogene, 2008, 27(15): 2128-2136.
[7]Xiao F, Zuo Z, Cai G, et al. miRecords: an integrated resource for microRNA target interactions[J]. Nucleic Acids Research, 2009, 37(suppl 1): 105-110.
[8]Lewis B P, Burge C B, Bartel D P. Conserved seed pairing, often flanked by adenosines, indicates that thousands of human genes are microRNA targets[J]. Cell, 2005, 120(1): 15-20.
[9]Friedman R C, Farh K K H, Burge C B, et al. Most mammalian mRNAs are conserved targets of microRNAs[J]. Genome Research, 2009, 19(1): 92-105.
[10]Grimson A, Farh K K H, Johnston W K, et al. MicroRNA targeting specificity in mammals: determinants beyond seed pairing[J]. Molecular Cell, 2007, 27(1): 91-105.
[11]Garcia D M, Baek D, Shin C, et al. Weak seed-pairing stability and high target-site abundance decrease the proficiency of lsy-6 and other microRNAs[J]. Nature Structural & Molecular Biology, 2011, 18(10): 1139-1146.
[12]Krek A, Grün D, Poy M N, et al. Combinatorial microRNA target predictions[J]. Nature Genetics, 2005, 37(5): 495-500.
[13]Betel D, Wilson M, Gabow A, et al. The microRNA. org resource: targets and expression[J]. Nucleic Acids Research, 2008, 36(suppl 1): 149-153.
[14]Buscaglia L E B, Li Y. Apoptosis and the target genes of miR-21[J]. Chinese Journal of Cancer, 2011, 30(6): 371.
[15]Frankel L B, Christoffersen N R, Jacobsen A, et al. Programmed cell death 4 (PDCD4) is an important functional target of the microRNA miR-21 in breast cancer cells[J]. Journal of Biological Chemistry, 2008, 283(2): 1026-1033.
[16]Gurbuz V, Konac E, Varol N, et al. Effects of AG490 and S3I201 on regulation of the JAK/STAT3 signaling pathway in relation to angiogenesis in TRAILresistant prostate cancer cells in vitro[J]. Oncology Letters, 2014, 7(3): 755-763.
[17]Chen J, Hou R, Zhang X, et al. calycosin suppresses breast cancer cell growth via ERβ-dependent regulation of IGF-1R, p38 MAPK and PI3K/Akt pathways[J]. PloS One, 2014, 9(3): e91245.
[18]Ma X, Choudhury S N, Hua X, et al. Interaction of the oncogenic miR-21 microRNA and the p53 tumor suppressor pathway[J]. Carcinogenesis, 2013, 34(6): 1216-1223.
[19]Zhong Y, Feng J, Chen B, et al. Signal transducer and activator of transcription 3 (STAT3) gene polymorphisms are associated with treatment outcomes in acute myeloid leukemia[J]. International Journal of Laboratory Hematology, 2012, 34(4): 383-389.
[20]Shen L, Ling M, Li Y, et al. Feedback regulations of miR-21 and MAPKs via Pdcd4 and Spry1 are involved in arsenite-induced cell malignant transformation[J]. PloS One, 2013, 8(3): e57652.
