三疣梭子蟹(Portunus trituberculatus)RXR基因克隆及其在蜕皮过程中的表达分析*
2014-03-19吴旭干刘智俊成永旭
王 伟 吴旭干 楼 宝 徐 蕾 刘智俊 成永旭①
(1. 上海海洋大学 水产种质资源发掘与利用教育部重点实验室 上海 201306; 2. 上海海洋大学 上海高校知识服务平台水产动物遗传育种中心 上海 201306; 3. 浙江省海洋水产研究所 舟山 316100)
三疣梭子蟹(Portunus trituberculatus)是我国一种重要的经济蟹类, 在海洋捕捞和海水养殖中都占有重要地位(谢忠明等, 2002)。尽管2012年我国三疣梭子蟹的养殖产量已经高达9.96万t (农业部渔业局,2013), 但是三疣梭子蟹育苗和养殖过程中的蜕皮综合征(molting death syndrome, MDS)一直是困扰该产业健康发展的重要问题(Takeuchiet al, 1999a, b; Wuet al, 2010a, b; 路允良等, 2012)。MDS是指蟹类不能顺利完成蜕皮, 在蜕皮过程中或蜕皮不久便大量死亡的一种综合症状, 这给经济蟹类养殖生产造成了巨大的经济损失(Suprayudiet al, 2004), 其形成原因非常复杂, 初步研究结果表明MDS可能与饵料营养及环境因子有密切关系(Danet al, 2011; 路允良等,2012)。深入了解MDS的形成机制, 首先必须研究三疣梭子蟹蜕皮的生理机制, 从而为三疣梭子蟹养殖过程中MDS的控制提供理论参考和实践依据。
甲壳动物的蜕皮过程受包括蜕皮激素、眼柄神经多肽类和甲基法尼酯等多种激素的联合调控, 它们有着不同的生理功能和调控路径(Riddifordet al,2003)。蜕皮激素在蜕皮过程中扮演着十分重要的角色, 其含量在蜕皮过程中呈周期性变动, 蜕皮前含量最高, 蜕皮间期含量最低(Mykles, 2011; Techaet al,2013)。蜕皮激素对甲壳动物蜕皮过程的生理调控是主要通过与蜕皮激素受体(EcR)和类维甲酸X受体(RXR)的二聚体结合来实现的。蜕皮激素与此二聚体的结合体可直接与细胞核上的激素应答原件结合,调节下游基因E74及E75等的转录, 从而调控甲壳动物的蜕皮过程(Riddifordet al, 2003; Kimet al, 2005;Techaet al, 2013)。RXR是核受体超家族的一员, 具有核受体家族的典型特征, 在结构上分为A、B、C、D、E、F六区, 组成四个功能结构域(A/B域、C域、D域和E/F域)。目前有关甲壳动物的RXR研究主要集中在RXR的基因克隆及其表达模式分析, 功能研究较少(Wuet al, 2004; Kimet al, 2005; Priyaet al,2009)。先前的研究结果表明, 不同甲壳动物的RXR基因序列及结构存在较大的差异(Duricaet al, 1996;Wuet al, 2004; Kimet al, 2005), 且RXR在不同甲壳动物蜕皮过程中的表达模式也有所不同(Asazumaet al, 2007; 王文青等, 2010; 王瑶等, 2013)。通过研究RXR基因在甲壳动物不同组织及不同蜕皮阶段的表达情况, 可以初步了解RXR的表达部位、调控位点及其与蜕皮过程的关系, 从而为深入研究RXR的基因功能奠定基础。迄今为止有关三疣梭子蟹RXR基因克隆和表达模式的研究报道极少, 仅见NCBI中登录的一条三疣梭子蟹RXR基因cDNA序列。因此, 本研究在通过三疣梭子蟹转录组文库获得RXR部分cDNA序列(contig698)的基础上, 进一步通过反转录PCR(RT-PCR)和cDNA 末端快速扩增(RACE)技术,克隆得到了三疣梭子蟹RXR2基因cDNA全长, 并进行相关生物信息学分析, 进一步通过荧光定量PCR(qRT-PCR)技术研究了该基因在三疣梭子蟹不同组织及不同蜕皮阶段的表达情况, 结果可以为深入研究三疣梭子蟹RXR的基因功能和阐明蟹类蜕皮的调控机制提供基础资料, 同时丰富甲壳动物发育生物学的研究内容。
1 材料与方法
1.1 材料与采样
三疣梭子蟹幼蟹于2011年8月购自浙江舟山宏福水产养殖场, 甲壳宽为7—9cm, 初始体重50—80g,挑选肢体健全、活力较好、处于蜕皮间期的雌体60只, 活体运输到研究室, 在室内循环水系统中暂养一周, 暂养水族箱体积为150 L(长×宽×高 = 75cm×53cm×47cm), 箱底部放置无毒PVC管(直径15cm,长15cm)作为隐蔽物, 每箱放8—10只幼蟹, 暂养期间每日下午19:00按照蟹体重的5%—10%投喂章鱼,次日上午10:00清理残饵和粪便。
实验用蟹暂养一周后, 挑选32只肢体健全、活力较好、处于蜕皮间期(C期)或蜕皮前期(D期)的个体用于实验。由于三疣梭子蟹极易自相残杀导致成活率偏低, 因此正式实验期间每只幼蟹均单独饲养于小型循环水族箱(水体体积40L, 长×宽×高 = 53cm×18cm×45cm)中。实验期间自然光照, 水体盐度为24,水温24—26°C, pH 7.0—9.0, 溶氧>5mg/L; 氨氮<0.5mg/L,亚硝酸盐<0.15mg/L。投喂和日常管理与暂养期间基本一致。根据沈洁等(2011)形态学方法将三疣梭子蟹蜕皮周期分为蜕皮期(E期)、蜕皮后期(AB期), 蜕皮间期(C期)和蜕皮前期(D期), 每日检查每只蟹所处的蜕皮阶段, 并记录。对到达特定蜕皮时期的三疣梭子蟹进行解剖, 采集该蜕皮时期三疣梭子蟹肌肉、肝胰腺、心脏、胃、肠、鳃、Y器官、大颚器、眼柄、胸神经节、三角膜组织用于后续的实验。每个蜕皮期分别采集5—8只个体, 所有组织样品液氮速冻后保存于–80°C冰箱用于总RNA的提取。由于D期可以分为D0、D1、D2、D3和D4五个不同的亚期(沈洁等,2011), 考虑到D期每个亚期持续时间较短, 本实验中D期样品均来自较为典型的D2亚期。
1.2 RNA提取和cDNA全长的克隆
取冻存的三疣梭子蟹Y器官, 采用总RNA提取试剂盒(TaKaRa, Cat.D9108A)进行总RNA的提取, 分别采用1%琼脂糖凝胶电泳和紫外分光光度计法检测RNA的完整性和纯度。使用SMARTTMRACE cDNA Amplification Kit (Clontech, Cat.634923)进行第一链cDNA合成。各取1μg总RNA为反转录模板, 分别用于5’和3’端RACE扩增。进而采用 Advantage 2 PCR Kit (Clontech, Cat.639207)进行RACE扩增, 所用引物根据三疣梭子蟹转录组文库(NCBI登陆号:SRA051608)筛选到RXR基因核心序列(序列拼接号:contig698)设计而成, 5’-RACE和3’-RACE引物分别命名为PtRXR5-R1、PtRXR5-R2、PtRXR3-F1和PtRXR3-F2, 具体序列见表1。
所得PCR产物琼脂糖凝胶电泳后采用DNA回收试剂盒(TaKaRa, Cat.9763)进行回收纯化, 取1μL回收产物与pMD19-T载体(TaKaRa, Cat.D104A)连接,连接产物转化至大肠杆菌Top 10感受态细胞(天根,Cat.CB104-01)中, 在37°C、200r/min条件下振荡培养1—2h, 涂布接种后进行蓝白斑筛选, 随机挑选10个阳性克隆送上海生工生物工程有限公司进行测序。

表1 实验用PCR引物及序列Tab.1 Primers and their sequences used in the experiment
1.3 序列分析
利用DNAStar软件中的SeqMan程序对测序结果进行载体序列的去除和目的序列的拼接, 获得PtRXR全长cDNA; 采用Blast程序分析PtRXR基因与其它动物RXR基因的同源性和一致性; 采用ORF Finder程序(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)预测其开放阅读框(ORF)和编码氨基酸。使用ProtParam tool(http://web.expasy.org/protparam/)预测PtRXR编码氨基酸的物理参数, 分别用SignalP 4.1 Server(http://www.cbs.dtu.dk/services/SignalP/)和TMHMM 2.0程序(http://www.cbs. dtu.dk/services/TMHMM-2.0/)预测其信号肽和跨膜结构。最后采用ClustalX(http://www.clustal.org/)和MEGA 5.0软件(http://www.Megaso ftware.net/)进行多序列比对和NJ系统进化树构建。
1.4 实时荧光定量PCR(qRT-PCR)
采用总RNA提取试剂盒(TaKaRa, Cat.D9108A)对各采样组织进行总RNA的提取, 各取100ng总RNA为反转录模板, 采用反转录试剂盒(TaKaRa,Cat.D2639A)进行第一链cDNA合成。根据三疣梭子蟹RXR cDNA全长序列, 采用Primer Premier 5.0软件设计定量PCR用特异性引物PtRXR F1/R1及PtRXR F2/R2(表1)。以三疣梭子蟹18S-F/R(表1)为引物对内参基因18S rRNA进行扩增(Yanget al, 2005)。
按照TaKaRa定量PCR试剂盒(TaKaRa, Cat.DRR 420A)说明, 将模板cDNA梯度稀释后进行标准曲线扩增。当无非特异性扩增出现, 目标基因和内参基因的扩增效率在95%—105%, 标准曲线R2大于0.99时,确定qRT-PCR反应体系与条件, 最终采用PtRXR F1/R1作为RXR定量PCR扩增引物。qRT-PCR的反应体系见表2, 反应条件为: 95°C预变性30s; 95°C变性3s, 60°C退火30s, 共进行40个循环。qRT-PCR反应体系与条件确定后, 以C期三疣梭子蟹肌肉、肝胰腺、心脏、胃、肠、鳃、Y器官、大颚器、眼柄、胸神经节和三角膜11种组织样品的cDNA作为模板,研究PtRXR在各组织中表达情况。根据PtRXR在各组织中的表达情况, 对Y器官、大颚器、眼柄、胸神经节、肝胰腺、鳃和肌肉中不同蜕皮阶段PtRXR基因的表达变化进行研究。每个样品重复4次, 每个蜕皮阶段重复5个体。
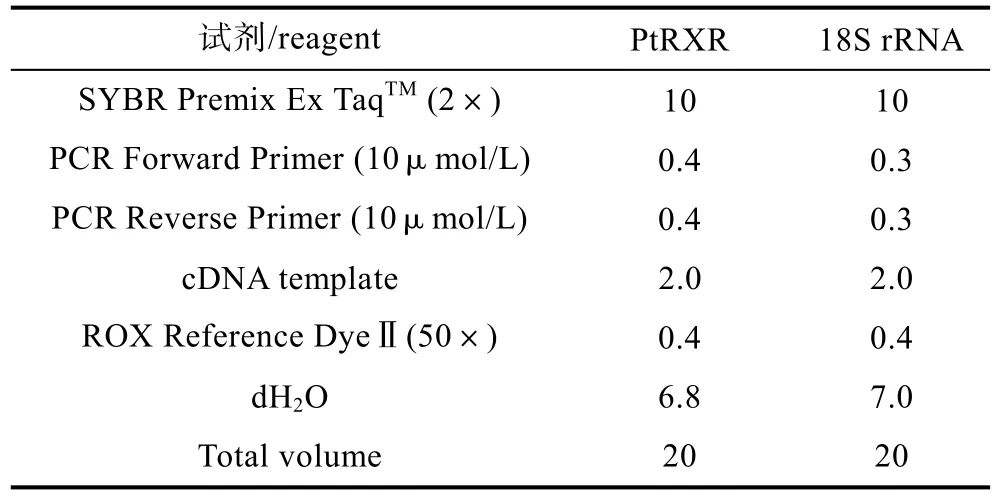
表2 PtRXR与18S rRNA基因荧光定量PCR反应体系中各试剂添加量(μL)Tab.2 The volume (μL) of each reagent added to the PCR mixture used for qRT-PCR of PtRXR and 18S rRNA
1.5 数据分析
采用18S rRNA作为内参基因, 计算△Ct, 通过公式△△Ct =△Ct样品-△Ct对照计算△△Ct, 2–ΔΔCt法计算PtRXR-mRNA的相对表达水平。采用SPSS 17.0软件对qRT-PCR数据进行统计分析, 所有数据采用平均值±标准差表示, 采用Levene’s法进行方差齐性检验, 当不满足齐性方差时对数据进行反正弦或平方根处理, 采用ANOVA对实验结果进行方差分析, 采用Tukey’s法进行多重比较, 当P<0.05时为差异显著。
2 结果
2.1 PtRXR cDNA全长克隆与序列分析
根据三疣梭子蟹转录组文库(NCBI登陆号: SRA 051608)中RXR的cDNA片段(序列拼接号: contig698),设计多条特异性引物进行RACE扩增, 直到3’和5’端分别出现poly A和起始密码子及非编码区, 将所得序列进行拼接和验证, 确定本研究中三疣梭子蟹RXR基因cDNA序列全长为1718bp, 命名为PtRXR2,GenBank登录序列号为KF914662。序列分析结果表明, 该基因包括5’非编码区(5’-UTR)141bp、3’-UTR 209bp和开放阅读框(ORF)1368bp, 编码455个氨基酸。ORF编码氨基酸的预测分子式为C2176H3479N605O671S28,分子量为49.75kDa, 理论等点6.79。进一步分析表明该蛋白不存在信号肽序列和跨膜结构区, 因此推测该蛋白不属于分泌蛋白。整体上, PtRXR2和NCBI上已经登录的PtRXR(AGV08303)存在较大差异, 两者不仅5’-UTR的长度不同, 且PtRXR在ORF中存在168bp和60bp的核苷酸缺失, 其它多处存在共15个核苷酸的序列差异。
PtRXR2的氨基酸序列从N端到C端具有4个典型功能结构域: A/B域(转录激活域)、C域(DNA结合域, DBD)、D域(铰链域)、E/F域(含有配体结合域,LBD)。C域具有P-box和D-box, D域中存在T-box (图1)。不同甲壳动物RXR的氨基酸比对结果表明, 三疣梭子蟹PtRXR2、招潮蟹(Uca pugilator)UpRXR、凡纳滨对虾(Litopenaeus vannamei)LvRXR和美洲鳌虾(Homarus americanus)HaRXR1 在氨基端(25个氨基酸)序列相对保守, 而这些氨基酸序列在PtRXR、HaRXR2、黑背陆地蟹(Gecarcinus lateralis)GlRXRa、褐虾(Crangon crangon) CcRXR1和中华绒螯蟹(Eriocheir sinensis)EsRXRb的A/B域中缺失(图2中实线方框标出); 5种虾蟹类RXR的C域高度保守, 均含有保守的P-box与D-box, DBD中含有两个C2C2型锌指结构和两个α螺旋; D域具有保守的T-box, 但PtRXR2、PtRXR、UpRXR、LvRXR和HaRXR1等的T-box中存在六个氨基酸残基缺失(图2中实线方框标出); E/F域的E区较为保守, 5种虾蟹类在该区均含有氨基酸序列完全一致的转录激活结构域(AF-2); F区较短且保守性最低, 但PtRXR和PtRXR2的F区推断氨基酸序列完全一致(图2)。

图1 PtRXR2基因cDNA全长及预测的氨基酸序列Fig.1 Full cDNA sequence and its deduced amino acid sequence of PtRXR2
整体上, PtRXR2和PtRXR一致性最高, 两者差异主要在于PtRXR的A/B域中存在两段氨基酸序列的缺失(图2中上划线标示), 对应于PtRXR ORF中缺失的核苷酸序列; PtRXR2与蓝蟹(Callinectes sapidus)两种RXR亚型的一致性最高, 分别为99%和98%; 与滨蟹(Carcinus maenas)RXRⅠ序列的一致性为97%;与黑背地蟹三种RXR亚型的一致性分别为87%、86%和86%; PtRXR2与虾类RXR的氨基酸序列比对结果显示, PtRXR2与褐虾RXR 三种亚型的氨基酸序列一致性较高, 在84%—85%之间; 与凡纳滨对虾、中国明对虾(Fenneropenaeus chinensis)和日本囊对虾(Marsupenaeus japonicus)RXR氨基酸序列一致性相对较低, 分别为76%、75%和74%。

图2 三疣梭子蟹与其它甲壳动物的RXR氨基酸序列比对Fig.2 Comparison of amino acid sequences of RXR between P. trituberculatus and other crustacean species
基于20种节肢动物(甲壳动物和昆虫)的RXR/USP氨基酸序列, 采用MEGA(version 5.0)软件邻接法(Neighbor-Joining)构建系统进化树, 结果显示昆虫纲中国蜜蜂(Apis cerana)、沙蟋(Gryllus firmus)和太平洋折翅蠊(Diploptera punctata)聚为一支, 它们与甲壳动物亲缘关系更近, 与甲壳动物RXR聚为一大支,其余聚为另外一支。甲壳动物中虾蟹类的RXR, 在进化上更为接近, 飞马哲水蚤(Calanus finmarchicus)在甲壳纲为独立一支。三疣梭子蟹、普通滨蟹与滨蟹的RXR亲缘关系最近, 它们聚为一支(图3)。
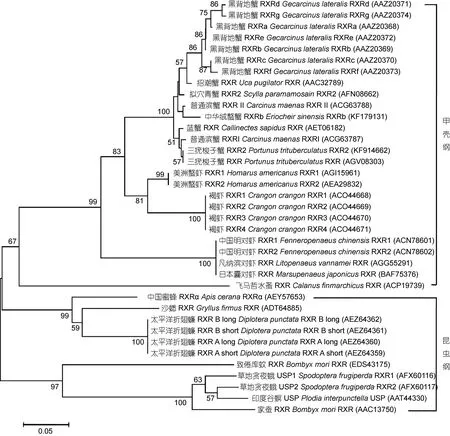
图3 RXR氨基酸序列的NJ进化树Fig.3 The neighbor-joining phylogenetic tree for RXRs
2.2 PtRXR2在蜕皮间期不同组织中的表达情况
定量PCR结果表明, PtRXR2在三疣梭子蟹C期的肌肉、肝胰腺、心脏、胃、肠、鳃、Y器官、大颚器、眼柄、胸神经节和三角膜中均有表达, 且各组织中的PtRXR2-mRNA表达水平差异显著(P<0.05, 图4)。Y器官中的PtRXR2-mRNA表达水平最高, 其余依次为鳃>大颚器>眼柄>三角膜>胃>胸神经节>心脏>肠>肌肉>肝胰腺, 其中肝胰腺中的PtRXR2-mRNA表达水平仅为Y器官的10.92%。
2.3 PtRXR2在不同蜕皮阶段的表达情况
根据PtRXR2在不同组织中的表达情况, 采用Y器官、眼柄、胸神经节、大颚器、肌肉、肝胰腺和鳃7个主要组织, 研究PtRXR2-mRNA在三疣梭子蟹蜕皮周期中的表达变化情况。结果表明, PtRXR2-mRNA在7种组织中的表达水平均在不同蜕皮阶段变化显著(P<0.05), 但不同组织呈现不同的变化模式(图5)。Y器官中PtRXR2-mRNA的表达水平在E期最低, 蜕皮后AB期显著升高, 其相对表达量是C期的5倍以上, C期和D期的表达水平显著下降, 但仍显著高于E期; 在整个蜕皮周期中, 眼柄、胸神经节和肝胰腺中PtRXR2-mRNA表达变化模式基本一致, 为E期表达量较低, AB期显著升高, C期显著降低, D期显著升高; 与上述三个组织的PtRXR2-mRNA变化趋势相反,大颚器中PtRXR2-mRNA在AB期的表达水平最低,其余三个阶段相对表达量无显著差异; 鳃中PtRXR2-mRNA在C期表达量最高, D期最低, E期和AB期表达量处于两者之间, 且各期间的相对表达量差异显著(P<0.05); 在整个蜕皮周期中, 肌肉中PtRXR2-mRNA表达水平相对较低, 各期相对表达量依次为AB期>C期>E期>D期(图5)。
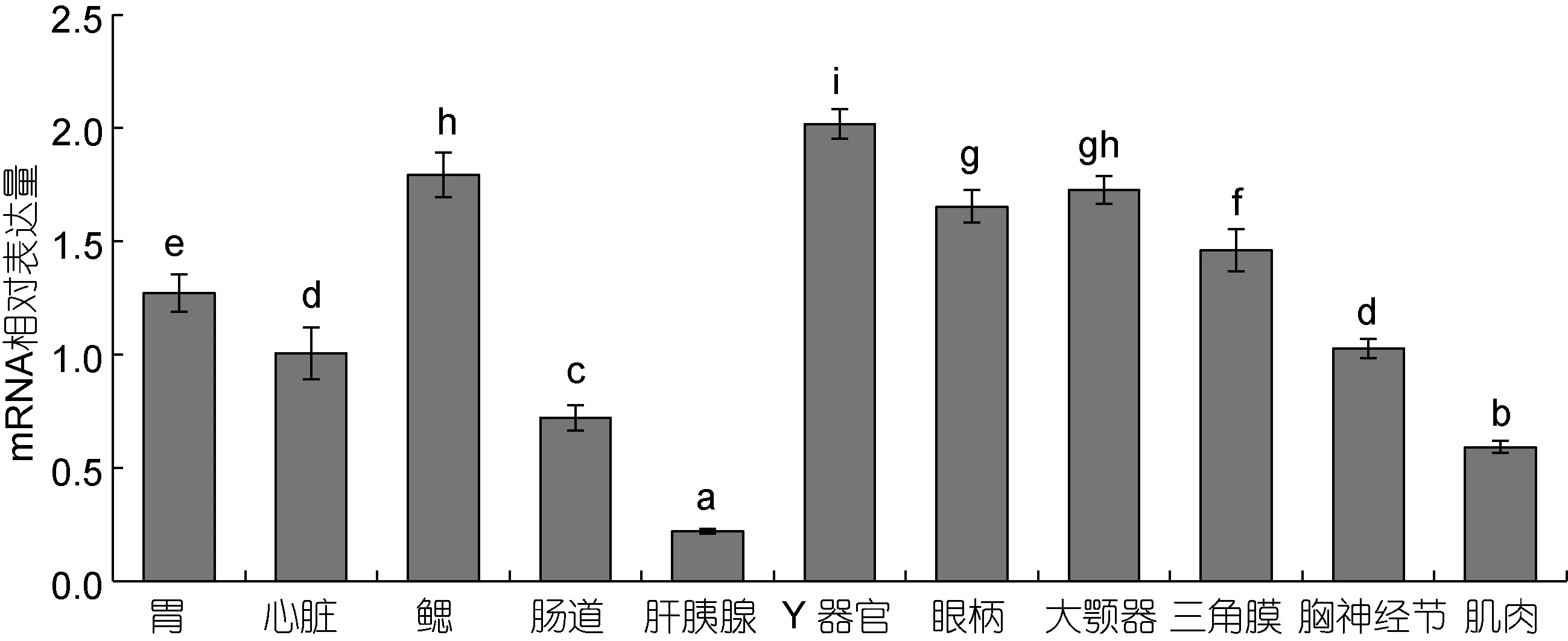
图4 蜕皮间期三疣梭子蟹各组织中PtRXR2-mRNA的表达差异分析Fig.4 Analysis of expression difference of PtRXR2-mRNA in various tissues of P. trituberculatus at intermolt stage
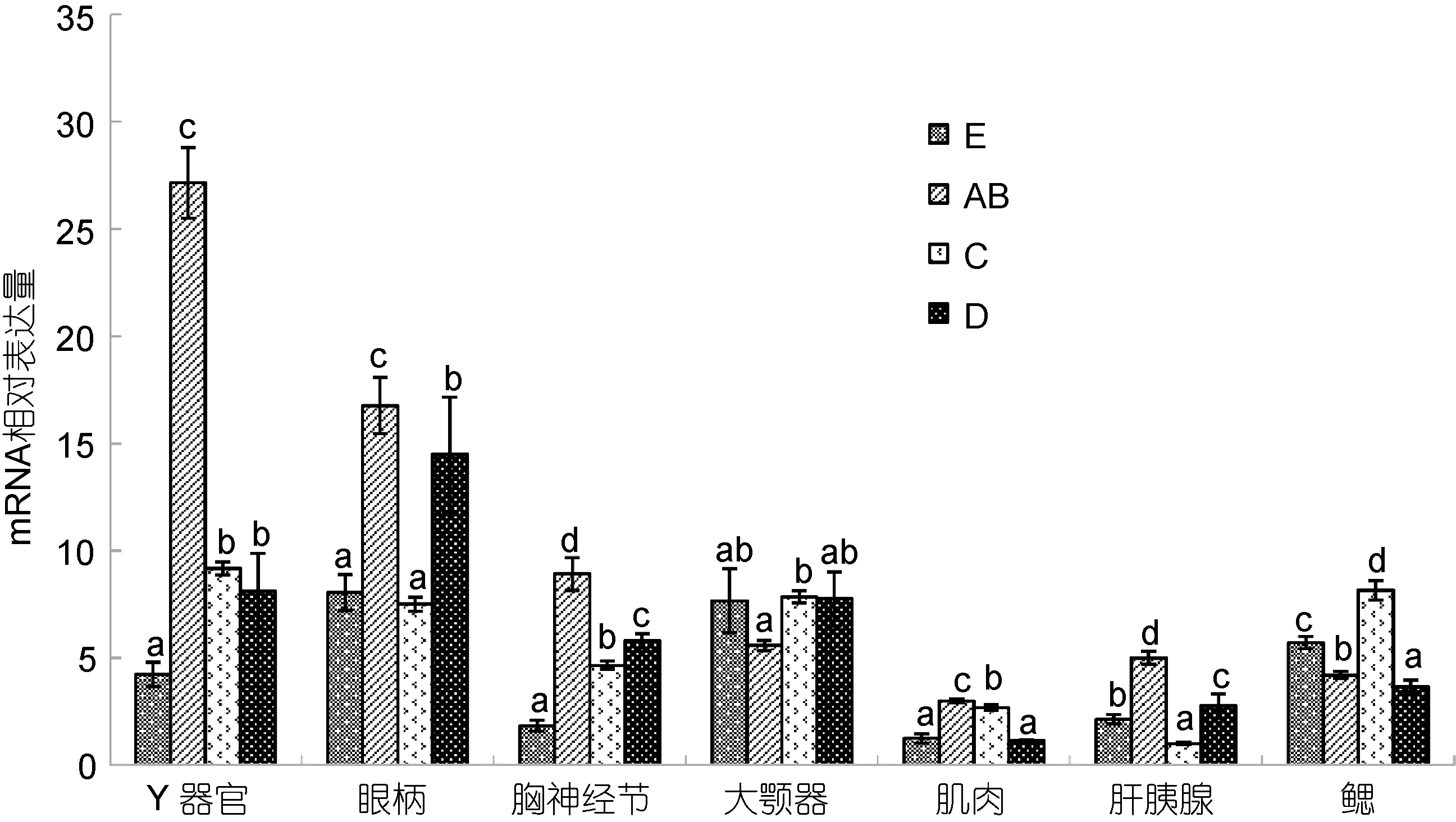
图5 不同蜕皮阶段三疣梭子蟹七种组织中的PtRXR2-mRNA表达差异分析Fig.5 Analysis of expression difference of PtRXR2-mRNA in seven tissues of P. trituberculatus at different molting stages
3 讨论
3.1 PtRXR2序列结构分析
本研究通过转录组测序和RACE技术, 获得PtRXR2基因的cDNA全长序列, 该基因与NCBI已登录的PtRXR基因在5’-非编码区和ORF中存在多处核苷酸和预测氨基酸序列差异, 且PtRXR在A/B域存在两段氨基酸序列的缺失, 长度分别为56和20个氨基酸(图2中上划线标示), 对应于PtRXR ORF序列中缺失的长度分别为168bp、60bp的两段核苷酸序列。因此, 初步推断PtRXR2和PtRXR为三疣梭子蟹RXR的两个不同基因, 而非选择性剪切造成的不同转录本或亚型, 将此基因暂命名为PtRXR2。整体上,不同甲壳动物的RXR较为保守, 已知虾蟹类的RXR与PtRXR2氨基酸序列一致性高达70%以上, 特别是C、D功能结构域和E区保守性较高。常见甲壳动物RXR的C域均含有高度保守的P-box和D-box典型结构, 其中P-box与RXR作用元件的DNA结合有关,D-box与同型二聚体的形成有关, 从而保证RXR作用元件及二聚体的形成和调控作用的发挥(Devarakondaet al, 2003; Asazumaet al, 2007); D域也存在DNA识别的T-box(Kimet al, 2005; Priyaet al, 2009), 但是部分种类的RXR或不同转录本存在6个氨基酸缺失,有趣的是在T-box中存在这6个氨基酸的物种在A/B域N端都存在25个氨基酸残基缺失, 造成这种差异的原因可能与进化或RNA的选择性剪切有关(王文青等, 2010); E/F域含有保守的配体结合域(LBD), 所有物种均具有一个保守的转录激活结构域(AF-2, 见图2)(Wanget al, 2000)。然而, 不同虾蟹类RXR的A/B域和E/F域的F区通常存在一定的差异。A/B域为转录激活域, 不同物种的RXR在该序列上差异较大,同一物种的不同RXR亚型也有所不同(Huet al, 2003;Kimet al, 2005; Techaet al, 2013), 这暗示不同RXR基因及同一RXR基因的不同亚型生理作用可能有所不同, 这种功能差别可能是通过不同信号通路的转录调控实现的(王文青等, 2010; Dawsonet al, 2012)。尽管RXR的F区较短, 但不同物种间F区的差别较大, 其氨基酸序列存在较大变化或缺失, 同一物种的不同RXR基因F区氨基酸序列及长度基本一致(图2),但是同一RXR基因亚型可能会通过选择性剪切形成不同的转录本, 造成F区氨基酸长度不同甚至缺失(Kimet al, 2005; Techaet al, 2013)。迄今为止, 有关RXR的F区生物学功能研究极少, 推测认为该区域可以调节配体与LBD的结合力、二聚体的形成及参与调节其它调控因子间的交互作用(Wuet al, 2004),如在哺乳动物中, F区的部分氨基酸序列与转录调控有关(Dawsonet al, 2012)。
3.2 PtRXR2的生理功能及其与蜕皮的关系
绝大部分动物体内都存在RXR/USP(昆虫中的超气门蛋白, 类似于甲壳动物的RXR)基因, 该基因家族在细胞动物生长发育、形态发生、细胞增殖分化、新陈代谢和细胞内环境平衡稳定等过程中起着非常重要的调控作用(Mangelsdorfet al, 1990; Kimet al,2005; Dawsonet al, 2012)。有关大鼠的研究结果表明,鼠类存在三种不同的RXR基因, 分别命名为RXRα、RXRβ和RXRγ, 这三种RXR蛋白的DBD和LBD极为保守, 但其A/B域的氨基酸序列差异较大, 推测它们可能参与了不同的转录调控过程, 起着不同的生理作用(Mangelsdorfet al, 1992)。进一步的研究表明,RXRα在肝脏等代谢器官中表达水平较高, 主要参与脂类和维生素A的代谢调控, 这是因为RXRα的DNA反应元件是细胞视黄醇结合蛋白(CRBPII)和载脂蛋白(apoAI)基因启动子的一部分, 而CRBPII和apoAI主要参与维生素A和脂类的运输和代谢; RXR β与老鼠的神经鞘质损伤和睾丸生理功能有关, 但该基因的表达部位较多(Huanget al, 2011); RXRγ主要在神经系统、肌肉和上皮组织中表达, 主要调控细胞的增殖分化和神经信号传导等(Königet al, 2012)。
迄今为止, 有关甲壳动物的RXR基因功能研究较少。现有研究表明, 甲壳动物的RXR基因与蜕皮调控有关, 如中国明对虾和中华绒螯蟹Y器官中的RXR-mRNA在蜕皮后表达水平逐步升高, 在蜕皮前达到最大值(Priyaet al, 2009; 王瑶等, 2013)。而甲壳动物和昆虫体内的RXR/USP具体如何参与蜕皮调控则非常复杂, 如: 对中国明对虾中RXR的RNAi实验表明, RXR正向调控中国明对虾几丁质酶的基因表达(Priyaet al, 2009)。甲壳动物蜕皮前通常需要高含量的几丁质酶水解旧壳中的甲壳素以便旧壳裂开, 从而有利于蜕皮的进行(沈洁等, 2011); 昆虫体内20-羟基蜕皮酮(20-E)-EcR-USP复合体可以调控Br-C、E74、E75和E93等20-E初级应答基因的表达, 这些基因触发次级应答基因表达后最终会引起昆虫变态和蜕皮过程中的细胞自噬和凋亡(Liuet al, 2009; 李康等,2011)。值得注意的是, 同一甲壳动物的一种RXR基因可以通过选择性剪切得到不同的转录本及蛋白,从而发挥不同的调控作用(Kimet al, 2005; Techaet al,2013)。
本研究结果表明, PtRXR2在三疣梭子蟹蜕皮间期的11个组织中都有表达, 且在内分泌器官(Y器官和大颚器)、神经系统(眼柄和胸神经)、上皮组织发达的三角膜、肠道和胃, 以及肌肉组织中表达水平相对较高, 在肝胰腺中的表达水平最低(图4), 这与哺乳动物的RXRγ在不同组织中的表达特性基本一致(Mangelsdorfet al, 1992; Huanget al, 2011)。此外, 根据PtRXR2与哺乳动物的RXR氨基酸序列比对结果发现, PtRXR2也是与RXRγ一致性最高。因此, 作者推测PtRXR2可能类似于哺乳动物RXRγ型。先前研究表明, 甲壳动物的RXR均在Y器官中表达水平较高, 这是因为Y器官主要分泌蜕皮酮调节蜕皮过程。就不同甲壳动物种类而言, RXR在其它器官中的表达水平不尽一致。RXR在中华绒螯蟹、日本囊对虾和中国明对虾的肝胰腺中表达水平仅次于Y器官(Asazumaet al, 2007; Priyaet al, 2009; 王瑶等, 2013),但本研究PtRXR2在肝胰腺中的表达水平最低。造成这种差异的原因可能是由于不同虾蟹中发现的RXR基因种类或转录本有所不同, 它们具有不同的生理功能, 因而在不同组织中的表达模式存在差异。如RXRα主要调节哺乳动物的脂类和维生素A代谢,因此在肝胰腺等代谢器官中表达水平较高, 而RXRγ则调控细胞的增殖分化和神经信号传导等, 故在神经系统、肌肉和上皮组织中表达较高(Mangelsdorfet al, 1992; Königet al, 2012)。本研究结果表明, PtRXR2在三疣梭子蟹的Y器官、眼柄和胸神经节中均是AB期表达水平最高, 但是蜕皮过程中的E期最低, 这可能是因为上述内分泌器官和神经组织的细胞增殖主要发生在AB期(Changet al, 2011), 而PtRXR2可能在此阶段参与这些细胞增殖过程; 肌肉中的PtRXR2表达水平在AB期和C期较高, E期和D期最低, 与肌肉的生长和营养物质积累主要发生这两个阶段相对应(沈洁等, 2011); 肝胰腺中的PtRXR2在AB期最高, 暗示肝胰腺中的上皮细胞增殖和分化主要发生在此阶段, 肝胰腺中C期表达水平最低, 也暗示PtRXR2主要作用不是参与肝胰腺中的脂类营养代谢过程; 而蜕皮周期中, PtRXR2-mRNA在鳃和大颚器中的变化原因不详。尽管本研究克隆了PtRXR2的cDNA全长, 根据其生物学信息学分析及其在蜕皮过程中表达分析结果, 推测该基因可能类似于哺乳动物的RXRγ型, 但由于缺乏相关的功能验证, 故PtRXR2在三疣梭子蟹蜕皮过程中的准确生理功能、调控机制及其基因分类有待进一步深入研究。
王 瑶, 杨志刚, 郭子好等, 2013. 中华绒螯蟹RXR基因全长cDNA克隆及表达分析. 水产学报, 37(12): 1761—1769
王文青, 朱小玲, 2010. 日本沼虾不同亚型维甲酸类X受体(RXR)cDNA克隆及组织表达分析. 淡水渔业, 40(1): 4—10
农业部渔业局, 2012. 2012年中国渔业统计年鉴. 北京: 中国农业出版社, 33
李 康, 李 胜, 曹 阳, 2011. 蜕皮激素与其受体EcR-USP的转录调控机制. 昆虫学报, 5(8): 933—937
沈 洁, 朱冬发, 胡泽辉等, 2011. 三疣梭子蟹蜕皮周期的分期. 水产学报, 35(10): 1481—1487
谢忠明, 刘洪军, 冯 蕾, 2002. 海水经济蟹类养殖技术. 北京: 中国农业出版社, 15—27
路允良, 王 芳, 赵卓英等, 2012. 盐度对三疣梭子蟹生长、蜕皮及能量利用的影响. 中国水产科学, 19(2): 237—245
Asazuma H, Nagata S, Kono Met al, 2007. Molecular cloning and expression analysis of ecdysone receptor and retinoid X receptor from the kuruma prawn,Marsupenaeus japonicas.Comp Biochem Physiol B Biochem Molecular Biology,148(2): 139—150
Chang E S, Mykles D L, 2011. Regulation of crustacean molting:A review and our perspectives. General and Comparative Endocrinology, 172: 323—330
Dan S, Hamasaki K, 2011. Effects of salinity and dietary n-3 highly unsaturated fatty acids on the survival, development,and morphogenesis of the larvae of laboratory-reared mud crabScylla serrata(Decapoda, Portunidae). Aquaculture International, 19: 323—338
Dawson M I, Xia Z B, 2012. The retinoid X receptors and their ligands. Biochimica et Biophysica Acta (BBA)-Molecular and Cell Biology of Lipids, 1821: 21—56
Devarakonda S, Harp J M, Kim Yet al, 2003. Structure of the heterodimeric ecdysone receptor DNA-binding complex.The EMBO Journal, 22: 5827—5840
Durica D S, Hopkins P M, 1996. Expression of the genes encoding ecdysteroid and retinoid receptors in regenerating limb tissues from the fiddle crab,Uca pugilator. Gene, 171:237—241
Hu X, Cherbas L, Cherbas P, 2003. Transcription activation by the ecdysone receptor (EcR/USP): identification of activation functions. Molecular Endocrinology, 17: 716—731
Huang J K, Jarjour A A, Oumesmar B Net al, 2011. Retinoid X receptor gamma signaling accelerates CNS remyelination.Nature Neuroscience, 14: 45—53
König R, Stillfried M, Aperdannier Pet al, 2012. Expression of retinoid X receptor beta is induced in astrocytes during corpus callosum demyelination. Journal of Chemical Neuroanatomy, 43(2): 120—132
Kim H W, Lee S G, Mykle D L, 2005. Ecdysteroid-responsive genes, RXR and E75, in the tropical land crab,Gecarcinus lateralis: Differential tissue expression of multiple RXR isoforms generated at three alternative splicing sites in the hinge and ligand-binding domains. Molecular and Cellular Endocrinology, 242: 80—95
Liu Y, Sheng Z T, Liu Het al, 2009. Juvenile hormone counteracts the bHLH-PAS transcription factors MET and GCE to prevent caspase-dependent programmed cell death inDrosophila. Development, 136: 2015—2025
Mangelsdorf D J, Ong E S, Dyck J Aet al, 1990. Nuclear receptor that identifies a novel retinoic acid response pathway. Nature, 345(6272): 224—229
Mangelsdorf D J, Borgmeyer U, Heyman R Aet al, 1992.Characterization of three RXR genes that mediate the action of 9-cis retinoic acid. Genes & Development, 6(3): 329—344
Mykles D L, 2011. Ecdysteroid metabolism in crustacean.Journal of Steroid Biochemistry and Molecular Biology,127(3—5): 196—203
Priya T A, Li F, Zhang Jet al, 2009. Molecular characterization and effect of RNA interference of retinoid X receptor (RXR)on E75 and chitinase gene expression in Chinese shrimpFenneropenaeus chinensis. Comparative Biochemistry and Physiology, Part B, 153: 121—129
Riddiford L M, Hiruma K, Zhou X Fet al, 2003. Insights into the molecular basis of the hormonal control of molting and metamorphosis fromManduca sextaandDrosophila melanogaster. Insect Biochemistry and Molecular Biology,33(12): 1327—1338
Takeuchi T, Nakamoto Y, Hamasaki Ket al, 1999a. Requirement of n-3 highly unsaturated fatty acids for larval swimming crabPortunus trituberculatus. Nippon Suisan Gakkaishi, 65:797—803
Takeuchi T, Satoh N, Sekiya Set al, 1999b. The effect of dietary EPA and DHA on the molting rate of larval swimming crabPortunus trituberculatus. Nippon Suisan Gakkaishi, 65: 998—1004
Techa S, Chung J S, 2013. Ecdysone and retinoid-X receptors of the blue crab,Callinectes sapidus: cloning and their expression patterns in eyestalks and Y-organs during the molt cycle. Gene, 527(1): 139—153
Wang S F, Ayer S, Segraves W Aet al, 2000. Molecular determinants of differential ligand sensitivities of insect ecdysteroid receptors. Molecular and Cellular Biology, 20:3870—3879
Wu X G, Cheng Y X, Zeng C Set al, 2010a. Reproductive performance and offspring quality of wild-caught and pond-reared swimming crab (Portunus trituberculatus)brookstock. Aquaculture, 301:78—64
Wu X G, Cheng Y X, Zeng C Set al, 2010b. Reproductive performance and offspring quality of the first and the second brood of female swimming crab (Portunus trituberculatus)brookstock. Aquaculture, 303: 94—100
Wu X H, Hopkins P M, Palli S Ret al, 2004. Crustacean retinoid-X receptor isoforms: distinctive DNA binding and receptor-receptor interaction with a cognate ecdysteroid receptor. Molecular and Cellular Endocrinology, 218: 21—38
Yang F, Xu H T, Dai Z Met al, 2005. Molecular characterization and expression analysis of vitellogenin in the marine crabPortunus trituberculatus. Comparative Biochemistry, 142(4):456—464
