舟山海域一株产碱性蛋白酶海洋放线菌的鉴定、选育及发酵条件的初步研究*
2014-03-19苗增良王健鑫
李 鹏 苗增良 王健鑫
(浙江海洋学院海洋科学与技术学院 舟山 316022)
碱性蛋白酶(Alkaline protease)是指在pH在碱性条件下水解蛋白质肽键的酶类, 广泛存在于动、植物及微生物体中, 是一类非常重要的工业用酶。碱性蛋白酶应用很广泛, 在洗涤剂、丝绸、饲料、饲料、制革、食品、医药、环保等领域广泛应用(Bhaskaret al,2007; Wanget al, 2008; Jellouliet al, 2009), 在全世界范围内占工业酶制剂市场的65%以上(Baniket al,2004), 具有很重要的工业和经济价值(Guptaet al,2002)。微生物由于生长速度快、生长条件较简单、代谢过程特殊等特点, 使之成为蛋白酶的重要来源。微生物蛋白酶均为胞外酶, 其从微生物中提取, 不受资源、环境和空间的限制, 具有动物蛋白酶和植物蛋白酶所不可比拟的优越性(梅承芳等, 2005)。
目前对碱性蛋白酶的研究主要集中在3个方面:通过诱变、培养条件优化、异源表达等提高酶产量;分子结构研究和定点、定向突变以提高酶的性能; 筛选新型碱性蛋白酶和产酶菌株。在筛选新型碱性蛋白酶和产酶菌株方面, 近年来已报道了具有较高pH适应性的碱性蛋白酶(郝建国等, 2010), 水解多种底物的碱性蛋白酶(肖昌松等, 2001), 碱性弹性蛋白酶(Kumaret al, 1999), 具有耐表面活性剂、耐热、耐氧化剂等特性的碱性蛋白酶等(Banerjeeet al, 1999;Johnveslyet al, 2001)。现阶段所报道的产酶菌株主要为芽孢杆菌属(Bacillus)。碱性蛋白酶仍然存在品种单一的问题, 酶的活性不高, 价格昂贵等不足, 加上各国都十分注重对已有的重要微生物资源的保护, 使得优质产酶菌株的筛选和选育有着十分重要的研究前景。
海洋微生物与陆生微生物相比其生存环境更加恶劣, 如高渗、低温、高压、低营养等, 也造就其独特的代谢途径, 因此海洋微生物所产蛋白酶可能具有更加独特的酶学性质和构造。
Sreeja等(2011)从海洋微生物Engyodontium albumBTMFS10中分离到碱性丝氨酸蛋白酶, 该酶除了具有很好的耐高温(60°C酶活最高)、嗜碱(pH 11时酶活最高)以外, 还具有很好的稳定性, 如其在烃类、天然油脂、表面活性剂及大部分的有机溶剂中均表现很好的稳定性, 在洗涤剂工业具有很好的应用前景。
Anjali等(2014)从海洋微生物Bacillus tequilensisP15中发现耐受绝大部分有机溶剂的碱性蛋白酶, 试验结果显示该酶对于去除衣物上的血渍、脱毛及除去感光胶片上的明胶具有很好的效果。
国产碱性蛋白酶主要在酶活、成本和酶学性质等方面不能满足工业发展的要求, 大规模工业用碱性蛋白酶主要还依赖于进口, 希望通过本次试验研究,能发现高产蛋白酶的海洋微生物, 并为下一步的研究, 如: 发酵条件的优化、蛋白酶基因的克隆、工程菌的构造等方面的研究打下基础。
1 材料与方法
1.1 实验材料
分别在浙江舟山的三江码头、鸭蛋山码头、东极岛、朱家尖、东港等地的潮间带采集海水、海泥。
1.1.1 培养基
(1) 高氏一号改良培养基: 可溶性淀粉20g,KNO31g, K2HPO40.5g, MgSO4⋅7H2O 0.5g, NaCl 0.5g,FeSO40.01g, 琼脂20g, 人工海水1000mL, pH 7.4—7.6。
(2) 脱脂奶培养基(沈萍等, 1999): 牛肉膏5.0g,蛋白胨10.0g, 氯化钠1.0g, 琼脂20.0g, pH值7.2—7.4,加热溶解于海水中, 121°C高压灭菌20min, 备用。脱脂牛奶10.0g, 溶解于海水中, 115°C灭菌30min。两者在无菌环境下混匀得到1000mL的培养基。
(3) 酪素琼脂改良培养基: 酪蛋白4g, CaCl20.002g, MgSO4⋅7H2O 0.5g, K2HPO4⋅7H2O 1.07g, KH2PO40.36g, ZnCl20.014g, NaCl 0.16g, FeSO40.0002g, 水解干酪素0.05g, 琼脂18g, 人工海水1000mL, pH 8.0。
(4) 放线菌发酵培养基, 分别记为1—6号:
① 淀粉5g, 黄豆粉10g, KH2PO41g, 人工海水1000mL, pH 8.5。
② 胰蛋白胨10g, 酵母膏5g, 玉米粉5, Na2HPO4⋅12H2O 4g, KH2PO40.3g, Na2CO31g, 人工海水1000mL,pH 8.5。
③ 酪蛋白10.0g, 牛肉膏3.0g, NaCl 5.0g, K2HPO42.0g, 人工海水1000mL, pH 8.5。
④ 可溶性淀粉4.0%, 酵母膏0.5%, 牛肉膏1.0%蛋白胨1.5%, NaCl 0.5%, K2HPO40.35%, NaH2PO40.03%, Na2CO30.056%, 人工海水1000mL, pH 8.5(沈萍等, 1999)。
⑤ 蛋白胨5.0g, 酵母膏3.0g, 葡萄糖5.0g, 人工海水 1000mL, pH 8.5 (周德庆, 2006)。
⑥ 牛肉膏5.0g, 蛋白胨10.0g, NaCl 1.0g, 脱脂奶10.0g, 人工海水1000mL, pH 8.5。
1.1.2 实验试剂 细菌基因组DNA提取试剂盒(TIANamp BacteriaDNA Kit)由天根生化科技(北京)有限公司提供, 常规化学试剂DNA Marker购自TaKaRa公司, 其余均购于国药集团。
1.2 实验方法
1.2.1 菌株的分离与纯化 将采样得到的潮间带海泥进行梯度稀释, 在高氏I号培养基平板上涂布培养, 按照菌落形态、大小、颜色等不同挑选单菌落在高氏I号培养基平板上划线, 倒置25°C培养3—5d,继续挑取单菌落, 重复划线。重复上述步骤2—3次,共得到纯菌株50株。
1.2.2 目标菌株的筛选 将分离纯化得到的放线菌在脱脂奶平板上进行点种, 25°C培养, 观察有无透明圈的产生。
1.2.3 酶活测定 将筛选到的菌株接种于放线菌发酵培养基50/250mL摇瓶中进行培养, 温度25°C,转速120r/min培养3—5d后测酶活。
1.2.3.1 标准曲线的制作 用酪氨酸配置0—100μg/mL的标准溶液, 酪氨酸在275nm下有特征光吸收, 通过建立酪氨酸浓度与该浓度下的酪氨酸在275nm下光吸收关系的标准曲线, 通过紫外分光光度法测定碱性蛋白酶促蛋白质消化反应的生成物在275nm下的吸光度, 就可以计算反应生成酪氨酸的量,根据酶活力的定义计算碱性蛋白酶的活力。按表1制备不同浓度的酪氨酸溶液。
用分光光度计于680nm波长下测各管的光吸收值(A), 进行测量, 用“1”号管作为空白对照, 以于680nm波长下测各管的A值为纵坐标, 每管的酪氨酸含量为横坐标, 绘制酪氨酸的标准曲线。
1.2.3.2 碱性蛋白酶活力的测定 将目标菌株发酵酶液进行5000r/min离心去除菌体后, 取粗酶液适当稀释后按照表2进行酶活测定, 每种样品做2次平行试验。其中试管1为空白对照。具体步骤见表2。
蛋白酶活力: 以酪蛋白为底物, 每分钟水解产生1μg酪氨酸的酶量为1个酶活力单位。

式中,Up表示蛋白酶活力,A680表示由样品测得的吸光度值,K为吸光系数(从标准曲线中得到), 4为反应试剂的总体积, 10为反应时间,n为稀释倍数。
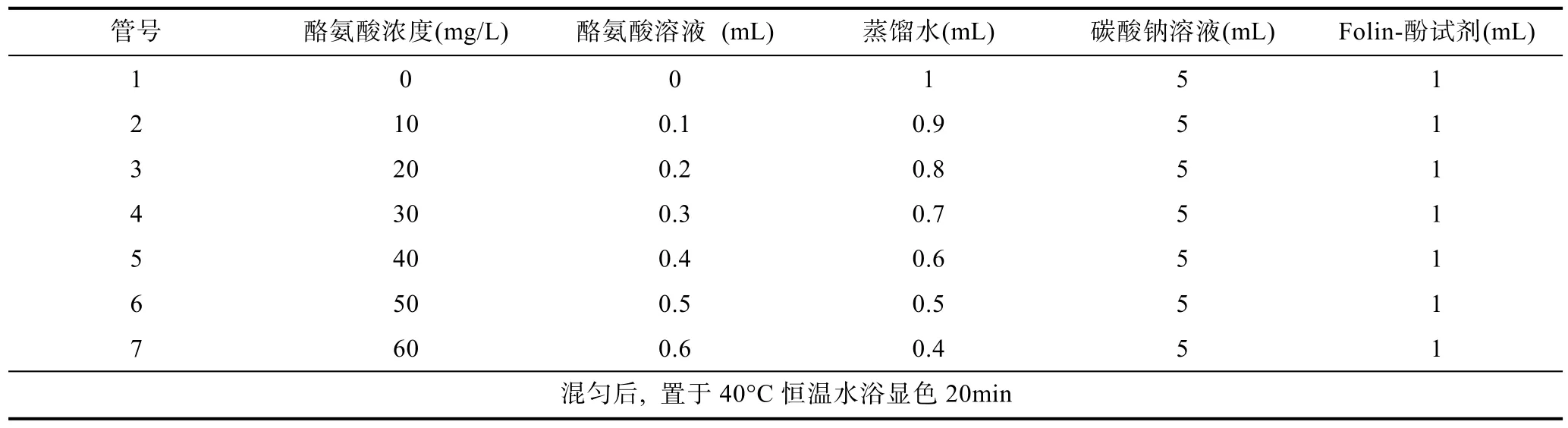
表1 标准曲线的制作Tab.1 Data for making standard curves

表2 测样品酶活的步骤Tab.2 Steps of measuring enzyme activity in the samples
1.2.4 菌株形态观察 利用插片法对目标菌株进行形态观察。
1.2.5 菌株A20的生理生化试验 采用不同的糖作为碳源, 对目标菌株的碳源利用情况进行观察。将筛选得到的目标菌株进行常规项目的生理生化实验,观察结果。
1.2.6 菌株A20的16S rDNA分子鉴定 用细菌基因组DNA 提取试剂盒提取目标菌株的DNA, 以总DNA为模板进行PCR扩增(Kunamneniet al, 2003),PCR产物经过1%琼脂糖凝胶电泳检测分析, PCR产物纯化试剂盒回收1500bp的PCR产物, 产物以Seq Forward、Seq Reverse、Seq Internal为引物进行16S rDNA测序, 测序工作由宝生物工程(大连)有限公司完成。
1.2.7 菌株A20的系统进化树的绘制 利用Blast对得到的序列在数据库中进行相似序列的搜索, 获得与目标菌株相似性较高的菌株(97%以上相似性),对同源性较好的序列进行比对(Clustalx 1.8), 绘制系统进化树(PhyloDraw V8.0)。
1.2.8 不同发酵培养基对菌株A20的酶活影响分别采用淀粉培养基、胰蛋白胨培养基、蛋白胨培养基等六种培养基进行发酵试验, 得到最适产酶培养基。
1.2.9 菌株的诱变 先将目标菌株进行紫外诱变, 获得的高产株再分别进行DES诱变, 获得最终的高产菌株。
1.2.9.1 紫外诱变 取2mL的菌悬液放入6cm培养皿中, 垂直照射距离为30cm。先盖盖子照射1min,打开盖子照射处理, 同时用磁力搅拌器进行搅拌, 处理时间为30、60、90、120、150、180s。在各个处理时间点将处理的菌液取100μL用无菌生理盐水进行梯度稀释, 稀释至10–3浓度, 取0.1mL在脱脂奶培养基上进行涂布, 3个重复, 同时对未经紫外处理的对照菌液也进行稀释后平板涂布, 计算致死率, 致死率 =(1-诱变处理后每0.1mL菌落数/对照每0.1mL菌落数)×100%。涂布后的平板在暗处培养3—5d后观察, 选取HC比值(透明圈直径/菌落直径)较对照明显大的单菌落进行发酵, 25°C, 120r/min, 50mL/ 250mL装量, 做3个平行, 测其酶活。
1.2.9.2 DES诱变 挑取经紫外诱变后的斜面保种突变菌株于液体高氏1号培养基, 50mL/250mL接种, 25°C、120r/min培养5d。诱变处理将处于对数生长期的菌液在室温下5000r/min离心10min, 弃上清液, 收集菌体。用10mL 0.1 mol/L 磷酸缓冲液(pH 7.0)洗涤2次, 然后重悬于pH 7.0的磷酸缓冲液中, 调整菌液浓度为108个/mL(调整OD600= 0.5左右即可)。移取4mL菌液到16mL磷酸钾缓冲溶液(pH 7.0)中,加入0.2mL硫酸二乙酯, 分别振荡处理10、20、30、40、50、60min, 最后加入0.5mL 25%硫代硫酸钠中止反应。将诱变处理10、20、30、40、50、60min的菌液稀释到10–3稀释度, 做3个重复, 取0.1mL平板涂布, 以未经化学诱变处理的出发菌为对照, 25°C恒温培养箱中培养5d (邱雁临等, 2008)。将培养好的平板取出, 菌落计数, 计算致死率。挑取HC比值大的单菌落进行发酵(同时斜面保种), 做3个平行重复,测其酶活。
1.2.9.3 稳定性试验 将诱变得到的高产菌株连续传代10代, 测其酶活。
1.2.10 最适温度的选择 将诱变得到的菌株分别在28、37、45、50、55、60°C下摇瓶培养, 分别测酶活。
1.2.11 最适pH选择 将诱变得到的菌株分别接种于pH为7.5、8、8.5、9、9.5、10的发酵培养液中,分别测酶活。
1.2.12 最适培养时间选择 将诱变得到的菌株分别接种于pH 7.5的发酵培养液中, 28°C分别培养24、36、48、60、72、84、96h。分别测酶活。
2 结果与分析
2.1 筛选结果
得到2株产较大透明圈的菌株, 分别记A8和A20。筛选结果如图1和图2所示。透明圈大小情况如表3所示。
2.2 酶活测定结果

图1 菌株A8平板图Fig.1 Shape of Strain A8
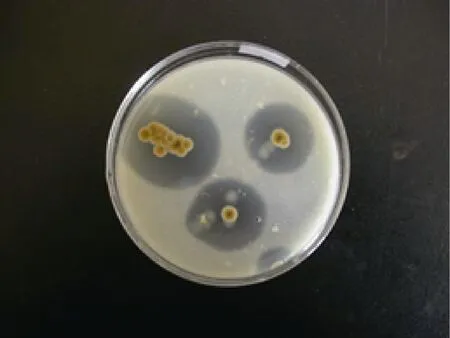
图2 菌株A20平板图Fig.2 Shape of Strain A20

表3 两株放线菌所形成透明圈直径大小Tab.3 The size of diameter of transparent zone
2.2.1 标准曲线的制作 用100μg/mL的酪氨酸标准溶液配制标准系列溶液, 在OD680nm处进行测定,并绘制标准曲线。不同含量的酪氨酸的吸光度值结果见表4。
以光吸收值(A)为纵坐标, 酪氨酸含量为横坐标,绘制酪氨酸的标准曲线, 见图3。回归方程y=0.0088x-0.0111, 相关系数R2= 0.9977, 具有较好的线性关系。

表4 不同含量的酪氨酸吸光度值结果Tab.4 Results of different levels of tyrosine absorbance
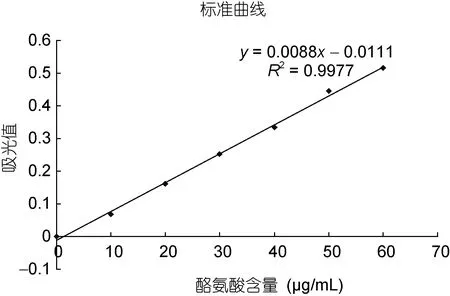
图3 酪氨酸标准曲线Fig.3 Standard curve of tyrosine
2.2.2 目标菌株酶活测定 对菌株A8、A20进行两组平行测定酶活, 最后取平均值作为最终结果, 如表5所示。
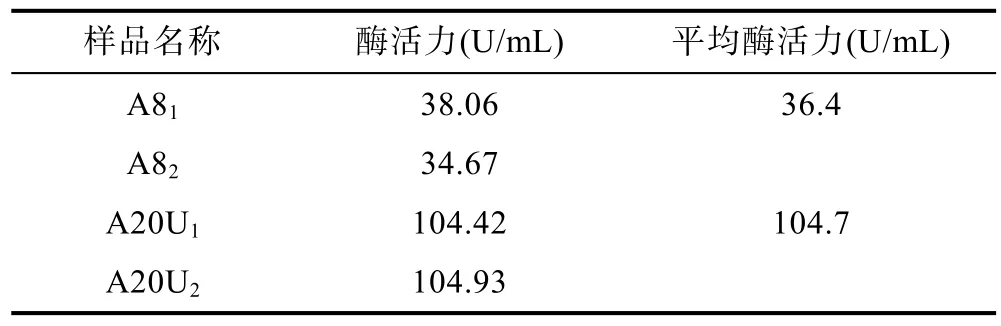
表5 碱性蛋白酶活力测量结果Tab.5 Results of alkaline protease activity
菌株A20的酶活较高, 因此选取菌株A20作为研究对象, 进行紫外线和DES诱变处理以获得高产菌株。
2.3 菌株A20的形态观察
利用插片法观察A20形态, 菌丝呈链状, 断裂,无卷曲状分布。显微观察结果如图4所示。
2.4 菌株A20的生理生化试验
菌株A20在不同培养基条件下的生长3d情况如表6所示, 对不同碳源的利用情况如表7所示, 生理生化结果如表8所示。
由生理生化试验结果可知, 菌株A20可以利用除了除了鼠李糖之外的常见碳源, 并都能使硝酸盐还原, 还具有水解淀粉的能力。
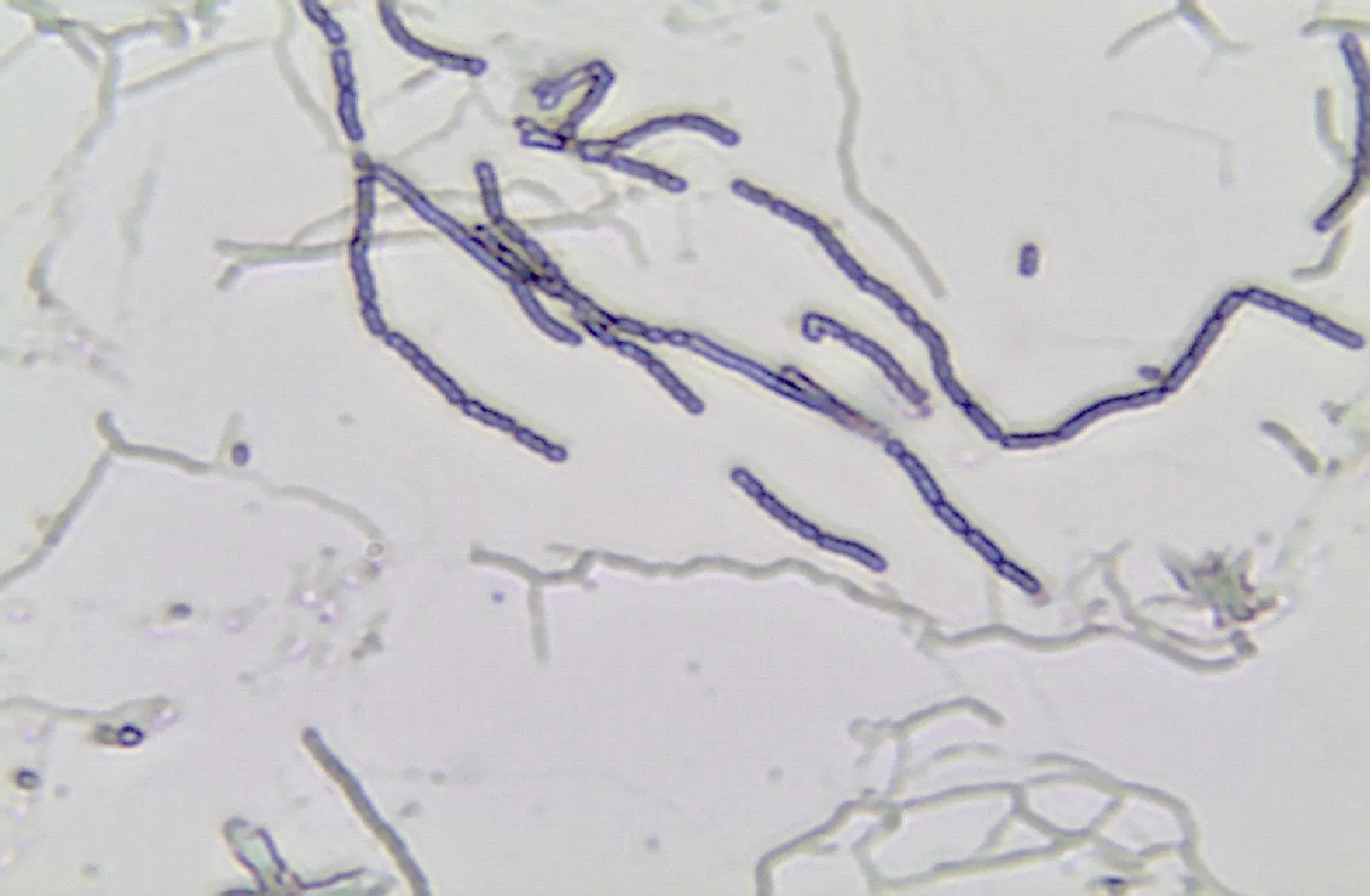
图4 菌株A20显微观察图Fig.4 Hyphal morphology of Strain A20
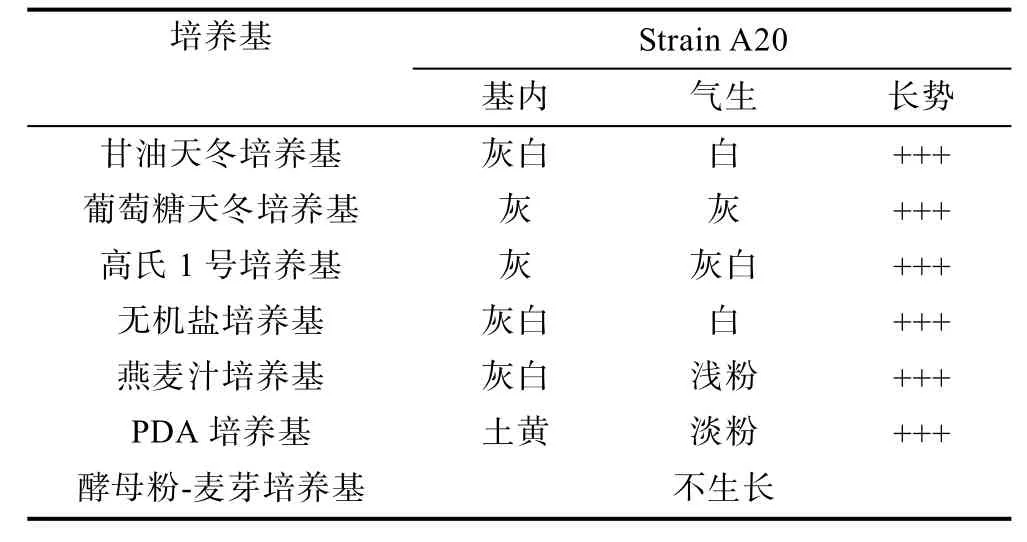
表6 菌株在不同培养基上培养3d的生长情况Tab.6 Strain A20 in different culture media on the growth after 3d

表7 菌株A20对不同碳源的利用情况Tab.7 Strain A20 on the utilization of different carbon sources

表8 菌株A20的生理生化试验结果Tab.8 Physiological and biochemical tests on Strain A20
2.5 16S rDNA序列分析结果
16S rDNA分子鉴定结果显示菌株A20的核苷酸长度为1361bp, 将测序得到的核苷酸序列上传至NCBI的GenBank数据库获得登录号为GU263841。
2.6 系统进化树绘制
利用Blast软件, 从GenBank数据库中查找同源性较高的菌株, 来构建系统进化树, 系统进化树如图5所示。系统进化树显示菌株A20与Streptomyces roseoviridisJS-9(玫瑰绿链霉菌)和Streptomyces roseusJS-18(浅玫瑰色链霉菌)的进化位置最近, 相似性达99.8%, A20不产水溶性色素, 并在燕麦汁、PDA培养基中培养时产生浅玫瑰色菌落, 菌株A20很可能为一种浅玫瑰色链霉菌。
2.7 不同发酵培养基对菌株A20的酶活影响
在1—6号不同发酵培养基培养条件下菌株A20的酶活大小如表9所示。结果显示1号培养基(淀粉培养基)培养条件下酶活较大。因此诱变均在1号培养基作为发酵培养基条件下进行。
2.8 诱变结果
2.8.1 紫外诱变结果 紫外诱变致死率情况如表10所示, 结果显示紫外线处理时间应控制在60—150s。
在120s处理的平板上得到HC比值最大的菌株(HC比值为5.3)进行发酵培养, 最终得到酶活为182.8U/mL。相比于初始酶活104.7U/mL, 酶活提高74.6%, 并将得到的菌株进行DES诱变。

图5 根据16S rDNA序列构建的菌株A20以及相关种、属的系统进化树Fig.5 The NJ evolutionary tree based on completed sequence of Strain A20 16S rDNA
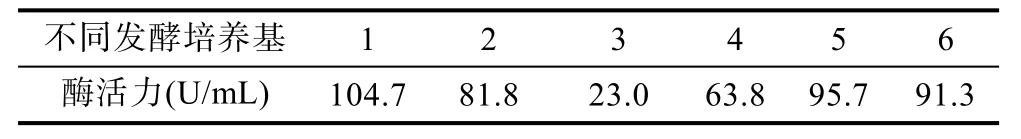
表9 碱性蛋白酶活力测量结果Tab.9 Alkaline protease activity measurement results
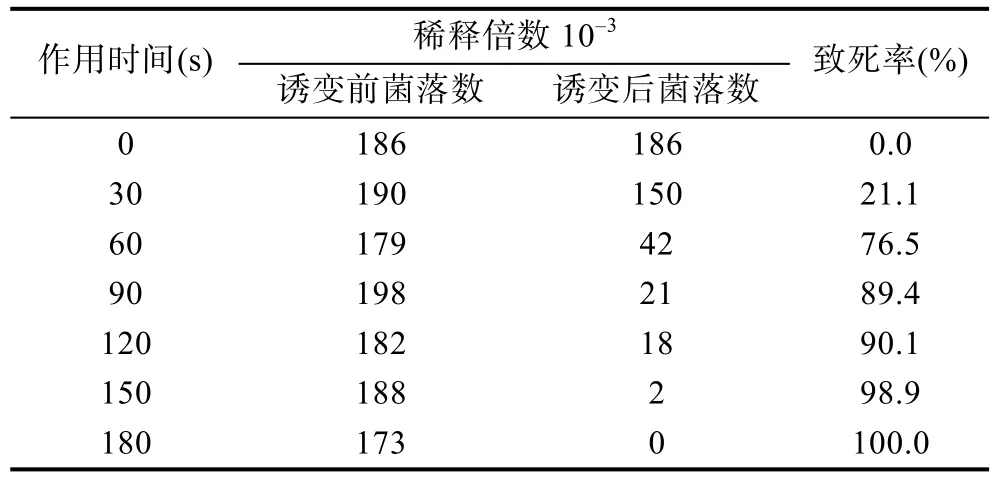
表10 紫外线诱变结果Tab.10 The result of ultraviolet mutation
2.8.2 DES诱变结果 DES诱变结果致死率如表11所示, 结果显示处理时间应控制在30—50min。

表11 DES处理结果Tab.11 The result of DES mutation
在处理50min的平板上获得HC比值最大的菌株(HC比值为5.9), 进行发酵培养, 最终得到酶活为227.5U/mL, 酶活提高51.8%。
2.9 传代试验
将DES处理过的酶活为227.5U/mL的A20菌株进行传代试验, 转接种10次, 测试酶活结果如表12所示。

表12 传代10次的酶活测试结果Tab.12 Results of enzyme activity test after ten times of subculture
连续传代10代结果显示酶活最低为第10代的208.9U/mL, 酶活下降8.17%, 酶活下降较少, 诱变后的菌株较为稳定。
2.10 最适温度
各温度下酶活测试结果如图6所示。结果显示发酵温度为50°C具有最高酶活, 酶活为227.9U/mL。
2.11 最适pH值
各pH下酶活如图7所示。结果显示当pH 9.0时酶活最高。

图6 最适温度试验Fig.6 The optimal temperature test

图7 最适pH试验Fig.7 The optimal pH test
2.12 最适培养时间
培养时间为36h之前为菌体生长阶段, 此时产酶较少, 酶活较低, 当培养时间超过48h以后, 菌体进入产酶阶段, 酶活逐渐增加, 当培养时间为72h时, 酶活最高, 为228.5U/mL。详见图8。

图8 最适培养温度试验Fig.8 Testing for the optimal culture time
3 讨论
从舟山潮间带海域中筛选得到产碱性蛋白酶的放线菌菌株A20, 生理生化试验和16S rDNA分析结果显示该菌株为Streptomyces roseus, 记为Streptomyces roseusA20, 该菌株在淀粉-黄豆粉发酵培养基中酶活较高, 这可能与黄豆粉属于慢速利用氮源有关。紫外诱变显示在处理时间为60—120s, 致死率为76.5%—90.1%时得到正突变株, 此时得到的诱变菌株透明圈明显较大, 经测定酶活为182.8U/mL, 与初始菌株相比酶活提高74.6%。再经过30—50min DES诱变处理,致死率为68.8%—93.8%时得到正突变株, 酶活为227.5U/mL, 比初始酶活104.7U/mL提高117.3%, 诱变效果较为明显。传代试验显示突变株具有较好的遗传稳定性, 在连续传代10次测定酶活发现该酶酶活无明显下降。单因素试验结果显示在发酵温度为50°C、pH值为9.0, 培养时间为72h时候酶活最高。
利用物理或者化学方法进行诱变育种是一种常规的获取高产菌株的方法, 尽管其具有一定的优点,如: 能够提高突变率, 在较短的时间内获得更多的优良变异类型。但是, 诱变育种也存在一些问题, 如:有益突变频率仍然较低, 变异的方向和性质难控制。因此提高诱变效率, 迅速鉴定和筛选突变体以及探索定向诱变的途径, 是当前研究的重要课题。
复合诱变的效果往往要好于单种诱变剂的诱变效果, 这可能是由于先用物理诱变剂处理以后, 微生物细胞细胞膜的完整性或者渗透性有所改变, 然后再用化学试剂进行处理, 就能更好的与细胞染色体位点进行接触从而使得诱变效率增加。Ahmed等(2013)先后用伽马辐射和EMS复合诱变菌株Penicillium chrysogenumNRRL 792, 得到产最高酶活诱变体菌株EMS-1, 试验结果还显示突变体菌株所产碱性蛋白酶的稳定性(pH, 热稳定性)要大大好于野生型。电泳结果显示野生型与突变型的蛋白酶分子量大小也不同, 诱变结果改变了碱性蛋白酶的分子结构。
目前诱变育种已经与基因工程进行很好的结合,从而获得更加稳定而高产的突变菌株。Wang等(2010)从嗜冷产碱性蛋白酶海洋微生物菌株Pseudomonassp.中, 利用反向PCR技术克隆该酶的结构基因, 发现1443bp的OFR编码一组无信号肽的463氨基酸, 序列比对结果显示该酶属于丝氨酸类型的金属蛋白酶,重组蛋白在Escherichia coli成功进行表达。Jasmin等(2010)从海洋真菌Engyodontium album中克隆碱性蛋白酶基因(Eap), 并对其三维结构进行了推测。
碱性蛋白酶主要在洗涤工业上有广泛的应用。尤其新型的耐氧化、耐高温、耐碱性、耐高盐度、耐有机溶剂、及在低温环境下具有较高酶活的一类碱性蛋白酶更是其中研究的热点。
Tsuyoshi等(2004)研究了枯草杆菌产碱性蛋白酶的抗氧化性稳定性, 并应用于洗涤剂行业。Kunamneni等(2003)研究了枯草芽孢杆菌PE-11产碱性蛋白酶的热稳定性, 发现该酶在60°C处理了350min后仍保持100%酶活力。Sudhir等(2010)在一株新颖的嗜高温菌株Paenibacillus tezpurensissp. nov. AS-S24-II发酵液中发现清洁稳定性良好的碱性蛋白酶。WANG等(2012)在印尼温泉中筛选到一株产碱性蛋白酶菌株Brevibacillussp. PLI-1, 试验结果显示在70°C时该酶具有最高活性。
Raja等(2006)对耐有机溶剂的碱性蛋白酶进行了酶学性质研究, 利用硫酸铵盐析及层析方法纯化酶,酶活力提高了近124倍。Subramani等(2009)筛选到一株海洋放线菌MML1614, 发现其具有很好的耐热与耐碱性, 与洗涤剂结合后使洗涤效果大幅增加。Hames-Kocabas等(2007)从海泥样品中筛选得到碱性蛋白酶生产菌株MA1-1, 在pH 9.0、50°C有较高酶活, 同时鉴定出此碱性蛋白酶属于丝氨酸基团。Jignasha等(2009)在印度沿海筛选到一株嗜碱放线菌Mit-1, 其产碱性蛋白酶不仅耐盐耐高温(70°C最适宜温度), 且耐受有机溶剂, 此类蛋白酶的报道极为罕见。Iram等(2012)在克什米尔地区的冰川土壤中筛选到一株食单胞菌属(Stenotrophomonassp.)菌株, 该菌株所产碱性蛋白酶在较低温度(15°C)时具有最高酶活, 而低温清洗能对一些高档丝绸织物起到很好的保护作用。
在某些方面, 蛋白酶甚至被用在回收利用废弃的贝壳类动物中(Anil, 2010), 在处理蓖麻壳的浪费上(Madhuriet al, 2012)也能得到应用。Feng等(2013)在菌株Kocuria kristinaeF7的发酵液中分离到一种很罕见的碱性丝氨酸蛋白酶, 该蛋白酶除了耐盐耐高温, 还能改善大豆蛋白质水解产物的口味, 在食品工业中具有较好的应用前景。蛋白酶可以被用作脱毛剂使用, Vijay等(2011)从Bacillus altitudinisGVC11分离到一种新颖的丝氨酸蛋白酶, 试验发现该酶作用于山羊毛具有很好的脱毛效果, 却不会损坏羊毛中的胶原蛋白也不影响羊毛的完整性。
影响菌株产酶的因素有很多, 如: 温度、pH、碳源、氮源、培养温度、盐度、溶氧(转速)等等, 选择合理的粗酶分离纯化过程, 从而很好保持甚至提高其酶活也非常重要。因此, 如何优化培养方案、选择合适的分离纯化工艺, 将是后续研究的重点。
肖昌松, 吕 健, 田新玉, 2001. 嗜碱芽孢杆菌XE22-4-1碱性弹性蛋白酶发酵条件的研究. 微生物学报, 41(5): 611—616
邱雁临, 梁 亮, 王 亮, 2008. 紫外线与氯化锂复合诱变选育L-组氨酸产生菌. 现代食品科技学报, 03: 217—219
沈 萍, 范秀容, 李广武, 1999. 微生物学实验. 北京: 高等教育出版社, 45—46
周德庆, 2006. 微生物学实验教程. 北京: 高等教育出版社, 75—76
郝建国, 薛燕芬, 马延和, 2010. 一株产蛋白酶嗜碱菌株的分离、鉴定及酶学特性. 微生物学报, 50(1): 54—59
梅承芳, 江晓路, 牟海津, 2005. 碱湖高产碱性蛋白酶菌的选育和产酶条件研究. 中国海洋大学学报, 35(4): 613—617
Ahmed F Afifi, Heba I Abo-Elmagd, Manal M Housseiny, 2013.Improvement of alkaline protease production byPenicillium chrysogenumNRRL 792 through physical and chemical mutation, optimization, characterization and genetic variation between mutant and wild-type strains. Ann Microbiol, 35: 147—156
Anil Kumar Singh H S, 2010. Optimization of protease production byStreptomycessp. A6 using statistical approach for reclamation of shellfish waste. World J Microbiol Biotechnol, 26: 1631—1639
Anjali Bose, Vishal Chawdhary, Haresh Keharia, 2014. Production and characterization of a solvent-tolerant protease from a novel marine isolateBacillus tequilensisP15. Ann Microbiol, 64: 343—354
Banerjee U C, Sani R K, Azmi W, 1999. Thermostable alkalineprotease fromBacillus brevisand its characterizationas a laundry detergent additive. Process Biochemistry, 35(1/2): 213—219
Banik R M, Prakash M, 2004. Laundry detergent compatibility of the alkaline protease fromBacillus cereus. Microbiol Res,159: 135—140
Bhaskar N, Sudeepa E S, Rashmi H Net al, 2007. Partial purification and characterization of protease ofBacillus proteolyticusCFR3001 isolated from fish processing waste and its antibacterial activities. Bioresour Technol, 98: 2758—2764
Feng Zhen, Chen Xi, Li Juan-juanet al, 2013. An alkaline protease fromKocuria kristinaeF7: properties and characterization of its hydrolysates from soy protein. Eur Food Res Technol, 236: 293—301
Gupta R, Beg Q K, Lorenz P, 2002. Bacterial alkaline proteases:molecular approaches and industrial applications. Applied Microbiology and Biotechnology, 59(1): 15—32
Hames-Kocabas E Esin, Atac Uzel, 2007. Alkaline protease production by an actinomycete MA1-1 isolated from marine sediments. Annals of Microbiology, 57(1): 71—75
Iram Saba Parvaiz H, Qazi Shabir A, Rather Refaz Aet al, 2012.Purification and characterization of a cold active alkaline protease fromStenotrophomonassp., isolated from Kashmir,India. World J Microbiol Biotechnol, 28: 1071—1079
Jasmin C, Sreeja Chellappan, Rajeev K Sukumaranet al, 2010.Molecular cloning and homology modeling of a subtilisin-like serine protease from the marine fungus,Engyodontium albumBTMFS10. World J Microbiol Biotechnol, 26: 1269—1279
Jellouli K, Bougatef A, Manni Let al, 2009. Molecular and biochemical characterization of an extracellular serineprotease fromVibrio metschnikoviiJ1. J Ind Microbiol Biotechnol, 36: 939—948
Jignasha T Thumar, Satya P Singh, 2009. Organic solvent tolerance of an alkaline protease from salt-tolerant alkaliphilicStreptomyces clavuligerusstrain Mit-1. J Ind Microbiol Biotechnol, 36: 211—218
Johnvesly B, Naik G R, 2001. Studies on production of thermostable alkaline protease from thermophilic and alkaliphilicBacillussp. JB-99 in a chemically defined medium. Process Biochemistry, 37(2): 139—144
Kumar C G, Tiwari M P, Jany K D, 1999. Novel alkaline serineproteases from alkalophilicBacillusspp. purification and some properties. Process Biochemistry, 34(5): 441—449
Kunamneni Adinarayana, Poluri Elllaiah, Davuluri Siva Prasad,2003. Purification and partial characterization of thermostable serine alkaline protease from a newly isolatedBacillus sublitisPE-11. AAPS Pharm Sci Tech, 4(4): 65—72
Madhuri A, Nagaraju B, Harikrishna Net al, 2012. Production of Alkaline Protease byBacillus altitudinisGVC11 using Castor Husk in Solid-State Fermentation. Appl Biochem Biotechnol, 167: 1199—1207
Raja Noor Zaliha Raja Abd Rahman, Lee Poh Geok, Mahiran Basri, 2006. An organic solvent-stable alkaline protease fromPseudomonas aeruginosastrain K: Enzyme purification and characterization. Enzyme and Microbial Technology, 7: 1484—1491
Sreeja Chellappan C, Jasmin Soorej M, Basheer Archana Kishore Ket al, 2011. Characterization of an extracellular alkaline serine protease from marineEngyodontium albumBTMFS10.Microbiol Biotechnol, 38: 743—752
Subramani Ramesh, Mahalingam Rajesh, Narayanasamy Mathivanan, 2009. Characterization of a thermostable alkaline protease produced by marineStreptomyces fungicidicusMML1614. Bioprocess Biosyst Eng, 32: 791—800
Sudhir K Rai, Jetendra K Roy, Ashis K Mukherjee, 2010.Characterisation of a detergent-stable alkaline protease from a novel thermophilic strainPaenibacillus tezpurensissp. nov.AS-S24-II. Appl Microbiol Biotechnol, 85: 1437—1450
Tsuyoshi Nonaka, Masahiro Fujihashi, Akiko Kita Katsuhisa,2004. The crystal structure of an oxdatively-stable subtilisin alkaline serine protease, KP-43, with a C-terminal β-barrel domain. J Biol Chem, 45: 47344—47351
Vijay Kumar E, Srijana M, Kiran Kumar Ket al, 2011. A novel serine alkaline protease fromBacillus altitudinisGVC11 and its application as a dehairing agent. Bioprocess Biosyst Eng,34: 403—409
Wang Fang, Hao Jianhua, Yang Chengyeet al, 2010. Cloning,expression, and identification of a novel extracellular cold-adapted alkaline protease gene of the marine bacterium strain YS-80-122. Appl Biochem Biotechnol, 162: 1497—1505
Wang S L, Yang C H, Liang T Wet al, 2008. Optimization of conditions for protease production byChryseobacterium taeanenseTKU001. Bioresour Technol, 99: 3700—3707
WANG Shuai, LIN Xuezheng, HUANG Xiaohanget al, 2012.Screening and characterization of the alkaline protease isolated from PLI-1, a strain ofBrevibacillussp. collected from Indonesia, hot springs. Oceanic and Coastal Sea Research, 11(2): 213—218
