双齿围沙蚕Cu/Zn-SOD cDNA基因的克隆及序列分析
2014-02-08岳宗豪樊鑫赵欢任洪伟张旭峰周一兵
岳宗豪,樊鑫,赵欢,任洪伟,张旭峰,周一兵
(大连海洋大学辽宁省海洋生物资源恢复与生境修复重点实验室,辽宁大连 116023)
双齿围沙蚕Cu/Zn-SOD cDNA基因的克隆及序列分析
岳宗豪,樊鑫,赵欢,任洪伟,张旭峰,周一兵
(大连海洋大学辽宁省海洋生物资源恢复与生境修复重点实验室,辽宁大连 116023)
根据已知多毛类Alitta succinea铜锌超氧化物歧化酶 (Cu/Zn-SOD)基因序列设计引物,利用同源克隆及RACE方法首次从双齿围沙蚕Perinereis aibuhitensis中克隆得到Cu/Zn-SOD基因全长cDNA序列。结果表明:双齿围沙蚕Cu/Zn-SOD基因cDNA全长870 bp,其中包括156 bp的5'端非编码区,261 bp 3'端非翻译区和453 bp开放阅读框,编码150个氨基酸;该蛋白序列具有典型的Cu2+和Zn2+结合位点,并具有两处Cu/Zn-SOD蛋白家族标签序列。通过生物信息学分析表明,该蛋白属于胞内Cu/Zn-SOD,理论相对分子质量为15 249 900,等电点为5.66,无信号肽和跨膜区,推测为亲水性蛋白。同源性分析表明,双齿围沙蚕Cu/Zn-SOD氨基酸序列与部分软体动物、鱼类和昆虫的Cu/Zn-SOD蛋白序列具有很高的相似性。该研究结果为后续研究Cu/Zn-SOD与环境污染物之间的剂量效应关系奠定了基础,为研究双齿围沙蚕机体的防御机制提供了基础资料。
双齿围沙蚕;铜锌超氧化物歧化酶;生物信息学分析;同源性分析
铜锌超氧化物歧化酶 (copper-zinc superoxide dismutase,Cu/Zn-SOD)属于超氧化物歧化酶家族,广泛存在于真核和原核生物中。Cu/Zn-SOD一般是由2个基本相似的亚基组成的二聚体,且每个亚基含有1个铜原子和1个锌原子[1]。其中铜与催化活性有关,铜的缺失将会导致其活性完全丧失,进而危害人类和动物体的健康,产生多种疾病[2-5]。Cu/Zn-SOD的主要功能是清除体内多余的超氧阴离子自由基,保护机体免受氧化损伤。由于Cu/Zn-SOD的存在,健康水生动物体内的活性氧自由基能够维持在有利无害的较低水平[6]。此外,Cu/Zn-SOD还与免疫有关。Sook等[7]研究发现,注射脂多糖的螃蟹Callinectes sapidus体内Cu/Zn-SOD被显著诱导,表明Cu/Zn-SOD与免疫防御系统有关。Bao等[8]研究发现,对海湾扇贝Argopecten irradians注射鳗弧菌后,其血细胞中Cu/Zn-SOD表达水平增加,表明Cu/Zn-SOD在对鳗弧菌的免疫反应中可能起着重要作用。
双齿围沙蚕Perinereis aibuhitensis隶属于环节动物门Annelida、多毛纲Polychaeta,是广泛分布于中国沿海潮间带及河口潮滩的优势底栖动物,属于重要的经济多毛类[9]。由于其生活在极易富集污染物的沉积质中,更容易受到污染物的影响,所以双齿围沙蚕常作为污染物暴露的指示生物。目前,已经在软体动物[10-12]、节肢动物[13-14]、脊索动物[15-16]等中获得了 Cu/Zn-SOD 序列,但有关双齿围沙蚕Cu/Zn-SOD序列的研究国内外尚未见报道。
从分子水平深入开展沙蚕Cu/Zn-SOD基因克隆研究,有助于探索污染物在沙蚕体内的代谢途径及其解毒机理,并为进一步研究双齿围沙蚕的生态毒理学提供基础依据和新思路。本研究中,利用同源克隆及RACE方法首次从双齿围沙蚕体壁组织中克隆得到Cu/Zn-SOD基因,并对该基因进行了生物信息学分析。
1 材料与方法
1.1 材料
试验用双齿围沙蚕采自辽宁省盘锦市双台子河口,挑选体质量为1.5~2.5 g的个体,带回辽宁省海洋生物资源恢复与生境修复重点实验室暂养一周。暂养期间,每24 h更换一次海水,水温保持在 (19±0.5)℃,盐度为31~32,pH为8.25±0.10。试验海水中 Cu(Ⅱ)、Hg(Ⅱ)、Zn(Ⅱ)、Pb(Ⅱ)和Cd(Ⅱ)的背景浓度分别为0.38、0.01、75.00、0.27、0.06 μg/L。
1.2 方法
1.2.1 总RNA的提取 剪取0.1 g双齿围沙蚕体壁放入预冷灭菌研钵中,加入液氮将组织迅速研磨至粉末状,按照RNAisoTMPlus(TaKaRa公司)说明书中的相关步骤提取总RNA。将提取的总RNA溶于DEPC处理水中,于-80℃下保存备用。用10 g/L琼脂糖凝胶电泳检测总RNA的完整性,使用超微量分光光度计测定其浓度。
1.2.2 cDNA第一链的合成 以提取的双齿围沙蚕总RNA为模板,按照Reverse Transcriptase MMLV(RNase Hˉ)反转录试剂盒 (TaKaRa公司)说明书进行第一链的合成。cDNA第一链合成反应条件如下:70℃下反应10 min,放到冰上急冷2 min,然后在42℃下反应60 min,70℃下反应15 min。
1.2.3 引物设计 依据已知多毛类Alitta succineaCu/Zn-SOD基因 (HM563679.1)CDS保守区设计简并引物,利用同源克隆获得双齿围沙蚕Cu/Zn-SOD基因的特异性片断。根据得到的Cu/Zn-SOD特异性片段设计3'RACE和5'RACE引物。UPM由Clontech SMARTTM RACE cDNA Amplification Kit试剂盒提供,其余引物利用Primer 5.0软件设计,由TaKaRa公司合成。本试验中所用引物见表1。
1.2.4 特异性片段扩增 按照TaKaRa Ex TaqⓇHot Start Version试剂盒说明书,利用设计的简并引物进行特异性片段的扩增。特异性片段PCR反应体系共 25 μL,包含:TaKaRa Ex Taq(5 U/μL)0.2 μL,10 × Ex PCR Buffer(Mg2+plus)2.5 μL,dNTP Mixture(2.5 mmol/L)2.0 μL,引物各 0.5 μL,模板 2.0 μL,ddH2O 17.3 μL。反应条件如下:94℃下预变性2 min;94℃下变性30 s,55℃下退火30 s,72℃下延伸30 s,共进行35个循环;最后在72℃下再延伸10 min。取4.0 μL PCR产物用10 g/L琼脂糖凝胶电泳检测,用TIANgel Midi Purification Kit切胶回收试剂盒切胶回收目的条带。回收后的产物连接到pMDTM18-T Vector(TaKaRa公司)载体上,连接产物热转化至DH5α感受态细胞中,然后涂布于含有Amp(Ampicillin)的LB平板培养基上,37℃下倒置培养12~16 h。挑选独立的较大白色单菌落至LB液体培养基 (含Amp)中,于37℃下培养4 h,取1.0 μL菌液进行菌落PCR检测,选取阳性克隆送上海生物工程有限公司测序。
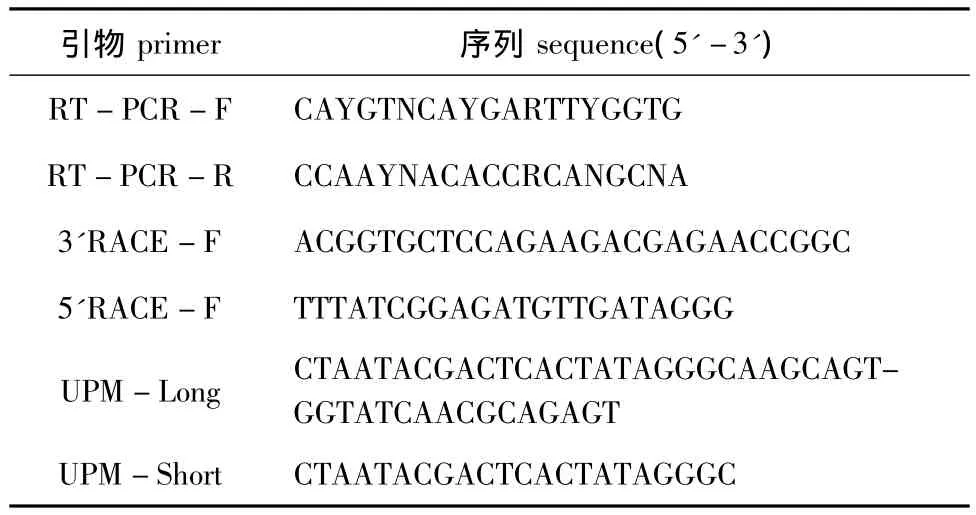
表1 双齿围沙蚕Cu/Zn-SOD基因全长扩增引物Tab.1 The primers used in the amplication of Cu/Zn-SOD in sandworm Perinereis aibuhitensis
1.2.5 RACE扩增 按照Clontech SMARTTMRACE cDNA Amplification Kit说明书进行3'RACE和5'RACE扩增。3'RACE和5'RACE PCR反应体系共50 μL,包含:10 ×Adantage 2 PCR Buffer 5.0 μL,dNTP Mix 1.0 μL,50 × Adantage 2 Polymerase Mix 1.0 μL,3'RACE -Ready cDNA(或5'RACE -Ready cDNA)2.5 μL,3'RACE(或5'RACE)引物 1.0 μL,10 × UPM Mix 5.0 μL,ddH2O 34.5 μL。反应条件如下:94℃下变性30 s,72℃下延伸3 min,共进行5个循环;94℃下变性30 s,70℃下退火30 s,72℃下延伸2 min,共进行5个循环;94℃下变性30 s,68℃下退火30 s,72℃下延伸2 min,共进行25个循环。取4.0 μL PCR产物用10 g/L琼脂糖凝胶电泳检测,利用“1.2.4”节的方法进行切胶回收和转化连接。挑取阳性克隆送上海生物工程有限公司测序。利用DNAStar软件对中间片段、3'RACE和5'RACE测序结果进行拼接。
1.2.6 序列分析 应用DNAStar软件查找双齿围沙蚕Cu/Zn-SOD基因开放阅读框 (ORF),并将其翻译成氨基酸序列;利用Conserved Domains软件预测氨基酸序列保守区域,使用ScanProsite软件分析氨基酸序列的潜在修饰位点,使用ProtParam工具预测氨基酸序列的理化性质,使用PSORTⅡ软件进行细胞内定位预测,使用Signal P Server软件进行信号肽预测,使用TMHMM软件进行跨膜区分析,使用ProtScale工具进行疏水性分析,使用SOPMA软件和SWISS-MODEL软件分别预测氨基酸序列的二级和三级结构;使用Clustal 2.1进行双齿围沙蚕Cu/Zn-SOD多重序列比对,使用Mega 5.0软件构建同源关系进化树,自展检测重复为1000次。
2 结果与分析
2.1 特异性片段扩增及RACE扩增
依据已知多毛类Alitta succineaCDS保守区设计简并引物,同源克隆得到326 bp的特异性片段。3'RACE扩增获得一条512 bp的条带,5'RACE PCR产物经切胶回收测序为453 bp。使用DNAStar软件进行序列拼接,得到870 bp的双齿围沙蚕cDNA全序列,提交至 GenBank,登录号为KF199864。
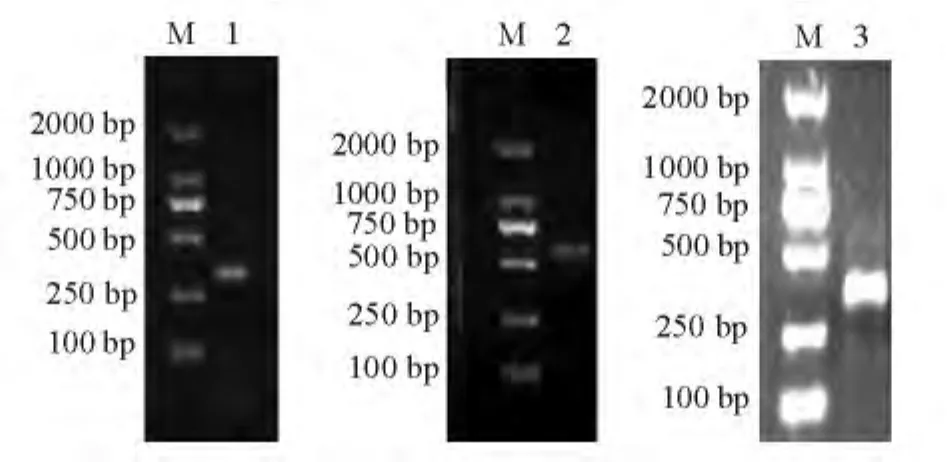
图1 双齿围沙蚕Cu/Zn-SOD特异性片段扩增及RACE扩增结果Fig.1 RACE amplification product of Cu/Zn-SOD fragment from sandworm Perinereis aibuhitensis
2.2 生物信息学分析
2.2.1 双齿围沙蚕Cu/Zn-SOD基本理化信息分析 双齿围沙蚕Cu/Zn-SOD cDNA基因全长870 bp,其中5'端非翻译区 (5'UTR)为156 bp,3'端非翻译区 (3'UTR)为261 bp,开放阅读框(ORF)为453 bp,编码一个由150个氨基酸组成的蛋白质 (图2)。使用ProtParam工具推测Cu/Zn-SOD氨基酸序列分子式为C655H1043N195O217S4,原子数为2114,理论相对分子质量为15 249 900,等电点为5.66,氨基酸序列中带负电荷的氨基酸(Asp+Glu)17个,占11.33%,带正电荷的氨基酸 (Arg+Lys)11个,占7.33%。根据ProtScale预测结果,Cu/Zn-SOD氨基酸疏水性最大值为2.200,最小值为-2.644,整个多肽链中亲水性氨基酸多于疏水性氨基酸,推断该蛋白为亲水性蛋白。
2.2.2 双齿围沙蚕Cu/Zn-SOD亚细胞定位分析预测结果显示,该蛋白位于细胞质中的可能性为65.2%,位于细胞核内的可能性为21.7%,在其他细胞器中的可能性较小。对Cu/Zn-SOD蛋白进行信号肽的预测,未发现明显的信号肽位点,也未发现该蛋白有跨膜区,表明Cu/Zn-SOD蛋白既不是跨膜蛋白也不是分泌蛋白。
2.2.3 双齿围沙蚕Cu/Zn-SOD功能区预测 通过Conserved Domains预测氨基酸序列保守区域发现,该蛋白具有典型的Cu2+结合位点 His-43、His-45、His-59和His-117,以及Zn2+结合位点His-59、His-68、His-77和 Asp-80(图 2)。此外,该氨基酸序列还含有两处 Cu/Zn-SOD蛋白家族标签序列:GFHVHQFGDNT(41~51)和GNAGGRLACGVI(135~146)(图2)。
2.2.4 双齿围沙蚕Cu/Zn-SOD二级和三级结构预测 对双齿围沙蚕Cu/Zn-SOD基因编码的蛋白二级结构进行预测,结果显示:α-螺旋 (Alpha helix)占整个蛋白质的4.67%,无规则卷曲(Random coil)占54.67%,延伸链 (Extended strand)占32.00%,而β-转角 (Beta turn)占8.67%。Cu/Zn-SOD三级结构预测结果见图3。
2.2.5 双齿围沙蚕Cu/Zn-SOD同源序列比对及系统进化分析 同源性比较结果表明,双齿围沙蚕Cu/Zn-SOD与琥珀刺沙蚕Alitta succinea同源性最高,达79%,与部分软体动物、鱼类和昆虫也具有较高的同源性,同源性为71%~78%。其中与太平洋牡蛎Crassostrea gigas同源性为78%,与日本盘鲍Haliotis discus discus同源性为76%,与庞贝蠕虫Alvinella pompejana同源性为75%,与日本鳗鲡Anguilla japonica同源性为72%,与烟粉虱Bemisia tabaci同源性为71%(图4)。系统进化分析结果表明,双齿围沙蚕和琥珀刺沙蚕首先聚为一簇,表明其亲缘关系较近 (图5)。
3 讨论

图2 双齿围沙蚕Cu/Zn-SOD cDNA基因序列及推导的氨基酸序列Fig.2 Nucleotide and deduced amino acid sequence of Cu/Zn-SOD cDNA in sandworm Perinereis aibuhitensis
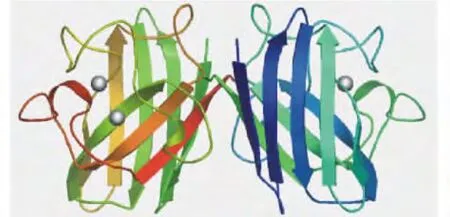
图3 双齿围沙蚕Cu/Zn-SOD三级结构预测图Fig.3 Predication of the tertiary structure of Cu/Zn SOD from sandworm Perinereis aibuhitensis

图4 双齿围沙蚕Cu/Zn-SOD的氨基酸序列与其他物种蛋白氨基酸序列的比对Fig.4 Comparison of deduced amino acid sequence alignment of Cu/Zn-SOD in the sandworm with that in the other species
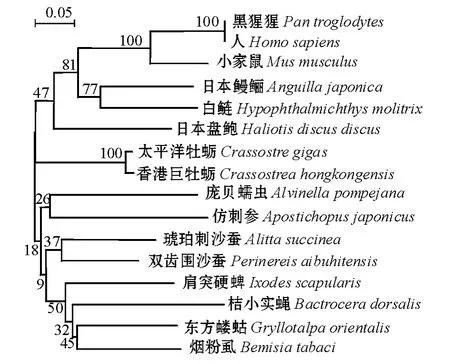
图5 双齿围沙蚕Cu/Zn-SOD的系统进化树Fig.5 Phylogenetic tree of Cu/Zn-SOD from sandworm Perinereis aibuhitensis
本研究中,首次报道了双齿围沙蚕Cu/Zn-SOD基因的cDNA序列全长。序列分析结果显示,双齿围沙蚕Cu/Zn-SOD cDNA全长870 bp,包括156 bp的5'端非翻译区,261 bp的3'端非翻译区和453 bp的阅读框,编码150个氨基酸。Rhee等[17]克隆并分析琥珀刺沙蚕Cu/Zn-SOD序列时发现,该序列包含4个Cu2+结合位点 (His-45、His-47、His-62和 His-119)和4个 Zn2+结合位点 (His-62、His-70、His-79和 Asp-82),同时还具有两个Cu/Zn-SOD特征序列 (GFHVHEFGDNT、GNAGGRLACGVI)。Wang 等[18]在紫贻贝Mytilus galloprovincialisCu/Zn-SOD蛋白序列中也发现,具有Cu2+、Zn2+结合位点和特征序列。本研究中,在获得的双齿围沙蚕SOD序列中亦发现了4个Cu2+结合位点 (His-43、His-45、His-59和His-117)和4个 Zn2+结合位点 (His-59、His-68、His-77和 Asp-80),并确定具有两处Cu/Zn-SOD蛋白家族标签序列 (41~51,135~146)。因此,推断该序列为双齿围沙蚕Cu/Zn-SOD序列。此外,Cu/Zn-SOD氨基酸序列具有色氨酸和酪氨酸缺乏,甘氨酸含量较高的特点[19-20]。对获得的序列进行分析发现,不存在色氨酸和酪氨酸,而甘氨酸的比例达到16.7%,这进一步表明获得的序列为双齿围沙蚕Cu/Zn-SOD。
生物体内含有胞外和胞内两种Cu/Zn-SOD序列,其中胞外Cu/Zn-SOD通常含176~251个氨基酸残基,N端具有信号肽序列;而胞内Cu/Zn-SOD含147~167个氨基酸残基,N端无信号肽序列[21-22]。本研究中获得的双齿围沙蚕 Cu/Zn -SOD蛋白质序列包含150个氨基酸,且N端无信号肽,细胞定位预测其存在于细胞质中的可能性为65.2%,故推测其为胞内Cu/Zn-SOD序列。
多序列同源比对分析结果,通过NJ法进行系统进化树分析。结果显示,多毛类双齿围沙蚕和琥珀刺沙蚕首先聚为一簇,然后与东方蝼蛄、肩突硬蜱、桔小实蝇等昆虫纲聚为一簇,再与软体动物、脊椎动物哺乳类和鱼类聚在一起。这说明Cu/Zn-SOD基因在物种中具有高度保守性,并且在一定程度上是按照该基因所属物种的进化而进化的。但是,姜冰等[6]和朱丹等[23]的研究表明,Cu/Zn-SOD的系统进化并不严格按照该基因所属物种的进化而进化。这可能是由于物种差异造成的,有的物种中Cu/Zn-SOD是以物种进化而进化的,而有的则不是。
赵双菁等[24]研究表明,2.000 ~ 8.653 μL/L Pb2+能够上调水丝蚓Limnodilus claparedianuSOD酶活性,12.481 μL/L Pb2+则显著抑制 SOD酶活性。据Kim 等[25]报道,1 μg/L 4- 壬基酚 (4-NP)能够上调日本虎斑猛水蚤Tigriopus japonicusCu/Zn-SOD和 Mn-SOD mRNA的表达。鉴于SOD对环境污染物的敏感性,利用SOD作为分子生物标记已经成为了近年来的研究热点。双齿围沙蚕Cu/Zn-SOD基因的克隆目前尚属首次,双齿围沙蚕Cu/Zn-SOD基因的获得及对该序列的分析,为研究其体内免疫和机体防御机制提供了基础资料,也为Cu/Zn-SOD作为生物标志物指示和监测环境污染奠定了基础。
[1] 张立颖,赵萌,王跃智.水生生物超氧化物岐化酶的研究进展[J].江西农业大学学报,2012,34(4):800 -804.
[2] Mavelli I,Ciriolo M R,Rossi L,et al.Favism:a haemolytic disease associated with increased superoxide dismutase and decreased glutathione peroxidase activities in red blood cells[J].European Journal of Biochemistry,1984,139(1):13 -18.
[3] Brown D R,Schmidt B,Groschup M H,et al.Prion protein expression in muscle cells and toxicity of a prion protein fragment[J].European Journal of Cell Biology,1998,75(1):29 -37.
[4] Noor R,Mittal S,Iqbal J.Superoxide dismutase - applications and relevance to human diseases[J].Medical Science Monitor,2002,8(9):210-215.
[5] Anju A,Jeswin J,Thomas P C,et al.Molecular cloning,characterization and expression analysis of cytoplasmic Cu/Zn-superoxide dismutase(SOD)from pearl oyster Pinctada fucata[J].Fish &Shellfish Immunology,2013,34(3):946 -950.
[6] 姜冰,鲍相渤,张明,等.虾夷扇贝铜锌超氧化物歧化酶基因的克隆与转录表达特性分析[J].生物技术通报,2011,12:150-156.
[7] Sook Chung J,Bachvaroff T R,Trant J,et al.A second copper zinc superoxide dismutase(CuZnSOD)in the blue crab Callinectes sapidus:cloning and up-regulated expression in the hemocytes after immune challenge[J].Fish & Shellfish Immunology,2012,32(1):16-25.
[8] Bao Y,Li L,Wu Q,et al.Cloning,characterization,and expression analysis of extracellular copper/zinc superoxide dismutase gene from bay scallop Argopecten irradians[J].Fish & Shellfish Immunology,2009,27(1):17 -25.
[9] 李颖,周一兵,万良,等.双齿围沙蚕Hsp70 cDNA基因的克隆及序列分析[J].大连海洋大学学报,2012,27(6):502 -507.
[10] Kim K Y,Lee S Y,Cho Y S,et al.Molecular characterization and mRNA expression during metal exposure and thermal stress of copper/zinc-and manganese superoxide dismutases in disk abalone,Haliotis discus discus[J].Fish & Shellfish Immunology,2007,23(5):1043 -1059.
[11] Zhang G,Fang X,Guo X,et al.The oyster genome reveals stress adaptation and complexity of shell formation[J].Nature,2012,490:49-54.
[12] Ni D,Song L,Gao Q,et al.The cDNA cloning and mRNA expression of cytoplasmic Cu,Zn superoxide dismutase(SOD)gene in scallop Chlamys farreri[J].Fish & Shellfish Immunology,2007,23(5):1032-1042.
[13] Lyu K,Zhu X,Wang Q,et al.Copper/Zinc superoxide dismutase from the cladoceran Daphnia magna:molecular cloning and expression in response to different acute environmental stressors[J].Environmental Science & Technology,2013,47(15):8887-8893.
[14] Cheng W,Tung Y H,Liu C H,et al.Molecular cloning and characterisation of copper/zinc superoxide dismutase(Cu,Zn - SOD)from the giant freshwater prawn Macrobrachium rosenbergii[J].Fish & Shellfish Immunology,2012,21(1):102 -112.
[15] Zhang Z W,Li Z,Liang H W,et al.Molecular cloning and differential expression patterns of copper/zinc superoxide dismutase and manganese superoxide dismutase in Hypophthalmichthys molitrix[J].Fish & Shellfish Immunology,2011,30(2):473 -479.
[16] Chakravarthy N,Aravindan K,Kalaimani N,et al.Intracellular copper zinc superoxide dismutase(icCuZnSOD)from Asian seabass(Lates calcarifer):molecular cloning,characterization and gene expression with reference to Vibrio anguillarum infection[J].Developmental & Comparative Immunology,2012,36(4):751-755.
[17] Rhee J S,Won E J,Kim R O,et al.Expression of superoxide dismutase(SOD)genes from the copper- exposed polychaete,Neanthes succinea[J].Marine Pollution Bulletin,2011,63(5/12):277-286.
[18] Wang Q,Yuan Z Y,Wu H F,et al.Molecular characterization of a manganese superoxide dismutase and copper/zinc superoxide dismutase from the musselMytilus galloprovincialis[J].Fish &Shellfish Immunology,2013,34(5):1345 -1351.
[19] 包永波.海湾扇贝超氧化物歧化酶家族基因结构表达和多态性分析[D].青岛:中国科学院海洋研究所,2009.
[20] 田春美,钟秋平.超氧化物歧化酶的现状研究进展[J].中国热带医学,2005,5(8):1730 -1732.
[21] Lin Y C,Vaseeharan B,Chen J C.Identification of the extracellular copper-zinc superoxide dismutase(ecCuZnSOD)gene of the mud crab Scylla serrata and its expression following β-glucan and peptidoglycan injections[J].Molecular Immunology,2008,45(5):1346-1355.
[22] Parker J D,Parker K M,Keller L.Molecular phylogenetic evidence for an extracellular Cu Zn superoxide dismutase gene in insects[J].Insect Molecular Biology,2004,13(6):587 -594.
[23] 朱丹,李宏俊,高祥刚,等.文蛤胞内铜锌超氧化物歧化酶基因的克隆与序列分析[J].生物技术通报,2010(11):123-128.
[24] 赵双菁,李艳秋,柳郁滨,等.Pb2+对水丝蚓的急性毒性及超氧化物歧化酶活性的影响[J].中国农学通报,2012,28(8):87-89.
[25] Kim B M,Rhee J S,Park G S,et al.Cu/Zn - and Mn -superoxide dismutase(SOD)from the copepod Tigriopus japonicus:molecular cloning and expression in response to environmental pollutants[J].Chemosphere,2011,84(10):1467 -1475.
Cloning and sequence analysis of Cu/Zn-SOD cDNA from sandworm Perinereis aibuhitensis
YUE Zong-hao,FAN Xin,ZHAO Huan,REN Hong-wei,ZHANG Xu-feng,ZHOU Yi-bing
(Key Laboratory of Marine Bio-resources Restoration and Habitat Reparation in Liaoning Province,Dalian Ocean University,Dalian 116023,China)
A full length cDNA of Cu/Zn-SOD was firstly cloned in sandwormPerinereis aibuhitensisby homology cloning and RACE techniques based on the partial copper-zinc superoxide dismutase(Cu/Zn-SOD)gene from polychaeteAlitta succinea.The full length of the cDNA was found to be 870 bp including a 156 bp 5'untranslated region,a 261 bp 3'untranslated region and 453 bp open reading frame encoding 150 amino acids.There were typical Cu2+and Zn2+binding sites as well as two Cu/Zn-SOD protein family tag sequences in the deduced protein which was within the intracellular Cu/Zn-SOD with relative molecular mass of 15 249 900 and the isoelectric point of 5.66 by bioinformatic analysis.No signal peptide and transmembrane domain were observed in the deduced protein,indicating that it belonged to the hydrophilic protein.Multiple sequences alignment analysis revealed that the deduced amino acids had high homology to the proteins of partial molluscs,fishes and insects.The findings will provide basis for research of dose-response between gene expression and environmental pollutants,and defense mechanism of the sandworm.
Perinereis aibuhitensis;Cu/Zn-SOD;bioinformatic analysis;homology analysis
Q78;S917
A
10.3969/J.ISSN.2095-1388.2014.04.006
2095-1388(2014)04-0354-06
2013-12-03
国家海洋公益性行业科研专项 (201305002,201305043);国家海洋局重点实验室开放基金资助项目 (201217);国家自然科学基金资助项目 (41306138)
岳宗豪 (1989—),男,硕士研究生。E-mail:yzh642324521@163.com
周一兵 (1957—),男,教授。E-mail:ybzhou@dlou.edu.cn
