海洋动物Toll样受体的研究进展
2013-12-23孙红娟周遵春崔军王秀利
孙红娟 周遵春 崔军 王秀利
(1.大连海洋大学水产与生命学院,大连 116023; 2.辽宁省海洋水产科学研究院 辽宁省海洋水产分子生物学重点实验室,大连 116023)
动物界中有大约80%的动物,共25个门类生活在海洋中。对海洋低等无脊椎动物免疫系统的研究有助于人类探索先天性免疫系统的起源,对硬骨鱼免疫系统的研究可以展现脊椎动物先天性免疫系统的进化途径。先天性免疫系统作为机体的第一道防线,主要通过细胞表面的模式识别受体(Pattern recognition receptor,PRR)特异地识别病原相关分子模式(Pathogen-associated molecular pattern,PAMP)。例如,蛋白、糖类、脂质、核酸及这些生物大分子的衍生物[1]。TLR是一个在进化上高度保守的免疫受体家族,属于I型跨膜蛋白,主要由胞外区,跨膜区和胞质区组成。TLR胞外区是由亮氨酸重复序列(Leucine-rich repeat,LRR)形成的螺线管状结构,根据LRRs结构差异可以将TLR分为:脊椎动物类TLR(Vertebrate-like TLR,V-TLR)和原口动物类TLR(Protostome-like TLR,P-TLR)(图1)。包括后口动物在内的V-TLR胞外区含有一组“LRRNTLRRs-LRRCT”(N-terminal LRR,LRRNT; C-terminal LRR,LRRCT)序列,P-TLR 胞外区含有一组额外的LRRCT-LRRNT基序[2,3]。TLR胞质区是一个与IL-1(Interleukin-1)受体家族具有高度同源性的TIR(TLR and IL-1 receptor domain)结构,能够募集含有TIR结构的接头蛋白参与信号转导。目前已经发现了5种接头蛋白包括:MyD88(Myeloid differentiation primary response gene 88)、MAL(MyD88 adaptorlike)、TRIF(TIR-domain containing adaptor inducing IFN-β)、TRAM(TRIF related adaptor molecule)和SARM(Sterile α- and armadillo-motif-containing protein),表明机体在受到病原体刺激后会产生与特定接头蛋白相对应的免疫应答[4]。TLR信号途径主要是MyD88依赖途径,由胞内接头蛋白MyD88介导,下 游 的IRAK(IL-1 receptor associated kinase)和TRAF(TNF receptor associated factor)依次参与到信号传递过程,最后NF-кB(Nuclear factor Kappa B)因子被激活而产生免疫应答。另一种是针对TLR3和TLR4的MyD88非依赖途径,这两个受体能通过TRIF进行信号转导。TLR3能和TRIF直接接触,而TLR4通过TRAM将信号传递给TRIF,最终都激活转录因子IRF(Interferon regulatory factor)[5,6](图2)。
第一个Toll蛋白是在果蝇体内发现的,其不仅对果蝇生长发育具有调控作用,还参与免疫反应[7]。随后在人体内也发现了TLR[8],研究热点迅速转向了哺乳动物。到目前为止,从最原始的后生动物海绵到高等的鸟类,共在59个物种中发现了TLR[9],其中脊椎动物体内至少存在23种TLRs。为阐明脊椎动物TLR的起源,对占据过渡地位的棘皮动物、头索动物、尾索动物和无颌脊椎动物TLR的研究是至关重要的。本文按照海洋动物由低到高的进化顺序对TLR及其信号通路相关分子进行介绍(表1),并对TLR基因在进化过程中表现出的结构变化和多态性进行探讨。
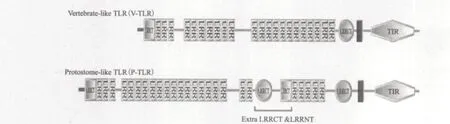
图1 TLR的两种结构类型[2]

图2 TLR信号通路[6]
1 不同海洋动物中TLR及其信号通路分子
1.1 海绵动物
海绵动物代表着最原始的后生动物类群,是研究先天性免疫系统起源的理想对象。早在1907年,Wilson[10]研究发现,硅质海绵(Siliceous sponge)可以识别非己成分,并且体内存在大量免疫分子。近年来对寻常海绵纲中两个物种TLR的研究为揭示后生动物TLR家族的起源具有重要意义。

表1 海洋动物中已发现的TLR及信号通路分子
2007年,Wiens等[11]在居蟹皮海绵(Suberites domuncula)体内发现了3个和脊椎动物高度同源的免疫应答元件:类TLR、IRAK4和细胞凋亡蛋白。类TLR的胞外区仍没有明显的LRRs结构,TIR区和脊椎动物表现出高度的同源性,推测居蟹皮海绵体内的类TLR只是脊椎动物TLR的一部分。在受到LPS刺激后,居蟹皮海绵细胞表面特有的LIP(LPS-interacting protein)可以识别LPS(Lipopolysaccharide)与MyD88结合启动下游信号途径。2010年,Srivastava等[12]构建了澳大利亚大堡礁海绵(Amphimedon queenslandica)的基因组草图。尽管没有检测到TLR,但是发现两个含有TIR区的受体以及TLR信号通路中的MyD88和Tollip(Toll interacting protein)。这4个蛋白在胚胎时期和幼虫时期的动态表达说明它们参与了发育和免疫过程[13]。
1.2 刺胞动物
刺胞动物又叫腔肠动物,包括海蜇、水螅、海葵、珊瑚等。它和两侧对称动物处于平行的进化分支上,是研究两侧对称动物免疫系统起源的重要参考模型。目前已经完成对大乳头水螅(Hydra magnipapillata)、星海葵(Nematostella vectensis)和指轴孔珊瑚(Acropora digitifera)的基因组测序。刺胞动物的形态结构比较简单,但它们的免疫系统仍包含着复杂的信息。在星海葵和指轴孔珊瑚中发现了TLR以及胞内信号通路成分NF-κB[14]。虽然在大乳头水螅体内没有发现典型的TLR以及NF-κB ,但是鉴定得到一个Rel的同源物和4种含有TIR区的蛋白,功能研究表明它们都参与免疫应答[15]。通过3种刺胞动物体内TLR、IL-1受体以及只含有TIR区蛋白的结构比对发现它们在指轴孔珊瑚体内要比星海葵和大乳头水螅中的复杂[16]。
1.3 软体动物
软体动物处于原口动物进化的初始阶段,以贝类为主。免疫学的研究发现双壳类体内存在保守的进化途径,基因多态性和基因丢失几率相对较低,适于研究原口动物免疫系统的起源和进化。目前已经完成了太平洋长牡蛎(Crassostrea gigas)、虾夷扇贝(Patinopecten yessoensis)和贻贝(Mytilus edulis)这3种双壳类动物的大规模转录组测序,并对地中海贻贝(Mytilus galloprovincialis)数据库中免疫相关基因进行了系统分析。在太平洋长牡蛎[17]、虾夷扇贝[18]和地中海贻贝[19]中都已经发现了两个TLR片段,然而在贻贝体内发现了27个可能编码TLR的基因,其中有8个可翻译成具有完整的TLR结构的蛋白[20]。这一结果表明在双壳类体内存在着复杂的先天性免疫系统。
另外,在栉孔扇贝(Chlamys farreri)体内鉴定到一个Toll(CfToll-1)和TLR信号通路的4个关键基因,即MyD88、TRAF6、IкB(Inhibitor of Kappa B)和NF-кB。经过LPS刺激后,它们的表达量都上升。这些结果表明,栉孔扇贝体内存在保守的MyD88依赖途径,可以激活下游抗氧化、抗菌和细胞凋亡等反应[21]。在太平洋牡蛎(Crassostrea gigas)体内除了克隆到一个全长Toll(CgToll-1),还发现了5个信号通路的核心基因,即MyD88、IRAK、TRAF6、IкB和Rel[22]。
1.4 海洋节肢动物
节肢动物门是动物界中最大的一个门,鲎和虾是海洋节肢动物免疫学研究的重点对象。
在中华鲎(Tachypleus tridentatus)血细胞中鉴定到的Toll和果蝇Toll的同源性最高,胞外域LRR上没有明显的PAMP结合位点,也没有组织表达特异性。这些结果表明颗粒状血细胞表面的Toll不具有识别LPS的功能[23]。随后在圆尾鲎(Carcinoscorpius rotundicauda)体内发现了TLR的接头分子SARM,在结构上和人体中SARM具有同源性[24]。到目前为止,在对虾中已经鉴定到6个TLR,其中3个都是在南美白对虾(Litopenaeus vannamei)中发现的,分别命名为LvToll、LvToll2和LvToll3[25]。最先发现的LvToll具有P-TLR的典型结构。在受到溶藻弧菌(Vibrio alginolyticus)刺激后,3个基因的表达量发生了变化,推测它们具有免疫功能。第四个TLR基因是在斑节对虾(Penaeus monodon)鳃的cDNA文库中检测到的,不具有组织表达特异性[26]。在中国对虾(Fenneropenaeus chinensis)和日本囊对虾(Marsupenaeus japonicus)体内分别发现了一个Toll基因[27,28],它们在结构和表达规律上与南美白对虾中的Tolls相似。最近在斑节对虾中又克隆得到一个Toll,它比第一次克隆到的Toll序列长[29]。
1.5 棘皮动物
棘皮动物是后口动物中进化地位最高的无脊椎动物,位于无脊椎动物与脊椎动物进化分支阶段,是研究两侧对称动物TLR进化过程中的重要物种。
海胆属于棘皮动物海胆纲,是组织胚胎学研究领域的模式生物。2006年完成了对紫海胆(Strongylocentrotus purpuratus)基因组的测序,发现了222个TLR基因(214个V-TLR和8个P-TLR),同时还发现了26个编码TLR接头蛋白的基因[30]。仿刺参(Apostichopus japonicus)属于棘皮动物中的海参纲,与海胆纲处于平行的进化分支。对仿刺参免疫基因的深入研究[31-34]有助于揭示棘皮动物先天性免疫系统的多态性。通过对仿刺参的转录组测序,将为遗传和基因组学研究提供更多信息[35,36]。在本实验室的转录组测序结果中发现了一些TLR片段,并且首次克隆得到两个TLR的cDNA全长,同时仿刺参体内也存在保守的MyD88信号通路成分(数据未发表)。
1.6 头索动物
脊索动物门是动物界最高等的门,共分为3个亚门:尾索动物亚门、头索动物亚门和脊椎动物亚门。系统进化分析表明头索动物是现存最古老的脊索动物,代表动物是文昌鱼[37]。2003年,美国文昌鱼(Branchiostoma floridae)的基因组草图绘制完成,检测到48个完整的TLR基因,由36个V-TLRs和12个P-TLRs组成[2]。同时,发现了40个含有TIR接头蛋白,包括MyD88、SARM、TIRAP和TICAM2等。根据TIR区构建系统发育树发现文昌鱼TLR主要分为3大类:一类和昆虫的V-TLR聚集在一起;另外两类和脊椎动物的TLR11家族聚集在一起。2009年,元少春等[38]在青岛文昌鱼(Branchiostoma belcheri tsingtauense)中 克 隆 到1个TLR(bbtTLR)和1个MyD88(bbtMyD88)。免疫刺激试验结果显示,bbtTLRl具有免疫功能,与bbtMyD88一起构成一条保守的信号途径。另外研究表明,青岛文昌鱼中的SARM对该TLR通路起到负调控作用[39]。
1.7 尾索动物
尾索动物是现存的和脊椎动物亲缘关系最近的物种,通过对其TLR分析有助于揭示脊椎动物TLR起源。玻璃海鞘作为尾索动物门的代表动物,它具有发育调控系统、神经肽和内分泌系统,已经成为免疫学研究的热点。对玻璃海鞘(Ciona intestinalis)基因组进行分析发现了3个TLR基因,以及参与TLR信号通路的MyD88、IRAK、TRAF、NF-κB和IκB[40]。目前已经在玻璃海鞘体内克隆得到两个TLR的cDNA全长(CiTLR1和CiTLR2)。它们具有典型的TLR结构,在玻璃海鞘细胞膜和细胞质中都发现了这两个基因,其病原体的识别机制和脊椎动物相似[41]。
1.8 无颌脊椎动物
脊椎动物亚门包括无颌总纲和有颌下门。七鳃鳗属于无颌脊椎动物,是最原始的脊椎动物类群,已经表现出适应性免疫系统的一些特征。2007年,在日本七鳃鳗(Lampetra japonica)中首次鉴定出两个TLR:TLR14a和TLR14b[42]。随后,在海七鳃鳗(Petromyzon marinus)基因组最新拼接结果中发现了16个TLR基因,系统发育树分析表明海七鳃鳗的TLRs是由鱼类TLRs和哺乳动物TLRs组成,同时还鉴定得到了TLR信号通路下游的MyD88、TICAM和SARM[43]。尽管海七鳃鳗体内含有适应性免疫系统,但是主要是通过先天性免疫系统中TLR来应对病原体入侵。
1.9 软骨鱼
软骨鱼纲属于脊椎动物亚门,处在衔接无颌类和有颌类的过渡地位,成为研究脊椎动物适应性免疫系统起源和进化的重点对象。在软骨鱼中已经发现了具有适应性免疫特征的细胞因子及其受体,如IL-1、IL-8、IL-1R(Interleukin-1 receptor)等[44]。同时对于软骨鱼先天性免疫系统的研究也取得了一定的进展,在大象鲨鱼(Callorhinchus milii)基因组中发现了TLR信号通路中的相关成分:TICAM(Toll-like receptor adapter molecule)和TIRAP(TIR domain containing adapter protein)。和其它物种TLR信号通路中的接头分子TICAM-1/2和接头蛋白TIRAP比对发现大象鲨鱼是非哺乳动物中唯一一个含有TICAM-2 基因的[45]。
1.10 硬骨鱼
硬骨鱼是水生脊椎动物中进化最成功的,被称为地球上的水域征服者。目前已经完成了对红鳍东方鲀(Takifugu rubripes)、墨绿凹鼻鲀(Tetraodon nigroviridis)、斑马鱼(Danio rerio)、虹鳟(Oncorhynchus mykiss)、青鳉(Oryzias latipes)、大西洋鲑(Salmo salar)和三刺鱼(Gasterosteus aculeatus)的大规模基因组或转录组测序,发现硬骨鱼TLRs以及信号通路的成分和哺乳动物的TLR系统同源性较高[9]。近年来对斑马鱼和红鳍东方鲀的TLR研究比较深入,分别发现了14种[46]和12种TLR基因[47]。非哺乳动物类的TLR14、TLR21和TLR22在两种鱼中都存在。TLR4b、TLR19和TLR20只存在于斑马鱼中,而可溶性TLR5(soluble TLR5,TLR5s)和TLR23只出现在红鳍东方鲀中。目前已经在31种鱼类中发现了16种TLRs,有8种是鱼类特有的。通过序列相似性比对和系统发生分析结果将鱼类TLRs分为8个家族[48]。
2 TLR基因结构域混编
通过对海洋动物基因组和转录组测序,大量只含有LRR区或TIR区的序列被发现。要阐明TLR的起源和进化,首先要解决的问题是:TLR结构中TIR区和LRR区哪个先出现;两个结构何时结合在一起形成功能性的TLR;它们的结构在进化过程发生了怎样的变化。通过对最原始后生动物体内TLRs、LRRs和TIRs序列进行分析发现,刺胞动物中出现了脊椎动物类TIR(Vertebrate-like TIR,V-TIR)和 原口动物类Toll(Protostome-like Toll,P-Toll)[14],说明在非两侧对称动物体内出现了原口动物类TIR(Protostome-like TIR,P-TIR)和原口动物类LRR(Protostome-like LRR,P-LRR)的结合,V-TIR区要比功能性的V-TLR出现的早。对后口动物海胆和文昌鱼中TLRs、LRRs和TIRs序列分析推断,在V-TLRs形成过程中脊椎动物类LRR(Vertebrate-like LRR,V-LRR)和V-TIR之间出现了两轮结合。第一次出现在两侧对称动物和非两侧对称动物出现分化以后;另一次出现在原始脊椎动物进化过程中[49]。同时,在后口动物中也发现P-TLR,说明P-TLR在两侧对称动物的祖先体内是存在的,随后在脊椎动物中消失[30]。
3 TLR基因的多态性
处于不同进化地位的海洋动物中无脊椎动物占大多数,它们主要依靠模式识别受体来有效对抗大量病原体的入侵,因此无脊椎动物体内的模式识别受体基因表现出高度的多态性。在贻贝、海胆和文昌鱼中所发现的TLR基因家族为研究先天性免疫系统多态性提供了参考依据。
海胆TLR基因的高度多态性主要表现在胞外LRRs区,这表明海胆TLR对PMAP识别具有特异性,可以直接和配体结合而不需要通过中介物。海胆LRRs区产生多态性的机制包括:单个氨基酸替换或小片段的插入或缺失,LRR基序内部或不同LRR基序之间大片段的插入,以及少数完整LRR基序的插入[3]。在同属于原始后口动物的美国文昌鱼中也发现了具有高度多态性的TLR基因家族,推测在后口动物祖先中的先天性免疫系统普遍存在多态性[2]。这种多态性机制一方面扩大了过渡物种识别病原体的范围,更重要的是有助于适应脊椎动物复杂的发育过程和形态结构[50]。将双壳类贻贝中TLR基因和海胆TLR基因多态性相结合,推测它们先天免疫系统的复杂性可能和这4个因素相关:缺少适应性免疫系统;拥有复杂而庞大的体型;底栖生活并且寿命较长[20]。从进化的角度来看,TLR基因所表现出的高度多态性说明这些动物体内的TLR基因正处于一个动态进化阶段[3]。
4 小结
通过以上分析表明,在最古老的后生动物海绵中还没有出现完整的TLR,功能性TLR最早是在刺胞动物体内鉴定得到。随后在原始后口动物海胆和文昌鱼中发现了大量的TLR家族基因,据其所表现出的高度多态性推测,脊椎动物TLR起源于这种复杂的TLR系统。海洋动物TLR基因的多态性与其生存环境有密切联系,但是环境因子是如何影响TLR结构仍需要进一步的研究证明。目前对于TLR的研究主要停留在基因组水平上,对于该受体的功能和配体特异性识别机制的研究还不够深入。因此,加强对TLR特异性配体的鉴定可为人们揭示TLR基因多态性机制和确定TLR进化选择压力提供依据。同时,对海洋动物TLR功能的研究为研制新型基因疫苗、免疫调节剂和有效预防经济物种病害奠定理论基础。
[1] Kawai T, Akira S. The role of pattern-recognition receptors in innate immunity:update on Toll-like receptors[J]. Nat Immunol, 2010, 11(5):373-384.
[2] Huang SF, Yuan SC, Guo L, et al. Genomic analysis of the immune gene repertoire of amphioxus reveals extraordinary innate complexity and diversity[J]. Genome Res, 2008, 18(7):1112-1126.
[3] Hibino T, Loza-Coll M, Messier C, et al. The immune gene repertoire encoded in the purple sea urchin genome[J]. Dev Bio, 2006, 300(1):349-365.
[4] O’Neill LAJ, Bowie AG. The family of five:TIR-domain-containing adaptors in Toll-like receptor signaling[J]. Nat Rev Immunol, 2007, 7(5):353-364.
[5] Lin Q, Li MC, Fang D, et al. The essential roles of Toll-like receptor signaling pathways in sterile inflammatory diseases[J]. Int Immunopharmacol, 2011, 11(10):1422-1432.
[6] Keogh B, Parker AE. Toll-like receptors as targets for immune disorders[J]. Trends Pharmacol Sci, 2011, 32(7):435-442.
[7] Nusslein-Volhard C, Lohs-Schardin M, Sander K, et al. A dorsoventral shift of embryonic primordia in a new maternal-effect mutant of Drosophila[J]. Nature, 1980, 283(5746):474-476.
[8] Medzhitov R, Janeway Jr CA.Innate immunity:impact on the adaptive immune response[J]. Curr Opin Immunol, 1997, 9(1):4-9.
[9] Coscia MR, Giacomelli S, Oreste U. Toll-like receptors:an overview from invertebrates to vertebrates[J]. ISJ, 2011, 8:210-226.
[10] Wilson HV. On some phenomena of coalescence and regeneration in sponges[J]. J Exp Zool, 1907, 5(2):245-258.
[11] Wiens M, Korzhev M, Perovic-Ottstadt S, et al. Toll-like receptors are part of the innate immune defense system of sponges(demospongiae:Porifera)[J]. Mol Biol Evol, 2007, 24(3):792-804.
[12] Srivastava M, Simakov O, Chapman J, et al. The Amphimedon queenslandica genome and the evolution of animal complexity[J]. Nature, 2010, 466(7307):720-726.
[13] Gauthier ME, Du PL, Degnan BM. The genome of the sponge Amphimedon queenslandica provides new perspectives into the origin of Toll-like and interleukin 1 receptor pathways[J]. Evol Dev, 2010, 12(5):519-533.
[14] Miller DJ, Hemmrich G, Ball EE, et al. The innate immune repertoire in Cnidaria - ancestral complexity and stochastic gene loss[J]. Genome Biol, 2007, 8(4):R59.
[15] Bosch TCG, Augustin R, Anton-Erxleben F, et al. Uncovering evolutionary history of innate immunity:the simple metazoan Hydra uses epithelial cells for host defence[J]. Dev Comp Immunol, 2009, 33(4):559-569.
[16] Shinzato C, Shoguchi E, Kawashima T, et al. Using the Acropora digitifera genome to understand coral responses to environmental change[J]. Nature, 2011, 476(7360):320-323.
[17] De Lorgeril J, Zenagui R, Rosa RD, et al. Whole transcriptome profiling of successful immune response to vibrio infections in the oyster Crassostrea gigas by digital gene expression analysis[J]. PLoS ONE, 2011, 6(8):e23142. doi:10.1371/journal.pone.0023142.
[18] Hou R, Bao ZM, Wang S, et al. Transcriptome sequencing and de novo analysis for Yesso scallop(Patinopecten yessoensis)using 454 GS FLX[J]. PLoS One, 2011, 6(6):e21560. doi:10.1371/journal.pone.0021560.
[19] Venier P, Varotto L, Rosani U, et al. Insights into the innate immunity of the Mediterranean mussel Mytilus galloprovincialis[J]. BMC Genomics, 2011, 12:69.
[20] Philipp EER, Kraemer L, Melzner F, et al. Massively parallel RNA sequencing identifies a complex immune gene repertoire in the lophotrochozoan Mytilus edulis[J]. PLoS One, 2012, 7(3):e33091. doi:10.1371/journal.pone.0033091.
[21] Wang M, Yanga J, Zhoua Z, et al. A primitive Toll-like receptor signaling pathway in mollusk Zhikong scallop Chlamys farreri[J]. Dev Comp Immunol, 2011a, 35(4):511-520.
[22] Zhang LL, Li L, Zhang GF. A Crassostrea gigas Toll-like receptor and comparative analysis of TLR pathway in invertebrates[J]. Fish Shellfish Immunol, 2011, 30(2):653-660.
[23] Inamori K, Ariki S, Kawabata S. A Toll-like receptor in horseshoe crabs[J]. Immunol Rev, 2004, 198:106-115.
[24] Belinda LW, Wei WX, Hanh BT, et al. SARM:a novel Toll-like receptor is functionally conserved from arthropod to human[J]. Mol Immunol, 2008, 45(6):1732-1742.
[25] Wang PH, Liang JP, Gu ZH, et al. Molecular cloning, characterization and expression analysis of two novel Tolls(LvToll2 and Lv-Toll3)and three putative Spätzle-like Toll ligands(LvSpz1-3)from Litopenaeus vannamei[J]. Dev Comp Immunol, 2012, 36(2):359-371.
[26] Arts JA, Cornelissen FH, Cijsouw T, et al. Molecular cloning and expression of a Toll receptor in the giant tiger shrimp, Penaeus monodon[J]. Fish Shellfish Immunol, 2007, 23(3):504-513.
[27] Yang CJ, Zhang JQ, Li FH, et al. A Toll receptor from Chinese shrimp Fenneropenaeus chinensis is responsive to Vibrio anguillarum infection[J]. Fish Shellfish Immunol, 2008, 24(5):564-574.
[28] Mekata T, Kono T, Yoshida T, et al. Identification of cDNA encoding Toll receptor, MjToll gene from kuruma shrimp, Marsupenaeus japonicus[J]. Fish Shellfish Immunol, 2008, 24(1):122-133.
[29] Assavalapsakul W, Panyim S. Molecular cloning and tissue distribution of the Toll receptor in the black tiger shrimp, Penaeus monodon[J]. Genet Mol Res, 2012, 11(1):484-493.
[30] Rast JP, Smith LC, Loza-Coll M, et al. Genomic insights into the immune system of the sea urchin[J]. Science, 2006, 314(5801):952-956.
[31] 杨爱馥, 周遵春, 孙大鹏, 等.仿剌参铁蛋白ferritin基因的序列分析及表达[J]. 水产学报, 2010, 6(34):710-717.
[32] Yang AF, Zhou ZC, He CB, et al. Analysis of expressed sequence tags from body wall, intestine and respiratory tree of sea cucumber(Apostichopus japonicus)[J]. Aquaculture, 2009, 296:193-199.
[33] Yang AF, Zhou ZC, Dong Y, et al. Expression of immune-related genes in embryos and larvae of sea cucumber Apostichopus japonicus[J]. Fish Shellfish Immunol, 2010, 29:839-845.
[34] Zhou ZC, Sun DP, Yang AF, et al. Molecular characterization and expression analysis of a complement component 3 in the sea cucumber(Apostichopus japonicus)[J]. Fish Shellfish Immunol, 2011; 31:540-547.
[35] Sun LN, Chen MY, Yang HS, et al. Large scale gene expression profiling during intestine and body wall regeneration in the sea cucumber Apostichopus japonicus[J]. Comp Biochem Physiol, 2011, 6(2):195-205.
[36] Du HX, Bao ZM, Hou R, et al. Transcriptome sequencing and characterization for the sea cucumber Apostichopus japonicus(Selenka, 1867)[J]. PLoS One, 2012, 7(3):e33311. doi:10.1371/journal.pone.0033311.
[37] Delsuc F, Brinkmann H, Chourrout D, et al. Tunicates and not cephalochordates are the closest living relatives of vertebrates[J]. Nature, 2006, 439(7079):965-968.
[38] Yuan SC, Huang SF, Zhang W, et al. An amphioxus TLR with dynamic embryonic expression pattern responses to pathogens and activates NF-κB pathway via MyD88[J]. Mol Immunol, 2009, 46(11-12):2348-2356.
[39] Yuan SC, Wu K, Yang MY, et al. Amphioxus SARM involved in neural development may function as a suppressor of TLR signaling[J]. J Immunol, 2010, 184(12):6874-6881.
[40] Azumi K, Santis R, Tomaso A, et al. Genomic analysis of immunity in a Urochordate and the emergence of the vertebrate immune system:"waiting for Godot"[J]. Immunogenetics, 2003, 55(8):570-581.
[41] Sasaki N, Ogasawara M, Sekiguchi T, et al. Toll-like receptors of the ascidian Ciona intestinalis. prototypes with hybrid functionalities of vertebrate toll-like receptors[J]. Biol Chem, 2009, 284(40):27336-27343.
[42] Ishii A, Matsuo A, Sawa H, et al. Lamprey TLRs with properties distinct from those of the variable lymphocyte receptors[J]. J Immunol, 2007, 178(1):397-406.
[43] Kasamatsu J, Oshiumi H, Matsumoto M, et al. Phylogenetic and expression analysis of lamprey toll-like receptors[J]. Dev Comp Immunol, 2010, 34(8):855-865.
[44] Cateni C, Paulesu L, Bigliardi E, Hamlett WC. The interleukin I(IL-1)system in the uteroplacental complex of a cartilaginous fish, the smoothhound shark, Mustelus canis[J]. Reprod Biol Endncrinol, 2003, 1:25.
[45] Wu BJ, Xin B, Jin M, et al. Comparative and phylogenetic analyses of three TIR domain-containing adaptors in metazoans:implications for evolution of TLR signaling pathways[J]. Dev Comp Immunol, 2011, 35(7):764-773.
[46] Jault C, Pichon L, Chluba J. Toll-like receptor gene family and TIRdomain adapters in Danio rerio[J]. Mol Immunol, 2004, 40(11):759-771.
[47] Oshiumi H, Tsujita T, Shida K, et al. Prediction of the prototype of the human Toll-like receptor gene family from the pufferfish, Fugu rubripes, genome[J]. Immunogenetics, 2003, 54(11):791-800.
[48] Yniv P. Toll-like receptors in bony fish:From genomics to function[J]. Dev Comp Immunol, 2011, 35(12):1263-1272.
[49] Wu BJ, Huan TX, Gong J, et al. Domain combination of the vertebrate-like TLR gene family:implications for their origin and evolution[J]. J Genet, 2011, 90(3):401-408.
[50] Pancer Z, Cooper MD. The evolution of adaptive immunity[J]. Annu Rev Immunol, 2006, 24:497-518.
