胆固醇结石患者胆囊细菌的T-RFLP研究
2013-11-05叶吉云李洱花刘云霞田大广高波
叶吉云 李洱花 刘云霞 田大广 高波★
2.昆明医学院第二附属医院肝胆外科,云南,昆明 650101
末端限制性片段长度多态性(Terminal Restriction Fragment Length Polymorphism,T-RFLP)以分子生物学的手段,以微生物自身组成成分中的“家族特异性保守物质”(例如,DNA/RNA等)的分子进化史等为主要工作对象来研究微生物的群落生态[1],对微生物的检测不受微生物的状态和培养手段的限制[2],是目前被广泛采用的DNA指纹印记分析的一种方法。在国内,对于分子微生物生态学的研究也是刚刚起步。本文为进一步探讨细菌群落与胆囊结石的发病机理之间的关系,采用T-RFLP技术对胆囊结石微生物群落16S rDNA基因序列进行分析研究。
1 资料与方法
1.1 标本来源
取自2007年2月~2007年6月在昆明医学院第二附属医院接受择期胆囊切除术的胆囊结石患者,术前3个月内无急性胆道感染症状,HBsAg阴性。完成腹腔镜胆囊切除术,无菌注射器穿刺取胆汁3 mL~5 mL置于无菌试管内,同时收集胆囊结石,记录其大小、数目,用生理盐水冲洗干净。标本放在无菌容器内-20℃保存。依据红外吸收光谱法测定胆固醇含量纯胆固醇结石(CS):胆固醇含量≥90%,纳入本研究。胆汁细菌常规和厌氧培养阳性的病例被剔除。本组8例中男3例,女5例,年龄25~73岁,平均年龄55岁。诊断经病理证实。
1.2 微生物群落DNA 提取方法
1.2.1 结石微生物群落DNA提取
干燥至恒重后取150 mg结石粉末,加入 1% SDS 700μL在5 mL聚丙烯试管内,室温培养过夜。加入LiCl(7 mol/L),最终浓度为1.5 mol/L。静置15 min,震荡 1 min,离心(12 000 rpm,15 min);取上清液至一灭菌塑料离心管中,加入等体积酚/氯仿/异戊醇混合液抽提2次,取上清液,加1/10体积3 mol/L醋酸钠。再加无水乙醇(终体积比80%),-70℃静置2 h,将上部所得溶液全部倒入收集管内的吸附柱中,12 000 rpm离心5 min,将柱放入干净EP管中,加30~50μL TE缓冲溶液(含RNA酶)充分溶解DNA。静置1 min,12 000 rpm离心2 min,洗脱液为提取到的DNA,-20℃保存。
1.2.2 胆汁微生物群落DNA提取
对文献[3]介绍的DNA 提取方法进行适当修改后作为本研究的DNA 提取方法。取胆汁混合样10 mL,用聚碳酸酯膜(0.22μm,47 mm,北京华美公司)进行真空抽滤(-0.1 MPa),待水样抽干后,将滤膜取下,用无菌剪刀剪成小块,放入10 mL无菌离心管;加入1 mL Buffer I(0.15 mol/L NaCl,0.1 mol/L Na2EDTA,pH8),涡旋振荡,使buffer I与膜充分接触;加入100μL溶菌酶(50 g/L),温育1 h(37℃);将离心管放入液氮中5 min 后迅速移至65℃水浴锅中解冻3 min,这样反复冻融3次;加入0.5 mL buffer II(0.1 mol/L NaCl,0.5 mol/L Tris-HCl,10%SDS,pH8),上下摇匀,加入5μL蛋白酶K(20 g/L),水浴1 h(55℃);依据标准方法[4]进行酚仿抽提2次,取上清转移至新的10 mL离心管中;加入2倍体积无水冰乙醇沉淀DNA,-20℃过夜,离心(13 000 rpm,30 min);弃去上清,加入1 mL 70%乙醇,离心(13 000 rpm,15 min),弃上清;干燥后,加入30~50μL TE 缓冲溶液充分溶解DNA。
1.3 胆汁、结石细菌16S rDNA 基因扩增
对样品DNA进行适当稀释之后,进行细菌的16S rDNA的PCR扩增。扩增体系总体积50μL:(1)10×buffer:5μL;(2) 4×dNTP:4μL(2.5 mM);(3)MgCl2:3μL(25 mM);(4)8F-FAM:1μL(10 pmol/μL);(5)1492R:1μL(10 pmol/μL);(6)Taq 酶:0.3μL(TaKaRa,5 U/μL);(7)灭菌水:33.7μL;(8)DNA模板:2μL。热循环条件预变性:94℃,5 min;变性:94℃,1 min;退火:51℃,1 min;延伸:72℃,1.5 min;30个循环;4℃保存PCR产物。细菌的扩增引物采用细菌通用引物8F(5'-AGA GTT TGA TCC TGG CTC AG-3')和 1492R(5'-GGT TAC CTT GTT ACG ACT T-3'),在引物8F的5'端标记荧光物质(6-FAM)。用于构建克隆文库的PCR则采用不带荧光标记的引物。PCR产物利用ChargeSwitch纯化试剂盒(Invitrogen,USA)进行纯化。
1.4 T-RFLP分析
对细菌16S rDNA的PCR纯化产物分别用TaqI、65℃酶切 3 h 和 RsaI、37℃酶切 3 h;酶切产物进行脱盐后[5],用甲酰胺进行溶解;取3~5 μL酶切产物至10μL甲酰胺中,并加入0.2μL 内标Liz500(Applied Biosystems Incorporated,USA);将该混合物在95℃变性5 min,然后迅速转移至冰上,取10μL至96孔板中,在ABI 3130遗传分析仪中进行毛细管电泳;取荧光强度50RFU为基线,用Genemapper软件(Applied Biosystems Incorporated,USA)计算大于基线的峰高和出峰处的末端标记限制性片段(Terminal Restriction Fragment,T-RF)长度;将T-RF大于50 bp的峰面积进行标准化处理[6],计算相对应的丰度。
1.5 克隆文库的构建、测序和系统发育分析
利用非荧光标记的引物对细菌的16S rDNA片段进行PCR扩增,采用pGEM-T easy载体(Promega,USA)和E.coli DH5α菌株建立细菌的16S rDNA文库。利用PCR-T-RFLP分析对细菌克隆文库中随机挑选的46个阳性克隆进行分型筛选,挑选代表克隆进行测序(北方诺塞基因公司),获得细菌的16S rDNA部分序列。对这些序列利用Blast分析与GenBank中核酸数据进行序列比对(http://www.ncbi.nlm.nih.gov/blast)。
2 结果
2.1 胆汁、结石微生物群落DNA检出率
胆汁标本均未提取到胆汁细菌总DNA。结石标本中,纯胆固醇组细菌16S rDNA阳性率为37.5%(3/8)。
2.2 纯胆固醇组胆囊结石T-RFLP图谱分析
纯胆固醇组中有3例标本经PCR获得细菌16S rDNA,分别标为 L1、L2、L3通过对其在 ABI 3130遗传分析仪中进行毛细管电泳,用Genemapper软件计算大于基线的峰高和出峰处的T-RF长度得到T-RFLP 图谱(图 1、2、3)。
根据T-RF不同,挑选相应的克隆测序,结果表明,纯胆固醇结石中细菌主要为Enterobacteriaceae和Mircrococcaceae的微生物,其中克隆D13、D12、E13(T-RF 422 bp)分 布 在Enterobacteriaceae的Escherichia属,T-RF同为422 bp的E9则分布在Enterobacteriaceae的Salmonella属;克隆F4(T-RF 394 bp)为Enterobacteriaceae的Klebsiella属,克隆D10、G7、D8(T-RF 394 bp)则分布在Mircrococcaceae的Staphylococcus属。
T-RF同为422 bp的D15克隆则可能是属于新的微生物物种,因为目前在Genebank中尚未找到与其16S rDNA序列相似的细菌序列。(表1)。
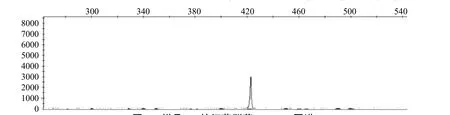
图1 样品L1的细菌群落T-RFLP图谱Figure 1 T-RFLP diagram of sample L1

图2 样品L2的细菌群落T-RFLP图谱Figure 2 T-RFLP diagram of sample L2

图3 样品L3的细菌群落T-RFLP图谱Figure 3 T-RFLP diagram of sample L3

表1 纯胆固醇结石中主要优势T-RF细菌克隆序列比对结果Table 1 Comparison of the clone seguence of T-RF bacteria in cholesterol stone
3 讨论
1995年Swidsinski[7]用NT-PCR方法,发现在常规细菌培养阴性的胆固醇结石中有较高的细菌DNA检出率,才将细菌感染引入到胆囊结石的研究中,由于方法学的限制,人们对引起胆囊结石的微生物认识有限,特别是对胆固醇含量超过90%的纯胆固醇结石与细菌感染的关系更是知之甚少。
分子微生物生态学就是运用分子生物学的方法和技术,在基因水平上估计种的个体丰度,查明种的变异情况以及探究群落中微生物间相互关系的科学。生物技术的应用,使我们不必培养微生物,而直接通过对环境中遗传物质的研究来达到目的,它为微生物生态学的研究开辟了新的途径,T-RFLP又被称为16S rDNA 基因的 T-RF分析技术,是一种新兴研究微生物多态性的分子生物学技术。该技术已被成功地应用到了各种微生物群落的比较分析、微生物群落多样性及结构特征等方面。T-RFLP是根据16S rDNA的保守区设计通用引物,对上游引物的5'端用荧光物质6-FAM标记,然后选择适当的限制性内切酶消化,再经毛细管电泳就可建立各细菌菌株特有的峰谱。本法能够检测难以培养的各种细菌。由于只有5'末端有标记,因此用GeneScan基因扫描仪只能测定5'末端带有荧光标记的限制性片段,从而克服了产生大量酶切片段的问题。利用T-RFLP方法分析16S rDNA片断中高度保守的家族特异性序列的种类、数量和相对丰度就可以了解样品中各种微生物的种类,种群数量和相对组成,从而比较准确、全面和客观地了解该环境中微生物群落的组成。其主要优势不仅避免了传统微生物富集培养的高度选择性、繁杂费时等限制,还可以同时进行多个样品的并行分析,速度快,信息量大,而且准确全面,易于实现自动化及互联网海量数据共享,从而探明微生物生态系统的功能和微生物群落结构的变化,有助于疾病分析及治疗。
本研究以利用T-RFLP技术对纯胆固醇结石细菌微生物群落16S rDNA基因序列进行分析,研究表明在胆汁中并未发现细菌16S rDNA存在的证据,而在结石中却有丰富的细菌16S rDNA。根据既往研究结果,我们认为即使胆汁中能够发现细菌16S rDNA存在的证据也仅能反映胆囊切除时细菌感染的情况,而结石中的细菌检出率则反映的是结石形成和生长过程中细菌感染的状况,二者在时间上无可比性。本研究也显示,在胆汁中无细菌16S rDNA存在的样本中,结石却能发现细菌16S rDNA,二者并无明显的联系。
研究表明纯胆固醇结石细菌16S rDNA阳性率为37.5%(3/8),T-RF主要为394 bp和422 bp。经克隆测序表明,细菌群落主要为肠杆菌(Enterobacteriaceae)和微球菌科(Mircrococcaceae)的微生物,这与已知的肠道菌群有较高的相似性,由此推断定植于肠道的正常菌群或条件致病菌细菌在某种因素的影响下可以突破机体的防御系统通过Oddi氏括约肌进入胆道到达胆囊定植,通过细菌本身和/或其代谢产物参与胆囊结石的形成。细菌群落在属水平涉及埃希氏菌属(Escherichia),沙门氏菌属(Salmonella),克雷伯氏菌属(Klebsiella)和葡萄球菌属(Staphlococcus),这些微生物群落在较高的分类学水平(例如科)的群落多样性并不很高,而在属水平表现出了不同的多样性,可能是由于胆囊对胆汁的浓缩功能,胆汁中含有一些物质,如胆汁酸盐、胆固醇和微量元素,可作为电子受体或营养物质支持细菌生长代谢,并使结石细菌群落表现出多样性。同时也应看出,有一定的未培养微生物(Uncultured bacterium clone)检出其多样性尚未纳入研究,对这类目前尚未培养的未知细菌的功能特性和生命活动规律的了解、还需更加深入的研究。
特别指出在本研究中检测到肠杆菌科(Enterobacteriaceae)的沙门氏菌属(Salmonella)。在体外研究发现,Salmonella能够形成生物被膜,抵御胆盐的侵袭,通过生物被膜与其他定植于胆囊的细菌相连接,共同构成一个相对稳定的细菌群落,将其生物被膜不断扩展以保护整个群落不受外界物质侵袭,而胆汁恰恰是生物被膜形成的最佳刺激信号。同时发现,Salmonella首先要在结石表面定植才能形成被膜,而且成膜时间平均为10~14天,而其他能够成膜的细菌平均成膜时间为几秒钟至几小时不等。但在体内由于胆囊内环境极为复杂,胆囊的排空功能在体外难以模拟,Salmonella能否也能形成生物被膜有待证实。
T-RFLP也有一定的局限性:(1)T-RFLP 是基于PCR 扩增的技术,因此就摆脱不了这类技术共同的缺陷;(2)该指纹印记技术只检测带荧光标记的末端限制性片段,造成对群落多样性的低估;(3)酶切后T-RF 的长度分布也会造成对复杂群落多样性的低估。因为测序仪检测500 bp以上的T-RF精度不够[8];(4)实验过程影响因素众多,使得T-RFLP图谱的解析存在一定的不确定性。比如DNA提取方法、PCR中的参数设置及限制性内切酶等的选择都可能直接对最终的T-RFLP图谱产生影响。本研究仅为初步探讨,具体细节还有待进行更广泛而深入的研究。
[1] Doolittle W F. Phylogenetic classification and the universal tree[J]. Sicnece, 1999, 284(5423): 2124-2129.
[2] Wu X L, Chin K J, Conrad R. Effect of temperature stress on the structure and function of the methanogenic archaeal community in a rice field soil[J]. FEMS Microbiol Ecol,2002, 39(3): 211-218.
[3] Sambrook J, Russell D W. Molecular cloning: a laboratory manual[M]. 3rd ed. New York: Cold Spring Harbor Laboratory Press, 2001: A8.9-A8.10.
[4] Watanabe K, Kodama Y, Syutsubo K, et al. Molecular characterization of bacterial populations in petroleumcontaminated groundwater discharged from underground crude oil storage cavities[J]. Appl Environ Microbiol, 2000,66(11): 4803-4809.
[5] Denaro R, D'Auria G, Di Marco G, et al. Assessing terminal restriction fragment length polymorphism suitability for the description of bacterial community structure and dynamics in hydrocarbon-polluted marine environments[J]. Environ Microbiol, 2005, 7(1): 78-87.
[6] McGuinness L M, Salganik M, Vega L, et al. Replicability of bacterial communities in denitrifying bioreactors as measured by PCR/T-RFLP analysis[J]. Environ Sci Technol,2006, 40(2): 509-515.
[7] Swidsinski A, Ludwig W, Pahlig H, et al. Molecular genetic evidence of bacterial colonization of cholesterol gallstones[J]. Gastroenterolgy, 1995, 108(3): 860-864.
[8] Marsh T L, Saxman P, Cole J, et al. Terminal restriction fragment length polymorphism analysis program, a webbased research tool for microbial community analysis[J].Appl Environ Microbiol, 2000, 66(8): 3616-3620.
