L-酪氨酸代谢工程研究进展*
2013-10-30姚元锋赵广荣
姚元锋,赵广荣
(天津大学化工学院制药工程系,天津,300072)
L-酪氨酸(L-tyrosine,Tyr)是一种重要的营养必需氨基酸,对人和动物的生长发育和新陈代谢起重要的作用,常作为营养补充剂以及L-多巴、对羟基肉桂酸、对羟基苯乙烯等医药化工产品的制备原料被广泛应用在食品、饲料、医药和化工等行业。传统的L-酪氨酸主要应用蛋白质水解法和酶法[1]来进行生产。但它们由于材料来源有限、反应过程中酶活性和稳定性差[2-3]、工艺繁琐、产品成分复杂等缺点而受到了严重制约。近年来随着代谢工程和先进生物技术迅猛发展,重新合理设计和优化微生物的代谢途径来更好地实现L-酪氨酸的发酵生产逐渐成为研究热点。
1 L-酪氨酸生物合成途径
L-酪氨酸生物合成途径属于芳香族氨基酸合成途径,只能由植物和微生物合成。目前L-酪氨酸的合成途径和调控机制以大肠杆菌(Escherichia coli)和谷氨酸棒状杆菌(Corynebacterium glutamicum)中研究得最多并阐释得最为清楚[4-5]。
如图1 所示,L-酪氨酸合成的前体物4-磷酸赤藓糖(Erythrose-4-phosphate,E4P)和磷酸烯醇式丙酮酸(Phophoenol pyruvate,PEP)在DAHP 合成酶(DS)的催化下缩合生成3-脱氧-D-阿拉伯庚酮糖酸-7-磷酸(DAHP),该反应也是L-酪氨酸生物合成途径的第一个限速步骤。在大肠杆菌中DAHP 合成酶包含AroG、AroF 和AroH 3 个同工酶,其表达和酶活分别受产物L-苯丙氨酸、L-酪氨酸和L-色氨酸的反馈阻遏和反馈抑制。从DAHP 到分支酸的7 步反应是所有芳香氨基酸合成的共同途径。分支酸是芳香族氨基酸合成途径的分支点,一个分支途径用于合成L-色氨酸,另一部分则在分支酸变位酶(chorism mutase,CM)和预苯酸脱水酶(prephenate dehydratase,PD)双功能酶TyrA 的作用下生成对羟基苯丙酮酸(4HPP),后者通过与L-谷氨酸的转氨作用生成L-酪氨酸,而TyrA 的表达和酶活同样受到产物L-酪氨酸的反馈阻遏和反馈抑制。
2 L-酪氨酸代谢途径改造
代谢工程作为一种修饰和优化微生物代谢网络与表达调控网络的强有力技术平台,在优化L-酪氨酸代谢途径,改善L-酪氨酸生产特性等方面得到广泛的应用并取得了很大成绩(如表1 所示)。而考虑到L-酪氨酸代谢途径和调控网络的特点,L-酪氨酸代谢途径的改造主要集中在以下几方面。
2.1 解除途径调控
L-酪氨酸合成途径中关键酶的酶活或表达强度受到代谢终产物L-酪氨酸强烈的反馈抑制或阻遏,并在转录水平受到调控蛋白TyrR 的调节(如图1 所示),使L-酪氨酸的积累受到严重制约。因此解除关键酶的反馈抑制(feedback-resistant,fbr)并解除转录负调控,有望提高微生物合成L-酪氨酸的能力。
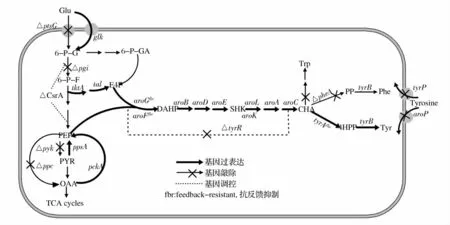
图1 酪氨酸生物合成途径及优化策略Fig.1 Biosynthesis pathway of tyrosine and its optimize strategy in E. coli

表1 代谢工程优化微生物生产酪氨酸Table 1 Metabolic engineering to optimize the production of Tyrosine
2.1.1 解除反馈抑制
在芳香族氨基酸合成途径中,DAHP 合酶DS 和双功能酶TyrA 受终产物L-酪氨酸的调控最为强烈。DS 酶的147 -149 位以及邻近的氨基酸残基和DS 的反馈抑制有关。Weaver 等[16]也发现AroF 蛋白的Pro-148-Leu 突变对L-酪氨酸不敏感。而Kikuchi等[17]的研究表明AroG 蛋白的Asp-146-Asn 突变可以解除苯丙氨酸的反馈抑制作用,而Pro-150-Leu 的突变则不仅不受L-苯丙氨酸的抑制,反而还可以被L-苯丙氨酸增强催化活性。同样Lutke 等[18]也发现TyrA 蛋白分支酸变位酶结构域的Met-53-Ile 突变和预苯酸脱氢酶结构域的Ala-354-Val 突变可以完全解除其对L-酪氨酸的敏感性。Ikeda 等[14]等克隆抗反馈抑制的DAHP 合酶DS II 和分支酸变位酶CM 并构建过表达质粒,转化生产色氨酸的谷氨酸棒杆菌KY10865 后,通过分批发酵获得了26 g/L 的L-酪氨酸产量。
2.1.2 缺失调控蛋白TyrR
大肠杆菌的tyrR基因编码芳香氨基酸生物合成和运输途径中的一种全局性调控蛋白,该蛋白质控制着包括自身编码基因tyrR在内的涉及苯丙氨酸、酪氨酸、色氨酸合成与运输的8 个转录单位的转录。当有酪氨酸存在时,TyrR 蛋白抑制包括tyrB、aroL、aroF、tyrA在内的等基因的转录[19]。因此TyrR 的敲除可以解除其对酪氨酸代谢途径的阻遏,从而促进L-酪氨酸的合成。Lutke 等[10]在过表达aroGfbr和tyrAfbr的大肠杆菌工程菌株中敲除tyrR,20 h 内在摇瓶培养可以合成346 mg/L 的L-酪氨酸。而在3L 的发酵罐中,通过分批补料发酵则可以获得3.8 g/L 的L-酪氨酸。Munoz 等[20]则发现在PTS+和PTS-的大肠杆菌菌株中,tyrR的敲除分别提高了1.6 倍和1.9倍的酪氨酸产率。
2.2 增加前体供应
在芳香族氨基酸合成途径中,PEP 和E4P 是2个重要的限制性底物,它们的供应量决定着中心代谢流的碳源能否有效地导向DAHP 的合成,从而为芳香族氨基酸的合成提供充足的前体物质。
2.2.1 提高PEP 合成
PEP 来自于糖酵解途径,是一个多酶竞争性底物。其中葡萄糖转运系统(PTS)消耗利用约50%的PEP,磷酸烯醇式丙酮酸羧化酶(PEP carboxylase,PEPC)和丙酮酸激酶(Pyruvate kinase,PYK)消耗约32%的PEP,大约只有3%的PEP 用于芳香氨基酸的合成[21]。因此改造PTS 系统和缺失ppc及pyk是提高细胞内PEP 含量的有效手段之一(如图1 所示)。Munoz 等[20]通过敲除酪氨酸生产菌株W3110-TIR 中的ptsI和ptsH来缺失PTS 系统,使酪氨酸的产率提高了3 倍达到33 mg/(g·h)。Gosset 等[22]在大肠杆菌中敲除pykA和pykF后,芳香氨基酸合成途径的碳代谢流增加了3.4 倍,继续缺失PTS 系统后则增加了5.8 倍。
加强PEP 合成途径关键酶的表达也可以提高PEP 的合成。磷酸烯醇式丙酮酸合酶(PEP synthase,PpsA)催化丙酮酸到PEP 的转化。Patnaik 等[23]等研究了过表达ppsA对DAHP 合成的影响。在大肠杆菌中过表达aroG和tktA的基础上,过表达PEP 合酶基因ppsA,细胞内的DAHP 含量提高了两倍,并接近最大理论产量。
增强PEP 合成的另一种策略是缺失全局碳调控因子csrA[24](如图2 所示)。贮碳因子(Carbon storage regulator ,CsrA)是一种从整体代谢水平上参与调控的RNA 结合蛋白,对负责分解PEP 的丙酮酸激酶pyk正调控,对负责PEP 生成的PEP 羧激酶(PckA)以及PEP 合成酶(PpsA)负调控。因此csrA的敲除可以解除其对这些有利于PEP 合成的酶的调控,从而提高PEP 水平[25-27]。
2.2.2 提高细胞内E4P 含量
E4P 是细胞内磷酸戊糖途径的代谢中间产物。因此过表达戊糖磷酸途径中的一些关键酶,如:转酮酶(Transketolase,TktA)[28]和转醛酶(Transaldolase,TAL)[29]等被用来优化L-酪氨酸代谢途径(如图2 所示)。Lutke 等[10]在L-酪氨酸生产菌株大肠杆菌T1的基础上单独过表达tktA,提高产量10%,单独过表达ppsA则可以使L-酪氨酸产量提高35%,而ppsA和tktA同时过表达则使酪氨酸的产量提高了80%,达到620 mg/L。Ikeda 等[11]在谷氨酸棒杆菌中报道了类似的结果,通过过表达棒杆菌的tktA基因,使酪氨酸产量提高了20%。
2.3 阻断竞争途径
由酪氨酸合成途径可以看出,苯丙氨酸和色氨酸合成途径是其竞争途径。阻断这2 条竞争性途径不仅可节约碳源,使代谢流更多的流向L-酪氨酸的合成,还可解除L-苯丙氨酸和L-色氨酸对合成途径中DAHP 合成酶(DS)的反馈抑制,有利于酪氨酸的积累。Olson 等[6]通过敲除负责L-苯丙氨酸合成酶基因pheA及其调控基因pheL,将一株生产L-苯丙氨酸的大肠杆菌改造成L-酪氨酸生产菌株,42 h 内L-酪氨酸产量为0.18 g/L。
2.4 调节转运系统
产物的转运和胞外分泌涉及到细胞对外界环境的感应,对于优化L-酪氨酸的代谢途径也很重要。Ikeda 等[21]过敲除谷氨酸棒杆菌中的转运蛋白基因aroP[30],缺失了菌株芳香族氨基酸的转运系统,使L-酪氨酸产量增加了30%,达到4.3 g/L。
3 L-酪氨酸代谢途径全局优化
传统的代谢工程方法主要改变的是生物合成途径中的关键的结构基因或调控基因,针对简单的代谢途径这是迅速而有效的。但是针对L-酪氨酸途径这样一个涉及多基因和多种调控的复杂途径,往往只能达到局部途径的最优化,而不是细胞的全局最优产量。因此从全局出发,综合考察涉及基因水平,转录水平和翻译水平对整个合成途径优化的影响是必须的。基于此,一些创新而有效的代谢工程新方法已经越来越多的应用于L-酪氨酸代谢途径的全局优化。
3.1 模块代谢工程优化策略
Juminaga 等[8]人采用模块化代谢工程(modular engineering)策略,将酪氨酸合成途径和中心碳代谢途径中的11 个关键基因,按照功能拆分成2 个较小的莽草酸合成模块和酪氨酸合成模块。莽草酸模块包含ydiB、aroD、aroB、aroGfbr、ppsA和tktA等6 个基因,代表由葡萄糖合成莽草酸这一局部代谢途径。L-酪氨酸模块包含tyrB、tyrAfbr、aroC、aroA和aroL等5个基因,代表由莽草酸合成L-酪氨酸另一局部代谢途径。然后分别对每个模块进行包含启动子、终止子、拷贝数、密码子等在内的优化,构建模块文库。然后通过2 个文库的不同组合,以终产物L-酪氨酸作为监测目标,进行模块之间适配性的优化。同时监测如奎宁酸、脱奎宁酸和莽草酸等中间代谢产物来进行模块内的微调。最终筛选得到由LacUV5 启动子控制aroE-aroD-aroB组成模块、由LtetO-1 启动子控制aroGfbr-ppsA-tktA组成模块,和由LacUV5 启动子控制tyrB-tyrAfbr-aroC组成模块、由Trc启动子控制的aroAaroL组成模块,组合为最优。最优菌株在24 h 内L-酪氨酸产量为2.6 g/L,达到理论产率的79%。对此菌株的模块进一步分析发现aroB,aroE和aroL3 个基因是优化此途径的瓶颈,这是传统代谢途径优化所没有发现的。由此可见模块化代谢工程不仅通过拆分简化了途径优化的复杂性而且还有助于发现一些未知的改造靶基因。
3.2 全局转录工程优化策略
代谢工程改造往往都是针对已知的代谢途径和调控因子,而对很多不明确的靶基因和调控因子无能为力。全局转录装置工程(global transcription machinery engineering ,gTME)[31]就是通过易错PCR、DNA shuffling 等方法对σ 因子和各种转录因子等转录元件进行多轮改组突变修饰,从而系统改变细胞转录组并有效地提高细胞的抗逆性能以及目标产物的合成能力。Santos 等[12]利用gTME 的方法,将大肠杆菌中编码σ70因子的rpoD基因和编码RNA 聚合酶α亚基的rpoA基因通过易错PCR 构建随机突变文库,并连接到低拷贝的表达载体上转化野生型大肠杆菌,筛选到rpoA突变的大肠杆菌rpoA14 菌株L-酪氨酸产量达13.8 g/L。测序显示rpoA14 菌株在RpoA 蛋白的α 亚基C 末端存在Val-257-Phe 和Leu-281-Pro 2 个突变位点。转录水平分析显示,此rpoA的突变导致几百个基因的上调和下调,这些基因大多与酸抗性调控因子evgA或者ppGpp 合酶relA有关。由此可以看出作为一种全新的代谢工程方法,gTME 是一种更加全局化的全细胞优化方式,其在多基因和多调控方式控制的复杂代谢途径优化上有无可比拟的优势。
3.3 sRNAs 工程优化策略
sRNAs(Small RNAs)是一类长度为几十到几百个核苷酸长度具有调控基因表达功能的非编码双链RNA[32]。在原核生物如大肠杆菌中,sRNA 主要是通过互补链结合或者与蛋白质相互作用在转录后水平对mRNA 的翻译进行调控[33]。针对大肠杆菌L-酪氨酸的生物合成中负作用基因tyrR、csrA、ppc和pgi,Dokyun 等[13]合理设计并人工合成了这4 个基因的负调控sRNA,并用于筛选L-酪氨酸高产菌株。为了促进sRNA 和靶mRNA 杂交以及靶mRNA 的降解,在sRNA 中引入了一个MicC[34]的骨架,用于结合RNA 分子伴侣Hfp 蛋白[35]。将这4 个合成的sRNA随机组合并构建了低拷贝表达载体,转化14 种不同的大肠杆菌进行筛选。最终发现了tyrR、csrA表达被sRNA 抑制的S17-1 菌株的酪氨酸高产菌株,在高密度培养下可达到21.9 g/L 的L-酪氨酸。
4 展望
L-酪氨酸作为3 大芳香氨基酸之一,在应用规模上一直远远小于L-苯丙氨酸和L-色氨酸[4]。但随着L-酪氨酸重要衍生物如L-多巴、黑色素、咖啡酸、丹参素等代谢途径的揭示和深入研究,L-酪氨酸逐渐发挥出一个平台型化合物的功能,而针对其代谢工程的研究也吸引了诸多研究者的关注。随着越来越多衍生物代谢途径及代谢调节机制的不断深入研究,加之新的代谢工程优化策略的开发和完善,相信L-酪氨酸的发酵生产和工业应用必将打开一个新局面。
[1] Kim do Y,Rha E,Choi S L,et al. Development of bioreactor system forL-tyrosine synthesis using thermostable tyrosine phenol-lyase[J]. J Microbiol Biotechnol,2007,17(1):116 -122.
[2] Rha E,Kim S,Choi S L,et al. Simultaneous improvement of catalytic activity and thermal stability of tyrosine phenol-lyase by directed evolution[J]. FEBS J,2009,276(21):6 187 -6 194.
[3] Kim J H,Song J J,Kim B G,et al. Enhanced stability of tyrosine phenol-lyase fromSymbiobacterium toebiiby DNA shuffling[J]. J Microbiol Biotechnol,2004,14(1):153 -157.
[4] Bongaerts J,Kramer M,Muller U,et al. Metabolic engineering for microbial production of aromatic amino acids and derived compounds[J]. Metab Eng,2001,3(4):289-300.
[5] Ikeda M. Towards bacterial strains overproducingL-tryptophan and other aromatics by metabolic engineering[J].Appl Microbiol Biotechnol,2006,69(6):615 -626.
[6] Olson M M,Templeton L J,Suh W,et al. Production of tyrosine from sucrose or glucose achieved by rapid genetic changes to phenylalanine-producingEscherichia colistrains[J]. Appl Microbiol Biotechnol,2007,74(5):1 031 -1 040.
[7] Santos C N,Stephanopoulos G. Melanin-based highthroughput screen forL-tyrosine production inEscherichia coli[J]. Appl Environ Microbiol,2008,74(4):1 190 -1 197.
[8] Juminaga D,Baidoo E E,Redding-Johanson A M,et al.Modular engineering ofL-tyrosine production in Escherichia coli[J]. Appl Environ Microbiol,2012,78(1):89 -98.
[9] Chavez-Bejar M I,Lara A R,Lopez H,et al. Metabolic engineering ofEscherichia coliforL-tyrosine production by expression of genes coding for the chorismate mutase domain of the native chorismate mutase-prephenate dehydratase and a cyclohexadienyl dehydrogenase fromZymomonas mobilis[J]. Appl Environ Microbiol,2008,74(10):3 284 -3 290.
[10] Lutke-Eversloh T,Stephanopoulos G.L-tyrosine production by deregulated strains ofEscherichia coli[J]. Appl Microbiol Biotechnol,2007,75(1):103 -110.
[11] Ikeda M,Okamoto K,Katsumata R. Cloning of the transketolase gene and the effect of its dosage on aromatic amino acid production inCorynebacterium glutamicum[J].Appl Microbiol Biotechnol,1999,51(2):201 -206.
[12] Santos C N,Xiao Wen-hai,Stephanopoulos G. Rational,combinatorial,and genomic approaches for engineeringLtyrosine production inEscherichia coli[J]. Proc Natl Acad Sci U S A,2012,109(34):13 538 -13 543.
[13] Na D,Yoo S M,Chung H,et al. Metabolic engineering ofEscherichia coliusing synthetic small regulatory RNAs[J]. Nat Biotech,2013,31(2):170 -174.
[14] Ikeda M,Katsumata R. Metabolic engineering to produce tyrosine or phenylalanine in a tryptophan-producingCorynebacterium glutamicumstrain[J]. Appl Environ Microbiol,1992,58(3):781 -785.
[15] Patnaik R,Zolandz R R,Green D A,et al.L-tyrosine production by recombinantEscherichia coli:fermentation optimization and recovery[J]. Biotechnol Bioeng,2008,99(4):741 -752.
[16] Weaver L M,Herrmann K M. Cloning of an aroF allele encoding a tyrosine-insensitive 3-deoxy-D-arabino-heptulosonate 7-phosphate synthase[J]. J Bacteriol,1990,172(11):6 581 -6 584.
[17] Kikuchi Y,Tsujimoto K,Kurahashi O. Mutational analysis of the feedback sites of phenylalanine-sensitive 3-deoxy-D-arabino-heptulosonate-7-phosphate synthase ofEscherichia coli[J]. Appl Environ Microbiol,1997,63(2):761 -762.[18] Lutke-Eversloh T,Stephanopoulos G. Feedback inhibition of chorismate mutase/prephenate dehydrogenase (TyrA)ofEscherichia coli:generation and characterization of tyrosine-insensitive mutants[J]. Appl Environ Microbiol,2005,71(11):7 224 -7 228.
[19] Pittard A,Davidson B. TyrR protein ofEscherichia coliand its role as repressor and activator[J]. Mol Microbiol,2006,5(7):1 585 -1 592.
[20] Munoz A J,Hernandez-Chavez G,de Anda R,et al.Metabolic engineering ofEscherichia colifor improvingL-3,4-dihydroxyphenylalanine (L-DOPA)synthesis from glucose[J]. J Ind Microbiol Biotechnol,2011,38(11):1 845 -1 852.
[21] Ikeda M,Katsumata R. Transport of aromatic amino acids and its influence on overproducton of the amino acids inCorynebacterium glutamicum[J]. J Ferment Bioeng,1994,78(6):420 -425.
[22] Gosset G,Yong-Xiao J,Berry A. A direct comparison of approaches for increasing carbon flow to aromatic biosynthesis inEscherichia coli[J]. J Ind Microbiol Biotechnol,1996,17(1):47 -52.
[23] Patnaik R,Liao J C. Engineering of Escherichia coli central metabolism for aromatic metabolite production with near theoretical yield[J]. Appl Environ Microbiol,1994,60(11):3 903 -3 908.
[24] Tatarko M,Romeo T. Disruption of a global regulatory gene to enhance central carbon flux into phenylalanine biosynthesis inEscherichia coli[J]. Curr Microbiol,2001,43(1):26 -32.
[25] Khamduang M,Packdibamrung K,Chutmanop J,et al.Production ofL-phenylalanine from glycerol by a recombinantEscherichia coli[J]. J Ind Microbiol Biotechnol,2009,36(10):1 267 -1 274.
[26] Rittmann D,Lindner S N,Wendisch V F. Engineering of a glycerol utilization pathway for amino acid production byCorynebacterium glutamicum[J]. Appl Environ Microbiol,2008,74(20):6 216 -6 222.
[27] Yakandawala N,Romeo T,Friesen AD,et al. Metabolic engineering ofEscherichia colito enhance phenylalanine production[J]. Appl Microbiol Biotechnol,2008,78(2):283 -291.
[28] Hernandez-Montalvo V,Martinez A,Hernandez-Chavez G,et al. Expression of galP and glk in aEscherichia coliPTS mutant restores glucose transport and increases glycolytic flux to fermentation products[J]. Biotechnol Bioeng,2003,83(6):687 -694.
[29] Lu Jia-ling,Liao J C. Metabolic engineering and control analysis for production of aromatics:Role of transaldolase[J]. Biotechnol Bioeng,1997,53(2):132 -138.
[30] Burkovski A,Kramer R. Bacterial amino acid transport proteins:occurrence,functions,and significance for biotechnological applications[J]. Appl Microbiol Biotechnol,2002,58(3):265 -274.
[31] Alper H,Moxley J,Nevoigt E,et al. Engineering yeast transcription machinery for improved ethanol tolerance and production[J]. Science,2006,314(5 805):1 565 -1 568.
[32] Huang H Y,Chang H Y,Chou C H,et al. sRNAMap:genomic maps for small non-coding RNAs,their regulators and their targets in microbial genomes[J]. Nucleic Acids Res,2009,37(1):D150 -154.
[33] Gottesman S. The small RNA regulators ofEscherichia coli:roles and mechanisms[J]. Annu Rev Microbiol,2004,58:303 -328.
[34] Urban J H,Vogel J. Translational control and target recognition byEscherichia colismall RNAs in vivo[J]. Nucleic Acids Res,2007,35(3):1 018 -1 037.
[35] Soper T,Mandin P,Majdalani N,et al. Positive regulation by small RNAs and the role of Hfq[J]. Proc Natl Acad Sci U S A,2010,107(21):9 602 -9 607.
