24个泥蚶EST-SSR标记的开发与分析
2013-10-13史松富姚韩韩林志华周小龙齐晓艳董迎辉
史松富, 姚韩韩, 林志华, 周小龙, 齐晓艳, 董迎辉
(1. 浙江万里学院 浙江省水产种质资源高效利用技术研究重点实验, 浙江 宁波 315100; 2. 上海海洋大学水产与生命学院, 上海 201306; 3. 宁波大学 海洋学院, 宁波 315211)
微卫星标记又称简单重复序列(simple seque ncerepeat, SSR), 其核苷酸序列是以2~6个核苷酸为一个单位重复排列。微卫星因其在基因组中分布广泛、多态性丰富、共显性遗传、重复性好以及实验操作简单等特点, 已被广泛应用于遗传多样性分析、分子标记辅助育种、遗传连锁图构建、QTL定位以及物种亲缘关系鉴定[1-3]。SSR标记的开发与分析在太平洋牡蛎(Crassostrea gigas)[4-5]、泥蚶(Tegillarca granosa)[6-7]、魁蚶(Scapharca brougtonii)[8]、栉孔扇贝(Chlamys farreri)[9-10]、文蛤(Meretrixmeretrix)[11]等海洋双壳贝类中已有报道。
泥蚶(Tegillarca granosa), 俗称血蚶、花蚶、粒蚶,为我国四大海水养殖贝类之一。泥蚶肉质鲜美、营养丰富, 市场前景广阔。关于泥蚶分子标记的研究已有很多报道, 如李太武等[12]利用RAPD标记技术分析了5个泥蚶群体的遗传多样性, 姚韩韩等[13]利用 AFLP标记技术对泥蚶4个快速生长家系的遗传变异进行了分析, 顾晓英等[14]通过磁珠富集法筛选了6对多态性SSR引物; 董迎辉等[6]利用泥蚶转录组文库开发了34个EST-SSR标记, Liu 等[7]开发了39个泥蚶EST-SSR位点。本研究在泥蚶转录组文库的基础之上, 探讨利用单拷贝序列筛选SSR标记的可行性及优缺点, 为泥蚶的种质资源保护、遗传多样性分析、遗传连锁图谱的构建以及种群鉴定提供有力工具。
1 材料与方法
1.1 实验材料与DNA提取
实验样品采自浙江宁波奉化海区, 随机取样 30粒, 活体解剖后取其闭壳肌, 保存于-20℃冰箱中备用。采用酚-氯仿法提取基因组DNA, 用1%的琼脂糖凝胶电泳检测 DNA的质量, 紫外分光光度计检测DNA的纯度与浓度, 调节DNA浓度最终为100 ng/μL。
1.2 候选SSR位点的获得与引物设计
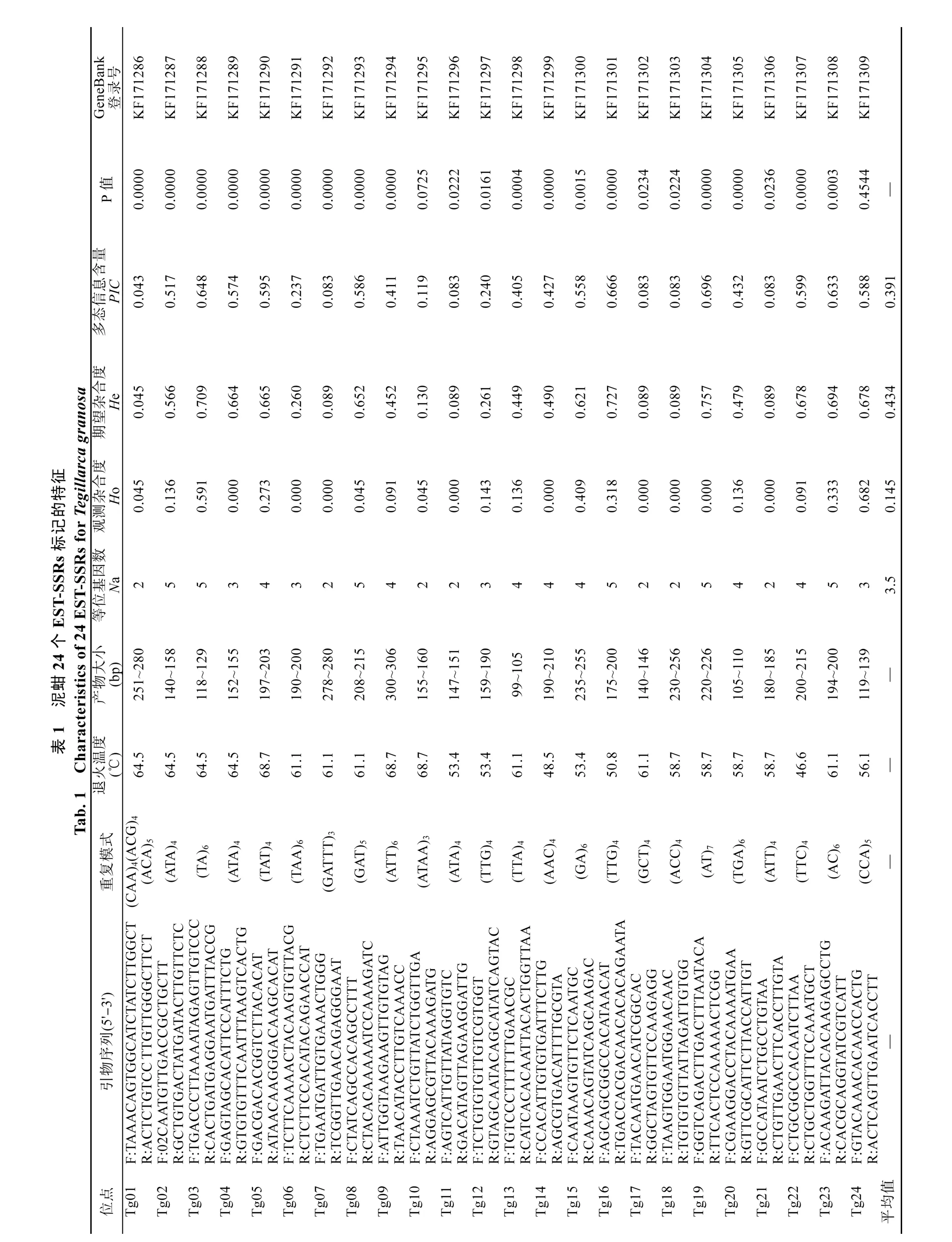
用SSR Hunter1.3软件在单拷贝序列中筛选微卫星位点, 筛选标准为二碱基、三碱基、四碱基、五碱基重复单元重复至少依次为 6、4、3、3次, 侧翼序列大于150 bp。利用软件Primer5.0设计引物, 引物长度18~24 bp, GC含量40%~60%, 引物由上海生工合成(表 1)。
1.3 泥蚶候选SSR位点检测与PCR扩增
随机取6个泥蚶DNA混成基因池, 对所有引物进行初步筛选。PCR反应体系为20 μL, 包括100 ng DNA, 0.2 mmol/L dNTP, 0.5U Taq 酶, 10×PCR buffer, 上游引物与下游引物各1 μmol/L, 1.5 mmol/L MgCl2。PCR反应条件: 94℃变性5 min; 94℃变性45 s,退火45 s, 72℃ 45 s, 共35循环; 72℃延伸8 min; 4℃保存。每对引物所需的退火温度见表1。用8%的非变性聚丙烯酰胺凝胶电泳检测PCR产物,电泳缓冲液为 1×TBE, 电压为 180 V, 电泳时间为3~4 h, EB染色, 用Biorad 凝胶成像系统检测结果。
?
1.4 泥蚶SSR位点分析
利用筛选的引物对泥蚶30个个体进行微卫星标记的多态性检测, 用8%的非变性聚丙烯酰胺凝胶电泳检测PCR产物。用CERVUS 3.0软件处理结果, 获得重要的遗传学参数: 等位基因数(Na)、期望杂合度(He)、观测杂合度(Ho)和多态信息含量(PIC)。用在线软件 GENEPOP进行哈迪-温伯格平衡(Hardy-Weinberg equilibrium, HWE)检测, 利用 Bonferroni correction法对结果进行校正。
2 结果
2.1 泥蚶SSR位点的筛选
利用SSR Hunter1.3软件搜索3000条单拷贝序列(singleton), 获得132条含有SSR位点的序列, 成功设计引物有 50条, 成功率为 37.88%; 利用 Primer5.0在各SSR位点两侧保守区设计引物, 41对引物获得稳定清晰的条带; 在泥蚶群体中对41对引物进行多态性检测, 24对引物的扩增产物表现为多态性。
在24个EST-SSR位点中, 三碱基重复类型的最多为18个, 占总SSR的75%; 其次二碱基重复类型的SSR有4个, 占总数的16.67%; 四碱基与五碱基重复类型的 SSR最少均为 1个, 均占总多态位点的4.17%(图 1)。
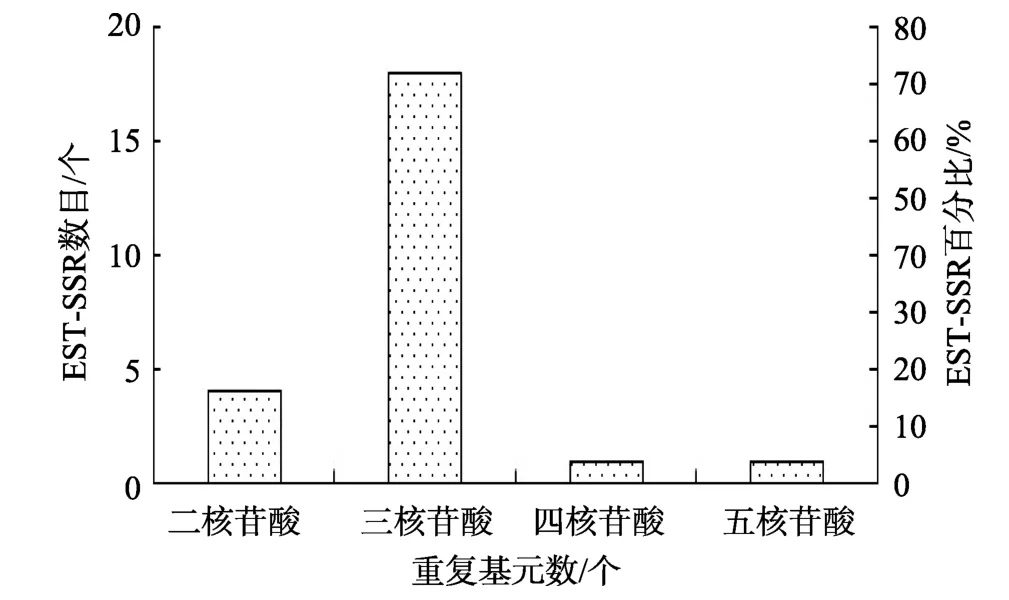
图1 泥蚶24个EST-SSR位点的核苷酸重复碱基的分布特征Fig. 1 Distribution of 24 EST-SSR motifs types in Tegillarca granosa
2.2 24个SSR位点对泥蚶群体的多态性评价
24个多态性位点在泥蚶 30个个体中共获得 84个等位基因, 多态位点的等位基因数为2~5个, 平均等位基因数为 3.5 个, 其中 Tg02、Tg03、Tg08、Tg16、Tg19、Tg23六个位点的等位基因数为5个。观测杂合度、期望杂合度分别为 0.000~0.591、0.045~0.727(表1)。PIC值是衡量群体变异程度的重要参数[15], 24个多态位点的PIC值介于0.043~0.696,平均值为 0.391, 其中有 11个高度多态位点(PIC>0.5)、 4个中度多态位点(0.25<PIC<0.5)、9个低度多态位点(PIC<0.25)。各多态位点经哈迪-温伯格平衡检测, 并用 Bonferroni correction法进行校正, 除Tg10、Tg11、Tg12、Tg17、Tg18、Tg21、Tg24 之外, 其余位点均显著偏离Hardy-Weinberg平衡。
3 讨论
目前SSR开发主要是基于构建的基因组文库和EST文库, EST-SSR的开发具有操作简单、成本低、高效率等特点, 越来越受到研究者的青睐。在海洋贝类中, 利用EST文库筛选SSR的研究已较深入, 如Wang等[16]利用从文蛤EST文库筛选出2970个候选SSR位点; Hou等[17]通过虾夷扇贝转录组文库得到2700个SSR候选位点。本研究在泥蚶转录组的单拷贝序列(singleton)中筛选SSR位点, 并将相关信息与从重叠群(contig)中筛选SSR位点的研究相比较, 分析singleton与contig筛选SSR位点的差异。
本研究利用泥蚶singleton筛选的SSR引物设计成功率为 37.88%, 董迎辉等[6]利用泥蚶 contig筛选SSR位点的引物设计成功率 73.97%, 引物设计成功率较低的原因有: (1)singleton的序列长度较短, 泥蚶singleton的平均长度为285.46 bp[18], 用于设计引物的区域相对较小, 而contig的平均长度为964.2 bp。(2)singleton中的一小部分被认为是在测序过程中被其他生物的序列污染或者是人工序列的产生[17]。
singleton来源的SSR同contig来源的SSR一样能反映生物群体的重要遗传学信息, 单拷贝序列的充分利用有利于群体的遗传多样性分析、种质资源的保护等, 如董迎辉等[6]利用 contig筛选 34个EST-SSR的研究中平均等位基因数为 3.59、平均多态信息含量为 0.396、平均期望杂合度为 0.447, Liu等[7]关于39个EST-SSR的研究中平均等位基因数、平均多态信息含量、平均期望杂合度分别为 3.9、0.402、0.449, 本实验利用singleton筛选的24个SSR多态位点的平均等位基因数为3.5、平均多态信息含量0.391、平均期望杂合度0.434。 目前EST-SSR筛选主要来自contig, 而关于singleton的开发应用以及与contig的比较研究较少。本研究证实利用singleton开发SSR标记是可行的, singleton在转录组文库中所占的比例较大且包含丰富的遗传信息, 特别在表达丰度较低基因的开发方面较 contig具有更重要的应用价值[17]。
[1]Somers D J, Isaac P, Edwards K. A high-density microsatellite consensus map forbread wheat (Triticum aestivum L.)[J]. Theoretical and Applied Genetics, 2004,109(6): 1105-1114.
[2]Morgante M, Hanafey M, Powell W. Microsatellites are preferentially associated with nonrepetitive DNA in plant genomes [J]. Nature Genetics, 2002, 30(2):194-200.
[3]McCouch S R, Teytelman L, Xu Y, et al. Development and mapping of 2240 new SSR markers for rice (Oryza sativa L.)[J]. DNA Research, 2002, 9(6): 199-207.
[4]Qi L, Wang Q Z, Qi M J, et al. Development,characterization, and inheritance of 113 novel EST-SSR markers in the Pacific oyster (Crassostrea gigas)[J].Genes & Genomics, 2011, 33:313-316.
[5]李慧娟, 亓海刚, 李莉, 等. 长牡蛎 17个 fosmid-SSR 标记的开发与分析[J], 水产学报, 2011,35(10):1464-1467.
[6]董迎辉, 吴国星, 姚韩韩, 等. 泥蚶34个EST-SSR标记的开发及在格粗饰蚶中的通用性检测[J]. 水产学报, 2013, 37(1):70-77.
[7]Liu B, Teng S S, Shao Y Q, et al. Isolation and characterization of 39 novel polymorphic EST-SSR loci for the blood clam,Tegillarca granosa[J]. Conservation Genet Resour, 2012, 4:375-378.
[8]吴彪, 梁超, 杨爱国, 等. 基于 SSR 标记的魁蚶(Scapharca broughtonii)不同群体遗传结构的分析[J].海洋与湖沼, 2012, 43(4):863-869.
[9]李红蕾, 宋林生, 王玲玲. 栉孔扇贝EST中微卫星标记的筛选[J], 高技术通讯, 2003: 72-75.
[10]Zhan A B, Bao Z M ,Hu X L, et al. Characterization of 95 novel microsatellite marker for Zhikong scallopChlamys farreriusing FIASCO-colony hybridization and EST database mining[J]. Fisheries Science, 2008,74(3): 516-526.
[11]朱东丽, 董迎辉, 林志华, 等. 利用微卫星标记对文蛤9个壳色花纹品系的遗传分析[J]. 水产学报, 2011,26(2): 202-209.
[12]李太武, 李成华, 宋林生, 等. 5个泥蚶群体遗传多样性的 RAPD分析[J]. 生物多样性, 2003, 11(2):118-124.
[13]姚韩韩, 董迎辉, 林志华, 等. 泥蚶 4个快速生长家系的遗传变异分析[J]. 水产学报, 2011, 35(3):350-357.
[14]顾晓英, 曾庆国, 尤仲杰, 等. 泥蚶(Tegillarca granosa)6个微卫星引物的分离和鉴定[J]. 海洋与湖沼, 2008, 39(6):661-664.
[15]Botstein D, White R L, Skolnick M, et al. Construction of a genetic-linkage map in man using restriction fragment length polymorphisms[J]. American Journal of Human Genetics, 1980, 32(3):314-331.
[16]Wang H X, Huan P, Lu X, et al.Mining of EST-SSR marker in clamMeretrix meretrixlarvae from 454 shotgun transcriptome[J]. Conservation Genetics Resource, 2011, 3(4):655-658.
[17]Hou R, Bao Z M, Wang S, et al. Transcriptome sequencing and de nove analysis for Yesso scallop(Patinopecten yessoensis)using 454 GS FLX[J]. Plos One, 2011, 6(6):e21560.
[18]董迎辉. 泥蚶高通量转录组分析及生长相关基因的克隆与表达研究[D]. 青岛:中国海洋大学, 2012:1-144.
