4个缢蛏群体遗传多样性和系统发生关系的微卫星分析
2013-10-13邵艳卿滕爽爽柴雪良张炯明肖国强
刘 博, 邵艳卿, 王 侃, 滕爽爽, 柴雪良, 方 军, 张炯明, 肖国强
(浙江省海洋水产养殖研究所 浙江省近岸水域生物资源开发与保护重点实验室, 浙江 温州325001)
缢蛏(Sinonovacula ConstrictaLamarck)俗称蛏子, 属软体动物门(Mollusca), 瓣鳃纲(Lamellibranchia)竹蛏科(solenidae)缢蛏属(sinonovacula)贝类。缢蛏为广温广盐性海产双壳类, 具有重要的经济价值, 是我国四大养殖贝类之一。缢蛏养殖历史悠久,过去主要集中在福建和浙江一带, 随着缢蛏养殖业的不断扩展, 山东、天津沿海地区开始从福建、浙江等地区引进苗种。浙江沿海为了能提早出苗, 每年9月份也大量从广东湛江和福建云霄等地购买亲贝催产育苗。无序的养殖、盲目引种、移养等使溢蛏种质资源混淆不清。因此, 分析不同地理群体遗传结构及种群间关系, 对缢蛏种质资源合理保护、开发和利用具有重要指导意义。
微卫星标记(SSR)与其他分子标记相比, 具有多态性高、多等位性、共显性、可重复性高、数量丰富和对基因组有很好的覆盖性等特点[1], 被广泛的用于种质资源及品种(系)鉴定、遗传结构分析、分子标记辅助选择等多方面的研究[2]。微卫星标记技术在太平洋牡蛎(Crassosttea Gigas)[3]、虾夷扇贝[4-5]、文蛤[6-8]、皱纹盘鲍(Haliotis discushanna)[9]等已有报道, 关于缢蛏在遗传多样性和遗传分化的研究已有线粒体COⅠ标记法, RAPD和同工酶标记等[10-12]; 目前, 缢蛏微卫星方面的研究仅局限在标记开发[13,14]和单一养殖群体的遗传多样性分析[15], 而关于用微卫星标记分析不同地理居群缢蛏遗传多样的报道至今还没有,本文首次利用SSR标记分析了4个缢蛏居群的遗传结构, 在分子水平上探讨了处于不同地理区域的缢蛏群体遗传多样性差异和遗传分化水平, 为其种质资源管理和遗传育种工作提供科学的理论依据。
1 材料与方法
1.1 实验材料
实验用4个缢蛏养殖群体, 于2011年9月分别采自福建云霄、广东湛江、天津塘沽及浙江乐清湾, 各群体随机取样30颗, 活体解剖取其水管、斧足放入无水乙醇固定保存, 带回实验室备用。
1.2 DNA提取
基因组 DNA的提取采用常规的蛋白酶K消化,苯酚、氯仿/异戊醇抽提, 无水乙醇沉淀的方法, 提取缢蛏肌肉组织的基因组 DNA, 用 TE缓冲液稀释至终浓度100 ng/μL用于实验分析。
1.3 PCR扩增及产物检测
微卫星位点的引物来自己发表的12对缢蛏引物序列(见表 1)[15]。PCR 反应体系为 15 μL, 包括 10×PCR buffer 1.5 μL, 25 mmol/L Mg2+1.5 μL, 2.5 mmol/L dNTP 1.2 μL, 10 μmol/L 上下游引物各 1.5 μL, Taq 酶(5U)0.075 μL, DNA 模板1.5 μL, 加灭菌双蒸水至15 μL。
PCR反应程序为: 94℃条件下变性5 min后, 进入30个PCR循环: 在94℃下变性30 s, 复性(温度见表1)30 s, 72 ℃下延伸45 s, 再最后72 ℃下延伸7 min,并将产物于4℃下保存。扩增产物用1.5%琼脂糖凝胶检测, 鉴定有扩增产物后再用8%的变性聚丙烯酰胺凝胶电泳检测, 银染。
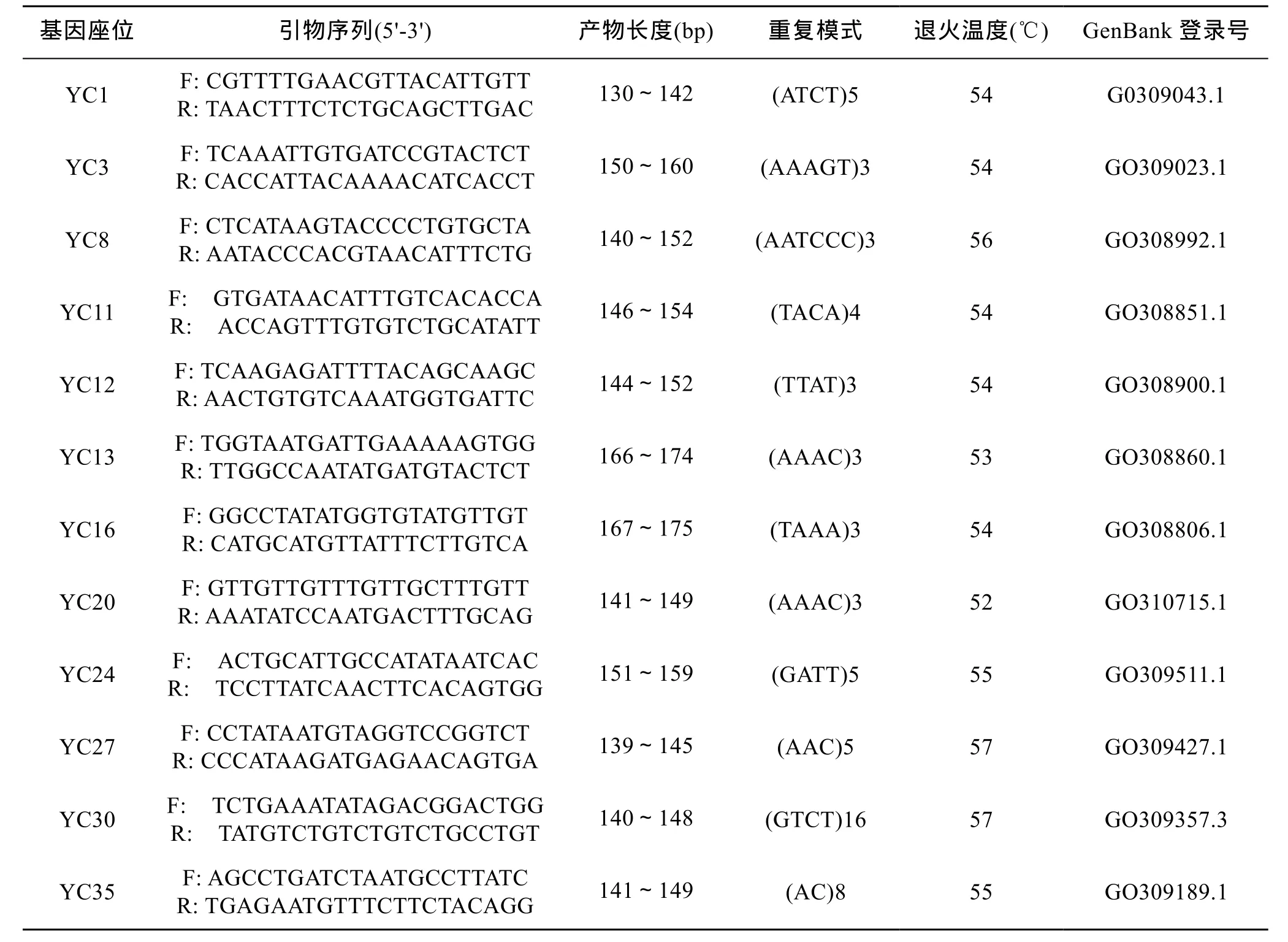
表1 12个多态微卫星引物序列Tab. 1 Primers sequences for the 12 polymorphic loci
1.4 数据分析
根据电泳图谱, 并参照引物设计时预期片段的大小统计条带, 应用 POPGENE32软件计算微卫星引物的期望杂合度(Expected heterozygosity,He)、观测杂合度(Ho)、哈代-温伯格平衡值(Hardy-Weinberg equilibrium,HWE)、FST、FIS及FIT等参数。根据遗传距离利用 MEGA4.0软件构建 UPGMA系统进化树。多态信息含量(Polymorphic information content,PIC)的计算公式如下:
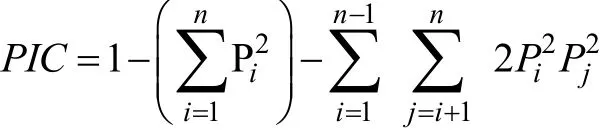
其中,Pi,Pj是某个位点第i、j个等位基因的基因频率,n为某个位点上的等位基因数。
2 结果
2.1 四个缢蛏群体的微卫星等位基因及多样性分析
12对引物共检测出 46个等位基因, 其中YC30位点等位基因数目最多为12个, 表现为高度多态; 每个位点等位基因数从2~12个不等, 平均每个位点观测等位基因数目为 3.83; 有效等位基因数从位点 YC8的 1.65到位点 YC30的 9.75, 平均有效等位基因数为3.03; 扩增片段长度在130~180 bp之间。
2.2 四个缢蛏群体内遗传变异
4个缢蛏群体的等位基因数(Ao)、有效等位基因数(Ae)、观测杂合度(Ho)、期望杂合度(He)、多态信息含量(PIC)及 Hardy-Weinberg检验结果见表 2。4个缢蛏群体的遗传差异较小, 平均等位基因数Ao在3.25~3.50, 有效等位基因数Ae在 2.57~2.69, 等位基因数最低的是天津缢蛏群体, 最高的是福建缢蛏群体, 平均值为 2.69。平均多态信息含量(PIC)在0.46~0.50之间, 以福建群体最高为0.50。群体观测杂合度Ho在 0.59~0.74之间, 最高的为天津群体,最低的为福建群体; 而4个群体的期望杂合度(He)差异不大, 在 0.55~0.58之间, 最高的是福建群体为0.58。Hardy- Weinberg平衡检验表明, 除福建云霄群体有4个位点偏离平衡外, 广东湛江群体、天津塘沽群体和浙江乐清湾群体中的大部分微卫星位点都偏离了Hardy- Weinberg平衡(表2)。
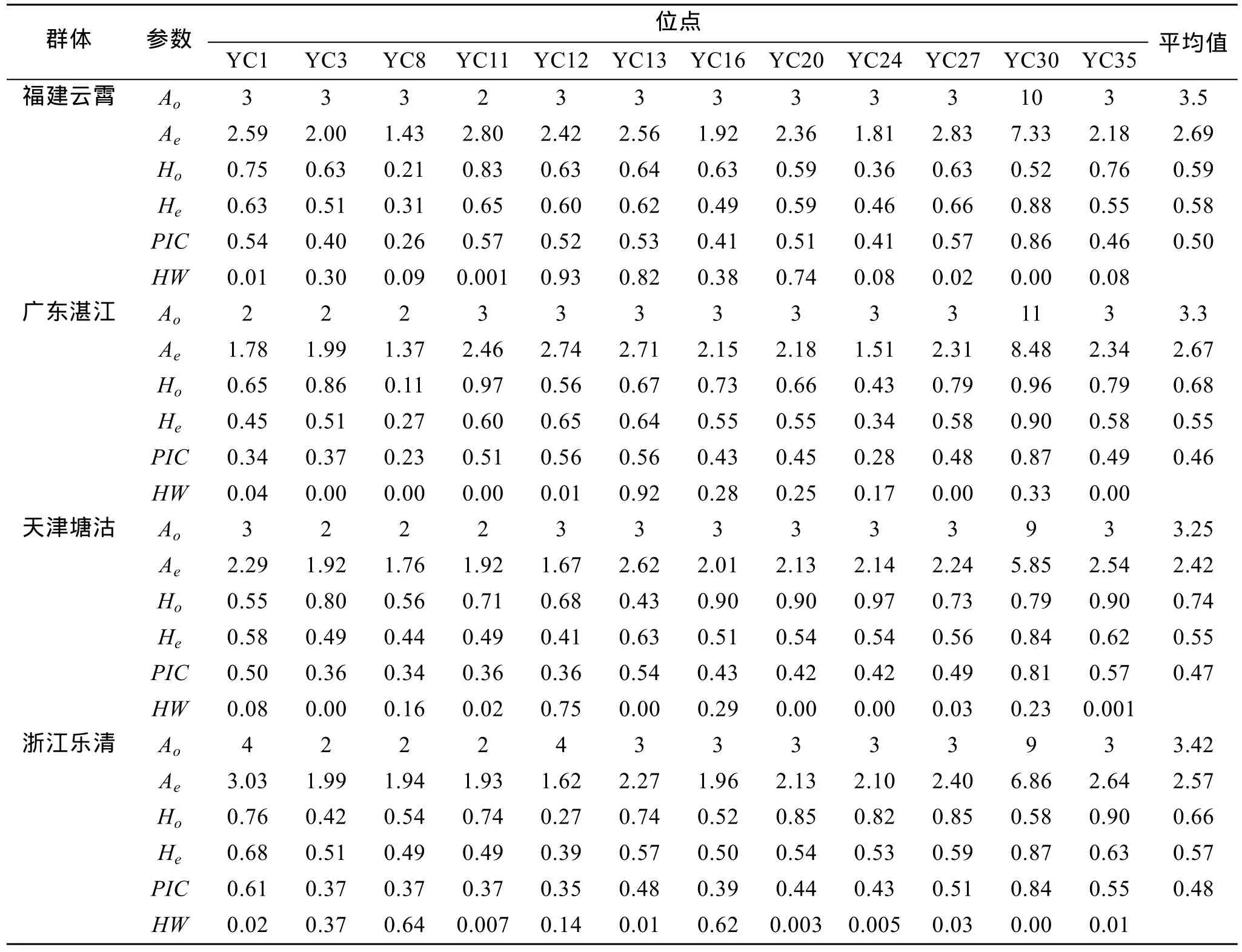
表2 12个微卫星位点在4个缢蛏群体中的遗传多样性及Hardy-Weinberg平衡检验Tab. 2 Genetic diversity and Hardy-Weinberg in four strains of S. constricta for the 12 polymorphic loci
2.3 不同缢蛏群体间遗传分化
应用Popgene32软件对4个缢蛏群体进行了F-统计量及基因流分析, 结果见表3。由表3可知, 4个缢蛏群体仅有 3个位点 YC8、YC12和 YC30的F-统计量为正值, 其余9个位点均为负值,Fis平均值为-0.21,Fit平均值为-0.31; 度量群体间遗传差异程度的Fst平均值为0.07。遗传分化系数(Fst)和基因流(Nm)分析不同群体的遗传分化情况, 发现缢蛏群体除了YC11、YC20、YC27与YC30等4个基因座固定指数小于0.05, 其余均大于种间无遗传分化的标准(Fst=0~0.05)[16], 基因流Nm平均值为3.33。
利用 MEGA4分析软件, 采用非加权的组平均法(UPGMA)对 4个群体进行聚类分析, 获得聚类树系图(图1), 4个缢蛏群体之间的Nei’s遗传距离和遗传相似度见表 4。4个缢蛏群体共分为 2大支系:浙江乐清湾群体和天津塘沽群体之间的遗传距离最小为 0.0344, 聚为第一支; 福建云霄群体和广东湛江群体之间的遗传距离为0.103, 聚为第二支。

表3 12个微卫星位点的固定指数及基因流Tab. 3 Fixation index and gene flow of 12 polymorphic loci

表4 Nei遗传相似度和遗传距离Tab. 4 Nei genetic similarity and genetic distance
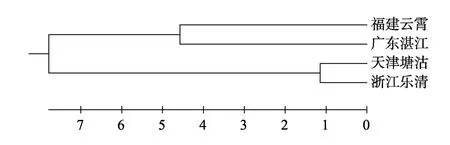
图1 根据群体间遗传距离用UPGMA法构建了4个泥蚶群体的系统进化树Fig. 1 Phylogenetic tree based on genetic distance of 4 four strains of S. constricta by UPGMA assay
3 讨论
3.1 缢蛏群体内遗传多样性
依据PIC判断标准:PIC<0.25为低度多态, 0.25≤PIC<0.5为中度多态,PIC≥0.5为高度多态[17], 可以看出本研究的 4个缢蛏群体的平均多态信息含量(PIC)基本一致在 0.46~0.50之间, 相差无几且接近于0.5, 表明4个缢蛏群体的遗传多样性处于中等偏上水平, 4个群体未出现明显的种质退化。
李成华等[18]和王冬群等[12]分别利用RAPD技术和同工酶技术分析了不同地理群体的野生缢蛏群体和养殖群体的遗传多样性, 野生群体的遗传多样性略高于养殖群体, 但与本实验相比, 以上各群体的遗传多样性明显低于本研究中 4个群体的, 分析原因主要与各实验采用的标记类型不同有关。显性标记RAPD与共显性标记SSR相比, 其无法区分显性纯合子和杂合子, 其多态信息含量仅是 SSR的 1/2;而 England等[19]通过对同工酶和微卫星标记多态性的比较发现, 同一群体的微卫星位点等位基因数为5.2个, 同工酶为 3.0个, 因此不同类型标记影响了对群体遗传多样性的分析。本研究中以天津群体的观测杂合度最高, 但从 4个缢蛏群体的平均等观测位基因数Ao来看, 天津群体的平均值最低为 3.25,等位基因数目最少, 基因型单一, 原因可能是这些基因型对环境的适应度较高, 经历了长时间的遗传漂变和适应性选择逐渐呈现出了高的基因频率, 进而产生了与其他地区群体不同的遗传结构[20]。
3.2 缢蛏群体间的遗传分化
群体Fst值在 0~0.05之间, 则表明其各亚群间不存在分化; 若Fst值在 0.05~0.15之间, 为中度分化; 若Fst值在 0.15~0.25之间, 则为高度分化。本研究结果群体间遗传差异程度的Fst=0.07, 表明4个缢蛏群体间的遗传变异为 7.0%, 而 93.0%为群体内的遗传变异, 两者比例悬殊, 说明所选4个缢蛏群体间的变异绝大部分来自群体内, 群体间的遗传分化程度不高, 而且这种差别也未影响到遗传水平。
固定指数Fis是检验群体杂合子缺失或过剩的指标,群体内固定指数Fis>0时, 表示品系内存在较严重的近交现象, 导致杂合子缺失;Fis<0时, 存在远交现象, 有杂合子过剩。本研究中发现12个位点中有9个位点存在杂合子过剩现象, 并且平均Fis=-0.21, 表示群体内并无明显的近交现象。一般认为杂合子过剩情况多的出现在研究对象为相对小的群体或者封闭群体, 如养殖群体中的子代群体往往由于亲本数量所限, 奠基者效应(Founder effect)会导致连锁不平衡现象, 继而造成杂合子过剩。分析本实验所出现的杂合子过剩的现象,可能与实验的样本量较小有关系。
当杂合子严重过剩即Fis明显偏离0时, 往往会引起群体显著地偏离Hardy-Weinberg平衡。本次研究中福建云霄群体、广东湛江群体、天津塘沽群体和浙江乐清湾群体的12个微卫星位点中分别有4、7、7和8个位点偏离了Hardy-Weinberg平衡。这种现象在其他海水贝类, 如类海湾扇贝[21]、栉孔扇贝[22]中也普遍存在。本研究中的4个缢蛏群体均处于不平衡状态, 这可能与群体间的无序养殖、迁移有关。浙江乐清湾是一个相对封闭的海湾, 但近年来人工育苗中, 为了提早培育出苗, 每年9至10月份都会从广东、福建等地大量采购亲贝进行人工繁殖, 而培育出的苗种除部分在本地中培和养殖外, 其余的又重新销往福建、广东、天津、辽宁等地, 因此迁移造成了对基因型的影响, 可能是导致这种不平衡的主要原因。
3.3 缢蛏群体间遗传距离与系统发生关系
遗传距离是研究物种遗传多样性的基础, Crawford等[23]指出由微卫星得出的遗传距离更能反映分化时间的长短, 能客观地反映品种间的遗传变异和分化。本研究中, 浙江乐清湾群体和天津塘沽群体之间的遗传距离最小为 0.0344, 聚为第一支; 福建云霄群体和广东湛江群体之间的遗传距离为 0.103, 聚为第二支。聚类结果与牛东红[24]利用ISSR标记对6个野生缢蛏群体的聚类结果差异较大, 并未表现出遗传距离和地理距离之间的正相关。出现这种情况可能与近年来缢蛏无序养殖有关, 福建、广东、浙江等沿海存在相互间盲目引种和移养的现象, 造成缢蛏资源混乱并使本土自然资源混淆不清, 目前已经很难找到未受人为因素干扰的野生缢蛏群体了。另外, 聚类结果的差异, 也可能与采用的分子标记类型不同有关。
3.4 缢蛏种质资源的保护与开发策略
由于本研究中的 4个群体均来源自当地的养殖群体, 可能已经受到了外来缢蛏群体的污染, 不能充分代表当地野生缢蛏种质资源状况, 因此建议在做好对当地原始野生缢蛏资源收集的情况下, 利用不同分子标记技术分析各地野生缢蛏原种间及野生原种和人工养殖群体的遗传差异, 根据主要遗传差异来源的不同(群体间还是群体内), 制定合理的保种策略。
针对本研究 4个缢蛏群体的变异绝大部分来自群体内, 群体间的遗传分化程度不高的情况, 笔者认为可以采取如下保种和开发的措施: (1)采取原位保存的措施, 通过建立原种场和自然保护区等方法加强野生原种的保护, 避免外来缢蛏群体对原种的污染。(2)由于4个群体的遗传变异主要来自群体内,说明合理有序的引种不会带来不利的遗传效应[25],因此, 在正确收集和保护好野生原种的基础上, 可以适当引进其他地理群体的优质缢蛏种质来提高当地种质资源的生产率。
[1]Powell W, Machray G C, Provan J. Polymorphism revealed by simple sequence repeats[J]. Trends Plant Science, 1996, 1: 215-222.
[2]Zane L, Bargelloni L, Patamello T. Strategies for microsatellites isolation: a review [J]. Molecular Ecology, 2002, 11: 1-16.
[3]李琪, 木岛明博.长牡蛎(Crassostrea gigas)微卫星克隆快速分离及特性分析[J].海洋与湖沼, 2004, 35(2):364-370.
[4]赵莹莹, 朱晓琛, 孙效文, 等.磁珠富集法筛选虾夷扇贝微卫星序列[J].中国水产科学, 2006, 13(5):749-755.
[5]李春艳, 丁君, 常亚青, 等. 虾夷扇贝微卫星标记的分离及其养殖群体的遗传结构分析[J]. 中国水产科学, 2009, 16( 1): 39-46.
[6]Lu X, Wang H X, Liu B Z, et al. Microsatellite-based genetic and growth analysis for a diallel mating design of two stocks of the clam,Meretrix meretrix[J]. Aquaculture Research, 2011: 15-18.
[7]朱东丽, 董迎辉, 林志华, 等. 利用微卫星标记对文蛤 4个壳色花纹品系的遗传分析.水产学报, 2012,36(2): 202-209.
[8]陈淑吟, 吉红九, 许广平, 等. 文蛤微卫星 DNA 的筛选及其特性分析[J]. 生物技术通报, 2009, 12:134-138.
[9]李莉, 孙振兴, 杨树德, 等. 用微卫星标记分析皱纹盘鲍群体的遗传变异[J]. 遗传, 2006, 28(12):1549-1554.
[10]牛东红, 陈慧, 王树亮, 等. 我国沿海缢蛏居群遗传结构的mtDNA-COⅠ分析[J]. 动物学杂, 2010, 45(2): 11-18.
[11]于颖, 孟祥盈, 王秀利, 等. 缢蛏遗传多样性的RAPD分析[J]. 生物技术通报, 2007, 6:138-143.
[12]王冬群, 李太武, 苏秀榕, 等. 象山缢蛏养殖居群和野生居群遗传多样性的比较[J]. 中国水产科学, 2005,12(2):138-143.
[13]Niu D H, Li J L, Liu B D. Polymorphic microsatellite loci for population studies of the razor clamSinonovacula constricta[J].Convser Genet, 2008, 9: 1393-1394.
[14]刘 博, 滕爽爽、邵艳卿, 等. 缢蛏(Sinonovacula constricta)EST-SSR分布特征及引物开发利用[J].海洋与湖沼, 2012, 43(1):132-137.
[15]刘博, 邵艳卿, 滕爽爽, 等. 乐清湾养殖缢蛏群体遗传结构的微卫星标记分析[J].中国农学通报,2012,28(2):69-73.
[16]Strauss W M. Preparation of genomic DNA from mammalian tissues. (In): Ausubel F.M, Brent R, Kingston R E, eds. Current protocol in molecular biology[M]. New York, John Wiley and Sons, 1989, 221-222.
[17]Botstein D, White R L, Skolnick M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. Am J Hum Genet,1980, 32:314-331.
[18]李成华, 李太武, 宋林生, 等. 4个缢蛏群体遗传结构的RAPD分析[J].水产科学, 2004,23(12):26-28.
[19]England P R, Briscoe D A, Frankham R. Microsatellite polymorphisms in a wild population of Drosophila melanogaster[J]. Genetical Research, 1996(67): 285-290.
[20]游翠红, 童金苟, 俞小牧. 大鳞副泥鳅 7个群体遗传变异的微卫星分析[J].水生态学杂志, 2012, 33 (1):84-91.
[21]Zhan A B, Hu J J, Wang X L, et al. A panel of polymorphic EST-derived microsatellite loci for the bay scallop (Argopecten irradians)[J]. Journal of Molluscan Studies, 2006, 72(4): 436-439.
[22]战爱斌, 胡景杰, 胡晓丽, 等. 富集文库-菌落原位杂交法筛选栉孔扇贝的微卫星标记[ J].水产学报, 2008,32(3): 353-361.
[23]Crawford A M, Littlepohn R P. The use of DNA marker in deciding conservation priorities in sheep and other livestock[J]. Animal Genetic Resources Information,1998, 23(1): 21-26.
[24]牛东红, 李家乐, 冯冰冰, 等. 缢蛏 6个群体遗传结构的ISSR分析[J]. 应用与环境生物学报, 2009, 15(3):332-336.
[25]Rossetto M, Weaver P K, Dixon K W. Use of RAPD analysis in devising conservation strategies for the rare and endangeredGrevillea scapigera(Proteaceae)[J].Molecular Ecology, 1995, 4: 321-329
