氨氧化古菌研究进展*
2013-08-16罗剑飞林炜铁
罗剑飞 林炜铁
(华南理工大学 生物科学与工程学院,广东 广州 510006)
百年来,作为生物硝化作用的限速步骤,由氨单加氧酶(AMO)催化的氨氧化反应一直被认为是一些化能自养氨氧化细菌(AOB)作用的结果;这些细菌主要包括来自β 变形菌纲的Nitrosomonas 和Nitrosospira属的细菌和γ 变形菌纲Nitrosococcus 属的细菌[1].然而,通过对海洋和陆地环境中氨氧化作用的原位分析,发现很多氨氧化作用经常在较低的底物浓度下进行,而这些底物浓度远远低于AOB 生长所需的浓度,因此推测环境中还存在未知的氨氧化微生物[2].
近年来,随着分子生物技术的发展,科学家们从构建的海水等环境样品的宏基因组文库中筛选到了参与氮素循环的泉古菌的克隆子,并通过高通量的测序技术发现这些古菌基因中含有编码AMO 的基因,且与细菌AMO 基因类似,因此推测这些基因编码的酶可能有氨氧化作用[3-5].而随着测序技术和基因组学的发展,在3 种目前已知的氨氧化古菌(AOA)的基因组序列中也发现含有类似细菌AMO 的基因[6-8].因此,基于AOA 的AMO 亚基的基因amo A 的特异性引物和探针,发现amo A 广泛分布于地球上各种环境中,包括海水、温泉、土壤、河口、沉积物、动物排泄物等,且很多环境中AOA 的数量是AOB 的几倍甚至数千倍,特别是在一些低溶氧的环境中[9-15].
AOA 的发现极大地丰富了氮的生物地球化学循环的知识,自AOA 和AOA amo 基因发现的不到十年间,国内外已做了大量的研究,国内也有多篇综述报道[16-19].本课题组长期从事自然生态和人工生态中氮、硫、磷等元素的生物地球化学循环的理论研究,通过对珠三角地区淡水水产养殖环境中的氨氧化微生物的研究,发现该环境中AOB 主要由β 变形菌纲的Nitosomonas 和Nitrosospira 组成,AOA 主要来自于Candidatus Nitrososphaera gargensis[20-21];通过稳定同位素标记(SIP)DNA 的研究,也进一步证明了上述结论[22];通过定量研究发现,该环境中古菌amo A 的数量是细菌amo A 的60~100 倍[23].文中根据笔者对AOA 的研究及认识,就当前AOA 在菌株和富集物、系统发育、氨氧化和反硝化作用等方面的研究进展进行了综述.
1 AOA 菌株和富集物
2005年,Könneke 等[6]通过富集和筛选的方法从热带海水水族缸中分离到了一株泉古菌SCM1,这是最早报道的一株可培养的AOA.SCM1 的基因序列与Nitrosopumilus maritimus 具有同源性,在此之前,N.maritimus 的amo A 基因在海水、海绵、污水处理系统、土壤等环境中均有发现[24-26].N.maritimus SCM1 可以氨氮为唯一能源进行生长,并将NH+4转化为,与AOB 相比,N.maritimus SCM1 的底物浓度阈值和半饱和常数是极其低的,对低氨氮浓度具有较强的适应性[27];N.maritimus SCM1 的这一生长特性可以适应各种寡营养的海水或极端环境,因此也解释了为何很多自然环境中AOA 的数量是AOB 的数十倍,甚至数万倍.
2011年,Tourna 等[28]从菜园土壤中富集和分离到了一株AOA,并命名为Nitrososphaera viennensis EN76,这也是迄今为止分离到的第二株可培养AOA,是唯一的一株来源于土壤的AOA 菌株.与海洋型N.maritimus SCM1 相比,N.viennensis EN76 生长的最高浓度可达20 mmol/L,是N.maritimus SCM1的10 倍;除氨氮外,N.viennensis EN76 可以尿素为唯一能源进行生长,在其基因组中含有编码脲酶的基因ureABCDEFG,这些基因也在Cenarchaeum symbiosum 中被发现[28-29];而N.maritimus SCM1 由于缺乏编码脲酶和转导尿素的蛋白的基因,只能在无机培养基中生长,不能利用尿素等有机氮源[30].
AOA 匮乏的生长条件和生理生化信息阻碍了从环境中获取其纯化的菌株.目前除可培养的N.maritimus SCM1 和N.viennensis EN76 外,通过富集的方法,分别从温泉、土壤、沉积物、海绵等环境中得到了Candidatus N.gargensis、Nitrosocaldus yellowstonii HL72、Candidatus Nitrosopumilus NM25、Candidatus Nitrosoarchaeum koreensis MY1、Candidatus Nitrosoarchaeum limnia SFB1、Candidatus Nitrosoarchaeum limnia BG20、C.symbiosum、Candidatus Nitrosotalea devanaterra、Candidatus Nitrosopumilus salaria BD31、Archaeon SJ 的富集物(如表1 所示).尽管不是纯培养物,但这些富集物中AOA 的丰度均较高,特别是Candidatus N.devanaterra 富集物,其AOA 细胞浓度高达99%,这样的富集物可用于AOA 氨氧化等特性的研究.

表1 AOA 菌株和富集物的系统发育和基因组信息1)Table 1 Phylogenetic and genome information of AOA strains or enrichments
2 AOA 的系统发育
根据16S rRNA 基因的同源性,1990年Woese等[41]将已培养的嗜极端环境的古菌分为广古菌门(Euryarchaeota)和泉古菌门(Crenarchaeota).随着分子生物技术的发展,发现古菌也广泛分布在很多中温环境和土壤环境中,这些古菌因此被称为中温泉古菌(mesophilic Crenarchaeota).这些中温古菌多样性大、丰度高,比较基因组学分析表明这些中温古菌在古菌的系统发育中形成独立的分支;根据16S rRNA基因序列的系统发育树分析,所有已知的AOA 都属于这个分支.
2008年,Brochier-Armanet 等[42]基于核糖体蛋白氨基酸序列和16S rRNA 基因序列,将包括AOA在内的中温古菌从泉古菌门和广古菌门分离,划分为一个新的门,作为古菌域的第三个类群——Thaumarchaeota(如图1 所示).

图1 Thaumarchaeota 的系统发育[43]Fig.1 Phylogeny of Thaumarchaeota
张丽梅等[19]将Thaumarchaeota 译为奇古菌门.自此,目前已知的AOA 中来源于温泉的N.yellowstonii 归为Group Ⅲ,N.gargensi 和来源于土壤的N.viennensis归为Group 1.1b,来自海洋环境的C.symbiosum,N.maritimus,N.limnia 和来自农田根际土壤的N.koreensis 被归为Group 1.1a.Candidatus N.devanaterra 的16S rRNA 基因和amo A 基因与Group1.1a 的同源性高于Group1.1b,因此将其归为Group1.1a,但与其他已知的Group1.1a 的序列比较又形成独立的分支,因此最终将其归为一类新的分支——Group1.1a-associated(如图2 所示).
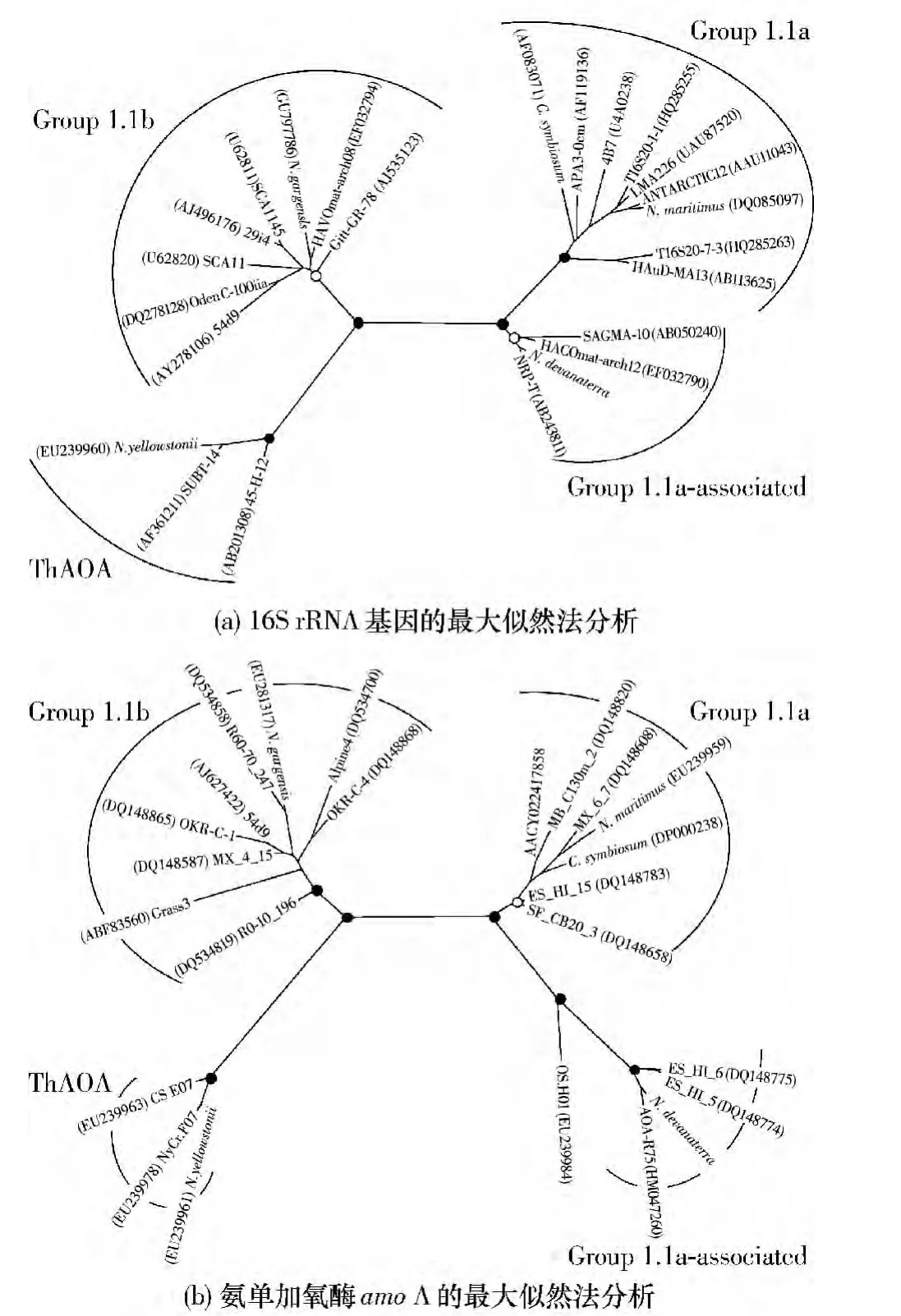
图2 N.devanaterra 系统发育分析[40]Fig.2 Phylogenetic analysis of N.devanaterra.
3 AOA 与氨氧化作用
基于特异性核苷酸标记16S rRNA 基因和amo A的研究,发现AOA 广泛分布于土壤、沉积物、海洋、河口、淡水、热泉、废水处理系统等各种生态环境中.特别地,对这些基因丰度的研究结果表明,大多数生态中AOA 的数量是AOB 的数十几倍甚至上万倍.海洋生态中AOA 多样性高、分布广、数量多,N.maritimus在一些海水中的数量占总海洋微生物数量的20%~30%[24-25];同时,N.maritimus 和几种海洋型AOA 表现出比AOB 高的氨氮亲和力,可以在低氨氮浓度的环境中生长代谢,对环境有较强的适应性[27,44],丰度和氨氧化特性的研究表明,AOA可能是海洋中主要的氨氧化作用贡献者.
然而在淡水和土壤环境中,AOA 和AOB 的丰度因生态环境而各异,它们的氨氧化能力受环境温度、盐度、溶解氧、氨氮浓度等理化因子的影响.Zhang等[45]利用12CO2和13CO2分别作为农田土壤样品中AOA 和AOB 的碳源,以amo A 和CO2固定的关键基因hcd 为标记,发现随着富集的进行,AOA 的13Camo A 和13C-hcd 基因的丰度增加,而AOB 的DNA中一直没有检测到13C-amo A,表明只有AOA 固定CO2进行自养生长.Pratscher 等[46]同样用13CO2分别作为农田土壤样品中AOA 和AOB 的碳源,amo A为标记,开展了基于RNA 稳定同位素的探测(RNASIP),结果表明AOA 和AOB 都能固定CO2和氨氧化作用,而基于DNA 稳定同位素探测(DNA-SIP)的结果表明只有AOB 而不是AOA 固定CO2进行自养生长,同样的研究方法却得到相反的结果.尽管很多研究证明了AOA 的氨氧化特性,但它们对环境中氨氧化作用的贡献是否像它们的数量一样,仍需要进一步深入地研究.
4 AOA 与反硝化作用
N.maritimus SCM1 是最早分离得到的AOA,也是世界上公认的AOA 模式菌株,研究者们以SCM1菌株出发进行了其系统进化、基因结构、代谢路径、氨氧化动力学等的研究和分析,相关研究工作如图3(根据文献[6,27,30,47]进行绘制)所示.海洋环境中,温室气体一氧化二氮(N2O)的排放常被认为是细菌硝化和反硝化作用的结果,AOB 在通过“硝化-反硝化”途径将NH+4氧化为NO-2的过程中,同时将生成的NO-2还原为N2O[48].基于SCM1 纯菌株的研究表明:N.maritimus 在低溶氧条件下具有很高的N2O 释放能力,而相同条件下AOB 生成N2O 的能力却很低[49];Santoro 等[49]利用稳定同位素技术对AOA 的富集物进行了研究,证明其在氨氧化的过程中也会产生N2O,AOA 对海洋环境N2O 产生的贡献远大于细菌的硝化和反硝化作用;对该富集物中AOA 的amo A 和16S rRNA 的基因分析表明,其与N.maritimus有很高的同源性[36].这些研究说明N.maritimus 等AOA 对自然界N2O 的产生起重要的作用,是海洋环境反硝化作用的主要贡献者.
尽管对纯培养物和富集物的研究证明了AOA具有释放N2O 的功能,但至今在AOA 乃至古菌中仍未发现催化N2O 生成的酶,也未能在原位进行AOA 氨氧化和反硝化的研究,也许基于基因组的生物信息学分析能揭开或预测AOA 氨氧化和反硝化的特性.通过分析N.maritimus SCM1 基因组,发现该菌缺少编码羟胺(NH2OH)氧化还原酶的基因,推测AMO 催化NH+4氧化的过程中不产生NH2OH,而是生成一种类似硝酰基化合物(例如HNO)的中间产物,HNO 分子被氧化为NO-2 ,或转化成N2O 溢出[47].另外,在Nitrosopumilus sp.MY1 基因组中发现含有与反硝化有关的编码亚硝酸盐还原酶和一氧化氮氧化还原酶的基因[35].
反硝化细菌通过nirK 编码的亚硝酸盐还原酶催化NO-2生成NO,是反硝化过程中的关键酶之一,AOB 中也发现有nirK,其编码的NirK 通过反硝化途径生成N2O[50-51].有趣的是,在土壤Thaumarchaeota文库[4]、环境宏基因组[52]和AOA 基因组序列[30,37]中都发现了与反硝化菌和AOB nirK 同源的基因序列.AOA 的NirK 序列与细菌NirK 仅有30%的相似性,虽然都是铜离子型的酶,但AOA 的NirK 序列在N-端可能编码一个跨膜蛋白,在C-端有180 个氨基酸编码的amicyanin 铜蛋白[53].Lund 等[54]基于nirK序列研究了海洋和沿海环境中nirK 的多样性和丰度,结果表明Thaumarchaeal nirK 的丰度是amo A 的10~100 倍,nirK 序列揭示的AOA 多样性比amo A要高,说明nirK 广泛存在于AOA 中.该结果更进一步证明了AOA 的反硝化特性.AOA 普遍具有生成N2O 的能力,它可能是海洋甚至陆地和淡水环境中N2O 释放的主要动力.
5 展望
基于特异性标记16S rRNA 基因和amo A 的研究揭示了环境中AOA 的分布、多样性、丰度、种群结构等生态学;基于纯培养物和富集物的研究揭示了AOA 的氨氧化特性、反硝化特性等理化特性;基于基因组的研究揭示了AOA 的系统进化发育、物质和能量代谢途径等.AOA 的发现极大地丰富了生态环境中关于氨氧化微生物、氨氧化作用和氮的生物地球化学循环的知识.在不到10年的研究中,AOA 一直是微生物生态学和生物地球化学循环研究的热点.然而,对于AOA 的研究刚处于起步阶段,还有很多的未知问题亟待科研工作者的潜心研究,例如:1)AOA 的AMO 蛋白结构与氨氧化功能,以及AOA反硝化产生N2O 的作用机理;2)原位环境的AOA氨氧化和反硝化作用,以及与AOB 相比对环境中氨氧化作用的贡献;3)AOA 的营养代谢类型,有机物对其生长的影响;4)AOA 与亚硝酸盐氧化菌(NOB)之间的生态关系;5)来源于淡水生态的AOA 菌株的
分离纯化,其基因组和功能基因与海洋型AOA 的比较分析.

图3 N.maritimus 的相关研究工作Fig.3 Related researches on N.maritimus
[1]Koops H P,Purkhold U,Pommerening-Röser A,et al.Prokaryotes:an evolving electronic resource for the microbiological community [M].Dworkin M ed.New York:Springer-Verlag,2003.
[2]Prosser J I.Autotrophic nitrification in bacteria[J].Adv Microb Physiol,1989,30:125-181.
[3]Schleper C,Jurgens G,Jonuscheit M.Genomic studies of uncultivated archaea[J].Nature Rev Microbiol,2005,3(6):479-488.
[4]Treusch A H,Leininger S,Kietzin A,et al.Novel genes for nitrite reductase and Amo-related proteins indicate a role of uncultivated mesophilic crenarchaeota in nitrogen cycling [J].Environ Microbiol,2005,7(12):1985-1995.
[5]Venter J C,Remington K,Heidelberg J F,et al.Environmental genome shotgun sequencing of the Sargasso Sea[J].Science,2004,304(5667):66-74.
[6]Könneke M,Bernhard A E,de la Torre J R,et al.Isolation of an autotrophic ammonia-oxidizing marine archaeon[J].Nature,2005,437(7058):543-546.
[7]de la Torre J R,Walker C B,Ingalls A E,et al.Cultivation of a thermophilic ammonia oxidizing archaeon synthesizing crenarchaeol[J].Environ Microbiol,2008,10(3):810-818.
[8]Hatzenpichler R,Lebedeva E V,Spieck E,et al.A moderately thermophilic ammonia-oxidizing crenarchaeote from a hot spring [J].Proc Natl Acad Sci USA,2008,105(6):2134-2139.
[9]Caffrey J M,Bano N,Kalanetra K,et al.Ammonia oxidation and ammonia-oxidizing bacteria and archaea from estuaries with differing histories of hypoxia [J].ISME J,2007,1(7):660-662.
[10]Ingalls A E,Shah S R,Hansman R L,et al.Quantifying archaeal community autotrophy in the mesopelagic ocean using natural radiocarbon[J].Proc Natl Acad Sci USA,2006,103(17):6442-6447.
[11]Wuchter C,Abbas B,Coolen M J L,et al.Archaeal nitrification in the ocean [J].Proc Natl Acad Sci USA,2006,103(33):12317-12322.
[12]Adair K L,Schwartz E.Evidence that ammonia-oxidizing archaea are more abundant than ammonia-oxidizing bacteria in semiarid soils of northern Arizona,USA[J].Microb Ecol,2008,56(3):420-426.
[13]Herrmann M,Saunders A M,Schramm A.Archaea dominate the ammonia-oxidizing community in the rhizosphere of the freshwater macrophyte Littorella uniflora[J].Appl Environ Microbiol,2008,74(10):3279-3283.
[14]Leininger S,Urich T,Schloter M,et al.Archaea predominate among ammonia-oxidizing prokaryotes in soils[J].Nature,2006,442(7104):806-809.
[15]Santoro A E,Francis C A,De Sieyes N R,et al.Shifts in the relative abundance of ammonia-oxidizing bacteria and archaea across physicochemical gradients in a subterranean estuary [J].Environ Microbiol,2008,10(4):1068-1079.
[16]胡安谊,焦念志.氨氧化古菌——环境微生物生态学研究的一个前沿热点[J].自然科学进展,2009,19(4):370-379.Hu An-yi,Jiao Nian-zhi.Ammonia-oxidizing archaea——Hot topics in environmental microbiology[J].Progress in Natural Science,2009,19(4):370-379.
[17]贾仲君,翁佳华,林先贵,等.氨氧化古菌的生态学研究进展[J].微生物学报,2010,50(4):431-437.Jia Zhong-jun,Weng Jia-hua,Lin Xian-gui,et al.Microbial ecology of archaeal ammonia oxidation——A review[J].Acta Microbiologica Sinica,2010,50(4):431-437.
[18]刘晶静,吴伟祥,丁颖,等.氨氧化古菌及其在氮循环中的重要作用[J].应用生态学报,2010,21(8):2154-2160.Liu Jing-jing,Wu Wei-xiang,Ding Ying,et al.Ammoniaoxidizing archaea and their important role in nitrogen biogeochemical cycling,A review[J].Chinese Journal of Applied Ecology,2010,21(8):2154-2160.
[19]张丽梅,贺纪正.一个新的古菌类群——奇古菌门(Thaumarchaeota)[J].微生物学报,2012,52(4):411-421.Zhang Li-mei,He Ji-zheng.A novel archaeal phylum:Thaumarchaeota——A review[J].Acta Microbiologica Sinica,2012,52(4):411-421.
[20]高利海,林炜铁.南美白对虾养殖底泥中氨氧化细菌与氨氧化古菌多态性分析[J].微生物学报,2011,51(1):75-82.Gao Li-hai,Lin Wei-tie.Diversity of β-proteobacterial ammonia-oxidizing bacteria and ammonia-oxidizing archaea in shrimp farm sediment[J].Acta Microbiologica Sinica,2011,51(1):75-82.
[21]李敬源,林炜铁,罗剑飞,等.典型对虾养殖水体中参与硝化与反硝化过程的微生物群落结构[J].微生物学报,2012,52(4):478-488.Li Jing-yuan,Lin Wei-tie,Luo Jian-fei,et al.Community structure of microbes involved in nitrification and denitrification in typical shrimp-farming water[J].Acta Microbiologica Sinica,2012,52(4):478-488.
[22]蔡小龙.基于稳定性同位素探测技术的养殖水体氮素循环微生物的多样性研究[D].广州:华南理工大学生物科学与工程学院,2011.
[23]高利海.南美白对虾养殖底泥中氨氧化细菌与氨氧化古菌多态性分析.[D].广州:华南理工大学生物科学与工程学院,2010.
[24]Herndl G J,Reinthaler T,Teira E,et al.Contribution of Archaea to total prokaryotic production in the deep Atlantic Ocean [J].Appl Environ Microbiol,2005,71(5):2303-2309.
[25]Karner M B,DeLong E F,Karl D M.Archaeal dominance in the mesopelagic zone of the Pacific Ocean[J].Nature,2001,409(6819):507-510.
[26]Massana R,DeLong E F,Pedros-Alio C.A few cosmopolitan phylotypes dominate planktonic archaeal assemblages in widely different oceanic provinces[J].Appl Environ Microbiol,2000,66(5):1777-1787.
[27]Martens-Habbena W,Berube P M,Urakawa H,et al.Ammonia oxidation kinetics determine niche separation of nitrifying archaea and bacteria[J].Nature,2009,461(7266):976-979.
[28]Tourna M,Stieglmeier M,Spang A,et al.Nitrososphaera viennensis,an ammonia oxidizing archaeon from soil[J].Proc Natl Acad Sci USA,2011,108(20):8420-8425.
[29]Hallam S J,Mincer T J,Schleper C,et al.Pathways of carbon assimilation and ammonia oxidation suggested by environmental genomic analyses of marine Crenarchaeota[J].PLoS Biol,2006,4(4):520-536.
[30]Walker C B,de la Torre J R,Klotz M G,et al.Nitrosopumilus maritimus genome reveals unique mechanisms for nitrification and autotrophy in globally distributed marine crenarchaea[J].Proc Natl Acad Sci USA,2010,107(19):8818-8823.
[31]Matsutani N,Nakagawa T,Nakamura K,et al.Enrichment of a novel marine ammonia-oxidizing archaeon obtained from sand of an Eelgrass zone[J].Microbes Environ,2011,26(1):23-29.
[32]Park B,Park S,Yoon D,et al.Cultivation of autotrophic ammonia-oxidizing archaea from marine sediments in coculture with sulfur-oxidizing bacteria[J].Appl Environ Microbiol,2010,76(22):7575-7587.
[33]Stein J L,Marsh T L,Wu K Y,et al.Characterization of uncultivated prokaryotes:isolation and analysis of a 40-kilobase-pair genome fragment from a planktonic marine archaeon[J].J Bacteriol,1996,178(3):591-599.
[34]Hallam S J,Konstantinidis K T,Putnam N,et al.Genomic analysis of the uncultivated marine crenarchaeote Cenarchaeum symbiosum[J].Proc Natl Acad Sci USA,2006,103(48):18296-18301.
[35]Kim B K,Jung M,Yu D S,et al.Genome sequence of an ammonia-oxidizing soil archaeon“candidatus Nitrosoarchaeum koreensis”MY1[J].J Bacter,2011,193(19):5539-5540.
[36]Jung M Y,Park S J,Min D,et al.Enrichment and characterization of an autotrophic ammonia-oxidizing archaeon of mesophilic crenarchaeal Group I.1a from an agricultural soil [J].Appl Environ Microbiol,2011,77(24):8635-8647.
[37]Blainey P C,Mosier A C,Potanina A,et al.Genome of a low-salinity ammonia-oxidizing archaeon determined by single-cell and metagenomic analysis [J].PloS ONE,2011,6(2):1-12.
[38]Mosier A C,Allen E E,Kim M,et al.Genome sequence of“Candidatus Nitrosoarchaeum limnia”BG20,a lowsalinity ammonia-oxidizing archaeon from the san francisco bay estuary [J].J Bacter,2012,194(8):2119-2120.
[39]Mosier A C,Allen E E,Kim M,et al.Genome sequence of“Candidatus Nitrosopumilus salaria”BD31,an ammonia-oxidizing archaeon from the San Francisco Bay Estuary[J].J Bacter,2012,194(8):2121-2122.
[40]Lehtovirta-Morley L E,Stoecker K,Vilcinskas A,et al.Cultivation of an obligate acidophilic ammonia oxidizer from a nitrifying acid soil[J].Proc Natl Acad Sci USA,2011,108(38):15892-15897.
[41]Woese C R,Kandler O,Wheelis M L.Towards a natural system of organisms:proposal for the domains Archaea,Bacteria,and Eucarya [J].Proc Natl Acad Sci USA,1990,87(12):4576-4579.
[42]Brochier-Armanet C,Boussau B,Gribaldo S,et al.Mesophilic crenarchaeota:proposal for a third archaeal phylum,the Thaumarchaeota [J].Nature Reviews Microl,2008,6(3):245-252.
[43]Pester M,Schleper C,Wagner M.The Thaumarchaeota:an emerging view of their phylogeny and ecophysiology[J].Current opinion in Microbiology,2011,14(3):300-306.
[44]Schleper C.Ammonia oxidation:different niches for bacteria and archaea?[J]ISME J,2010,4(9):1092-1094.
[45]Zhang L M,Offre P R,He J Z,et al.Autotrophic ammonia oxidation by soil thaumarchaea[J].Proc Natl Acad Sci USA,2010,107(40):17240-17245.
[46]Pratscher J,Dumont M G,Conrad R.Ammonia oxidation coupled to CO2fixation by archaea and bacteria in an agricultural soil[J].Proc Natl Acad Sci USA,2011,108(10):4170-4175.
[47]Santoro A E,Buchwald C,McIlvin M R,et al.Isotopic signature of N2O produced by marine ammonia-oxidizing archaea[J].Science,2011,333(6047):1282-1285.
[48]Miranda K M,Ridnour L,Esprey M,et al.Comparison of the chemical biology of NO and HNO:an inorganic perspective[M].Hoboken:John Wiley & Sons,Inc,2005.
[49]Löscher C R,Kock A,K nneke M,et al.Production of oceanic nitrous oxide by ammonia-oxidizing archaea[J].Biogeosciences,2010,9(7):2419-2429.
[50]Dundee L,Hopkins D.Different sensitivities to oxygen of nitrous oxide production by Nitrosomonas europaea and Nitrosolobus multiformis [J].Soil Biol Biochem,2001,33(11):1563-1565.
[51]Shaw L J,Nicol G W,Smith Z,et al.Nitrosospira spp.can produce nitrous oxide via a nitrifier denitrification pathway[J].Environ Microbiol,2006,8(2):214-222.
[52]Hollibaugh J T,Gifford S,Sharma S,et al.Metatranscriptomic analysis of ammonia-oxidizing organisms in an estuarine bacterioplankton assemblage[J].ISME J,2011,5(5):866-878.
[53]Bartossek R,Nicol G W,Lanzen A,et al.Homologues of nitrite reductases in ammonia-oxidizing archaea:diversity and genomic context [J].Environ Microbiol,2010,12(4):1075-1088.
[54]Lund M B,Smith J M,Francis C A.Diversity,abundance and expression of nitrite reductase (nirK)-like genes in marine thaumarchaea[J].ISME J,2012,6(10):1966-1977.
