长春地区部分医院革兰阴性杆菌氨基糖苷类修饰酶基因的检测
2013-08-11周佳琦卢佳睿孙延波
周佳琦,卢佳睿,徐 花,韩 放,孙延波
(吉林大学白求恩医学院病原生物学系,吉林 长春 130021)
*吉林大学白求恩医学院临床医学七年制2008级
自抗菌素问世以来,各种感染性疾病所造成的多种急重症的治疗已不再是难题,然而,抗生素的不合理使用所引起的细菌耐药性问题日益突出。细菌对氨基糖苷类抗菌素的耐药是由于细菌产生氨基糖苷类修饰酶(aminoglycoside modifying enzyme,AME)以及氨基糖苷类药物的作用靶位16SrRNA的基因突变[1]。2006年初,美国学者 Robicsek等[2]发现了不仅对氨基糖苷类药物有修饰作用,而且对喹诺酮类药物也有修饰作用的新氨基糖苷类修饰酶aac(6′)-Ⅰb-Cr型。国内虽有对 AME基因的研究[3-4],但未见有关长春地区的文献报道。肠杆菌科细菌和某些非发酵革兰阴性杆菌是造成医院感染重要的致病菌[5-6],因此,为了解本地区革兰阴性杆菌中AME基因存在状况,本文作者对从临床分离的69株革兰阴性杆菌进行了药敏试验,并检测了6种 AME 基因,即aac(3)-Ⅰ、aac(3)-Ⅱ、aac(6′)-Ⅰ b、aac(6′)-Ⅱ、ant(3")-Ⅰ 和ant(2")-Ⅰ。
1 材料与方法
1.1 菌株和药品 69株革兰阴性杆菌来自长春地区部分医院检验科,包括大肠埃希菌(Escherichia coli)31株,鲍曼不动杆菌(Acinetobacter baumannii)15株,肺炎克雷伯菌(Klebsiella pneumoniae)11株,阴沟肠杆菌(Enterobacter cloacae)12株。上述菌株经法国生物梅里埃公司VITEK-32全自动微生物分析系统鉴定。阿米卡星由中国药品生物制品检定所提供,批号:130335-200204。庆大霉素由北京鼎国昌盛生物技术有限公司提供,批号:1B310330。
1.2 药敏试验 采用琼脂稀释法测定69株革兰阴性杆菌对阿米卡星和庆大霉素的敏感性,在药物浓度超过16mg·L-1时,细菌仍生长判定为耐药,介于2~8mg·L-1为中介,低于2mg·L-1为敏感。
1.3 细菌处理 取菌液1mL置于1.5mL离心管,10000r·min-1离心1min,弃上清加入100μL TE Buffer(1mol·L-1Tris-HCl pH 8.0,500mmol·L-1EDTA pH 8.0),加入等体积酚/氯仿/异戊醇(25∶24∶1),涡旋震荡 30s,10000r·min-1离心5min,上清液即为基因检测的模板,-20℃冰箱保存备用。
1.4 AME基因的引物设计及检测 根据GenBank发布的各型AME基因序列自行设计引物,6种靶基因引物序列和产物长度见表1。聚合酶链反应(PCR)扩增体系:P1、P2引物各1μL(0.4μmol·L-1),Premix Taq 13μL(Taq DNA poL 1.25U·25μL-1,dNTPs 0.4mmol·L-1,PCR buffer),ddH2O 9μL,模板1μL,反应体系25μL。PCR产物长度>500bp的PCR反应条件:93℃ 初始变性2min;93℃ 变性1min,55℃退火1min,72℃延伸1min;35个循环,72℃最后延伸5min。PCR产物长度<500bp的PCR反应条件:93℃初始变性5min;93℃变性30s,55℃退火30s,72℃延伸1min;35个循环,72℃最后延伸5min。PCR产物经2% 琼脂糖凝胶电泳后,用凝胶成像仪观察结果。
1.5 PCR产物测序 PCR产物送至北京六合华大基因科技股份有限公司测序,利用Chromas读序软件将测得序列与GenBank发布的序列进行比对。
2 结 果
2.1 药敏试验结果 69株革兰阴性杆菌中有16株大肠埃希菌、1株肺炎克雷伯菌、3株阴沟肠杆菌和11株鲍曼不动杆菌对阿米卡星呈现耐药,即最低抑菌浓度超过16mg·L-1(表2)。庆大霉素的耐药试验显示:26株大肠埃希菌、11株肺炎克雷伯菌、8株阴沟肠杆菌和15株鲍曼不动杆菌呈现耐药(表3)。
2.2 AME基因检测结果 在69株革兰阴性杆菌中检 出 aac(3)-Ⅰ、aac(3)-Ⅱ、aac(6′)-Ⅰb、ant(3")-Ⅰ和ant(2")-Ⅰ基因,其阳性株数分别为3(4.3%)、56(81.2%)、30(43.5%)、23(33.3%)和9(13.0%),而aac(6′)-Ⅱ基因为阴性(表4)。经分析发现:1株鲍曼不动杆菌同时携带4种AME基因,15株细菌(5株鲍曼不动杆菌、3株阴沟肠杆菌、5株大肠埃希菌和2株肺炎克雷伯菌)同时携带3种基因,25株(5株鲍曼不动杆菌、3株阴沟肠杆菌、11株大肠埃希菌和6株肺炎克雷伯菌)同时携带2种基因(表5)。22株细菌仅1种基因阳性,其余6株未检出任何1种基因。5种AME基因的PCR扩增产物电泳结果见图1。
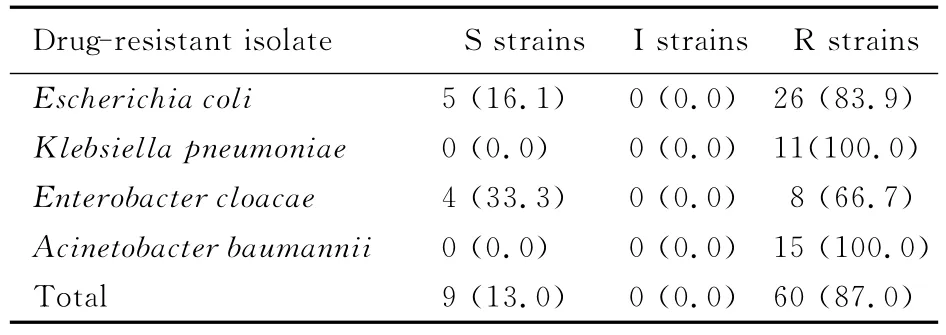
表3 革兰阴性杆菌对庆大霉素的药敏率Tab.3 The susceptibilities of gram-negative bacilli to gentamicin [n(η/%)]
2.3 AME基因序列分析 5种AME基因测序结果见图2~6。测序结果与GenBank数据库比对,序列完全一致。

表4 AME基因检测结果Tab.4 The detection results of AME gene [n(η/%)]
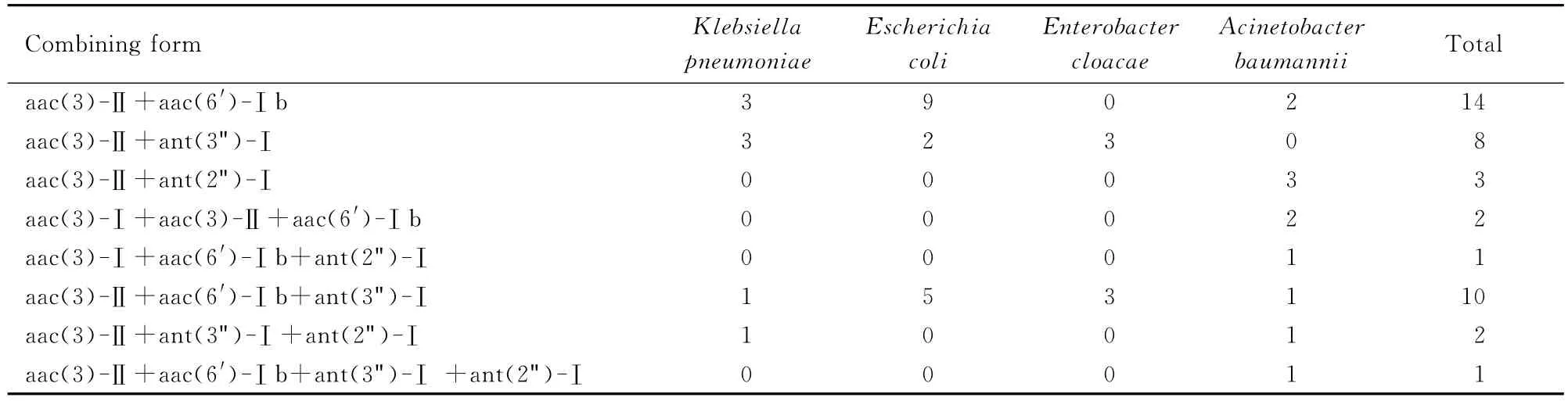
表5 AME基因组合形式Tab.5 The combination form of AME gene
3 讨 论
氨基糖苷类抗生素(aminoglycosides)是由氨基糖与氨基环醇连接而成的氨基糖苷类抗生素,可来自链霉菌的链霉素和小单孢菌的庆大霉素等天然氨基糖苷类。氨基糖苷类抗生素通过与细菌核糖体30S亚基的16SrRNA的A位解码区核苷酸形成氢键,干扰细菌蛋白质的合成,从而发挥抗菌作用。

图2 aac(3)-Ⅰ基因测序图Fig.2 Sequencing diagram of aac(3)-Ⅰ gene
图3 aac(3)-Ⅱ基因测序图Fig.3 Sequencing diagram of aac(3)-Ⅱ gene
自1944年首次发现链霉素以来,因此类药物抗菌谱广、疗效卓越,在医学临床和畜牧兽医业得到广泛应用。但由于逐年泛用和过度使用,各种细菌对氨基糖苷类抗菌素耐药问题也日益严重,细菌对该类抗生素耐药可通过酶修饰、细胞膜渗透性改变、外排泵活性、phoP-phoQ系统和16SrRNA甲基化酶等机制[5-7]来实现。在所有机制中,质粒或染色体介导的AME是最普遍的。AME由质粒或染色体编码,通常由可移动DNA片段携带,质粒的交换和转座子作用均有利于耐药基因渗入到敏感菌株[8]。这些修饰酶包括乙酰转移酶(AAC)、磷酸转移酶(APH)和核苷转移酶(ANT)3类[9-11]。目前已发现的编码AME的基因已有30余种,在革兰阴性杆菌中常见的有10种,除本文作者检测的6种外,还有aac(3)-Ⅲ、aac(3)-Ⅳ、aac(6′)-Ⅰ和aph(3′)-Ⅵ[12]。这些耐药基因经常借助可移动性元件如质粒、转座子和整合子等在同种或不同种细菌间传播[13-14]。
图4 aac(6′)-Ⅰ b基因测序图Fig.4 Sequencing diagram of aac(6′)-Ⅰ b gene
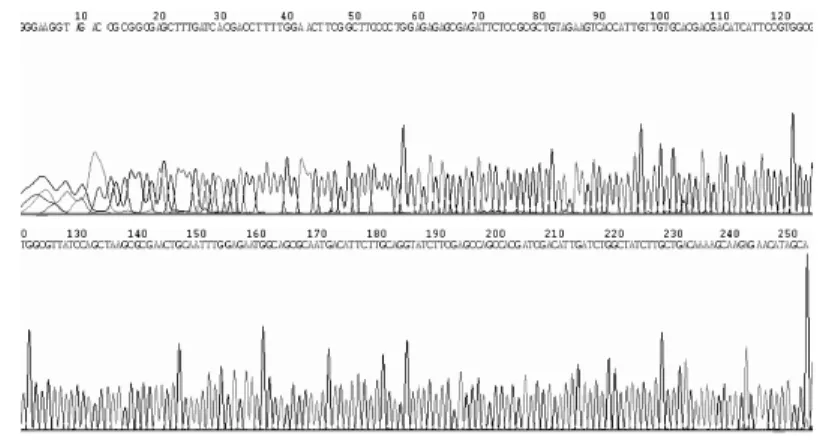
图5 ant(3")-Ⅰ基因测序图Fig.5 Sequencing diagram of ant(3")-Ⅰ gene
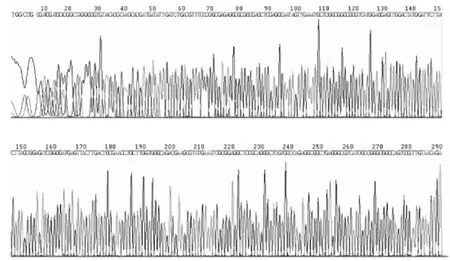
图6 ant(2")-Ⅰ基因测序图Fig.6 Sequencing diagram of ant(2")-Ⅰ gene
本研究分离的革兰阴性杆菌对阿米卡星与庆大霉素的耐药率分别为44.9%和87.0%,除肺炎克雷伯菌与阴沟杆菌对阿米卡星耐药率稍低(9.1%、25.0%),余者对这2种抗生素的耐药率均高于50%,说明革兰阴性杆菌对氨基糖苷类抗生素耐药率已经很高。在69株细菌中有63株检测出AME基因,占91.3%,说明革兰阴性杆菌携带氨基糖苷类耐药基因较为严重,与耐药表型相符。本研究中AME基因阳性率较高并居前3位的分别为aac(3)-Ⅱ(81.2%)、aac(6′)-Ⅰ b(43.5%)和ant(3")-Ⅰ(33.3%);有41 株 (59.4%)的菌株携带2种及以上的修饰酶基因,多种耐药基因的组合在细菌对氨基糖苷类抗生素耐药机制中发挥了重要作用。携带2种及以上的修饰酶基因中也是aac(3)-Ⅱ比例最高,这种修饰酶基因的存在显然与菌株对氨基糖苷类抗生素的高水平耐药密切相关。9株敏感菌株中2株检测出ant(2")-Ⅰ,1株检测出aac(3)-Ⅱ,原因可能是与AME基因表达强弱或者无表达有关,耐药基因盒的表达不仅依赖于启动子的强弱,而且与启动子的距离远近有关;上游基因表达强,下游基因表达弱甚至可能不表达[15],有待进一步研究。同时,携带aac基因盒和碳青霉烯酶的整合子将会使细菌耐药问题越来越严重[16-17],本研究中检出率最高的是aac(3)-Ⅱ基因,值得高度关注。抗生素泛用和滥用是导致相关耐药基因传播的重要原因,因此临床上要注意合理使用抗生素。
[1]糜祖煌,陆亚华 .氨基糖苷类修饰酶及其基因检测 [J].现代实用医学,2004,1(6):13-16.
[2]Robicsek A,Strahilevitz J,Jacobk GA,et al.Fluoroquinolone modifying enzyme:a new adaptation of a common aminoglycoside acetyltransferase [J].Nature Med,2006,12(1):83-88.
[3]糜祖煌,秦 玲.泛耐药铜绿假单胞菌16SrRNA甲基化酶、氨基糖苷类修饰酶基因研究[J].中华医院感染学杂志,2008,18(12):1656-1658.
[4]陈 杰,王卫华,汪 丽,等﹒大肠埃希菌中同时发现携带aac(6′)-Ⅰb及aac(6′)-Ⅰb-Cr氨基糖苷类修饰酶基因[J].中华医院感染学杂志,2011,21(6):1064-1067.
[5]吕增春,何连德,霍云燕.医院外感染抗生素合理应用临床分析[J].药物流行病学杂志,1997,6(2):91-93.
[6]李蓓涵,张学英,李菁华,等.长春地区部分医院革兰阴性杆菌超广谱β-内酰胺酶的基因类型及分布 [J].吉林大学学报:医学版,2010,36(1):205-209.
[7]Islam S,Oh H,Jalal S,et al.Chromosomal mechanisms of aminoglycoside resistance in Pseudomonas aeruginosa isolates from cystic fibrosis patients[J].Clin Microbiol Infect,2009,15(1):60-66.
[8]Macfarlan E,Kwasnicka A,Hancock R. Role of Pseudomonas aeruginosa PhoP-PhoQ in resistance to antimicrobial cationic peptides and aminoglycosides [J].Microbiology,2000,146(10):2543-2554.
[9]Doi Y,Ghilardi AC,Adams J,et al.High prevalence of metallo-β-lactamase and 16SrRNA methylase coproduction among imipenem-resistant Pseudomonas aeruginosa isolates in Brazil[J].Antimicrob Agents Chemother,2007,51(9):3388-3390.
[10]Wachino J,Arakawa Y.Exogenously acquired 16SrRNA methyltransferases found in aminoglycoside-resistant pathogenic gram-negative bacteria:an update[J].Drug Resist Update,2012,15(3):133-148.
[11]Shaw KJ,Rather PN,Hare RS,et al.Molecular genetics of aminoglycoside resistance genes and familial relationship of the aminoglycoside modifying enzymes[J].Microbiol Rev,1993,57(1):138-163.
[12]Smith CA,Baker EN.Aminoglycoside antibiotic resistance by enzymatic deactivation[J].Curr Drug Targets-Infect Disord,2002,2(2):143-160.
[13]Yamane K,Wachino J,Doi Y,et al.Global spread of multiple aminoglycoside resistance genes [J].Emerg Infect Dis,2005,11(6):951-953.
[14]李智山,周乐翔,赵建忠.大肠埃希菌新的氨基糖苷类修饰酶基因研究[J].中华医院感染学杂志,2007,17(8):914-916.
[15]黄支密,糜祖煌,秦 玲.20株阴沟肠杆菌耐药性及氨基糖苷类修饰酶基因分析[J].中华微生物学和免疫学杂志,2005,25(5):428-431.
[16]Park YJ. Aminoglycoside resistance in gram-negative bacilli[J].Korean J Clin Microbiol,2009,12(2):57-61.
[17]Poirel L,Le Thomas I,Naas T,et al.Biochemical sequence analyses of GES-1,a novel class A extended-spectrumβlactamase,and the class 1integron In52from Klebsiella pneumoniae[J].Antimicro Agents Chemother,2000,44(3):622-632.
[18]Strateva T,Yordanov D. Pseudomonas aeruginosa - a phenomenon of bacterial resistance [J].J Med Microbiol,2009,58(9):1133-1148.
[19]Ramirez MS,Tolmasky ME. Aminoglycoside modifying enzymes[J].Drug Resist,2010,13(3):151-171.
