不同热处理对沙门氏菌DNA消亡的影响
2013-08-07李苗云赵改名柳艳霞高晓平黄现青张秋会
刘 杰,李苗云*,赵改名,柳艳霞,高晓平,田 玮,黄现青,张秋会
(河南农业大学食品科学技术学院,河南 郑州 450002)
沙门氏菌广泛分布于自然界,是肠杆菌科中一种常见、重要的人畜共患病原菌之一,对人类和动物具有极大危害,沙门氏菌污染能使人类发生伤寒、副伤寒、败血症、肠胃炎和食物中毒等病症[1-3]。其中肉、蛋、乳等营养丰富的食品是沙门氏菌的主要传播介质,直接影响着人们的饮食健康安全[4-5]。近年来,核酸分析被广泛应用于病原菌的检测,具有检测范围广、敏感度高、特异性好等优点,大大提高了检测的效率,缩短了检测流程,但其存在假阳性偏高等问题[5-9]。鲁玉侠等[10]研究了121℃、15min 处理后沙门氏菌的PCR扩增效果,结果显示PCR产物电泳条带很明显。Malorny[11]、Nocker[12]、Oliveira[13]等研究指出死菌DNA会长期存在。另外,很多研究者[14-18]指出死菌DNA的存在使基因水平的检测方法高估了样品中活菌的水平。而我国肉类食品有低温、高温、涮、煎、烤等多种加工和消费方式,常用的热处理方式对沙门氏菌致死情况和DNA的破坏程度不是很清楚。本实验模拟肉品在加工和消费过程中常用的几种加热处理过程,以研究不同热处理对沙门氏菌致死效果以及对沙门氏菌DNA消亡的影响,为肉品消费时的安全性和沙门氏菌检测技术的研究提供依据。
1 材料与方法
1.1 菌株与试剂
沙门氏菌标准菌株(ASI.1859F3) 双汇集团技术中心提供。
U N I Q-1 0 柱式细菌基因组抽提试剂盒 生工生物工程(上海)有限公司;2×PCR Solution Premix PrimeSTAR®HS 宝生物工程大连有限公司;平板计数琼脂培养基 北京路桥技术有限责任公司。
1.2 仪器与设备
HIRAYAMA高压灭菌锅 日本Hirayama公司;UVP凝胶成像系统 美国UVP公司;梯度PCR仪 德国Biometra公司;HH-501恒温水浴锅 金坛市杰瑞尔电器有限公司;DYY-12型电泳仪 北京六一仪器厂。
1.3 方法
1.3.1 菌株培养
取沙门氏菌标准菌株ASI.1859F3接种到5mL液体LB培养基中。37℃、180r/min振荡培养14~16h,使菌液达到对数生长期。平板计数得到初始菌浓。
1.3.2 不同热处理对沙门氏菌存活状况的影响
模拟肉品在加工和消费过程中常用的热处理过程,取1mL过夜培养的标准菌液加入到1.5mL PE管中,分别按照65℃保温20min、85℃保温30min、沸水浴2min、沸水浴3min、121℃保温15min进行加热处理。处理后的菌液进行平板计数,每种处理3个平行。
1.3.3 基因组DNA的提取
取菌液1mL,10000r/min离心1min,弃上清收集菌体,UNIQ-10柱式细菌基因组抽提试剂盒抽提基因组DNA,制备的DNA样品溶解于50μL TE缓冲液中,保存备用。
1.3.4 热处理后沙门氏菌不同放置时间对基因组DNA消亡的影响
对不同热处理后完全失活的组别进行室温放置,每隔1d取菌液1mL提取基因组DNA,1.5%的琼脂糖凝胶电泳120V,25min,EB染色后紫外分析仪进行分析。
1.3.5 热致死后沙门氏菌残存DNA的PCR扩增
致死后的菌液经过一段时间放置后按1.3.3节方法提取基因组DNA,进行PCR扩增。引物根据已发表的鼠伤寒沙门氏菌特异性inva基因核苷酸序列(GenBank AE008832)设计,上游引物(5’-GTG AAA TAA TCG CCA CGT CGG GCA A-3’);下游引物(5’-TCA TCG CAC CGT CAA AGG AAC C-3’),扩增片段长度为285bp[19]。引物由宝生物工程大连有限公司合成。采用20μL反应体系:2×PCR Solution Premix PrimeSTAR®HS 10μL、1μmol/L Forward Primer 2μL、1μmol/L Reverse Primer 2μL、DNA模板 2μL、dH2O(灭菌蒸馏水)4μL。反应参数:95℃、30s;95℃、5s;64℃、34s进行40个循环,最后72℃延伸2min。取3μL PCR产物进行电泳,并进行紫外光谱分析。
2 结果与分析
2.1 不同加热处理后对沙门氏菌存活状况的影响

表 1 不同加热处理对沙门氏菌的致死情况Table 1 The death status of Salmonella under different heat treatments
从表1可知,所采取的5种热处理方式均能有效杀死菌液中的沙门氏菌,其中65℃ 20min、85℃ 30min、沸水浴3min、121℃ 15min处理能完全杀死菌液中沙门氏菌,沸水浴2min处理后虽不能完全杀死菌液中的沙门氏菌,但其菌落形态已发生明显变化,处理前菌落边缘光滑,呈乳白色圆形菌落,处理后则菌落较小,颜色暗淡,边缘呈不规则齿状或放射状的怪异形态,继续培养发现菌落生长缓慢。肉品在食用过程中经常会在沸水中短时间处理,如火锅中常见的“涮”,通常时间较短,对于沙门氏菌沸水中处理2min尚不能将其杀死,需要至少3min以上。因此,在肉品消费过程中,需要提高杀菌时间以提高其安全性。
2.2 致死后沙门氏菌不同放置时间对基因组DNA消亡的影响
经过平板计数后发现,65℃ 20min、85℃ 30min、沸水浴3min、121℃ 15min处理后的沙门氏菌完全失活。对以上4种方式处理的菌液进行室温放置,每隔1d提取细菌基因组DNA,电泳后进行分析。
2.2.1 65℃、20min处理后电泳结果
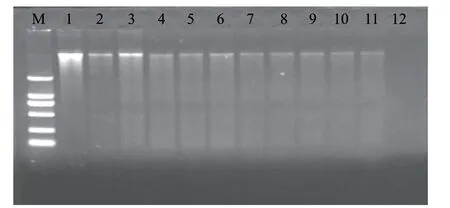
图 1 65℃、20min处理后沙门氏菌DNA电泳图谱Fig.1 Electrophoresis results of the DNA from Salmonella subjected to heat treatment at 65 ℃ for 20 min
由图1可知,与原菌液相比,65℃、20min处理后的菌液DNA的条带亮度明显变弱,但仍能看出清晰的条带。说明65℃、20min处理会破坏部分基因组DNA,影响DNA的提取效果,但不能完全使其降解。随着放置时间的延长,DNA条带慢慢变弱,说明致死后的细菌DNA会逐渐降解,但到18d为止,变化并不是很明显。
2.2.2 85℃、30min处理后电泳结果
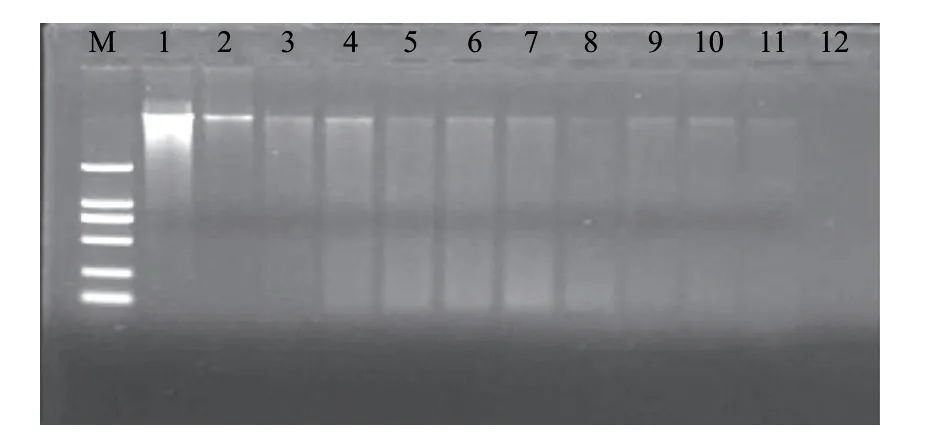
图 2 85℃、30min处理后沙门氏菌DNA电泳图谱Fig.2 Electrophoresis results of the DNA from Salmonella subjected to heat treatment at 85 ℃ for 30 min
由图2可知,与原菌液相比,85℃、30min处理后的菌液DNA的条带亮度明显变弱。说明85℃、30min处理会破坏部分基因组DNA,随着放置时间的延长,DNA条带逐渐模糊,与65℃、20min处理相比变化更为明显,说明85℃、30min致死后的细菌DNA降解速度更快,到18d为止,条带已经很模糊,但仍有完整的基因组DNA存在。
2.2.3 沸水浴3min处理后电泳结果
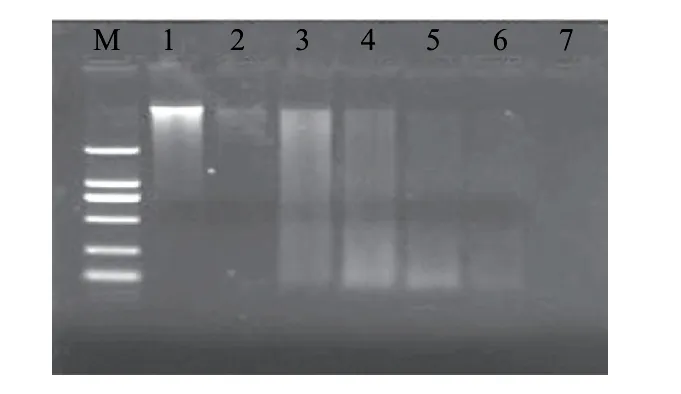
图 3 沸水浴3min处理后沙门氏菌DNA电泳图谱Fig.3 Electrophoresis results of the DNA from Salmonella subjected to boiling for 3 min
由图3可知,沸水浴3min处理后的菌液在放置1、2、4d后可以看到很微弱DNA的条带,放置6d后条带消失。这说明沸水浴3min处理对基因组DNA的破坏程度很大,且对DNA提取效果的影响很大。随着放置时间的延长,DNA降解速度很快,到第6天时基因组DNA已经大部分降解,无可见条带。
2.2.4 121℃、15min处理后电泳结果
由图4可知,菌液经过121℃、15min处理后,基因组DNA条带完全消失。说明121℃、15min处理会严重破坏基因组DNA,处理后已经没有完整的基因组DNA存在。
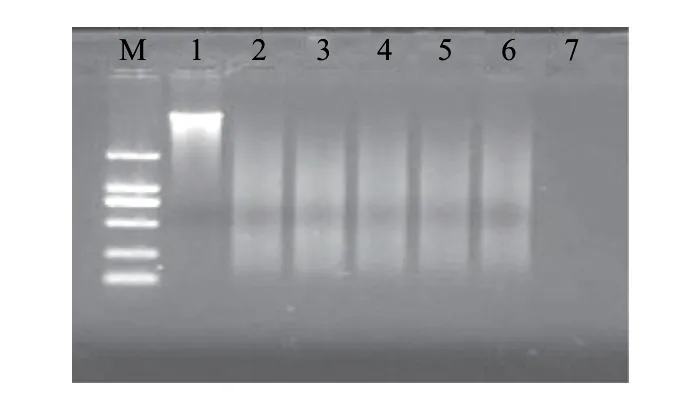
图 4 121℃、15min处理后沙门氏菌DNA电泳图谱Fig.4 Electrophoresis results of the DNA from Salmonella subjected to sterilization at 121 ℃ for 15 min
2.2.5 长时间放置后死菌残存DNA的PCR扩增结果
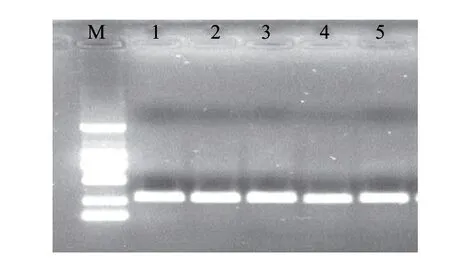
图 5 长时间放置后死菌残存DNA的PCR扩增结果Fig.5 PCR amplifi cation of DNA in dead bacilli after long storage period
由图5可知,沙门氏菌在经过65℃ 20min、85℃ 30min、沸水浴3min、121℃ 15min处理致死并放置相当长一段时间后,其DNA扩增条带依然很明显。沸水浴3min和121℃、15min处理后DNA的扩增条带比65℃、20min和85℃、30min处理后的有些暗淡。这说明虽然DNA电泳条带已经很模糊或消失,但死亡的细胞中仍存在有大量的目的基因片段存在,且这些基因片段消亡速度很慢,会在很长一段时间内对PCR扩增产生极大的作用。高温处理对DNA的破坏则更为明显。
由图1~5可知,经过不同温度处理后,完整的基因组DNA会发生不同程度的降解,在经过沸水浴3min和121℃、15min处理后的细胞中已经很少有完整的大分子质量的DNA存在,而是降解为了各种不同大小的基因碎片。沙门氏菌受到较低温度处理时,其基因组DNA的完整性受到的破坏程度相对较小,但DNA会随着时间的延长也逐渐降解为分子质量较小的片段。这些片段就包括引起PCR特异性扩增的目的基因片段。这些分子质量相对较小的片段会长期存在于死细胞中并且很不容易消亡,从而对基于DNA技术进行的病原菌检测等产生干扰作用。沙门氏菌DNA消亡速度与温度高低和处理的时间长短有关,加热温度越高、时间越长,沙门氏菌DNA消亡速度越快。
沙门氏菌是鲜肉及其制品中常见的,也是我国对肉品主要检测和控制的食源性病原菌之一。肉类食品有多种加工和消费方式,如“涮”、“煎”等,通常时间较短,本实验研究表明,对于沙门氏菌沸水中处理2min尚不能将其杀死,所以,需要提高杀菌时间以提高其安全性。而目前,PCR等分子技术在病原菌检测方面的应用越来越多,但从本实验结果来看,各种加工肉制品在经过不同温度杀菌以后,虽然肉品中的细菌被灭活,但加热处理并不能使细菌基因组DNA完全降解消亡,大量的基因片段仍然残存在死亡的细胞当中,而且消亡速度很慢,尤其在较低温度处理的肉品中更为明显,这与死菌DNA会长期存在的研究结果一致[11-13]。121℃、15min处理后沙门氏菌的PCR研究结果与鲁玉侠等[10]一致。本实验结果表明,加热温度越高、时间越长,沙门氏菌DNA消亡速度越快,这对于经过不同加工方式处理后的产品,如果应用PCR等技术分析检测沙门氏菌,会对检测结果产生很大的干扰作用,直接影响检测结果的准确性。
3 结 论
肉品在沸水中加热时至少在3min以上才能使沙门氏菌完全灭活;加热温度越高、时间越长,沙门氏菌DNA消亡速度越快;不同温度加热后,菌液中仍存在大量的目的基因片段,对应用DNA分析检测沙门氏菌会产生很大的干扰作用,影响检测结果的准确性。
[1] TIRADO C, SCHMIDT K. WHO surveillance programme for control of foodborne infections and intoxications, results and trends across greater Europe[J]. Journal of Infection, 2001, 43: 80-84.
[2] WALLACE D J, GILDER T V, SHALLOW S, at al. Incidence of foodborne illnesses reported by the foodborne diseases active surveillance network (foodnet)-1997. foodnetworking group[J]. Journal of Food Protection, 2000, 63: 807-809.
[3] MALORNY B, HOORFAR J, BUNGE C, et al. Multicenter validation of the analytical accuracy of Salmonella PCR: towards an international standard[J]. Applied and Environmental Microbiology, 2003, 69: 290-296.
[4] ZHANG H M, SHI L, GUO S Y, et al. Identification and characterization of class 1 integron resistance gene cassettes among Salmonella strains isolated fromhealthy humans in China[J]. Microbiology and Immunology, 2004, 48(9): 639-645.
[5] MALORNY B, BUNGE C, HELMUTH R. A real-time PCR for the detection of Salmonella enteritidis in poultry meat and consumption eggs[J]. Journal of Microbiological Methods, 2007, 70(2): 245-251.
[6] MINAM H, SRINIVASAN V, GILLESPIE B E, et al. Application of SYBR green real-time PCR assay for specific detection of Salmonella spp. in dairy farm environmental samples[J]. International Journal of Food Microbiology, 2005, 102: 161-171.
[7] KNUT R, BIRGITTEM, SIGNEM D, et al. Use of ethidiummonoazide and PCR in combination for quantification of viable and dead cells in complex samples[J]. Applied and Environmental Microbiology, 2005, 71(2): 1018-1024.
[8] PUSTERLA N, BYRNE B A, HODZIC E, et al. Use of quantitative real-time PCR for the detection of Salmonella spp. in fecal samples from horses at a veterinary teaching hospital[J]. The Veterinary Journal, 2010, 186: 252-255.
[9] PERELLE S, DILASSER F, MALORNY B, et al. Comparison of PCR-ELISA and lightcycler real-time PCR assays for detecting Salmonella spp. in milk and meat samples[J]. Molecular and Cellular Probes, 2004, 18: 409-420.
[10] 鲁玉侠, 郭祀远, 石磊. EMA-LAMP方法快速检测死/活的食源性沙门氏菌[J]. 食品科学, 2009, 30(22): 324-327.
[11] MALORNY B, TASSIOS P T, COOK N, et al. Standardization of diagnostic PCR for the detection of foodborne pathogens[J]. Food Microbiol, 2003, 83(1): 39-48.
[12] NOCKER A, CHEUNG C Y, CAMPER A K. Comparison of propidium monoazide with ethidium monoazide for differentiation of live vs. dead bacteria by selective removal of DNA from dead cells[J]. Journal of Microbiological Methods, 2006, 67: 310-320.
[13] OLIVEIRA S D, SANTOS L R, SCHUCH D M T, et al. Detection and identification of Salmonella from poultry-related samples by PCR[J]. Veterinary Microbiology, 2002, 87: 25-35.
[14] SEO K H, VALENTIN-BON I E, BRACKETT R E. Detection and enumeration of Salmonella enteritidis in homemade ice creamassociated with outbreak: comparison of conventional and real-time PCR methods[J]. Journal of Food Protection, 2006, 69(3): 639-643.
[15] WOLFFS P, NORLING B, RADSTRFM P. Risk assessment of falsepositive quantitative real-time PCR results in food, due to detection of DNA originating from dead cells[J]. Journal of Microbiological Methods, 2005, 60: 315-323.
[16] NOGVA H, DROMTORP S, NISSEN H, et al. Ethidium monoazide for DNA-based differentiation of viable and dead bacteria by 5V-nuclease PCR[J]. BioTechniques, 2003, 34(4): 804-813.
[17] WHYTE P, GILL K M, COLLINS J, et al. The prevalence and PCR detection of Salmonella contamination in raw poultry[J]. Veterinary Microbiology, 2002, 89: 53-60.
[18] LUO Jianfei, LIN Weitie, GUO Yong. Method to detect only viable cells in microbial ecology[J]. Appl Microbiol Biotechnol, 2010, 86: 377-384.
[19] MALORNY B, HOORFAR J, BUNGE C, et al. Multicenter validation of the analytical accuracy of Salmonella PCR: towards an international standard[J]. Applied and Environmental Microbiology, 2003, 69: 290-296.
