基于组合核的蛋白质交互关系抽取
2013-04-23李丽双黄德根
李丽双,刘 洋,黄德根
(大连理工大学 计算机学院,辽宁 大连 116023)
1 引言
随着信息数字化和生物医学的快速发展,生物医学文献正以指数级的速度增长,要从这些浩如烟海的文献中找出所需的信息越来越难。作为生物医学信息抽取领域的一个重要部分,蛋白质交互关系抽取具有很高的应用价值,尤其是对蛋白质知识网络的建立、蛋白质关系的预测、新药的研制等均具有重要意义。
目前用于PPI抽取的方法可以分为三种,基于词共现的方法,基于模式匹配的方法和基于统计机器学习的方法。基于词共现的方法将在同一个句中出现的词进行统计,根据统计学原理判断是否存在蛋白质交互关系。由于基于同现的方法简单而且灵活,这种方法被广泛应用于关系抽取,并且能获得很高的召回率。文献[1]把同现统计应用到句子水平的关系抽取,抽取性能得到了提高。但是这种方法由于缺乏对新关键词的统计,所以很难发现新的PPI。
基于模式匹配的方法是利用事先定义好的模式和规则进行关系抽取。文献[2]提出利用动态规划算法自动从文献中获得匹配模式的方法进行蛋白质交互关系抽取,取得了较好效果。文献[3]提出了一个规则学习方法,并且证明了这种规则获得方法比手工方法好。但基于模式匹配的方法并不能涵盖全部的模式,也不能产生新的模式。
基于机器学习的方法弥补了上述两种方法的缺陷,将关系抽取转化为分类问题。机器学习方法又可分为基于特征向量和基于核函数的方法,如文献[4]在词汇、 基本短语块等特征中融入依存树特征,采用SVM进行蛋白质交互关系抽取,在AIMED语料上获得的F值为54.7%,取得了较好的效果。但基于特征的方法无法用于复杂的结构,研究者采用核函数代替特征向量内积运算计算两个对象的相似度,并具有很好复合性。文献[5]应用卷积树核实现了英文命名实体关系抽取,并提出最短路径闭包树,经过实验验证其用于关系抽取效果最好,并尝试将平面核与树核结合,在ACE2004上得到了P/R/F为76.1%/68.4%/72.1%的最好结果。文献[6]提出用图核的方法完成蛋白质交互关系的自动抽取,把潜在的关系定义成一个图表示法,为了有效地从图中学习,定义了一个基于图的核函数,在AIMED上得到了P/R/F为52.9%/61.8%/56.4%的结果。文献[7]提出了一种组合核的方法进行关系抽取,把基于几个句法分析器的核组合起来,抽取了更多有用的信息,在AIMED上取得了P/R/F为58.7%/66.1%/61.9%的最好结果。文献[8]使用了一种集成核的方法从生物文本中抽取蛋白质交互关系,把词等特征与自定义的路径核相结合,在IEPA上达到了P/R/F为73.03%/82.09%/74.38%的结果。总之核函数的方法在关系抽取领域取得了很好的效果。
本文首先用完全树(Complete Tree,CT)、最小完全树(Minimum Complete Tree,MCT)、最小树(Minimum Tree,MT)和最短路径闭包树(Shortest Path Enclosed Tree,SPT)等简单的句法解析树在AIMED上进行PPI抽取实验,经验证使用SPT效果最好,F值为53.89%;但在提取SPT树的过程中,发现一些对PPI抽取有用的结构化信息可能被删减,所以当SPT树所含信息过少时对其进行动态拓展。本文提出一种动态拓展策略建立动态拓展树(Dynamic Extended Tree,DET),利用动态拓展树在AIMED进行10倍交叉实验,其精确率、召回率、F值分别达到了79.41%、43.99%、56.62%。最后将平面核与树核结合,在AIMED上进行10倍交叉实验,获得了较好的效果,其中将平面核与基于动态拓展树的核函数组合后精确率、召回率、F值分别达到了82.40%、51.30%、63.23%。结果要优于其他先进的方法。
2 SVM
设原始输入空间X⊆Rn(n为输入空间的维数),训练集S={(x1,y1),(x2,y2),...,(x,y)}xi∈X,yi∈{-1,1}是xi的标记,若xi属于正类,yi=1,若xi属于负类,yi=-1,为样本的个数。SVM就是寻找能够将训练数据划分为两类的最优超平面,该超平面可以通过求下面凸二次规划问题的解得到,如式(1)所示:
max
subject to
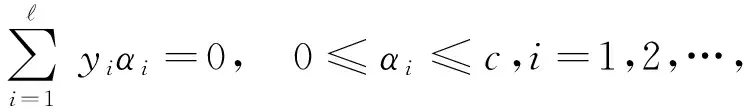
(1)
其中K(xi,xj)=φ(xi)·φ(xj)为Kernel函数,αi为与每个样本对应的Lagrange乘子,c>0是自定义的惩罚系数。给定一个测试实例x,它的类别由下面的决策函数(式2)决定:
sv为支持向量。
3 基于组合核函数的蛋白质交互关系抽取
3.1 特征向量的抽取
3.1.1 词特征
本文使用的词特征包括两个蛋白质名字中的词,两个蛋白质之间的词,蛋白质周围的词,以及表示两个蛋白质交互关系的交互词。下面以句子A: “The effects of theBbetachainoffibrinogenare mediated through cell surfacecalreticulin”为例。在这个句子中,Bbetachainoffibrinogen和calreticulin分别表示两个蛋白质。
(1) 两个蛋白质名字中的词(ProName)
即出现在两个蛋白质名字中的所有单词。在句子A中,Bbetachainoffibrinogen和calreticulin分别表示两个蛋白质的名字,那么名字中的每一个词都被当作一个特征。句子A中两个蛋白质名字分别被表示成p1_B, p1_beta,p1_chain,p1_of,p1_fibrinogen,p2_calreticulin。
(2) 两个蛋白质之间的词(Betwords)
指位于两个蛋白质之间的所有词。如果两个蛋白质之间没有别的单词,那么该特征被设置为NULL。句子A中,两个蛋白质之间的词为: are mediated through cell surface,那么对应的特征值就为: b_are,b_mediated,b_ through,b_cell,b_surface。
(3) 两个蛋白质周围的词(Surwords)
指位于第一个蛋白质左侧的n个词,和第二个蛋白质右侧的n个词。在本文实验中,n的取值为5。如果第一个蛋白质左侧没有单词,或者第二个蛋白质右侧没有单词,则把该向量设置为NULL。在句子A中,第一个蛋白质Bbetachainoffibrinogen左侧共有四个单词 the effect of the, 第二个蛋白质calreticulin右侧为0 个单词。在特征向量中分别被表示为: l_the,l_effect,l_of, l_the, r_null。
(4) 交互词特征(Keyword)
所谓交互词指的是能表示两个蛋白质之间交换关系的词(如: regulate, interact,modulate 等)。一个句子中是否含有交互词对判断两个蛋白质之间是否有交互关系具有重要的作用。它是两个蛋白质具有交互关系的必要条件。如果在两个蛋白质之间或者周围有一个交互词,则把这个词作为交互词特征。如果在一个句子中有两个或两个以上的交互词,则选择离两个蛋白质最近的那个词作为交互词;如果在一个句子中没有表示两个蛋白质交互关系的词,则把该特征设置为NULL。在句子A中,我们选择mediate 作为交互词。
3.1.2 链接特征
链接语法分析器(Link grammar parser)可以用来分析句子的链接语法。根据解析结果可以判断第一个蛋白质到第二个蛋白质之间是否存在一个解析路径。一般来说,两个蛋白质之间具有交互关系,那么很大程度上,它们之间存在一条链接路径。如果在两个蛋白质之间有一条链接路径,其特征值被设置为: LINK_Y,否则的话,特征值被设置为: LINK_N。
3.1.3 两个蛋白质之间的距离特征
在一个句子中,两个蛋白质离得越近,那么它们之间具有交互关系的可能性也就越大。因此蛋白质之间的距离可以作为判断两个蛋白质是否具有交互关系的一个因素。本文使用两种距离特征:
(1) TwoProDis: 这个特征是指两个蛋白质之间其他单词的个数。如果两个蛋白质之间单词的个数小于等于3,则把该特征值设为1,如果两个蛋白质之间的单词的个数大于3小于等于6,则把该特征值设为2。如果两个蛋白质之间的单词个数大于6小于等于9,则把该特征值设为3,否则的话,把该特征值设为4。
在句子A中,两个蛋白质之间具有5个单词,所以蛋白质之间的TwoProDis值为 2。
(2) ProNumDis: 两个蛋白质之间是否具有交互关系,除了受两个蛋白质之间单词的个数影响外,还与两个蛋白质之间是否有别的蛋白质有关。因此这种情形也被考虑进去,并称之为ProNumDis 特征。如果两个蛋白质之间没有其他的蛋白质,那么该特征的特征值被设为0,如果两个蛋白质之间有其他蛋白质,那么该特征的特征值为两个蛋白质之间其他蛋白质的个数。在句子A中,Bbetachainoffibrinogen和calreticulin之间没有其他的蛋白质,所以该特征值为0。
3.2 卷积树核
树核是将句子通过句法解析生成句法树后,计算任意两棵树之间的相似度。由Collins等人[9]提出的卷积树核是一个典型的树核。其通过计算两棵树之间的公共子树个数来计算相似度,如式(3)所示:
式(3)中Nj是句法解析树Tj的节点集,Δ(n1,n2)递归计算以n1和n2为根的公共子树的数量。计算公式(4):
1) 如果以n1和n2为根的上下文无关语法规则不同,Δ(n1,n2)=0;否则转到2)。
2) 如果n1和n2都是POS标签,那么Δ(n1,n2)=1*λ;否则转到3)。
3) 递归计算Δ(n1,n2):
其中#ch(n)表示节点n的孩子数量,ch(n,k)表示节点n的第k个孩子,λ(0<λ<1)为衰减因子,作用是在不同大小的子树间取得平衡。
3.3 卷积树核中句法树的剪裁及动态拓展
3.3.1卷积树核中句法树的剪裁
本文利用Stanford Parser[10]对句子进行解析,在解析之前用PROTAIN_1 和PROTAIN_2对蛋白质对进行替换,句子中其他的蛋白质则用PROTAIN替换。例如,句子B: “HvSPY coexpression largely abolishedGA3induced activity of analpha-amylasepromoter .” 其中GA3和alpha-amylase是蛋白质对。在解析这个句子之前,用PROTAIN_1 和PROTAIN_2取代两个蛋白质GA3和alpha-amylase。于是,原句子变为句子C: “HvSPY coexpression largely abolished PROTAIN_1 induced activity of an PROTEIN_2 promoter .”然后利用Stanford Parser 对句子C进行解析,得到其语法解析树,如图1所示。
在本文中将这种对一句话解析得到的整棵树叫做Complete Tree(CT),CT树所含信息很完整,但包含的冗余信息也很多,直接用其进行PPI抽取会引入过多噪声,会对抽取效果产生负面影响。所以我们按Zhang等[5]策略得到Minimum Complete Tree(MCT)和Shortest Path Enclosed Tree(SPT)。分别如图2和图3所示。

图1“HvSPY coexpression largely abolished GA3 induced activity of an alpha-amylase promoter .”经Stanford Parser解析出的CT树。

图2 MCT树

图3 SPT树
MCT是在CT基础上保留最近公共父节点的子树,SPT是在MCT的基础上删除连接两个蛋白质路径闭包之外的所有节点。若只保留路径上的节点,便得到了Minimum Tree(MT)[11],如图4所示。

图4 MT树
3.3.2 卷积树核中句法树的动态拓展
经验证,SPT树在以上几种基本句法树中效果最好,但一些含有蛋白质对的句子结构较简单,含有的结构化信息较少,如句子“PROTEIN_1 PROTEIN_2”和“PROTEIN_1 and PROTEIN_2”(其SPT结构如图5、图6所示),它们的SPT树中包含的信息不足以判断其中蛋白质对是否有关系,而表示两个蛋白质关系的交互词在很大程度上可能出现在以其最近公共父节点为根的树上,所以有必要在SPT树上进行拓展。考虑到这种信息量不足的SPT树节点数一般不多于7个,所以我们提出如下三种拓展策略:
(1) 当SPT中包含的节点个数少于7时,用MCT代替SPT;
(2) 当SPT中包含的节点个数少于7时,用SPT根节点的父节点的MCT代替SPT;
(3) 当SPT中包含的节点个数少于7时,若SPT与MCT不相同,用MCT代替SPT; 否则用SPT根节点的父节点的MCT代替SPT。
图5 “PROTEIN_1 PROTEIN_2”的SPT

图6 “PROTEIN_1 and PROTEIN_2”的SPT
3.4 组合核
基于特征向量的核充分利用了实例的平面特征却忽略了实例的结构化信息,而树核仅利用了结构化信息而忽略了平面特征。两种核各有优缺点而又互补。所以,为了最大限度获取PPI抽取的有用信息,我们将多项式核与卷积树核结合。本文使用Moschitti等人开发SVMlightTK工具包[12]。组合核函数定义如式(5)所示:
其中kt为上文已介绍过的卷积树核,kb为多项式核函数,定义如式(6)所示:
其中s,c,d为参数。
4 实验及结果
本文中所述实验均在AIMED语料上完成。AIMED语料来自PubMed摘要,其中蛋白质关系是根据DIP数据库识别的。在摘要中专家标注了人类的基因和蛋白质关系。本文从该语料中抽出4 500对蛋白质关系实例,其中有1 000对正例和3 500对负例。为提高实验的可靠性,本文中所涉及的实验均使用10倍交叉验证。SVM参数值全部选取工具包中的默认参数。
4.1 基于特征向量的PPI抽取
按上文3.1所述抽取特征,进行启发式实验,最终得到的性能指标如表1所示。

表1 多项式核的PPI抽取
由表1可知单独使用多项式核精确率达到了的82.83%,召回率达到了41.87%,F值为55.62%。
4.2 基于卷积树核的PPI抽取
4.2.1 四种基本句法树的有效性验证实验
按3.3中所述策略修剪出相应的句法树,使用相同的训练集和测试集, 用SVMlightTK工具包中的卷积树核进行训练和测试。表2 给出了不同句法树有效性验证的结果。

表2 不同句法树有效性验证
从表2中可以看出,经过修剪后,MCT树、SPT树、MT树的性能指标均要比CT树好。这是由于MCT树、SPT树和MT树比CT树中含有的冗余信息少,引入的噪声减少,结构化信息的比重提高,所以精确率、召回率和F值都有大幅提高。
三种经过修剪的句法树中,SPT树用于PPI抽取的效果最好。因为SPT树中包含了较完整的结构化信息及相对较少的冗余信息。其F值比CT树提高34.49%,比MCT树提高7.69%。MT树是在SPT树的基础上进行进一步修改而得到的,虽然其包含的冗余信息最少,但同时其包含的有用的结构化信息也被大幅删减,导致其召回率很低,只有12.22%,所以其抽取效果不如SPT树,但仍然比CT树的抽取效果好。
4.2.2 三种SPT拓展树的有效性验证实验
虽然SPT树在以上几种基本句法树中效果最好,但其仍有不足,需对其进行拓展,表3给出了采用三种拓展策略(见3.3.2)与SPT用于PPI抽取的结果比较。
从表3可以看出: 方法(1),方法(2)和方法(3)的效果都要比不拓展要好,方法(1)的精确率比不拓展提高了0.90%,召回率比不拓展提高了2.10%,F值比不拓展提高了2.01%。方法(2)的精确率比不拓展提高了0.65%,召回率和不拓展是相同的,F值比不拓展提高了0.16%。方法(3)的精确率比不拓展提高1.08%,召回率比不拓展提高2.91%,F值比不拓展提高2.73%。原因分析如下:
① 经方法(1),方法(2)和方法(3)拓展后,大部分如图5,图6所示的句法结构中增加了有用的结构化信息,使得召回率有较大提高,虽然方法(2)的召回率没有变化,但综合考虑这三种方法均比不拓展要好。
② 方法(2)考虑到一些句子的SPT树和MCT树相同,按方法(1)进行拓展时这类句子相当于没有扩充结构化信息,所以用上一层的MCT树进行拓展,但是SPT树和MCT树相同的句子在语料中所占的比例较低,虽然对这种特例进行了拓展,但对其他满足条件的句子进行拓展后,引入了比方法(1)多的噪声,所以结果没有方法(1)和方法(3)好。
③ 在这三种拓展策略中,方法(3)的效果最好,分析原因,是其结合了方法(1)和方法(2)的优点,能够对需要拓展的句法树进行拓展,同时尽量少的引入噪声。方法(3)的F值比不拓展提高2.73%,提高较为显著,说明拓展策略对PPI抽取有益。
方法(3)实际使用的是一种动态策略,不妨称其为Dynamic Extended Tree(DET)。
4.3 基于组合核的PPI抽取
用3.4介绍的方法构造组合核,表4给出了用组合核进行PPI抽取的结果。

表4 基于组合核的PPI抽取
由表4可知, 将多项式核与卷积树核组合后的抽取效果好于单个核函数。组合核的精确率比多项式核低0.43%、召回率提高9.43%、F值提高7.61%;组合核的精确率比卷积树核提高2.99%,召回率提高7.31%,F值提高6.61%。 这是因为特征核和树核从不同的方面计算两条实例的相似度,覆盖了更多的有用信息,即平面特征和句法树中的结构化特征具有互补性。
通过对基于组合核进行关系抽取的相关文献[5,13-15]分析发现,组合核较单个核函数的性能都有所提高。例如文献[5]将特征核与树核组合,在ACE语料上进行实体关系抽取,并验证了SPT树的抽取效果最好,该文采用线性核与树核组合后,在语料ACE2003上的F值为70.10%, 比单独使用特征核高15.70%,比单独使用树核高6.50%。文献[14]在SPT树的基础上提出一种动态的上下文敏感的拓展策略,并提出上下文敏感树核,实验证明这种树核优于基于SPT树的树核,最后将上下文敏感树核与线性核组合,在语料ACE2004上得到的F值为75.80%,比单独使用特征核高5.70%,比单独使用上下文敏感树核高2.60%。 文献[13]同样在Aimed语料上进行蛋白质交互关系抽取,将特征核与树核组合后,F值比单独使用特征核提高5.36%,比单独使用树核提高5.81%。
通过以上实验及分析可知,对基于组合核的关系抽取,其性能要高于单个核函数,并且其提高的程度可能不仅取决于单个核函数的性能,可能也与所用语料有关。
4.4 与其他先进方法比较
为了能更好地评价本文的方法,我们选择同样在AIMED上使用核函数方法进行PPI抽取的文献进行比较,结果如表5所示。
文献[6]中使用图核进行PPI抽取,未使用词特征等平面信息,本文F值比文献[6]提高6.83%。文献[7]中将深层句法解析器和浅层句法解析器结合使用,并将BOW(Bag Of Words)核、树核和图核组合,得到的P/R/F分别为58.70%/66.10%/61.90%。由于充分使用了结构化信息,所以得到了不错的效果,但其没有使用平面特征,所以F值比我们的方法低1.33%。文献[13]将特征核、树核、图核、语法路径核等多种核函数组合,得到了F值为64.41%的最好结果,本文只比较其特征核与树核的组合结果,其特征核与树核结合,F值为58.05%,本文较之提高5.18%。经以上比较,可以看出本文提出的PPI抽取方法是有优势的。

表5 与其他先进方法比较
5 结论
为提高PPI抽取系统的性能,本文首先对基本句法解析树进行裁剪并在卷积树核上进行测试,实验结果表明用SPT抽取PPI的效果最优,F值为53.89%。然后对性能最好的SPT采用三种策略进行拓展,条件是当SPT树的节点数少于7时,分别用MCT、以SPT根节点的父结点为根的MCT和DET代替SPT。DET取得了最好的效果,F值达到了56.62%。最后将基于特征的平面核与树核结合,在AIMED上进行了10倍交叉验证实验,得到了P/R/F为82.40%/51.30%/63.23%的最好结果。效果优于其他先进的PPI抽取系统。
考虑到现行的修剪策略虽然使得句法树包含较精练的信息,但还存在噪声,本文考虑下一步对句法树进行更精确修剪与扩充;并考虑从基因本体库中提取相关的语义信息来提高PPI抽取系统的性能。
[1] Bunescu R, Mooney R, Ramani A., et al. Integrating Co-occurrence Statistics with Information Extraction for Robust Retrieval of Protein Interactions from Medline[C]//Proceedings of BioNLP-2006, 2006: 49-56.
[2] Huang M, Zhu X, Hao Y, et al. Discovering patterns to extract protein-protein interactions from full biomedical texts [J]. Bioinformatics, 2004, 20(18):3604-3612
[3] Bunescu R C, Mooney R J. A shortest path dependency kernel for relation extraction[C]//Proceedings of the Human Language Technology Conference and Conference on Empirical Methods in Natural Language Processing, Vancouver, 2005:724-731.
[4] 刘兵,钱龙华,徐华,等. 依存信息在蛋白质关系抽取中的作用[J]. 中文信息学报. 2011, 25(2):21-26.
[5] Zhang M, Zhang J, Su J, et al.. A Composite Kernel to Extract Relation between Entities with both Flat and Structured Feature[C], in Proc.COLINGACL, Sydney,Australia,2006: 825-832.
[6] Airola A, Pyysalo S, Bjöne J, et al. A Graph for Protein-Protein Interaction Extraction[C]//Proceedings of BioNLP.Columbus,USA 2008.
[7] Miwa M, Satre R, Miyao Y, et al. Protein-protein interation extration by leveraging multiple kernels and parser[J]. Int.J.Med. Inform.2009,doi:10.1016/j.ijmedif.
[8] Lishuang Li, Jinyu Ping, Degen Huang. Protein-protein interacion extracion from biomedical literatures based on a combined kernel [J]. Journal of Information and Computational Science, 2010, 7(5): 1065-1073.
[9] Collins M, Duffy N. Covolution kernels for natural language[C].NIPS’2001,2001: 625-632.Cambridge,MA.
[10] De Marneffe M C,MacCartney B, Manning C D. Generating Typed Dependency Parses from Phrase Structure Parses[C]//In Proeeedings of the IEEE/ACL2006 Workshop on Spoken Language Technology.The Stanford Natural Language Proeessing Group.
[11] 孔芳. 指代消解关键问题研究[D]. 苏州:苏州大学,2009.
[12] http://disi.unitn.it/moschitti/Tree-Kernel.htm
[13] Yang Z H, Tang N, Zhang X, et al. Multiple kernel learning in protein-protein interaction extraction from biomedical literature[J]. Artif Intell Med (2011), doi:10.1016/j.artmed.2010.12.
[14] Zhou G D, Zhang M, Ji D H et al. (2007b). Tree kernel-based relation extraction with context-sensitive structured parse tree information[C]//Proceedings of the 2007 Joint Conference on Empirical Methods in Natural Language Processing and Computational Natural Language Learning (EMNLP/CoNLL-2007): 728-736.
[15] Qian L H, Zhou G D, Zhu Q M, et al. Exploiting constituent dependencies for tree kernel-based semantic relation extraction[C]//Proceedings of International Conference on Computational Linguistics (COLING’2008): 697-704.
