应用EMA-PCR检测沙门氏菌活菌的研究
2012-11-21赵素君李江凌王秋实叶勇刚罗丹丹叶健强廖党金
赵素君 曹 冶 谢 晶 李江凌 王秋实 叶勇刚 罗丹丹 叶健强 廖党金
(四川省畜牧科学研究院,成都 610066)
沙门氏菌(Salmonella)是肠杆菌科中一种重要的人畜共患病原菌,分布广泛,血清型繁多。常因污染饲料、水源、土壤、畜舍设施等造成畜禽感染。感染畜禽后可引起鸡白痢、鸡伤寒、副伤寒、仔猪副伤寒、流产等多种动物疾病。患病畜禽体内含有大量的沙门氏菌,在其排泄物中也经常带菌,因而成为新的传染源,如不及时检测发现和控制处理,则可导致畜禽间及养殖场间的水平传播,甚至垂直传播给子代畜禽。为有效地预防和控制疾病发生,建立快速、敏感而又特异的检测沙门氏菌的方法是非常必要的。长期以来,人们对沙门氏菌的检测大多采用传统的方法,即选择性或非选择性增菌、可疑细菌的分离、生化反应及血清学鉴定等常规方法,检测结果虽然在一定程度上准确、可靠,但检测过程烦琐、费时费力,而且肠杆菌科细菌间的生化反应多有交叉,因此,传统的检测方法在快速、敏感与特异性等方面存在一定的局限性[1,2]。
近年来,PCR技术以灵敏度高、特异性强、简便和快速等优点作为分子水平的快速检测技术越来越多的被应用于沙门氏菌的检测中[3-5]。沙门氏菌的侵袭蛋白决定细菌进入宿主上皮细胞的能力,与沙门氏菌的致病性密切相关。其中,invA基因编码的侵袭蛋白A是吸附和侵袭上皮细胞的表面抗原,是主要毒力因子,在沙门氏菌的致病过程中起着重要作用[6-8]。invA基因序列在沙门氏菌属具有很高的同源性,非常保守。是当前沙门氏菌检测的主要靶点[9]。
由于样品中常常存在死亡沙门氏菌,死菌虽然已经没有危害性,但在PCR检测中也能扩增出目的基因,从而使检测结果出现大量的假阳性,干扰了检测的真实性。所以区分死、活细菌方法的建立对沙门氏菌检测极其重要。本研究利用叠氮溴乙锭(Ethidium monoazide bromide,EMA)能穿过死细菌的细胞膜,在光激活的作用下与基因组DNA共价结合,从而能抑制样品中死菌体的DNA进行PCR扩增的特性,将EMA与PCR技术结合,通过特异性扩增编码侵袭蛋白A的invA基因,建立一种快速、有效的检测沙门氏菌活菌的方法,具有很好的实际应用价值。
1 材料与方法
1.1 材料
1.1.1 菌株 阳性菌株:猪霍乱沙门氏菌CVCC504、鼠伤寒沙门氏菌CVCC541、鸡白痢沙门氏菌CVCC525;对照菌株:金黄色葡萄球菌CVCC3055、CVCC3056;肠毒性大肠杆菌CVCC196、CVCC197、CVCC200、CVCC211、CVCC215,肠出血性大肠杆菌CVCC248,肠侵袭性大肠杆菌CVCC2072,肠致病性大肠杆菌CVCC251,均购自中国兽医药品检查所中国兽医微生物菌种保存管理中心。
1.1.2 主要试剂 EMA为Biotium公司产品;Taq DNA聚合酶、dNTPs、DL2000 DNA Marker购自Takara公司;其他化学试剂均为国产分析纯;引物合成及核酸测序由Invitrogen公司完成。
1.1.3 引物设计与合成 根据GenBank中沙门氏菌invA基因序列设计特异性引物,引物序列为F:5′-GATTCTGGTACTAATGGT GATGATC-3′;R:5′-GCCAGGCTATCGCCAATAAC-3′,扩增目的片段长度为285bp。
1.2 方法
1.2.1 沙门氏菌活菌和死菌的制备 接种沙门氏菌CVCC504,培养16 h后,取1ml菌液于EP管中,6 000 rpm,4℃,离心5 min,收集菌体沉淀,加入无菌生理盐水重悬沉淀,制成活菌悬液。再将活菌悬液72℃水浴15 min,得到死菌悬液。
1.2.2 EMA处理 取适量菌悬液于EP管中,加入终浓度为100μg/ml 的EMA,在黑暗处放置5min后,于650W卤素光源光照处理1min,光源放在离样品管20cm处,光照时EP管放在冰上,以免样品温度过高。EMA处理后的样品,用于下面的DNA模版的制备。
1.2.3 模版DNA的制备 取1 ml菌液,12 000 rpm离心5 min,去除上清液,然后用100 ul灭菌去离子水重悬。然后,100℃水浴5 min后,再10 000 rpm离心5 min,上清液用作PCR检测。
1.2.4 PCR反应体系及反应条件优化 PCR反应体系:10×Ex Taq Buffer(含MgCl2)2.5μl,dNTP mixture(各2.5mM)2μl,引物(20μM)各0.5μl,模板DNA 1μl,TaKaRa Ex Taq(5 U/μl)0.2μl,灭菌双蒸水加至25μl。
PCR反应条件优化主要通过退火温度来实现,为了获得特异性的扩增片段,采用Touchdown PCR方法确定PCR反应合适的退火温度,设定退火温度范围65℃~45℃。扩增结果经1%琼脂糖凝胶电泳检测。
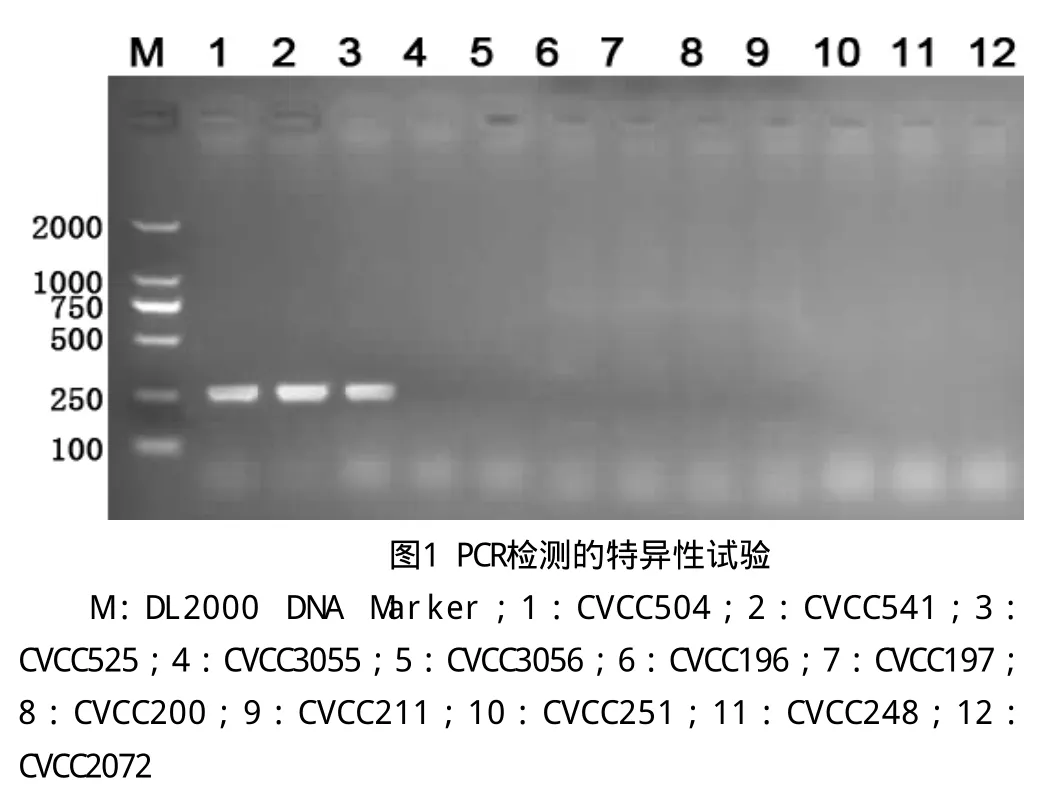
1.2.5 PCR特异性检测 以CVCC504、CVCC541、CVCC525作为阳性菌株;以CVCC3055、CVCC3056、CVCC196、CVCC197、CVCC200、CVCC211、CVCC248、CVCC251、CVCC2072作为对照菌株,进行PCR扩增,结果经琼脂糖凝胶电泳,用凝胶成像系统进行观察,出现特异性扩增条带,即为检测阳性,否则为阴性。
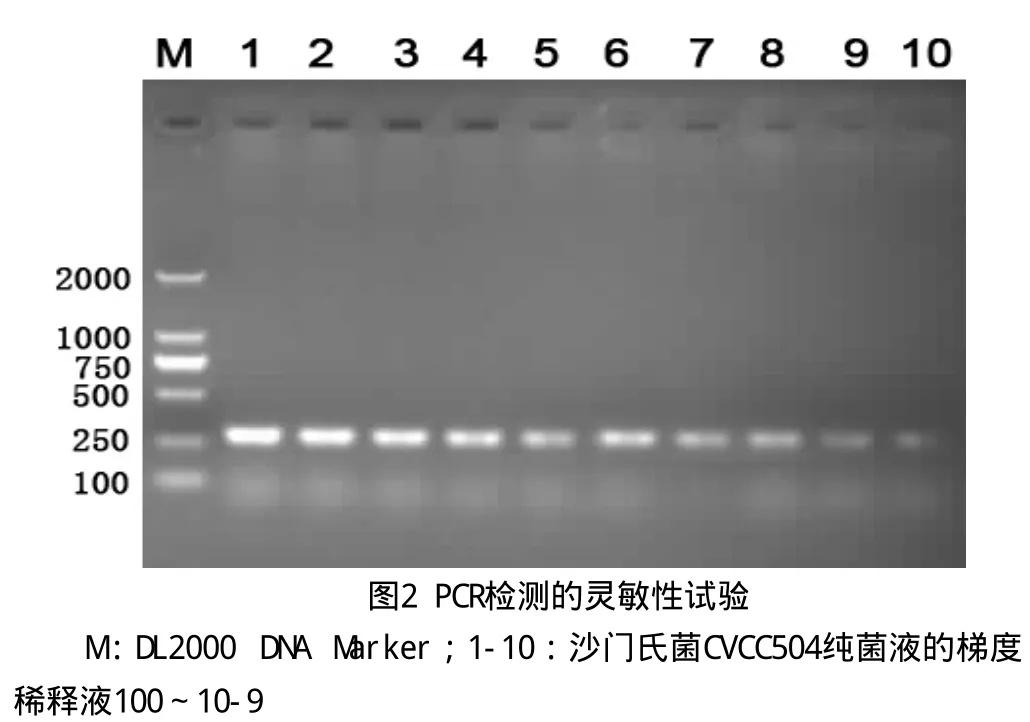
1.2.6 PCR灵敏度检测 接种沙门氏菌CVCC504,培养16 h,取1ml菌液于EP管中,递倍稀释10-1~10-9,进行如上模板DNA制备,PCR检测其灵敏度。同时,采用平板法进行细菌计数,通过牛肉膏蛋白胨琼脂平板进行对照计数。分别取递倍稀释100~10-9的菌液50μl进行涂板,37℃过夜培养,然后分别进行计数,计算检测灵敏度。
1.2.7 不同活细胞比例的混合菌悬液的EMA-PCR扩增 分别配制含有100%、75%、50%、30%、20%、10%、5%、4%、3%、2%、1%、0%的沙门氏菌CVCC504活细胞菌悬液混合体系,分别加入终浓度为100μg/ml 的EMA,在黑暗处放置5min后,样品置于冰上用650W卤素光源距离20cm处光照处理1min,经EMA处理后的菌液,用于如上DNA模版的制备,进行PCR扩增。
2 结果
2.1 PCR反应条件的优化结果
经过Touchdown PCR方法确定最佳的PCR反应条件为:94°C 4min;94°C 30sec、55°C 30sec、72°C 30sec,28个循环;72°C延伸5min。
2.2 沙门氏菌invA基因的PCR特异性检测结果
PCR扩增产物经琼脂糖凝胶电泳分析,结果显示(见图1),只有沙门氏菌CVCC504、CVCC541、CVCC525扩增出285bp的目的条带,DNA测序结果经BLAST分析,与GenBank上序列完全一致。而对照菌株CVCC3055、CVCC3056、CVCC196、CVCC197、CVCC200、CVCC211、CVCC248、CVCC251、CVCC2072均未检测到特异性条带,结果为阴性。
2.3 沙门氏菌invA基因的PCR检测灵敏度分析
将各梯度稀释液100~10-9分别进行invA基因的PCR扩增,结果经琼脂糖凝胶电泳分析,如图2所示其灵敏度检测低限为10-9,而相应的平板计数为11CFU/ml。
2.4 沙门氏菌EMA-PCR区分死菌与活菌的检测结果
EMA-PCR扩增结果经琼脂糖凝胶电泳分析,结果显示除了全部是死菌的组外,其他含活菌各种比例的混合菌液中,均出现特异性扩增条带,见图3。

3 讨论
叠氮溴乙锭(Ethidium monoazide bromide,EMA)是一种荧光插入型的核酸结合染料,能穿过死细菌的细胞膜,在光激活的作用下与基因组DNA共价结合,从而能抑制样品中死菌体的DNA进行PCR扩增;而完整未受损伤的活菌细胞膜能阻止EMA渗透,对活菌DNA不起作用[10,11]。PCR检测方法的特异性与灵敏度主要取决于相应检测靶点的选择,invA作为沙门氏菌的持家基因,具有很好的属内保守性与属间特异性,本研究选取invA基因作为靶序列设计特异性引物,将EMA与PCR技术结合,成功地建立了一种快速、有效的检测沙门氏菌活菌的方法。由于在检测过程中除去了死细胞DNA对PCR的干扰,从而大大提高了检测结果的准确性和真实性。由于沙门氏菌血清型较多,2001年就已经报道有2523种血清型[12]。本研究旨在提出一种鉴定沙门氏菌活菌的PCR检测方法,鉴定结果往往需要配合血清型分析鉴定器种类。该方法操作简单,大大缩短了传统常规方法的检测周期,对沙门氏菌的检测具有较好的应用价值。
[1]Pangloli P,Dje Y,Oliver SP,et al.Evaluation of methods for recovery of Salmonella from dairy cattle,poultry,and swine farms[J]. J Food Prot,2003,66(11):1987-1995.
[2]Nassib TA,El-Din MZ,El-Sharoud WM.Assessment of the presence of Salmonella spp. in Egyptian dairy products using various detection media[J]. Lett Appl Microbiol.2003,37(5):405-409.
[3]Upadhyay BP,Utrarachkij F,Thongshoob J,et al.Detection of Salmonella invA gene in shrimp enrichment culture by polymerase chain reaction[J]. Southeast Asian J Trop Med Public Health.2003,41(2):426-435.
[4]Mainar-Jaime RC,Atashparvar N,Chirino-Trejo M.Estimation of the diagnostic accuracy of the invA-gene-based PCR technique and a bacteriological culture for the detection of Salmonella spp. in caecal content from slaughtered pigs using Bayesian analysis[J].Zoonoses Public Health.2008,55(2):112-118.
[5]Gonzalez-Escalona N,Hammack TS,Russell M,et al.Detection of live Salmonella sp. cells in produce by a TaqMan-based quantitative reverse transcriptase real-time PCR targeting invA mRNA[J]. Appl Environ Microbiol.2009,75(11):3714-3720.
[6]Galan JE,Curtiss R.Distribution of the invA,-B,-C,and -D genes of Salmonella typhimurium among other Salmonella serovars:invA mutants of Salmonella typhi are deficient for entry into mammalian cells. Infect Immun.3rd. 1991.59(9):2901-2908.
[7]Penheiter KL,Mathur N,Giles D,et al. Non-invasive Salmonella typhimurium mutants are avirulent because of an inability to enter and destroy M cells of ileal Peyer’s patches[J]. Mol Microbiol.1997,24(4):697-709.
[8]Porter SB,Curtiss R.Effect of inv mutations on Salmonella virulence and colonization in 1-day-old White Leghorn chicks[j]. Avian Dis ,3rd. 1997. 41(1):45-57.
[9]D’Souza DH,Critzer FJ,Golden DA.Real-time reversetranscriptase polymerase chain reaction for the rapid detection of Salmonella using invA primers[J]. Foodborne Pathog Dis,2009. 6(9):1097-1106.
[10]Lee JL and Levin RE:Discrimination of viable and dead Vibrio vulnificus after refrigerated and frozen storage using EMA,sodium deoxycholate and real-time PCR[J]. J Microbiol Methods,2009,(79):184-188.
[11]Lee JL and Levin RE:A comparative study of the ability of EMA and PMA to distinguish viable from heat killed mixed bacterial flora from fish fillets[J]. J Microbiol Methods,2009,(76):93-96.
[12]Popoff MY,Bockemuhl J,Brenner FW,et al.Supplement 2000(no. 44)to the Kauffmann-White scheme[J]. Res Microbiol 2001,152(10):907-909
