新疆柯尔克孜族传统发酵饮料博扎中微生物群落结构的PCR-DGGE分析
2012-04-01努尔古丽热合曼华长春朱晓莹古丽苏木托克逊董明盛
努尔古丽·热合曼,华长春,朱晓莹,古丽苏木·托克逊,董明盛,*
(1.新疆师范大学生命科学学院,新疆 乌鲁木齐 830054;2.南京农业大学食品科技学院,江苏 南京 210095)
新疆柯尔克孜族传统发酵饮料博扎中微生物群落结构的PCR-DGGE分析
努尔古丽·热合曼1,华长春2,朱晓莹1,古丽苏木·托克逊1,董明盛2,*
(1.新疆师范大学生命科学学院,新疆 乌鲁木齐 830054;2.南京农业大学食品科技学院,江苏 南京 210095)
采用PCR-DGGE技术分析新疆柯尔克孜民族古老传统发酵饮料——博扎(Bozaa)中微生物种群结构,对博扎细菌和酵母菌DGGE图谱上主要条带的DNA进行测序和序列分析。结果表明,博扎中细菌组成包括植物乳杆菌(Lactobacterium plantarum)、短乳杆菌(Lactobacillus brevis)、蜡状芽孢杆菌(Bacillus cereus)、巴氏醋酸杆菌(Acetobacer pasteurianus)、啤酒酵母(Saccharomyces cerevisiae),初步成功解析评估了博扎中微生物的多样性。
博扎;变性梯度凝胶电泳;微生物区系
博扎(Bozaa)是一种用小米粉自然发酵而成的发酵饮料,其制作和保藏工艺天然传统,是一种酒精浓度较低,有CO2杀口感,低糖度,质地浓稠的天然发酵饮料。博扎起源可追述到居住于前奥托曼土耳其的古老名族,作为一种健康饮品延续至今,不仅在其原产地仍然盛行,也已经广泛传播至美洲、亚洲等地区,至今只有在我国新疆部分地区的柯尔克孜族家庭保留着制作和饮用博扎的习惯。作为传统自然发酵食品,独特的发酵方式决定了其中微生物区系的多样性,其中微生物菌系十分复杂,蕴藏着丰富的微生物资源,也是优良益生菌的来源。但有关新疆柯尔克孜族传统发酵饮料及其中的益生菌至今处在濒危状态,有关博扎的研究可以说还属于空白,国内外未见关研究报道。因此,本研究可以说是在工业化时代,对古老传统发酵饮料博扎中的微生物组分的挽救工作。
近年来,基于16S rDNA的分子生物学技术结合变性梯度凝胶电泳法(denaturing gradient gel electrophoresis,DGGE)为有效分析复杂微生物群落及其多样性提供了一种先进手段[1]。本研究采用PCR-DGGE指纹技术,解析博扎中微生物种群结构,揭示其中优势菌群组成,以为确定博扎饮料发酵优势微生物组分提供科学线索的同时,为进一步研究博扎的发酵机制、其质量控制以及此类发酵饮料今后的开发提供参考。
1 材料与方法
1.1 材料与试剂
博扎样品均采自新疆维吾尔自治区阿克苏温宿县柯尔克孜族家庭,自然发酵1d后放置冰盒内空运到实验室,并在第2天处理样品。
DNA提取试剂盒、PCR-supermix试剂 天根生化科技(北京)有限公司;DGGE试剂 美国Sigma公司;引物 上海生工生物工程技术服务有限责任公司合成。
1.2 仪器与设备
MyCycler梯度PCR 仪、Bio-Rad Dcode System 美国Bio-Rad公司;CDS8000凝胶成像分析系统 上海培清科技有限公司。
1.3 方法
1.3.1 样品总DNA的提取
用细菌基因组DNA提取试剂盒及酵母菌基因组提取试剂盒从博扎样品中直接提取细菌总DNA和酵母总DNA。经1.0%琼脂糖凝胶电泳检测后,-20℃冰箱保存备用。
1.3.2 PCR-DGGE分析
1.3.2.1 PCR扩增
细菌PCR扩增对象为细菌16S rDNA V6~V8可变区:上游引物为UGC 968- F(5'-CGC CCG GGG CGC GCC CCG GGC GGG GCG GGG GCA GGG GAA CGCGAA GAA CCT TAC-3'),下游引物为L1401R(5'-GCG TGT GTA CAA GAC CC-3')[1-2]。酵母菌PCR扩增对象为酵母菌26S rDNA的D1区;上游引物为NL1GC-f(5'-GCG GGC CGC GCG ACC GCC GGG ACG CGC GAG CCG GCG GCG GGC CAT ATC AAT AAG CGG AGG AAA AG-3'),下游引物为LS2(5′-ATT CCC AAA CAA CTC GAC TC-3')[3-4]。
PCR 反应体系(50μL):模板DNA 2μL,1×PCR缓冲液,0.2mmol/L dNTP,引物均为0.5μmol/L,Taq DNA 聚合酶1.25U,补充ddH2O至终体积50μL[3,5]。
PCR扩增采用梯度PCR仪。PCR条件采用降落PCR,反应程序为:94℃预变性4min,先20个循环(94℃ 变性30s,退火温度65~55℃,每个循环降低0.5℃,退火30s,72℃延伸30s),再于恒定退火温度条件下进行10个循环(94℃变性30s,55℃退火30s,72℃延伸30s),最终72℃延伸10min。PCR 产物经1.0%琼脂糖凝胶电泳检测后,-20℃冰箱保存备用[6]。
1.3.2.2 变性梯度凝胶电泳(DGGE)
8%聚丙烯酰胺凝胶 (丙烯酰胺、甲叉双丙烯酰胺体积比37.5:1)。变性梯度从40%到60%(100%变性剂含有7mol/L 尿素和40%甲酰胺),在1×TAE 缓冲液中,先200V 预电泳5~10min,然后在85V 的固定电压条件下电泳12~14h[7-8]。
1.3.2.3 染色(银染和EB染色)
银染是在电泳完成后将胶取出先置于一个干净的不锈钢或塑料容器中,加入200mL固定液,摇3 min;将固定液倒在容器中(后面操作使用);添加200mL银染溶液,摇10min;弃去银染溶液,用蒸馏水轻洗胶和容器;添加新鲜蒸馏水,摇2min;弃去蒸馏水,加显影溶液,直至出现清晰条带为止,结束显影,弃去显影溶液。加入使用过的固定液,摇5min;弃去固定液,加入蒸馏水,摇2min,弃去蒸馏水;加入保存溶液,摇7min;银染后用凝胶成像系统进行照相。图像用Quantity one分析软件进行分析[8-9]。
EB染色是在将DGGE胶在1×TAE(含0.5mg/L EB)中浸泡15min,弃去浸泡液,再在ddH2O中浸泡20min。然后用凝胶成像系统进行照相[9]。
1.3.2.4 DNA条带的回收
将EB染色的DGGE胶置于紫外灯下,切下不同位置的条带,将凝胶条剪成许多细小片,将碎片分别转移入1.5mL 离心管中;用洗脱缓冲液覆盖凝胶碎片并于37℃温育过夜;室温6000r/min离心10min,以沉淀凝胶碎片;移出上清液,注意不要混有聚丙烯酰胺碎片,另用洗脱缓冲液洗涤聚丙烯酰胺凝胶碎片,回收残存的DNA,必要时再离心,合并两次上清液;DNA用两倍体积100%乙醇沉淀,-20℃冷冻30min,12000r/min离心10min,DNA沉淀以100μL TE缓冲液重新溶解。往DNA溶液中加入10μL 3mol/L pH5.2的乙酸钠、两倍体积100%乙醇,重复沉淀过程,12000r/min离心10min回收DNA;沉淀物用70%乙醇洗涤、干燥、用TE缓冲液重新溶解[10]。
1.3.2.5 测序及相似性分析
以回收DNA为模板,进行PCR 扩增,细菌引物为:上游引物968- F(5'-AAC GCG AAG AAC CTT AC-3'),下游引物L 1401R(5'-GCG TGT GTA CAA GAC CC- 3');酵母菌引物为:NL1(5'-GCC ATA TCA ATA AGC GGA GGA AAA G -3')和LS2(5'-ATT CCC AAA CAA CTC GAC TC-3')。
扩增程序:94℃预变性4min,30个循环(94℃变性30s,55℃退火30s,72℃延伸30s),最终72℃延伸10min。细菌16S rDNA V6~V8可变区和酵母菌的26S rDNA的D1区PCR产物经1.0%琼脂糖凝胶电泳检验后,送上海生工生物工程技术服务有限公司测序。测序结果采用EditSeq程序进行拼接。登陆NCBI(www.ncbi.nlm. nih.gov/blast/)网站,将所得序列与数据库中已知序列进行比较。用Clustal1.83 和Mega 软件进行相似性分析及构建系统发育树,确定分类地位并将序列提交到GenBank核酸序列数据库得到登陆号[11]。
2 结果与分析
2.1 博扎中细菌种群结构的PCR-DGGE图谱分析
以提取的博扎中细菌总DNA为模板,用16S rDNA的V6~V8可变区引物(带GC夹)进行PCR扩增,经1.0%琼脂糖凝胶电泳检测,所有样品都检测到大小约为440bp的特异性扩增片段,结果表明本实验采用的DNA提取方法及PCR扩增条件是合适的(图1);用26S rDNA的D1 区引物(带GC 夹)进行PCR 扩增,获得300bp 的特异性扩增片段(图2)。

图 1 博扎中细菌16S rDNA V6~V8可变区PCR扩增产物电泳图Fig.1 PCR amplification products of 16S rDNA V6-CV8 fragments from Bozaa

图2 博扎中酵母菌26S rDNA D1区PCR 扩增产物电泳图Fig.2 PCR amplification products of yeast 26S rDNA D1 fragments from Bozaa
分别对得到反映博扎中细菌的16S rDNA V6~V8区扩增产物和酵母菌26S rDNA D1区扩增产物进行DGGE电泳检测,分别得到反映博扎中细菌种群结构的DGGE指纹图(图3)和反映博扎中酵母菌种群结构的DGGE指纹图(图4)。
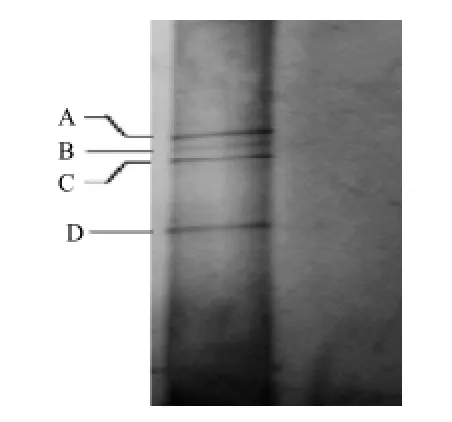
图3 博扎中细菌菌落PCR-DGGE指纹图谱菌相分析Fig.3 DGGE fingerprinting of bacterial communities in Bozaa

图4 博扎中酵母菌落PCR-DGGE指纹图谱菌相分析Fig.4 DGGE fingerprinting of yeast communities in Bozaa
从图3可以看出,细菌图谱中出现4条条带;从图4可以看出,酵母菌图谱中出现1条条带,在空白对照泳道中没有出现条带,说明出现的条带可以特异性代表博扎微生物种属。
2.2 割胶回收条带及其分子比对
将纯化DGGE胶上的DNA条带,并对此片段扩增、测序,经过Blast程序比对,确定条带A的序列与巴氏醋酸杆菌相似性达99%,条带B与植物乳杆菌相似性达99%,条带C与蜡状芽孢杆菌相似性达99%,条带D与短乳杆菌相似性达99%,条带I与啤酒酵母菌相似性达99%。本实验所测得的条带序列都已被GenBank数据库收录,其登录号见表1。

表1 博扎中细菌和酵母菌序列分析Table1 Results of sequence analysis of bacterial and yeast communities in Bozaa
3 讨 论
博扎主要产地为中亚、中东和非洲地区部分国家,作为一种传统发酵饮品,其发酵过程中的微生物构成复杂。国外有文献报道对产自土耳其等国家的博扎中的微生物采用传统方法分离与鉴定,从研究结果来看,其中蕴藏着丰富的对人体具有益生作用的特殊微生物资源,包括乳酸菌及酵母菌。在博扎饮料微生物组分研究方面,由于在国内,除了小米发酵饮料及酒方面有些研究报道,但与其制作工艺及风味等方面有一定的区别[12]。
在国外,关于博扎饮料微生物组成及其优势菌的分离鉴定方面有研究报道。Botes等[13]学者对土耳其发酵饮料博扎样品微生物组成研究表明,Lactobacillusparacasei subsp. paracasei、Lactobacillus pentosus、Lactobacillus plantarum、Lactobacillus brevis、Lactobacillus rhamnosus 和Lactobacillus fermentum等乳酸菌和Candida diversa、Candida inconspicua、Candida pararugosa、Issatchenkia orientalis、Pichia fermentans、Pichia guillliermondii、Pichia norvegensis、Rhodotorula mucilaginosa 和Torulaspora delbrueckii 等酵母菌参与博扎的发酵过程。
本研究结果初步得到新疆柯尔克孜族家庭博扎饮料中微生物组成包括:植物乳杆菌(Lactobacterium plantarum)、短乳杆菌(Lactobacillus brevis)、巴氏醋酸杆菌(Acetobacer pasteurianus)和蜡状芽孢杆菌(B a c i l l u s c e r e u s)等细菌;酵母菌为酿酒酵母(Saccharomyces cerevisiae)。此研究结果与之前国外学者相关研究结果也有共同点,植物乳杆菌(Lactobacterium plantarum)、短乳杆菌(Lactobacillus brevis)等菌不同地区博扎饮料中普遍存在;但是新疆柯尔克孜族家庭博扎中分离得到的巴氏醋酸杆菌(Acetobacer pasteurianus)和蜡状芽孢杆菌(Bacillus cereus)在以前的研究结果中未发现。此外关于酵母菌的结果也有不同之处,本研究结果中酵母菌发现酿酒酵母(Saccharomyces cerevisiae),这可能与本研究样品的数量有关,应该进一步对来自不同环境、不同家庭里发酵的博扎饮品作为材料进一步研究比较其中的优势菌,找出相对稳定的优势菌群。
从以上研究结果可见,不同国家、同一国家不同地区以及同一地区不同家庭发酵的博扎中的微生物组成有一定的差异,因此其风味也有所差异。这说明不同来源的博扎,因为地区差异、工艺及制作环境等方面不同等原因,其中微生物的种群结构具有一定差异,此外也可以说是不同研究方法所得到的结果差异,因此用分子生物学方法[16]解析其中所含的细菌及酵母菌对分离纯化发酵过程中优势微生物具有指导作用并具有更深刻的意义。
[1]ERCOLINI D. PCR-DGGE fingerprinting: novel strategies for detection of microbes in food[J]. Journal of Microbiological Methods, 2004, 56 (3): 297-314.
[2]ERCOLINI D, FRISSO G, MAURIELLO G, et al. Microbial diversity in natural whey cultures used for the production of Caciocavallo Silano PDO cheese[J]. International Journal of Food Microbiology, 2008, 124 (2): 164-170.
[3]COCOLIN L, AGGIO D, MANZANO M. An application of PCRDGGE analysis to profile the yeast populations in raw milk[J]. International Dairy Journal, 2002, 12(5): 407-411.
[4]AILIN B E H, FLEET G H, PRAKITCHAIWATTANA C, et al. Evaluation of molecular methods for the analysis of yeasts in foods and beverages [M]//HOCKING A D, PITT J I, SAMSON R A, et al. Advances in food mycology. USA: Springer Science+Business Media, Inc, 2006: 69-106.
[5]COCOLIN L, INNOCENTE N, BIASUTTI M, et al. The late blowing in cheese: a new molecular approach based on PCR and DGGE to study the microbial ecology of the alteration process[J]. International Journal of Food Microbiology, 2004, 90(1): 83-91.
[6]DOLCI P, ALESSANDRIA V, COCOLIN G, et al. Microbiological characterization of artisanal Raschera PDO cheese: analysis of its indigenous lactic acid bacteria[J]. Food Microbiology, 2008, 25(2): 392-399.
[7]LEE J S, HEO G Y, LEE J W, et al. Analysis of kimchi microflora using denaturing gradient gel electrophoresis[J]. International Journal of Food Microbiology, 2005, 102(2): 143-150.
[8]FLOREZ A B, MAYO B. Microbial diversity and succession during the manufacture and ripening of traditional, Spanish, blue-veined Cabrales cheese, as determined by PCR-DGGE[J]. International Journal of Food Microbiology, 2006, 110(2): 165-171.
[9]FASOLI S, MARZOTTO M, RIZZOTTI L, et al. Bacterial composition of commercial probiotic products as evaluated by PCR-DGGE analysis[J]. International Journal of Food Microbiology, 2003, 82(1): 59-70.
[10]奥斯伯F, 布伦特 R. 精编分子生物学实验指南[M]. 金友新, 包慧中, 译. 北京: 中国科技出版社: 31-32.
[11]罗静初. 生物信息学概论[M]. 北京: 北京大学出版社, 2002: 97-113.
[12]龚院生, 姚艾东, 王成中. 小米发酵饮料的研制[J]. 郑州粮食学院学报, 1999(3): 41-45.
[13]BOTES A, TODOROV S D, von MOLLENDORFF J W, et al. Identification of lactic acid bacteria and yeast from Bozaa[J]. Process Biochemistry, 2007, 42(2): 267-270.
[14]HANCIOGLU O.., KARAPINAR M. Microflora of Bozaa, a traditional fermented Turkish beverage[J]. International Journal of Food Microbiology, 1997, 35(3): 271-274.
[15]GOTCHEVA V, PANDIELLA S S, ANGELOV A, et al. Microflora identification of the Bulgarian cereal-based fermented beverage Bozaa[J]. Process Biochemistry, 2000, 36(1/2): 127-130.
[16]高蕙文, 吕欣, 董明盛. PCR-DGGE指纹技术在食品微生物研究中的应用[J]. 食品科学, 2005, 26(8): 465-468.
PCR-DGGE Analysis of Microbial Community Structure of Bozaa, a Traditional Kyrgyz Fermented Beverage
Nurgul RAHMAN1,HUA Chang-chun2,ZHU Xiao-ying1,Gulsum TOHSUN1,DONG Ming-sheng2,*
(1.School of Life Sciences, Xinjiang Normal Univery, Urumq 830054, China;2. College of Food Science and Technology, Nanjing Agricultural University, Nanjing 210095, China)
The microbial community structure of Bozaa, a traditional Kyrgyz fermented beverage, was analyzed by the PCRDGGE technique. PCR amplification of 16S rRNA gene V6-V8 region and 26S rDNA D1 region were performed for bacteria and yeast in the beverage, respectively. The main DNA bands in the DGGE profiles of the PCR products obtained were subjected to sequence analysis. The results showed that the microbial flora of Bozaa was composed of Lactobacterium plantarum, Lactobacillus brevis, Bacillus cereus, Acetobacer pasteurianus, and Saccharomyces cerevisiae, which was a successful primary elucidation of the microbial diversity of Bozaa.
Bozaa;denaturing gradient gel electrophoresis (DGGE);microflora
TS201.3
A
1002-6630(2012)01-0111-04
2011-08-17
新疆师范大学博士科研启动基金项目;国家自然科学基金项目(31050005)
努尔古丽·热合曼(1972—),女,副教授,博士,研究方向为食品微生物。E-mail:nurgul0716@sohu.com
*通信作者:董明盛(1961—),男,教授,博士,研究方向为食品微生物与生物技术。E-mail:dongms@njau.edu.cn
