EST-SSR标记在蔬菜作物中的开发和应用进展
2012-02-23吴海滨罗少波罗剑宁何晓莉
吴海滨 罗少波 罗剑宁* 龚 浩 何晓莉
(1 广东省农业科学院蔬菜研究所,广东广州 510640;2 广东省蔬菜新技术研究重点实验室,广东广州 510640)
DNA 分子标记在分子遗传学的建立和发展过程中起着举足轻重的作用,同时也是作物遗传育种的重要工具。其中SSR(Simple Sequence Repeats)又称简单序列重复,是一种在PCR 基础上发展起来的分子标记技术。与其他分子标记相比,它具有标记潜在数量丰富,等位位点变异多,信息含量高,共显性,操作简便,重复性好等优点。因此,20 世纪90年代以来,在一些大田作物中,SSR 标记已经被遗传学家和育种学家广泛应用于遗传多样性分析、亲缘关系鉴定、遗传图谱构建、功能基因的定位和克隆、分子标记辅助育种等研究领域(方宣钧 等,2000)。根据SSR 标记的序列性质不同,SSR 标记可分为基因组SSR(Genomic SSR,gSSR)和表达序列标签SSR(Expressed sequence tag SSR,EST-SSR),其中EST-SSR 是近年来建立起来的新型分子标记。
1 EST-SSR 标记概述
EST(Expressed Sequence Tags)即表达序列标签,是将mRNA 反转录成cDNA,构建成cDNA文库后,大规模随机挑选文库中cDNA 克隆,对其3′或5′端进行单向测序所获得的一段cDNA序列。EST 是表达基因的窗口,它能反映mRNA 的信息,可代表生物体某种组织或细胞在某一时间的一个表达基因(于凤池,2005)。目前,EST 数据库作为基因组学与后基因组学研究的桥梁和工具,已经被广泛应用于功能基因组分析、比较基因组分析、新基因的发现、基因表达调控及基因芯片的制作等研究领域。
研究表明,10%的 EST 序列中包含有简单重复序列,可以用作 EST-SSR 标记的开发(Yammanoto & Sasaki,1997;Hatey et al.,1998)。EST-SSR 标记的开发和应用原理与gSSR 相似,两者区别在于EST-SSR 来源于基因组的编码区,更能反映出生物功能基因的信息(Li et al.,2004)。与gSSR 相比,EST-SSR 标记还具有开发费用少和难度相对较低、通用性好、信息量大等优点。作为一种新的SSR 标记的来源,EST-SSR 标记已经在众多生物种类中得到证实和应用。
2 EST-SSR 标记在蔬菜作物中的研究进展
2.1 植物EST 数据库研究进展
植物EST 计划作为植物基因组计划的一个重要组成部分,已经在拟南芥、水稻、玉米、小麦、油菜等多种植物中得到开展。随着EST 计划在不同物种间的不断扩展和研究的深入,以及DNA 测序技术的不断进步,EST 的数量呈指数级的惊人速度增长。1991年各个公共数据库的EST数目尚不足2 000 条,但随着EST 数据的增多,1993年美国国家生物技术信息中心(National center of Biotechnology information,NCBI)建立了一个专门的EST 数据库dbEST(data base of EST)用来收集和保存所有的EST 数据。截止2011年2月28日,该数据库收集的水稻、玉米、小麦EST 序列数目已分别达到1.6×106、2.0×106、1.1×106条,蔬菜作物中黄瓜、甜瓜、番茄、辣椒的EST序列数目也已经分别达到8.1×103、3.6×104、2.9×105、1.1×105条。EST 资源数据库的不断扩充极大地方便和加快了人们在生命科学领域的研究,也为利用这些数据来开发EST 分子标记奠定了基础。
2.2 EST-SSR 标记在蔬菜作物中的研究现状
蔬菜是一大类作物的总称,种类多、范围广。初步统计,现在全国栽培的蔬菜种类(含亚种、变种)已有209 种,其中普遍栽培的种类也有60~120 种。目前,EST-SSR 标记在一些主要蔬菜作物中已经得到了一定程度的开发和应用(表1)。为了便于叙述,本文将主要蔬菜作物大致分为叶菜类、茄果类和瓜类进行综述。
2.2.1 叶菜类 在大白菜和普通白菜的EST-SSR 研究中,忻雅等(2006)对4 584 条大白菜和普通白菜EST 进行了SSR 搜索,共检索出474 个SSR 位点,检出率为10.3%。根据这些序列,设计了15 对EST-SSR 引物,在合适的PCR 反应体系下,以大白菜自交系A 的DNA 为模板,进行引物的筛选,发现15 对EST-SSR 引物都能扩增出产物。进一步用这些可扩增的引物对28 个大白菜和普通白菜品种进行PCR 扩增,发现7 对引物显示多态性,占引物总数的46.7%。李丽和郑晓鹰(2009)研制了由3 个引物对组成的6 套EST-SSR 复合标记组合,用于鉴定白菜〔Brassica campestrisL.ssp.chinensis(L.)Makino〕和大白菜〔Brassica campestrisL.ssp.pekinensis(Lour)Olsson〕品种,并形成了多重SSR 的最佳试验条件。这6 套标记组合完全鉴别了43 个白菜和大白菜品种,并且比较了单引物标记与复合标记的鉴定效率,确认其可以高效、准确和全面地鉴别白菜和大白菜品种的真实性和杂交种纯度。
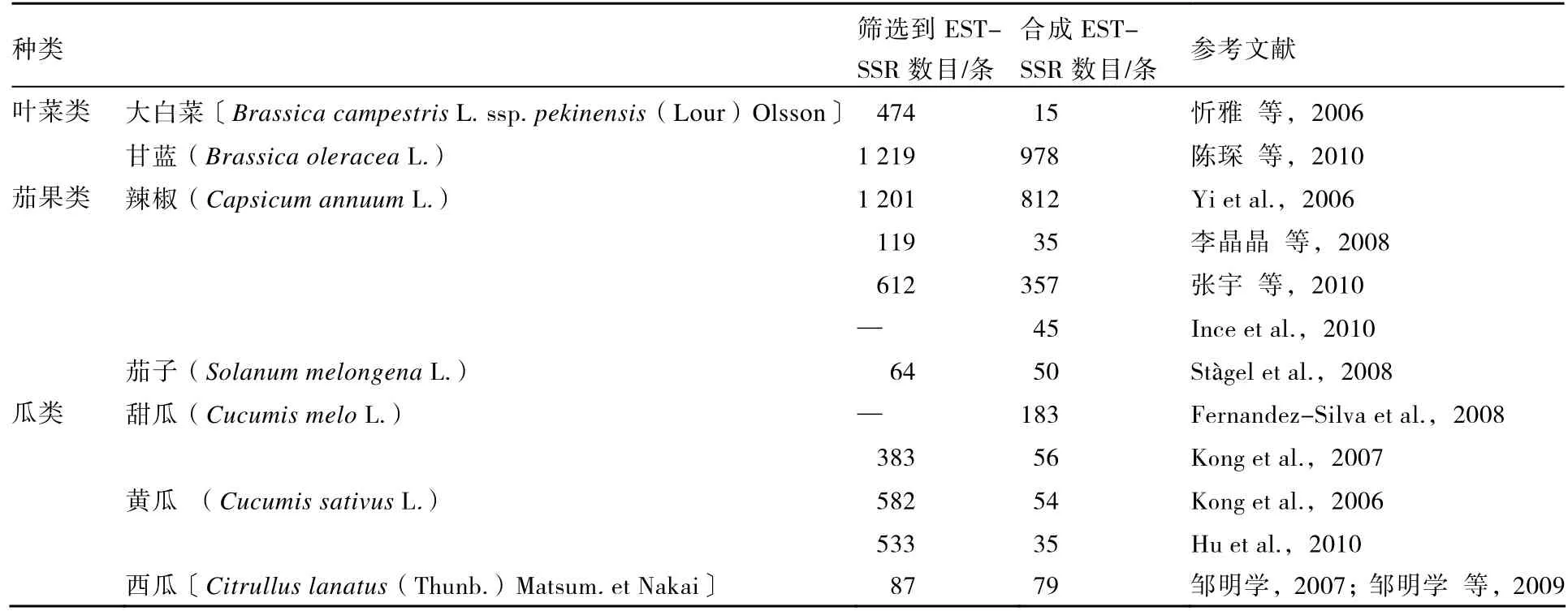
表1 蔬菜作物EST-SSR 标记开发进展
关于甘蓝的EST-SSR 研究,陈琛等(2010)从NCBI 数据库下载62 567 条甘蓝相关EST,处理后得到19 611 条无冗余的EST,通过对其进行SSR 搜索,共得到1 219 个SSR,分布在1 176条EST 上,分布密度为1/11.48 kb,包括273 种重复基元。针对这1 176 条含有SSR 的EST 序列,设计合成了978 对引物,用两份甘蓝自交系对978 对引物进行初筛,897 对引物有扩增产物,共扩增出1 026 条扩增带;128 对引物表现出多态性,共有258 条多态性带(占总带数的25.15%)。利用4 对多态性引物,初步构建了中甘11 号、8398、中甘15 号、中甘21 号甘蓝杂交新品种及其亲本的指纹图谱。以上结果说明从甘蓝相关EST 数据库中开发的SSR 引物有较好的可用性。
2.2.2 茄果类 在茄果类蔬菜中,有关辣椒的EST-SSR 分子标记研究相对较多。Yi 等(2006)对辣椒10 232 条非冗余的EST 序列进行SSR 搜索,获得1 201 个SSR 位点,平均每3.8 kb 有1个SSR 位点。利用所含SSR 的EST 序列,设计了812 对EST-SSR 引物,其中513 对引物(63.1%)有扩增产物,150 对引物(29.2%)在辣椒属种间不同品种 TF68(C.annuum)和 Habanero(C.chinense)显示出多态性。研究发现,包含AC 二核苷酸重复的EST-SSR 标记的多态性最高,多态性与基序长度和重复数成正比。同时将139 个多态性EST-SSR 引物整合于之前构建的遗传连锁图上,使得该连锁图包含了14 个连锁群,覆盖长度2 201.5 cM。这些EST-SSR 标记均匀分布于各个连锁群。这种基于SSR 标记的遗传连锁图谱对于辣椒属的比较作图和分子标记辅助育种作用很大。李晶晶等(2008)对4 000 条辣椒EST 序列进行筛选,获得了119 条含有SSR的EST,共设计出35 对EST-SSR 引物,占所有EST 序列的0.87%。以辣椒细胞质雄性不育系(CMS)21A,保持系21B 基因组DNA 为模板,利用新合成的EST-SSR 引物进行筛选,首次获得编号为Pe21、Pe26 和Pe27 的3 对与辣椒育性相关的特异引物。这些信息对进一步研究辣椒细胞质雄性不育分子机理有一定的促进作用。张宇等(2010)利用SSRIT 软件从NCBI 数据库中的19 173 条辣椒EST 序列中筛选到612 个SSR,检出率为3.19%。研究发现辣椒EST-SSR 以5~10 次重复为主,基序长度集中在15~20 bp 范围内。利用含SSR 的576 条EST 序列共设计357对引物,在合适的PCR 条件下,337 对(94.40%)引物扩增出清晰条带,75 对(21.01%)引物表现多态性,表明EST-SSR 标记在辣椒遗传分析中是有价值且可行的。Ince 等(2010)利用辣椒属EST 序列数据,设计了45 对EST-SSR 标记。将所设计的标记在辣椒属的多个种内进行验证,包括一年生辣椒(C.annuumL.)、灌木状辣椒(C.frutescensL.)、中国辣椒(C.chinense)、下垂辣椒(C.baccatum)和柔毛辣椒(C.pubescens)。结果显示,这些EST-SSR 标记在辣椒属内不同种间具有良好的通用性。同时,研究发现包含二核苷酸重复的SSR 标记比三核苷酸重复的SSR 标记具有更好的多态性。
Stàgel 等(2008)筛选了超过3 300 条茄属的EST 序列和基因组编码区序列,获得了64 条序列共含有70 个SSR 位点,其中50 条序列可以用于引物设计。用这些引物对38 个茄子栽培种和6 个茄属近缘种进行分析,其中39 对(78.0%)引物有扩增产物,31 对(79.5%)引物在茄属种间有多态性,仅有 11 对(28.2%)引物在茄属栽培种内显示多态性。通过试验证明这些EST-SSR 标记在遗传多样性分析和构建系统进化树方面作用显著。
2.2.3 瓜类 在葫芦科瓜类作物中,甜瓜、黄瓜、西瓜的EST-SSR 标记研究相对较多。Kong 等(2007)将GenBank 和Melon EST Database 数据库中收录的5 474 条来自甜瓜的EST 和mRNA序列进行拼接,共得到3 188 条Unigene,覆盖长度为1.8 Mb。对这些序列进行SSR 位点搜索,共获得分布于309 条Unigene 的383 个EST-SSR 位点,SSR 平均分布距离为1/4.7 kb。在这些鉴定出的SSR 位点中,191(49.9%)个位点为二核苷酸重复,167(43.6%)个位点为三核苷酸重复,12(3.1%)个位点为四核苷酸重复,8(2.1%)个位点为五核苷酸重复,5(1.3%)个位点为六核苷酸重复。选择合成56 对EST-SSR 引物,其中47 对引物能够扩增出预期产物,22 对引物在27 个甜瓜种质资源中扩增出特异性的多态性产物,平均每个位点能够检测到2.9 个等位基因。可转移性分析表明,有15 个引物对在黄瓜上获得了特异性扩增产物,占总数的68.2%。Fernandez-Silva 等(2008)根据甜瓜EST 序列和基因组文库序列设计了225 对SSR 引物,其中包括183 对EST-SSRs 引物和42 对gSSRs 引物。用这些引物在甜瓜种质资源中进行扩增验证,结果表明144 对(79%)EST-SSRs 引物和36 对(86%)gSSRs 引物有扩增产物。其中126 对EST-SSRs 引物和31 对gSSRs 引物在甜瓜种质资源中显示出多态性,多态性引物占有扩增产物引物的87%,EST-SSR 和gSSR 引物的多态率相似,分别为87.5%和89.0%。为了验证EST-SSR引物在葫芦科内部的通用性,将这些引物在南瓜属中的中国南瓜、美洲南瓜和印度南瓜中进行扩增验证。结果表明,有12.7%的引物至少在一种南瓜中有扩增产物,仅有5.4%的标记显示出多态性。并将其中121 个新开发的SSRs 标记整合到以往构建的甜瓜遗传图谱中。
Gonzalo 等(2005)对比了甜瓜的EST-SSR 标记和gSSR 标记的多态性差异,研究表明EST-SSR 的多态性比例为45.5%,gSSR 的多态性比例为51.2%,两者多态性值类似,并把EST-SSR 标记整合到甜瓜的遗传图谱上。
Kong 等(2006)从GenBank 中共获得了5 268 条来自于黄瓜的EST 和基因序列,通过对这些序列分别进行组装,获得3 558 条黄瓜的Unigene。以重复基序长度为2~6 bp、最低重复次数5 次为标准,在Unigene 上搜寻SSR 位点,在474 条Unigene 中共获得了582 个SSR 位点。在SSR 位点中,二核苷酸重复占60%,三核苷酸重复占36%。选择了54 个SSR 位点合成侧翼引物,在20 个黄瓜基因型上评价这些SSR 位点的多态性,其中有49 对引物有预期的扩增产物;20 对引物在20 个黄瓜种质材料中扩增出了特异性的具有多态性的产物。在多态性EST-SSR 位点上能够检测到2~6 个等位基因,平均每个位点能够检测到3.3 个等位基因。可转移性分析表明,有13 个引物对在甜瓜上有特异性扩增产物,占总数的65%,其中有7 个(35%)引物对在测试的甜瓜基因型上表现出多态性。Hu 等(2010)对GenBank 上公布的6 344 条黄瓜ESTs 进行SSRs 搜索,获得533 个SSRs 位点,这些SSRs 位点主要为二核苷酸和三核苷酸重复,其中以AG和AAG 重复单元为主。392 条包含SSRs 位点的Unigene 适合用于引物设计。选择合成了35 对SSR引物,28 对引物在黄瓜中有预期的特异性扩增产物,26 对引物在21 个黄瓜种质资源中扩增出特异性的多态性产物,平均每个位点能够检测到3.77 个等位基因。为了验证EST-SSR 引物在葫芦科内部的通用性,将28 对在黄瓜中有特异性扩增产物的引物在甜瓜、西瓜、南瓜和丝瓜中进行扩增验证。结果表明,分别有26(92.9%)、16(57.1%)、15 对(53.6%)和17 对(60.7%)引物有扩增产物。这表明EST-SSR 标记有很好的物种间通用性,适合用于比较基因组分析。
邹明学(2007)和邹明学等(2009)对820 条来自西瓜的EST 序列进行了SSR 序列的筛选,共发现SSR 序列87 条,占整个EST 数据库的10.6%。二核苷酸重复中AG、GA、CT 和TC 较高,三核苷酸重复以CAA 所占比例最大。根据筛选EST 数据库得到的SSR 序列设计并合成了79 对EST-SSR 引物。经过PCR 扩增和银染检测表明,36 对引物在西瓜作图亲本材料P1296341 和97103 中显示出稳定清晰的带型,并将其中的9 个EST-SSR 标记整合到以往构建的遗传图谱上。
3 EST-SSR 标记开发存在问题及解决方案
大量高质量的EST 序列信息是EST-SSR 标记开发的根本。目前,对于蔬菜作物而言,虽然网络公共数据库中已经积累了大量EST 数据,但是对于部分非大宗蔬菜作物而言,公共数据库中的可利用信息非常有限。例如,截止2011年10月2日,dbEST 数据库所收集的苦瓜、冬瓜、丝瓜EST 序列分别仅有180、19 条和1 条,如此少的序列信息将无法开发EST-SSR 标记。缺乏EST 序列信息是EST-SSR 标记在蔬菜作物中的大规模开发和应用的瓶颈。近年来,随着新一代高通量测序技术的快速发展,大规模测序的费用较以往已经大大降低,例如利用solexa 测序技术对一种新作物的转录组进行测序获得理想数据量的费用一般可以控制在5 万元以内。因此,对于某些特色蔬菜作物,即使通过自主测序来开发EST-SSR 标记,其费用对于大多数实验室而言也是可以接受的。
4 展望
蔬菜种类繁多,不同蔬菜作物间研究水平参差不齐。在分子标记研究领域中,除个别优势蔬菜作物外,大部分蔬菜作物研究水平薄弱。由于缺乏相应的序列信息,蔬菜中大量使用的分子标记主要分为两类:一类是随机引物标记,如RAPD 标记、ISSR 标记、SRAP 标记等(Singh et al.,2007;夏军辉和向长萍,2008;高山 等,2010;李怀志 等,2011),由于随机引物PCR所扩增的DNA 区段是无法预知的,具有很强的随机性和任意性,因此所获得的试验结果往往重复性差,准确度低;另一类是基于限制性酶切和PCR 技术的AFLP 标记(Gaikwad et al.,2008;杨衍 等,2009),该标记具有多态性条带丰富、信息量大、特异性高等优点。但是由于该技术步骤繁琐、操作难度大、耗时长等缺点,大大限制了该标记的应用。
近年来,随着测序技术的发展和各种生物体基因组、转录组测序的快速推进,单核苷酸多态性标记(single nucleotide polymorphism,SNP)越来越受到人们的重视。SNP 是指在基因组水平上单个核苷酸的变异所引起的DNA 序列多态性,它具有基因组分布密度高及数量巨大等优点,被认为是应用前景最好的遗传标记。目前,对于大多数蔬菜作物而言,SNP 的开发和检测还存在一定困难:首先,SNP 的开发需要依赖大量基因组或转录基因的序列信息,而蔬菜作物中大多数物种的基因组信息未知,因此标记的开发难度极大;其次,SNP 标记的检测难度相对较大,目前SNP 的检测方法主要有直接测序法、DNA 芯片技术、酶切PCR 检测法等。这些方法往往存在操作难度大、步骤繁琐、花费高等缺点,对于一般实验室而言,难以大规模应用。近年来,美国Idaho 公司开发出了高分辨熔解曲线检测系统(High Resolution Melting,HRM)突变筛查和基因分型分析系统,能够快速、大规模地检测SNP。但是该系统对样品和试验条件要求较高,同时大规模检测费用较高。因此,SNP 标记在蔬菜作物中的大规模应用还需要相当长的时间。
EST-SSR 标记与上述标记相比,具有操作简单、共显性、扩增稳定、多态性高、物种间通用性较好等突出优点,非常适合运用于蔬菜作物中。随着测序技术的发展,将会有越来越多蔬菜作物的转录组序列被测定,这将使得EST-SSR 标记的开发费用大大降低,将为其在蔬菜作物中的应用提供更加广阔的空间。到目前为止,EST-SSR 标记在一些蔬菜作物中已经得到了一定的应用,但是对于大多数蔬菜而言,其应用程度远远不够。基于EST-SSR 标记的诸多优点,大力开发蔬菜作物中EST-SSR 标记,对于提高分子标记在育种中的应用,加快蔬菜分子标记辅助育种步伐,进一步推进蔬菜产业的发展具有重要意义。
陈琛,庄木,李康宁,刘玉梅,杨丽梅,张扬勇,程斐,孙培田,方智远.2010.甘蓝EST-SSR 标记的开发与应用.园艺学报,37(2):221-228.
方宣钧,吴为人,唐纪良.2000.作物DNA 标记辅助育种.北京:科学出版社:28-31.
高山,林碧英,许端祥,傅睿清,林峰,林义章,潘东明.2010.苦瓜种质遗传多样性的RAPD 和ISSR 分析.植物遗传资源学报,11(1):78-83.
李怀志,张峻,李翔,陈火英.2011.应用SRAP 标记对茄子品种进行遗传多样性分析与指纹图谱构建.南京农业大学学报,34(4):18-22.
李晶晶,王述彬,刘金兵,潘宝贵,陈劲枫.2008.辣椒EST-SSR 标记的开发.分子植物育种,6(6):1219-1222.
李丽,郑晓鹰.2009.用于白菜和大白菜品种鉴定的EST-SSR 复合标记的建立.园艺学报,36(11):1627-1634.
忻雅,崔海瑞,卢美贞,姚艳玲,金基强,林容杓,崔水莲.2006.白菜EST-SSR 信息分析与标记的建立.园艺学报,33(3):549-554.
夏军辉,向长萍.2008.丝瓜种质资源遗传多样性的形态和RAPD 标记分析.中国蔬菜,(10):21-25.
杨衍,刘昭华,詹园凤,廖易,刘维侠.2009.苦瓜种质资源遗传多样性的AFLP 分析.热带作物学报,30(3):299-303.
于凤池.2005.EST 技术及其应用综述.中国农学通报,21(2):54-58.
张宇,张晓芬,陈斌,耿三省,李焕秀,李珍.2010.辣椒EST-SSR 信息分析及标记开发.西北农业学报,19(9):186-192.
邹明学.2007.西瓜EST-SSR 标记的开发及遗传图谱的构建与整合〔硕士论文〕.北京:首都师范大学.
邹明学,陈艳红,许勇,郭绍贵,张海英,宫国义.2009.西瓜EST-SSR 分子标记的筛选.长江蔬菜,(18):13-14.
Fernandez-Silva I,Eduardo I,Blanca J,Esteras C,Pico B,Nuez F,Arus P,Garcia-Mas J,Monforte A J.2008.Bin mapping of genomic and EST-derived SSRs in melon(Cucumis meloL.).Theor Appl Genet,118:139-150.
Gaikwad A B,Behera T K,Singh A K,Chandel D,Karihaloo J L,Staub J E.2008.Amplified fragment length polymorphism analysis provides strategies for improvement of bitter gourd(Momordica charantiaL.).HortScience,43(1):127-133.
Gonzalo M J,Oliver M,Garcia-Mas J,Monfort A,Dolcet-Sanjuan R,Katzir N,Arús P,Monforte A J.2005.Simple-sequence repeat markers used in merging linkage maps of melon(Cucumis meloL.).Theor Appl Genet,110:802-811.
Hatey,F,Tosser-klopp G,Martinato CC,Mulsant P,Gasser F.1998.Expressed sequence tags for genes:a review.Genet Sel Evol,30:521-554.
Hu J,Zhou X,Li J.2010.Development of novel EST-SSR markers for cucumber(Cucumis sativus)and their transferability to related species.Scientia Horticulturae,125:534-538.
Ince A G,Karaca M,Onus A N.2010.Polymorphic microsatellite markers transferable acrossCapsicumspecies.Plant Mol Biol Rep,28:285-291.
Kong Q,Xiang C,Yu Z.2006.Development of EST-SSRs inCucumis sativusfrom sequence database.Molecular Ecology Notes,6:1234-1236 .
Kong Q,Xiang C,Yu Z,Zhang C,Liu F,Peng C,Peng X.2007.Mining and charactering microsatellites inCucumis melonexpressed sequence tags from sequence database.Molecular Ecology Notes,7:281-283.
Li Y C,Korol A B,Fahima T,Nevo E.2004.Microsatellites within genes: structure,function,and evolution.Mol Biol Evol,21:991-1007.
Singh A K,Behera T K,Chandel D,Sharma P,Singh N K.2007.Assessing genetic relationships among bitter gourd(Momordica charantiaL.)accessions using inter-simple sequence repeat(ISSR)markers.The Journal of Horticultural Science & Biotechnology,82(2):217-222.
Stàgel A,Portis E,Toppino L,Rotino G L,Lanteri S.2008.Gene-based microsatellite development for mapping and phylogeny studies in eggplant.BMC Genomics,9:357.
Yammanoto K,Sasaki T.1997.Large-scale EST sequencing in rice.Plant Mol Biol,35:135-144.
Yi G,Lee J M,Lee S,Choi D,Kim B D.2006.Exploitation of pepper EST-SSRs and an SSR-based linkage map.Theor Appl Genet,114:113-130.
