进化代谢选育高渗透压耐受型产琥珀酸大肠杆菌
2012-02-09张常青苟冬梅梅佳军刘嵘明马江锋陈可泉朱建国姜岷
张常青,苟冬梅,梅佳军,刘嵘明,马江锋,陈可泉,朱建国,2,姜岷
1 南京工业大学 生物与制药工程学院 材料化学工程国家重点实验室,江苏 南京 211816
2 常茂生物化学工程股份有限公司博士后科研工作站,江苏 常州 213034
琥珀酸又名丁二酸,是一种较为常见的有机酸,是TCA循环的中间代谢产物以及厌氧碳代谢过程的终端还原产物。琥珀酸作为一种四碳二元羧酸,广泛应用于食品、农业、制造业等各个领域[1]。
相对于化学合成法,生物合成琥珀酸是利用细菌、真菌等各种微生物,以葡萄糖或其他各种水解液为碳源,经微生物发酵生产琥珀酸,其最大优势是原材料为可再生资源,并且具有消耗低、污染小、反应温和等优点,因此生物法合成琥珀酸成为近年来研究的热点[2-3]。
钠离子是维持细胞各项生理特性的最重要的离子之一[4],对菌体细胞的生长[5]、ATP的生成[6]、胞内物质运输以及扩散[7]、胞内 pH的调控[8]、基因转录[9]等都有一定的影响。对许多产琥珀酸菌体,当培养基中缺乏Na+时,会使菌株对葡萄糖的转运造成不良的影响[10],使得菌体无法正常代谢生长。然而当Na+浓度较高时,由于Na+的积累造成的渗透压升高,对菌体的生长代谢造成不利的影响[11-12]。Lee等[13]曾报道,在产琥珀酸厌氧螺菌Anaerobiospirillum succiniciproducens发酵过程中,当NaCl的浓度高于4 g/L时,菌体的生长以及产酸都受到抑制。Fang等[14]和徐敏等[15]曾报道通过筛选高浓度NaCl耐受性菌株,提高产琥珀酸放线杆菌Actinobacillus succinogenes对渗透压的耐受性,从而提高琥珀酸的产量。
在以NaOH和Na2CO3为酸中和剂进行的大肠杆菌 AFP111两阶段发酵生产琥珀酸的过程中,会有大量的Na+积累,从而导致体系中渗透压升高,从而严重抑制菌体的生长以及产酸。因此,选育一株具有高渗透压耐受型琥珀酸生产菌株显得尤为重要。
进化代谢是以一定的选择性压力为手段,利用菌体对环境的自适应机制,从细胞库中筛选出所需要的表型突变,使菌体选择性进化的方法[16]。进化代谢是一种非常高效的菌株筛选方法,它在生物进化研究和菌株选育中具有广泛的应用。利用基因组学与进化代谢技术相结合的方法,可以直观地了解到变异菌体整个基因组的变化,在菌体适应性进化的动力学和基因学基础方面也获得了巨大的成功[17-18],这对于研究生物进化是一种里程碑式的突破。因为细菌具有数量大,传代速度快,并且可以适应一定环境的优点,因此对于可以适应选择性压力的菌体,能够快速地适应外界环境并达到稳定的生长状态,从而在进化代谢的过程中获得生长优势,对于那些不能快速适应环境的菌体,由于其生长速度慢,则很快被优势菌体淘汰[17,19-20]。进化代谢方法主要用于以下几个方面的菌株选育,一是选择能够抵抗某种底物或产物压力的高抗逆菌株,如改善产琥珀酸放线杆菌在琥珀酸生产中铵根离子的抑制[21-22],二是选择可以高效利用某些底物的菌株,如改善菌体对某些糖的利用效率[23-27]。三是选育可以高效生产某一特定产物的菌体[28-29]。本研究以NaCl为渗透压调节剂,大肠杆菌AFP111为出发菌株,通过不断提高选择性培养基中NaCl的浓度,最终得到了一株高渗透压耐受型琥珀酸生产菌株。
1 材料与方法
1.1 菌株
大肠杆菌 Escherichia coli AFP111 [F+ λrpoS396 (Am) rph-1 △(pflAB::Cam) ldhA::Kan 95 ptsG],由David P. Clark 教授 (Southern Illinois University) 惠赠。
1.2 培养方法
1.2.1 种子培养基
蛋白胨10 g/L,酵母粉5 g/L,NaCl 5 g/L,pH 7.0,卡那霉素30 mg/L,氯霉素30 mg/L,过滤除菌后加入。
1.2.2 筛选平板培养基
在种子培养基的基础上,加入NaCl调节其最终浓度为0.2~0.8 mol/L,琼脂1.5%~2.0%。
1.2.3 发酵培养基
柠檬酸 3 g/L,Na2HPO4·12H2O 4 g/L,KH2PO48 g/L,(NH4)2HPO48 g/L,NH4Cl 0.2 g/L,(NH4)2SO40.75 g/L,MgSO4·7H2O 1 g/L,CaCl2·2H2O 10 mg/L,ZnSO4·7H2O 0.5 mg/L,CuCl2·2H2O 0.25 mg/L,MnSO4·H2O 2.5 mg/L,CoCl2·6H2O 1.75 mg/L,H3BO30.12 mg/L,Al2(SO4)31.77 mg/L,Na2MoO4·2H2O 0.5 mg/L,柠檬酸铁16.1 mg/L,121 ℃高压蒸汽灭菌15 min;维生素B120 mg/L,生物素2 mg/L,卡那霉素30 mg/L,氯霉素30 mg/L,过滤除菌后加入。
1.2.4 摇瓶发酵用培养基
30 mL发酵培养基 (如1.2.3),添加0.48 g碱式碳酸镁,30 g/L葡萄糖。
1.3 方法
1.3.1 种子培养
将保存于-80 ℃冻存管中的菌株以1%的接种量转接到装液量为 5 mL的试管中,37 ℃、200 r/min过夜培养作为一级种子。将试管中的一级种子,以1%的接种量转接到装液量为100 mL的500 mL三角瓶中,37 ℃、200 r/min培养6 h,作为二级种子。
1.3.2 两阶段摇瓶发酵
将二级种子培养的细胞8 000 r/min、4 ℃离心10 min,然后用30 mL新鲜发酵培养基将细胞重新悬浮到 100 mL厌氧血清瓶中,通无菌CO22 min以维持厌氧环境,37 ℃、200 r/min培养48 h。
1.3.3 发酵罐培养
采用发酵培养基,在7 L发酵罐 (BioFlo 110 fermenter;New Brunswick ScientificCo.,Edison,N.J.) 中进行两阶段发酵,装液量3 L,接种量5%,发酵温度37 ℃。在菌体有氧生长阶段,初始糖浓度为 15 g/L。当菌体浓度达到 OD600=15时,此时发酵罐中葡萄糖已经耗尽,流加600 g/L的葡萄糖,控制菌体比生长速率为0.7 h-1,有氧阶段,20% NaOH控制pH 6.8。当菌体密度达到 OD600=30,通入无菌过滤的 CO2转为厌氧发酵,通气量为 0.5 L/min,并改用 25% Na2CO3调节pH为6.4。流加补充葡萄糖,维持葡萄糖浓度在10 g/L左右[30-31]。
1.3.4 进化代谢选育
以一个自主设计的500 mL五口烧瓶作为进化代谢的反应器,装液量为200 mL,接种量为1%。水浴加热维持37 ℃,200 r/min磁力搅拌,0.5 L/min通入无菌空气,20% Na2CO3控制pH为 6.8。通过蠕动泵不断泵入新鲜培养基,当反应器中菌液体积超出200 mL时,将自动被空气压出。控制反应器中最初的稀释速率为0.02 h-1,当在一定的NaCl条件下,菌体密度和耗糖保持3个保留时间不变,则提高稀释速率到0.04 h-1,直至稀释速率达到0.153 h-1。新鲜培养基中NaCl的最初浓度为0.4 mol/L,逐渐增加至0.8 mol/L。在每次提高新鲜培养基中NaCl的浓度之前,从反应器中取样稀释1×104倍涂布在相应的筛选培养基平板上。
1.3.5 突变株的筛选
将进化代谢装置选育的突变株在筛选平板上多次传代培养,挑选生长性能稳定、生长迅速的菌株进行两阶段摇瓶发酵,37 ℃、200 r/min培养48 h,HPLC检测各菌株发酵性能。
1.3.6 分析检测
菌体密度测定:紫外可见分光光度计(Spectrumlab 752S),于600 nm处测定吸光值。
葡萄糖浓度测定:生物传感分析仪(SBA240C,山东省科学院生物研究所)。
发酵液中有机酸的测定[32]:用高效液相色谱法 (HPLC) 检测,色谱柱为 Prevail Organic Acid,流动相为25 mmol/L KH2PO4,pH 2.5,流速1.0 mL/min,紫外检测波长215 nm。
2 结果
2.1 渗透压对Escherichia coli AFP111产琥珀酸的影响
以NaCl为渗透压调节剂,考察了渗透压对E. coli AFP111生长性能和产酸的影响。由图1可知,在较低的NaCl浓度下,菌体的生长以及产酸受到的影响较小。然而,当培养基中 NaCl浓度超过0.4 mol/L时,菌体的生长明显受到影响,继续增加NaCl的浓度,则菌体生长严重被抑制,琥珀酸的产量也严重下降,尤其当培养基中NaCl浓度达到0.6 mol/L时,琥珀酸的产量仅有4.3 g/L。该结果说明渗透压对E. coli AFP111的生长以及产酸具有很大的影响,在高渗透压条件下,菌体的生长以及产酸都受到严重的抑制。因此,选育一株高渗透压耐受性并且高产琥珀酸的菌株是很有必要的。

图1 渗透压对菌体生长 (A) 和琥珀酸生产的影响 (B)Fig. 1 Effect of osmotic stress on cell growth (A) and succinic acid production by E. coli AFP111 (B). (A) The growth curve of E. coli AFP111 was measured under aerobic condition in LB media supplemented with different concentrations of NaCl. (B) Dual-phase fermentations of E. coli AFP111 after 48 h anaerobic fermentation supplemented with different concentrations of NaCl.
2.2 进化代谢系统的设计与操作
进化代谢育种系统是一个连续培养装置,通过不断补加含有高浓度NaCl的发酵培养基并逐步提高NaCl的浓度筛选耐高渗透压生产菌株。由图2可知,在504 h处,补料培养基中NaCl的浓度达到了 0.8 mol/L,稀释速率维持在0.153 h-1,该菌体密度的增加以及葡萄糖的消耗速率维持时间多于3个保留时间[21,33],表明菌体已经利用对环境的自适应机制,从细胞库中筛选出能所需要的表型突变,在高渗透压条件下进化代谢得到所需菌株,使其可以在高渗透压条件下快速生长。

图2 连续培养操作过程Fig. 2 The operational process of adaptive evolution.
2.3 突变菌株的筛选
进化代谢系统中菌体密度升高以及残糖的下降表明了耐高渗透压突变株已传代生长。从反应器中取适量菌体,涂布在含有 0.4~0.8 mol/L NaCl的筛选平板培养基中多次传代培养。选择菌体生长速度快,菌落直径较大的菌株进行两阶段摇瓶发酵。最终筛选到6株产酸性能较好的菌株XB1、XB2、XB3、XB4、XB5、XB6,其两阶段发酵厌氧阶段的产酸情况见表1。

表1 突变菌株与出发菌株AFP111产酸效果对比aTable 1 Succinic acid production in AFP111 compared with XB4a
由表1可以看出在0.4 mol/L NaCl的发酵培养基中,出发菌株AFP111的产酸明显受到抑制,琥珀酸的产量明显降低,并且菌体衰亡很快,而突变菌株的发酵性能在高浓度 NaCl中明显改善,琥珀酸的产量和收率都明显高于出发菌株,菌体的 OD600也要高于出发菌株。其中突变株XB4琥珀酸产量为23.7 g/L,比出发菌株提高了56%,基本达到了出发菌株在未添加NaCl的培养基中的产量 (24.5 g/L),琥珀酸相对于葡萄糖的收率为92%,也高于出发菌株的86%。
2.4 突变株XB4和出发菌株AFP111在不同浓度NaCl条件下的产酸效果对比
分别将突变株XB4和出发菌株AFP111在含有不同浓度NaCl的发酵培养基中进行两阶段摇瓶发酵,结果如图3所示。
由图 3可知,在两阶段摇瓶发酵过程中,NaCl的浓度对菌体的发酵产酸具有很大的影响。然而由高浓度 NaCl造成的高渗透压,突变株XB4与出发菌株AFP111相比,耐受性有了明显改善,尤其是当培养基中Na+的浓度大于0.6 mol/L时,突变株在产酸方面的优势更加明显。
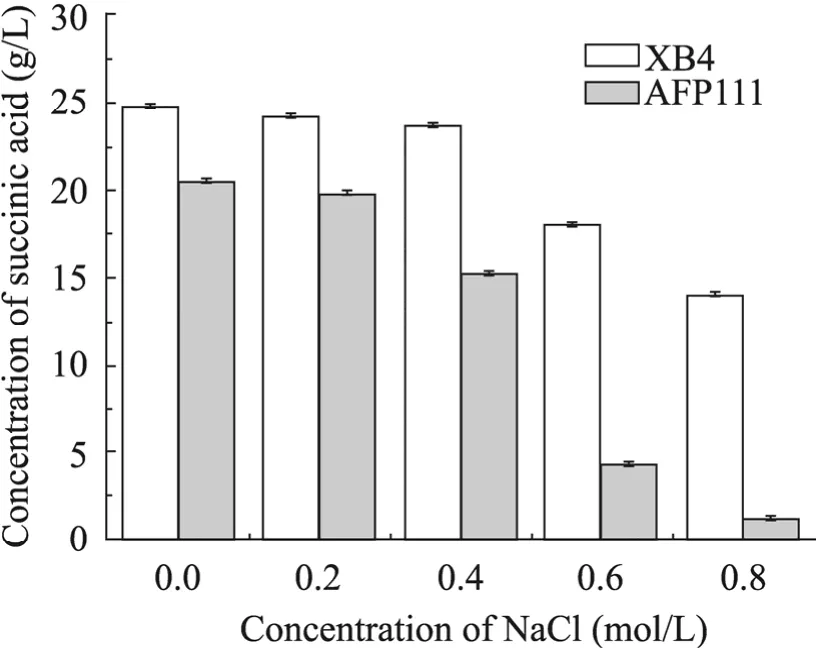
图3 突变菌株XB4和出发菌株AFP111在不同NaCl浓度下产酸效果对比Fig. 3 Effect of different concentration of NaCl on succinic acid production in AFP111 compared with XB4.
2.5 突变菌株与出发菌株在发酵罐中的两阶段分批补料发酵结果对比

图4 突变株XB4两阶段发酵菌体生长以及产酸结果Fig. 4 Time course of cell growth and production of organic acids in the dual-phase fermentation by XB4.
根据两阶段摇瓶发酵实验结果,选择 XB4作为最佳耐高渗透压生产菌株,并在7 L发酵罐中对XB4进行两阶段发酵,发酵结果如图4所示,对照菌的发酵过程曲线如图5所示。在菌体厌氧发酵阶段,琥珀酸在发酵液中积累,需要大量的 Na2CO3中和产生的有机酸,从而导致Na+大量积累,产生较高的渗透压,对菌体的活力以及产酸造成不良的影响。而突变株 XB4由于对由Na+产生的高渗透压具有很高的耐受性,因此在相同的条件下突变株 XB4发酵性能比出发菌株AFP111有了很大的提高。由表2可知,突变株XB4在两阶段培养条件下,厌氧发酵60 h,最终的琥珀酸产量达到了69.5 g/L,琥珀酸生产速率达到了 1.18 g/(L·h),分别比出发菌株AFP111提高了18.6%和20%。
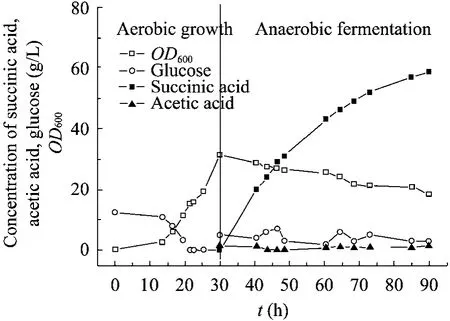
图5 E. coli AFP111两阶段发酵菌体生长以及产酸结果Fig. 5 Time course of cell growth and production of organic acids in the dual-phase fermentation by E.coli AFP11.

表2 大肠杆菌AFP111与突变株XB4两阶段发酵产琥珀酸结果对比Table 2 Succinic acid production in AFP111 compared with XB4 during dual-phase fermentation.
3 结论
本实验利用进化代谢育种方法,通过在高渗透压条件下对菌体进行连续培养,在高浓度NaCl梯度平板对突变菌株进行初筛,然后厌氧血清瓶发酵复筛,最终选到一株能够耐受 0.8 mol/L NaCl的突变株XB4。在以NaOH和Na2CO3为酸中和剂的两阶段发酵中,该突变株厌氧60 h产琥珀酸 69.5 g/L,琥珀酸生产速率达到了1.81 g/(L·h),分别比出发菌株 AFP111提高了18.6%和20%。
REFERENCES
[1] Willke T, Cukalovic KD. Industrial bioconversion of renewable resources as an alternative to conventional chemistry. Appl Microbiol Biotechnol, 2002, 66(2): 131−142.
[2] Cukalovic A, Stevens CV. Feasibility of production methods for succinic acid derivatives: a marriage of renewable resources and chemical technology. Biofuels Bioprod Biorefin, 2008, 2(6): 505−529.
[3] Song H, Lee SY. Production of succinic acid by bacterial fermentation. Enzyme Microb Technol, 2006, 39(3): 352−361.
[4] Strobel HJ, Russell JB. Role of sodium in the growth of a ruminal selenomonad. Appl Environ Microbiol, 1991, 57(6): 1663−1668.
[5] Datta R. Process for the production of succinic acid by anaerobic fermentation: US, 5143833. 1992-09-01.
[6] Hilpert W, Schink B, Dimroth P. Life by a new decarboxylation-dependent energy conservation mechanism with Na+as coupling ion. EMBO J, 1984, 3(8): 1665−1680.
[7] Record MT Jr, Courtenay ES, Cayley DS, et al. Responses of E. coli to osmotic stress: large changes in amounts of cytoplasmic solutes and water. Trends Biochem Sci, 1998, 23(4): 143–148.
[8] Padan E, Venturi M, Gerchman Y, et al. Na+/H+antiporters. Biochim Biophys Acta, 2001, 1505(1): 144−157.
[9] Gunasekera TS, Csonka LN, Paliy O. Genome-Wide transcriptional responses of Escherichia coli K-12 to continuous osmotic and heat stresses. J Bacteriol, 2008, 190(10): 3712−3720.
[10] Mika JT, Bogaart GVD, Veenhoff L, et al. Molecular sieving properties of the cytoplasm of Escherichia coli and consequences of osmotic stress. Mol Microbiol, 2010, 77(1): 200−207.
[11] Padan E, Krulwich TA. Sodium Stress. Washington, DC: ASM Press, 2000: 117−130.
[12] Kunte HJ, Crane RA, Culham DE, et al. Protein proQ influences osmotic activation of compatible solute transporter proP in Escherichia coli K-12. J Bacteriol, 1999, 181(5): 1537−1543.
[13] Lee PC, Lee WG, Lee SY, et al. Effect of medium components on the growth of Anaerobiospirillum succiniciproducens and succinic acid production. Process Biochem, 1999, 35(1/2): 49−55.
[14] Fang XJ, Li J, Zheng XY, et al. Enhancement of succinic acid production by osmotic-tolerant mutant strain of Actinobacillus succinogenes. World J Microbiol Biotechnol, 2011, 27(12): 3009−3013.
[15] Xu M, Zheng P, Ni Y, et al. Breeding of Na+-tolerant mutants of Actinobacillus succinogenes. Ind Microbiol, 2008, 38(5): 7−11.徐敏, 郑璞, 倪晔, 等. 琥珀酸放线杆菌的耐高浓度钠离子菌株选育. 工业微生物, 2008, 38(5): 7−11.
[16] Lee JW, Kim TY, Jang YS, et al. Systems metabolic engineering for chemicals and materials. Trends Biotechnol, 2011, 29(8): 370−378.
[17] Portnoy VA, Bezdan D, Zengler K. Adaptive laboratory evolution—harnessing the power of biology for metabolic engineering. Curr Opin Biotechnol, 2011, 22(4): 590−594.
[18] Conrad TM, Lewis NE, Palsson B. Microbial laboratory evolution in the era of genome-scale science. Mol Syst Biol, 2011, 7(509): 1−11.
[19] Charusanti P, Fong NL, Nagarajan H, et al. Exploiting adaptive laboratory evolution of Streptomyces clavuligerus for antibiotic discovery and overproduction. PloS ONE, 2012, 7(3): e33727.
[20] Unrean P, Srienc F. Metabolic networks evolve towards states of maximum entropy production. Metab Eng, 2011, 13(6): 666-673.
[21] Ye GZ, Li J, Xi YL, et al. Isolation of NH4+-tolerant mutants of Actinobacillus succinogenes for succinic acid production by continuous selection. J Microbiol Biotechnol, 2010, 20(8): 1219−1225.
[22] Çakar ZP, Turanlı-Yildiz B, Alkım C, et al. Evolutionary engineering of Saccharomyces cerevisiae for improved industrially important properties. FEMS Yeast Res, 2012, 12(2): 171−182.
[23] Jiang L, Li S, Hu Y, et al. Adaptive evolution for fast growth on glucose and the effects on the regulation of glucose transport system in Clostridium tyrobutyricum. Biotechnol Bioeng, 2012, 109(3): 708-718.
[24] Jantama K, Haupt MJ, Svoronos SA, et al. Combining metabolic engineering and metabolic evolution to develop nonrecombinant strains of Escherichia coli C that produce succinate and malate. Biotechnol Bioeng, 2008, 99(5): 1140−1153.
[25] Chen K, Iverson AG, Garza EA, et al. Metabolic evolution of non-transgenic Escherichia coli SZ420 for enhanced homoethanol fermentation from xylose. Biotechnol Lett, 2010, 32(1): 87−96.
[26] Cadière A, Ortiz-Julien A, C Camarasa, et al. Evolutionary engineered Saccharomyces cerevisiae wine yeast strains with increased in vivo flux through the pentose phosphate pathway. Metab Eng, 2011, 13(3): 263−271.
[27] Cai Z, Zhang B, Li Y. Engineering Saccharomyces cerevisiae for efficient anaerobic xylose fermentation: reflections and perspectives. Biotechnol J, 2012, 7(1): 34−46.
[28] Zhang X, Jantama K, Moore JC, et al. Metabolic evolution of energy-conserving pathways for succinate production in Escherichia coli. Proc Natl Acad Sci USA, 2009, 106(48): 20180−20185.
[29] Hong KK, Vongsangnak W, Vemuri GN, et al. Unravelling evolutionary strategies of yeast for improving galactose utilization through integrated systems level analysis. Proc Natl Acad Sci USA, 2011, 108(29): 12179−12184.
[30] Xu B, Jiang M, Ma JF, et al. Reuse of recombinant Escherichia coli to produce succinic acid by bioconversion. Chin J Biotech, 2010, 26(11): 1526−1531.徐冰, 姜岷, 马江锋, 等. 循环利用重组大肠杆菌细胞转化合成丁二酸. 生物工程学报, 2010, 26(11): 1526−1531.
[31] Jiang M, Liu SW, Ma JF, et al. Effect of growth phase feeding strategies on succinate production by metabolically engineered Escherichia coli. Appl Environ Microbiol, 2010, 76(4): 1298−1300.
[32] Ye GZ, Jiang M, Chen KQ, et al. Breeding of ammonium-tolerant mutants of Actinobacillus succinogenes for succinic acid production and effect of ammonium. Chin J Biotech, 2010, 26(2): 183−188.叶贵子, 姜岷, 陈可泉, 等. 耐铵型产琥珀酸放线杆菌的选育及铵离子对其生长代谢的影响. 生物工程学报, 2010, 26(2): 183−188.
[33] Balderas-Hernández VE, Hernández-Montalvo V, Bolívar F, et al. Adaptive evolution of Escherichia coli inactivated in the phosphotransferase system operon improves co-utilization of xylose and glucose under anaerobic conditions. Appl Biochem Biotechnol, 2011, 163(4): 485−496.