HLA-A*2402多肽复合物的两种构象——对Wolynes理论的补充
2012-02-09刘传省施一高福
刘传省,施一,高福
1 中国科学院微生物研究所 中国科学院病原微生物与免疫学重点实验室,北京 100101
2 中国科学院研究生院,北京 100049
在合适的条件下,蛋白质的一级结构包含了三级结构的所有信息,通常不必借助外力而自行折叠成有生物活性的状态[1]。至于蛋白质如何从一级结构中自发且准确地折叠成正确的状态,有很多观点。其中Wolynes及其同事提出的能量通道理论[2-3]获得了较为广泛认同。他认为,蛋白质的折叠沿能量减少的方向进行,直至一个最低的能量状态,也即有生物活性的状态。然而在达到最低能量状态的过程中,可能遇到局部最低的能量状态。因为这些局部最低的能量状态可以使蛋白质处于一个相对稳定但又不具生物活性状态,它们又被称为能量陷阱。这是有事实依据的,很多生物体内蛋白不正确折叠所导致的疾病[4-7]就是这一观点的佐证。显然这些不正确的折叠面临着巨大的选择压力。Wolynes认为自然选择会剔除蛋白折叠中的能量陷阱,使蛋白朝着有生物活性的稳定状态折叠[8-9]。后来的研究也证明了这一点[10]。于是又产生了另一个问题:会不会有一些不影响蛋白功能的能量陷阱在自然选择中保留下来?为了回答上述问题,本文对多肽HLA-A*2402复合物 (pHLA) 与T细胞受体 (TCR) 和辅助受体CD8αα的相互作用进行了研究。
pHLA在免疫系统中扮演着重要角色,是TCR的配体[11-15],并且可以和CD8αα结合[16-17]。由于和免疫识别密切相关,pHLA每一点结构的变化都会面临巨大的选择压力,因而是用于研究与自然选择相关的课题的好材料。最早用于研究的pHLA从细胞表面酶切获得[18],自复性方法获得有活性pHLA-A2后[19],酶切方法被摒弃。复性后的 HLA-A*2402多肽复合体 (pHLA) 可以与 TCR结合,通过对结合力的比较,可以在一定程度上反映天然状态下HLA-A*2402的活性。本文通过研究复性所得 pHLA-A*2402,发现它存在两种不同构象。进一步研究了这两种不同构象的生物活性,发现它们并无差别。由此给出结论:如果在不同的能量陷阱下折叠成的蛋白构象在活性上没有差别,那么它们就不会被自然选择区分,也就有可能同时被保留下来。
1 材料与方法
1.1 材料
质粒:HLA-A*2402重组质粒[20],TCRδ链重组pET-21a质粒,TCRβ链重组pET-21a质粒,CD8αα重组质粒[21]均由本实验室保存。
多肽:RYPLYFGWCF 由中科亚光生物技术有限公司合成。
菌株:大肠杆菌BL21(DE3) 由本实验室保存。
1.2 方法
1.2.1 包涵体的提取
转化有pET28a-HLA-A*2402质粒的大肠杆菌BL21过夜培养后,接种20 mL于2 L LB培养基中,37 ℃培养至OD600=0.4~0.6,加入IPTG至终浓度为1 mol/L,37 ℃继续培养3~5 h。按常规收菌 (接下去步骤都在低温下进行),用 60 mL左右1×PBS悬浮细菌,超声裂解 (超声6 s,间隔12 s,99次,300 W)。12 000 r/min离心30 min,用玻棒将细菌碎片拨掉,弃上清。用洗涤缓冲液(50 mmol/L Tris (pH 8.0),300 mmol/L NaCl,10 mmol/L EDTA,10 mmol/L DTT,0.5% Triton-100) 充分洗涤包涵体3次,12 000 r/min离心30 min,弃上清。用重悬缓冲液 (50 mmol/L Tris (pH 8.0),100 mmol/L NaCl,10 mmol/L EDTA,10 mmol/L DTT) 悬浮包涵体,12 000 r/min离心10~20 min,弃上清。按30 mg/mL将包涵体溶解在溶解缓冲液 (6 mol/L Gua-HCl,10% glycerol 50 mmol/L,Tris (pH 8.0),100 mmol/L NaCl,10 mmol/L EDTA,10 mmol/L DTT),4 ℃搅拌溶解,于-20℃保存备用。
1.2.2 pHLA-A*2402复性
用 5 mL的注射器向 2 L复性缓冲液(100 mmol/L Tris (pH 8.0), 400 mmol/L L-Arg HCl, 2 mmol/L EDTA, 5 mmol/L GSH,0.5 mmol/L GSSG) 中滴加β2m包涵体3 mL,复性8 h。取10 mg多肽,用200 μL DMSO溶解,并迅速注入复性缓冲液中,待完全溶解后,用5 mL的注射器向2 L复性缓冲液中滴加HLA-A24重链包涵体,每次3 mL,每隔约8 h一次,共3次。浓缩,换成20 mmol/L Tris,50 mmol/L NaCl (pH 8.0)缓冲液。将浓缩的蛋白用Hiload Superdex 200层析柱分离。收集复性的复合体蛋白,用Resource Q层析柱进一步纯化。
1.2.3 TCR复性
TCR复性采用透析复性的方法,将约60 mg的δ链,60 mg的β链包涵体加入到300 mL复性液 (400 mmol/L Arg-HCl,100 mmol/L Tris,2 mmol/L EDTA,2 mol/L Urea,调pH到8.5,5 mmol/L GSH,0.5 mmol/L GSSG),混匀,孵育过夜。然后,放入800 mL的水中,透析24 h。再放入4 L 10 mmol/L Tris (pH 8.5)溶液中透析24 h。浓缩到约40 mL,过Source 15Q层析柱。
1.2.4 表面等离子共振分析 (SPR)
将纯化好的pHLA-A*2402溶液换成HEPES缓冲液,并浓缩至150 μmol/L,梯度稀释成75、37.5、18.8、9.38 μmol/L。取冻存的CD8αα (制备方法见参考文献[21]),按上述方法稀释,浓度为7.5、3.75、1.88、0.94 μmol/L。将纯化的TCR换成HEPES缓冲液,浓度调至20 mg/mL,pH调至4.5。选用CM5芯片,固定TCR,进行SPR操作[22-23]。
2 结果与分析
2.1 pHLA-A*2402复合体存在两种不同的构象
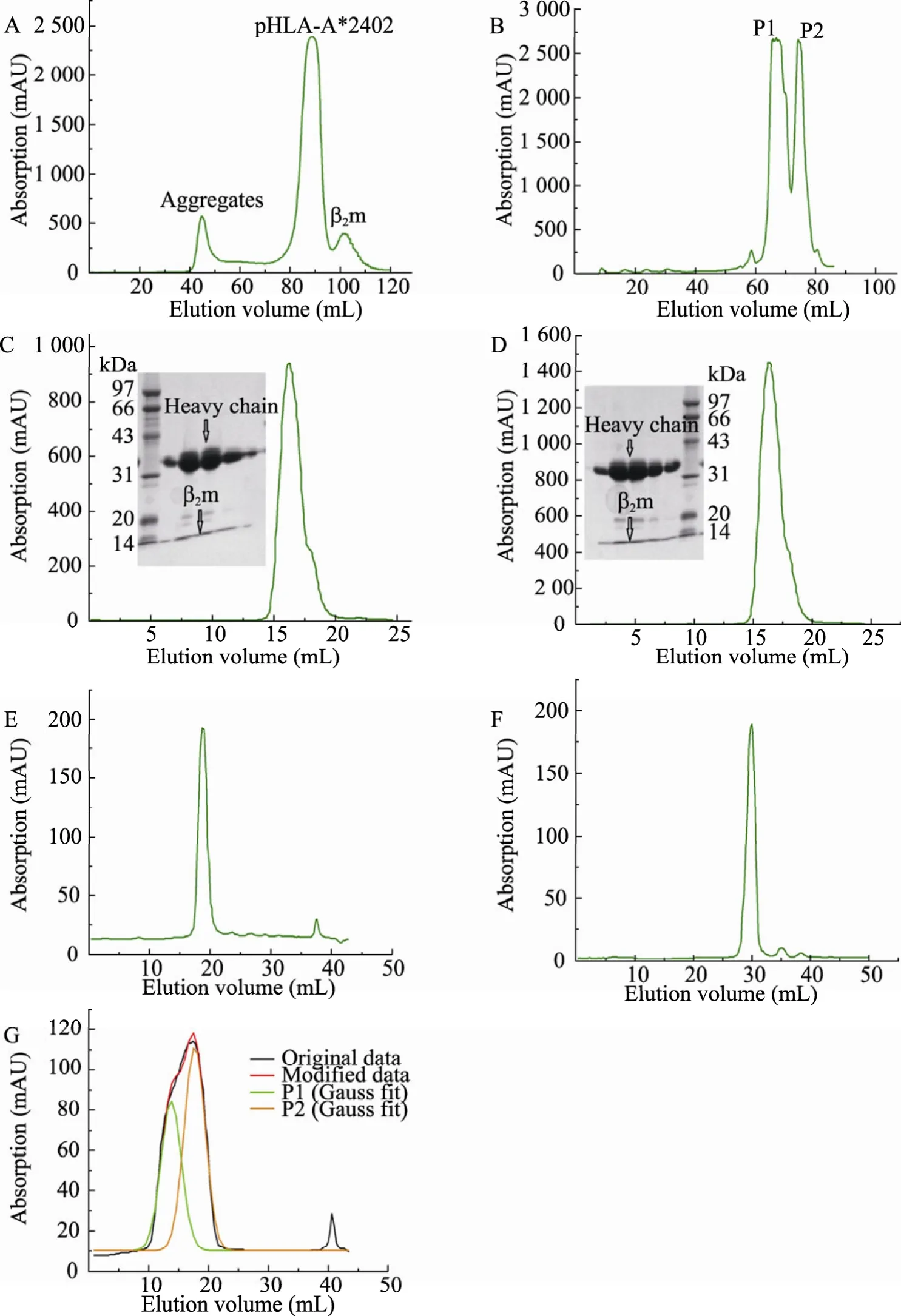
图1 pHLA-A*2402复合体的分离及电荷特征Fig. 1 Charts and pictures of pHLA-A2402. (A) Gel filtration of pHLA-A*2402. (B) Ion exchange of pHLA-A*2402 on column Resource Q, two peaks were eluded as P1 and P2. (C) Gel filtration of P1 and gel electrophoresis. (D) Gel filtration of P2 and gel electrophoresis. (E) Ion exchange of P1 after two months’ storage in 4 ℃. (F) Ion exchange of P2 after two months’ storage in 4 ℃. (G) Ion exchang of the mixture of P1 and P2 after two months storage and data fitting.
HLA-A*2402与β2m在多肽存在的情况下共复性,形成稳定的复合体。从图 1A看出,pHLA-A*2402复性效果很好,聚体峰和β2m峰都很小,说明绝大部分HLA-A*2402重链和β2m都参与形成了复合体。收集分离的复合体蛋白,进行离子交换层析,此时可以观察到,有两个独立的出峰位置。离子交换层析是利用在一定条件下蛋白表面所带电荷与层析柱中带相反电荷的离子交换试剂相互作用而对蛋白进行分离的一种方法。在相同溶液中,由于等电点不同蛋白所带电荷不同,与层析柱的亲和力不同,它们会被不同浓度的盐溶液洗脱下来。因此,一个均一的蛋白只有一个出峰位置。然而,从图1B看出,pHLA-A*2402复合体分成两个主峰。由此推断pHLA-A*2402可能存在两个等电点,也必然对应两种不同构象,分别标记为 P1、P2。将P1、P2两个组分的蛋白再次进行分子筛层析,两个组分出峰位置相同,表明分子量大小相同。SDS-PAGE显示两者均是HLA-A*2402,重链和β2m以 1∶1比例形成的复合体,分子量大小无差别 (图1C和图1D)。
收集到的P1、P2组分于4 ℃放置2个月后,再次进行离子交换层析,发现它们的出峰位置没有改变,表明它们的电荷特征没有改变 (图 1E和图1F)。从图1G中对P1和P2的混合峰的曲线拟合可以看出两者仍然为独立的两个峰,说明构象差异稳定存在。
2.2 TCR的复性与纯化
TCR δ链和β链在大肠杆菌中均以包涵体表达,通过透析的方法复性,共复性的蛋白通过离子交换法分离,出现一个主峰 (图2)。分离的蛋白在非还原情况下,由于δ链和β链之间二硫键的存在,使得电泳显示为一条带。在还原剂(DTT) 存在的情况下,二硫键被打断,电泳结果呈清晰的两条带,下面一条是δ链,上面一条是β链。以上结果表明TCR已经成功复性。
2.3 两种不同构象的pHLA-A*2402复合体与TCR和CD8具有相同的亲和力
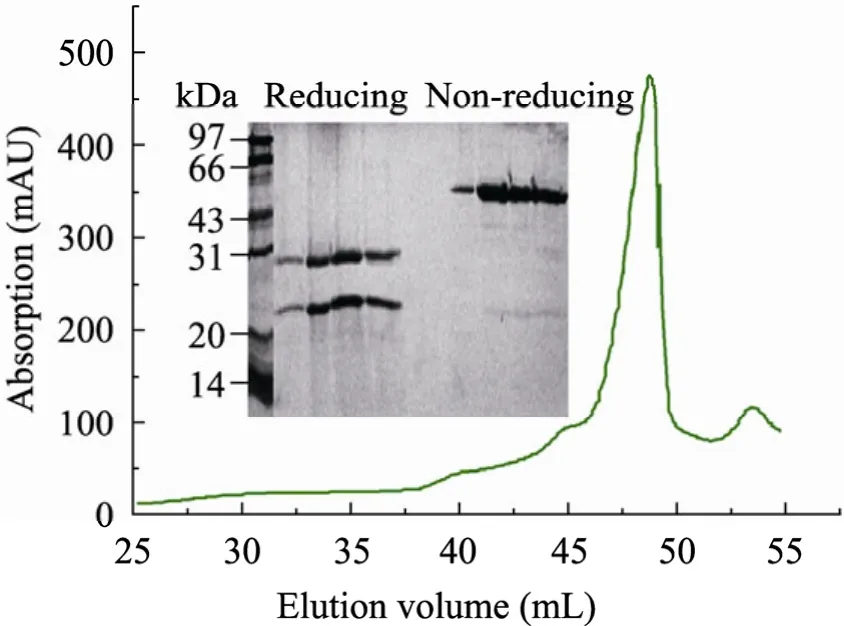
图2 复性TCR离子交换分离及鉴定Fig. 2 Ion exchange of refolded TCR, non-reducing electrophoresis and reducing electrophoresis
在T细胞与靶细胞的相互识别中,pHLA复合体与TCR特异的相互作用是关键的一步。在这个过程中,CD8作为辅助受体参与作用,并与pHLA复合体和TCR结合。因此,当观察到pHLA-A*2402复合体存在两种构象时,首先要回答的是,这两种构象的蛋白是否都具有与TCR和 CD8结合的功能?因此,本文利用表面等离子共振技术 (SPR) 检测了这两种构象的pHLA-A*2402复合体与TCR和CD8的亲和力。图3A和图3B的结果表明P1和P2与TCR的相互作用基本没有变化。P1的解离速率 kd为0.01891/s,P2为0.01691/s,相差10%左右,结合速率ka和解离常数KD也都在同一个数量级之内。图3C和图3D的结果表明P1和 P2在与CD8αα的相互作用基本没有变化,它们与CD8αα的结合速率 ka、解离速率 kd和解离常数 KD都在同一个数量级内。鉴于蛋白浓度测定的误差以及仪器本身所允许的误差范围,以上这些在同一数量级内的差异都可以视作无差异。同时从表1看出,所有的Chi2的值小于Rmax/10,说明用于计算动力学参数的 Langmuir模型是可信的。
综合以上结果,pHLA-A*2402复性后至少存在两种不同的稳定状态,这两种状态和 TCR及CD8αα相互作用时没有差别。
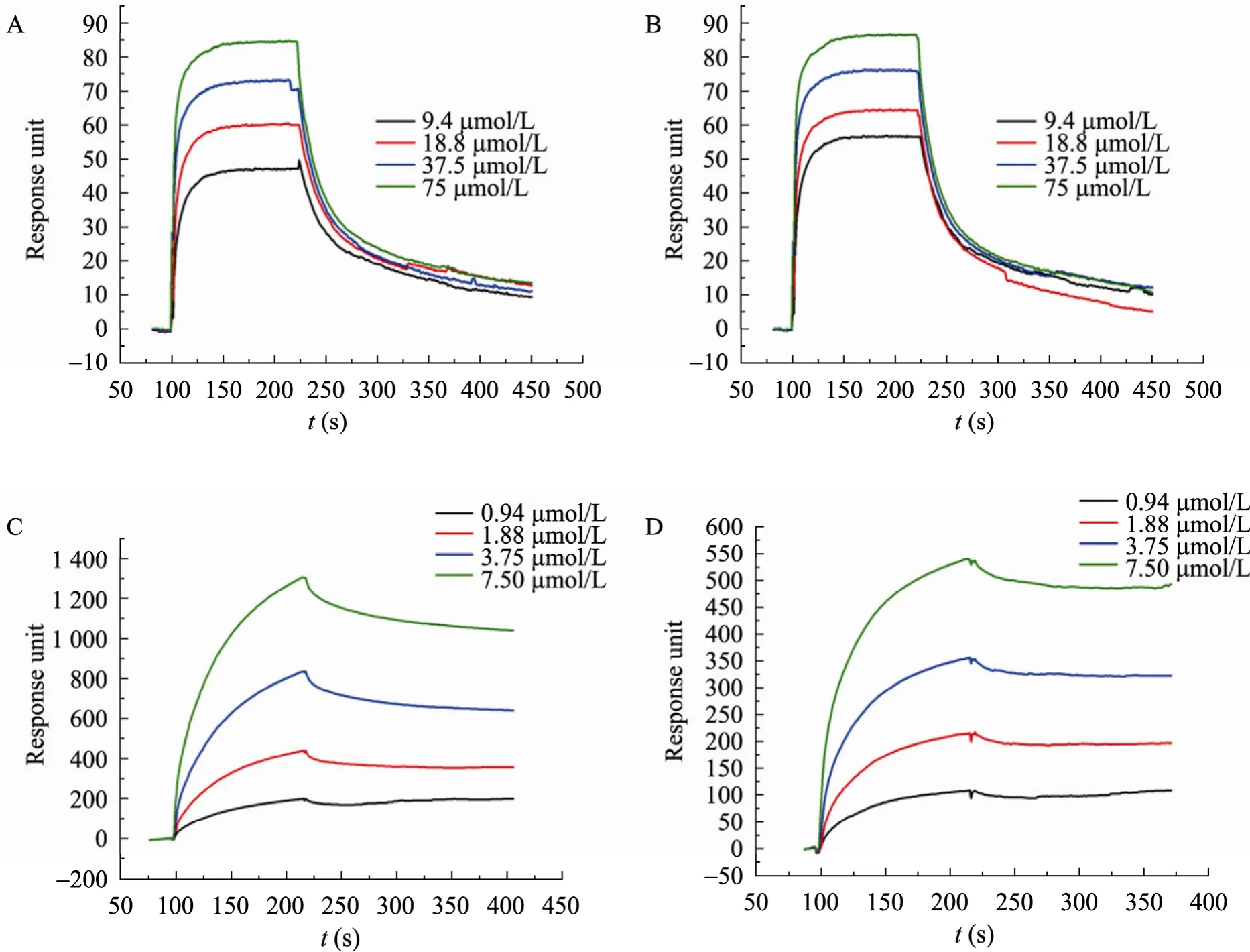
图3 两种不同构象的pHLA-A*2402与TCR和CD8αα的相互作用Fig. 3 Charts of BIAcoreÒ. (A) Interaction between P1 and TCR. (B) Interaction between P2 and TCR. (C) Interaction between P1 and CD8αα. (D) Interaction between P1 and CD8αα.
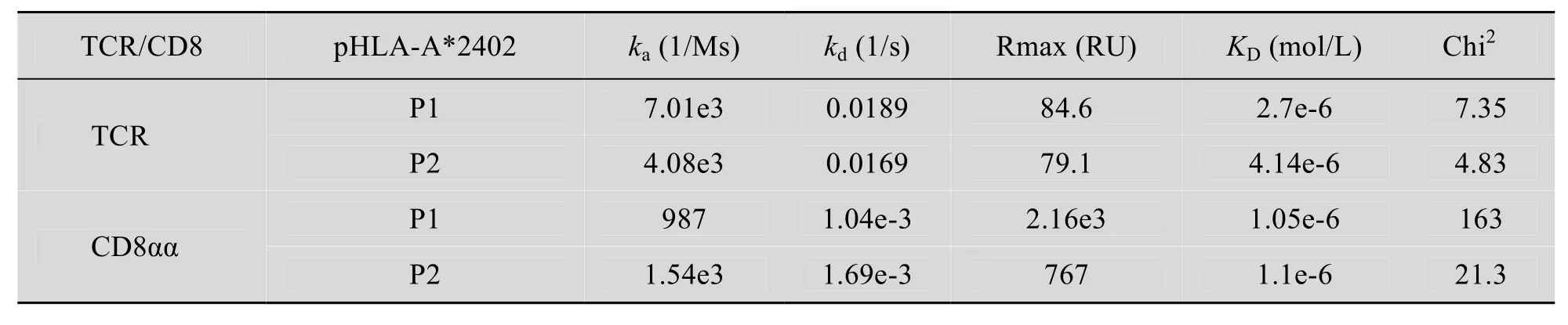
表1 两种不同构象的pHLA-A*2402与TCR和CD8αα亲和力Table 1 The affinity between the two conformations of pHLA-A*2402 and TCR, CD8αα
3 讨论
根据蛋白折叠的基本理论,复性后可以稳定存在的蛋白结构基本上反映了蛋白在生理状态的结构,所以用复性得到的蛋白来研究生理状态的蛋白基本上可靠[24]。这是本文的前提,也是所有以复性蛋白为材料进行研究的前提。
多数情况下相同蛋白的不同结构往往具有不同的功能[25],本文首次从复性的角度证明了同一蛋白在不同状态具有相同的功能。可以用能量陷阱和自然选择理论对上述实验结果进行一个合理的解释。当HLA进行折叠时,同样面临着能量陷阱,这就使得HLA具有两种不同的构象,然而由于它们在生物活性上并没有差别,自然选择无法对它们进行区分,于是就在进化中保留下来。虽然本文只是对HLA的折叠进行的研究,但是这个情形可以类似地推广到所有的蛋白质上。当蛋白质沿着能量降低的方向进行折叠时,有很多卡在能量陷阱之中,形成不具有生物活性的折叠,由于自然选择的压力,这些蛋白质的一级结构不断优化,直到这些能量陷阱从蛋白质折叠的过程中全部剔除。可是当掉入能量陷阱的蛋白质和“正确”折叠的蛋白质在生物活性上不具有差别时,它们完全可以逃脱自然选择的压力而得以保存。这就出现了同一个蛋白具有多个稳定构象,而这些构象又具有相同的功能。
上面的结论在对能量通道理论佐证和补充的同时,也为解析蛋白的结构提出了一些新的推测。当一个蛋白不能正常结晶,或者结晶后有部分区域的解析度不够高的时候,也许并不是因为这个蛋白的结构具有柔性,而是由于它可能同时具有多个相对稳定的状态。
REFERENCES
[1] Haber E, Anfinsen CB. Regeneration of enzyme activity by air oxidation of reduced subtilisin-modified ribonuclease. J Biol Chem, 1961, 236: 422–424.
[2] Bryngelson JD, Wolynes G. Spin glasses and the statistical mechanics of protein folding. Proc Natl Acad Sci USA, 1987, 84(21): 7524–7528.
[3] Frauenfelder H, Sligar SG, Wolynes PG. The energy landscapes and motions of proteins. Science, 1991, 254(5038): 1598–1603.
[4] Selkoe DJ. Folding proteins in fatal ways. Nature, 2003, 426(6968): 900–904.
[5] Dobson CM. Protein folding and its links with human disease. Biochem Soc Symp, 2001, 68: 1–26.
[6] Sherman MY, Goldberg AL. Cellular defenses against unfolded proteins: a cell biologist thinks about neurodegenerative diseases. Neuron, 2001, 29(1): 15–32.
[7] Sukhanova A, Poly S, Shemetov A, et al. Implications of protein structure instability: from physiological to pathological secondary structure. Biopolymers, 2012, 97(8): 577–588.
[8] Goldstein RA, Luthey-Schulten ZA, Wolynes G. Optimal protein-folding codes from spin-glass theory. Proc Natl Acad Sci USA, 1992, 89(11): 4918–4922.
[9] Goldstein RA, Luthey-Schulten ZA, Wolynes PG. Protein tertiary structure recognition using optimized Hamiltonians with local interactions. Proc Natl Acad Sci USA, 1992, 89(19): 9029–9033.
[10] Mirny LA, Abkevich VI, Shakhnovich EI. How evolution makes proteins fold quickly. Proc Natl Acad Sci USA, 1998, 95(9): 4976–4981.
[11] Bjorkman PJ. MHC restriction in three dimensions: a view of T cell receptor/ligand interactions. Cell, 1997, 89(2): 167–170.
[12] Rudolph MG, Stanfield RL, Wilson IA. How TCRs bind MHCs, peptides, and coreceptors. Annu Rev Immunol, 2006, 24(1): 419–466.
[13] Garcia KC, Adams EJ. How the T cell receptor sees antigen-a structural view. Cell, 2005, 122(3): 333–336.
[14] Garcia KC, Degano M, Robyn L, et al. An alphabeta T cell receptor structure at 2.5 A and its orientation in the TCR-MHC complex. Science, 1996, 274(5285): 209–219.
[15] Gakamsky DM, Luescher IF, Pecht I. T cell receptor-ligand interactions: a conformational preequilibrium or an induced fit. Proc Natl Acad Sci USA, 2004, 101(24): 9063–9066.
[16] Gao GF, Tormo J, Gerth UC, et al. Crystal structure of the complex between human CD8alpha (alpha) and HLA-A2. Nature, 1997, 387(6633): 630–634.
[17] Parel Y, Chizzolini C. CD4+ CD8+ double positive (DP) T cells in health and disease. Autoimmun Rev, 2004, 3(3): 215–220.
[18] Madden DR, Gorga JC, Strominger JL, et al. The three-dimensional structure of HLA-B27 at 2.1 Å resolution suggests a general mechanism for tight peptide binding to MHC. Cell, 1992, 70(6): 1035–1048.
[19] Garboczi DN, Ghosh P, Utz U, et al. Structure of the complex between human T-cell receptor, viral peptide and HLA-A2. Nature, 1996, 384(6605): 134–141.
[20] Shi Y, Qi J X. Plasticity of human CD8αα binding to peptide–HLA-A*2402. Molec Immunol, 2011, 48(15/16): 2198–2202.
[21] Zong L, Chen Y, Yan J, et al. Expression, purification, crystallization and preliminary X-ray diffraction analysis of rhesus macaque CD8αα homodimer. Acta Crystallogr Sect F Struct Biol Cryst Commun, 2010, 66(Pt4): 435–438.
[22] Pattnaik P. Surface plasmon resonance: applications in understanding receptor-ligand interaction. Appl Bioch Biotechnol, 2005, 126(2): 79–92.
[23] Healthcare GE. Interaction analysis product guide (Biacore systems and consumables) [EB/OL]. [2012-5-10]. https://www.gelifesciences.com/ gehcls_images/GELS/Related%20Content/Files/13 14774443672/litdoc28950947AA_2011083110404 7.pdf.
[24] Yon JM. Protein folding: concepts and perspectives. Cell Mol Life Sci, 1997, 53(7): 557–567.
[25] Parak FG. Proteins in action: the physics of structural fluctuations and conformational changes. Curr Opin Struct Biol, 2003, 13(5): 552–557.