基于光激活定位显微镜的膜蛋白鉴定技术
2012-02-09黄丽方维焕俞盈宋厚辉
黄丽,方维焕,,俞盈,宋厚辉
1 浙江农林大学动物科技学院,浙江 临安 311300
2 浙江大学动物预防医学研究所 浙江省动物预防医学重点实验室,浙江 杭州 310058
光激活定位显微成像技术 (Photo-activated localization microscopy,PALM) 是最近几年来才逐渐发展起来的、可以在活体水平进行的、且分辨率达到纳米级别、并优于激光共聚焦和双光子显微镜的新一代三维成像技术[1-2]。该技术的原理是采用荧光基团修饰技术,将各种常见的荧光蛋白进行突变,突变后的蛋白在光束下 (一般为405 nm激光或者紫外) 被活化,并释放出光子信号,然后利用光激活定位显微镜进行捕获和识别,最终可以捕捉到单分子信号,分辨率达到5~20 nm或者更高[2],其中分辨率的高低取决于荧光蛋白的特性。目前可以被光子激活(Photoactivatable,PA) 的荧光蛋白有:PAGFP[3]、 PAmCherry[4]、mEos2[5]、PATagRFP[6]等。如果这些蛋白不被405 nm激光激活,在普通荧光显微镜下不呈现任何荧光 (mEos2除外),在光激活定位显微镜下也不释放任何信号。但是这些荧光蛋白一旦被激活,PAGFP[3]、PAmCherry[4]、PATagRFP[6]等在普通显微镜下将呈现荧光,例如:PAGFP被激活后,在荧光显微镜下为绿色荧光。但对于在普通荧光显微镜下呈现绿色荧光的mEos2,被405 nm激光激活后,将表现出红色荧光 (即出现光转换现象,Photoconvertable),随着激发时间延长,绿色荧光越来越弱,而红色荧光越来越强,且释放出光子。
病原菌膜蛋白作为潜在的药物靶位、细胞受体和纳米通道越来越受到研究者的关注[7-9]。对于未知功能的膜蛋白,在研究的初始阶段仍然需要依赖生物信息学软件,通过已公布的基因组学信息,对蛋白结构和功能进行预测,进而通过实验设计进行验证,包括:膜蛋白表达、纯化、鉴定、分离和定位研究等[10]。其中,对细菌膜蛋白定位的研究,目前已经公布的方法有蛋白酶K实验和全细胞 ELISA实验[10-11]。这两种方法的原理是:如果膜蛋白暴露在细胞外部,将会被蛋白酶K裂解或者被相应的抗体识别。但是这两种方法共有的缺陷是:如果膜蛋白具有蛋白酶抗性;或者被多糖、脂类等包裹,利用蛋白酶K实验和全细胞ELISA鉴定膜蛋白都是无效的[11]。因此,如何设计一种精确的实验对细菌膜蛋白的定位进行直接观察,一直是困扰研究者的难题。
结核分枝杆菌 (Mycobacterium tuberculosis,Mtb) 是引起肺结核 (Tuberculosis, TB) 的致病菌,目前全球大约有1/3人口为结核阳性。根据WHO最新报告,2010年全球大约有145万人口死于TB[12]。由于Mtb可以通过空气传播,且需要严格的生物安全三级实验室才能进行病原学研究,为了加快Mtb蛋白组学的研究进展,研究人员往往采取非致病性的耻垢分枝杆菌(Mycobacterium smegmatis, Msmeg) 或者卡介苗分枝杆菌(M. bovis BCG) 作为模式菌,通过在非致病菌中表达Mtb蛋白,来间接研究Mtb蛋白功能[10,13-14]。
为了能够精确地对 Mtb膜蛋白的定位情况进行原位追踪,本研究采用无致病性的 Msmeg作为模式菌,以结核分枝杆菌外膜蛋白OmpA作为参考蛋白,应用光激活蛋白mEos2和光激活定位显微成像技术研究膜蛋白的定位。这是目前为止采用光激活定位显微成像技术研究膜蛋白定位的首次报道。
1 材料与方法
1.1 材料
1.1.1 化学试剂、菌株和质粒
本实验室所用的化学试剂均为分析纯,购自Sigma、USP和BD等公司。耻垢分枝杆菌Mc2155为本实验室保存。大肠杆菌克隆用菌株为DH5α。
1.1.2 细菌培养
大肠杆菌培养基为LB培养基,37 ℃培养。耻垢分枝杆菌培养基为 7H9培养基 (液体) 和7H10培养基 (固体),37 ℃培养。筛选用抗生素浓度:Hygromycin (潮霉素,50~200 mg/L)。
1.2 蛋白酶K实验
蛋白酶 K实验采用本实验室已报道的方法进行[10]。即:将pML003 (表达结核分枝杆菌外膜蛋白 OmpA) 质粒[10]转化入能够表达内膜蛋白PhoA (碱性磷酸酶) 的耻垢分枝杆菌ML1301中[15],裂解或者不裂解细菌,用蛋白酶 K处理后,分别用对应的抗体通过Western blotting检测OmpA (结核分枝杆菌外膜蛋白A)、PhoA (内膜蛋白,碱性磷酸酶)、MspA[16](耻垢分枝杆菌外膜蛋白,porin) 和RNA聚合酶 (RNAP,胞内蛋白) 的完整性。
1.3 mEos2和OmpA的融合表达
1.3.1 mEos2的基因修饰
由于分枝杆菌为高GC含量细菌,外源基因一般很难在其内进行表达。为此,我们将mEos2蛋白按照耻垢分枝杆菌密码子进行优化,在 3¢端加入了HA标签 (由GenScript公司进行合成),并命名为mEos2m。为了便于克隆,在mEos2m基因5¢和3¢端分别加入了PacⅠ和Hind Ⅲ酶切位点,直接克隆入pUC57载体中,命名为pUC57-mEos2m (由GenScript公司直接提供)。
1.3.2 mEos2m表达载体以及 mEos2m-OmpA融合表达载体的构建
mEos2m表达载体的构建:通过PacⅠ/Hind Ⅲ双酶切,将mEos2m片段从pUC57-mEos2m中切下,连接到pML003载体[10](含有分枝杆菌强启动子psmyc) 中,得到mEos2m表达载体pSL004。
OmpA-mEos2m融合表达载体的构建:采用OmpA-up-PacⅠ/OmpA linker-rev引物对,以pML003作为模板,PCR扩增OmpA片段;然后采用linker mEos2m-fwd/Eos2m-seq2引物对,以pUC57-mEos2m为模板,PCR扩增mEos2m片段;最后采用融合 PCR技术,以 OmpA-up-PacⅠ/ Eos2m-seq2为引物,以 OmpA片段和 mEos2m片段为模板,扩增OmpA-mEos2m片段。融合片段以 PacⅠ和 Hind Ⅲ酶切位点克隆入 pML003载体[10],得到 OmpA-mEos2m融合表达载体pSL005,其中OmpA和mEos2m片段之间含有柔性接头氨基酸:GSSGSSG。引物名称和序列如表1所示。

表1 引物名称和序列Table1 Primers used in this study
1.4 分枝杆菌感受态细胞的制备和转化
耻垢分枝杆菌感受态细胞的制备和电穿孔转化采用本实验室报道的方法进行[17]。即:将分枝杆菌在 100 mL 7H9液体培养基中培养至OD600为0.5,离心收获菌体,用10%预冷的甘油将菌体洗涤3次,最后用1 mL 10%甘油悬浮菌体 (感受态细胞)。电穿孔转换参数为:1 000 Ω,2.5 kV,25 µF。电击杯宽度为4 mm。
1.5 细菌的玻片固定
对于体视显微镜,细菌菌落不需要固定,可以直接进行观察。但是对于荧光显微镜和光激活定位显微镜,样品需要经过固定后才能进行观察。固定方法如下:准备200 µL新鲜的细菌液,10 000 r/min离心1 min,收集菌体,然后用PBS (8 g NaCl,0.2 g KCl,1.44 g Na2HPO4,0.24 g KH2PO4,定容至 1 L,pH 7.4) 洗涤 2次。用200 µL PBS (含 2%多聚甲醛+0.2%戊二醛) 悬浮细菌。室温静置10 min,离心收集菌体,并用PBS洗涤2次。用200 µL PBS (含0.1%硼氢化钠,在1 min内完成配制过程,直接使用) 悬浮细菌。室温静置7 min,离心收集菌体,并用PBS洗涤 2次,然后用 200 µL PBS悬浮细菌。取100 µL滴在多聚赖氨酸处理的载玻片 (Fisher公司) 上,室温静置15 min。用PBS轻轻洗涤载玻片,洗去未被吸附的菌体,覆上盖玻片。用擦镜纸压拭盖玻片,轻轻拭去多余的液体。用凡士林封片后,直接用荧光显微镜和光激活定位显微镜进行观察。
1.6 显微成像技术
本实验用的显微镜有:Zeiss体视荧光显微镜 (Stemi2000),Zeiss正置荧光显微镜 (Axio)和PALM光激活定位显微镜 (由美国HHMI的Hess实验室搭建,详细配置见文献[1])。光激活采用405 nm LED自制光源 (针对体视荧光显微镜和正置荧光显微镜) 或者 405 nm激光 (针对光激活定位显微镜)。mEos2m蛋白光转换吸收波峰为573 nm,发射波峰为584 nm。
2 结果与分析
2.1 外膜蛋白OmpA (细胞外loop) 和MspA对蛋白酶K不敏感
外膜蛋白通常会有部分 loop区域暴露在细胞外面并与外部环境接触。如果该loop区域被蛋白酶K降解,外膜蛋白将因失去完整性而失去活性。为此,我们利用经典的蛋白酶 K 实验[11-18]验证在分枝杆菌中已经被证实的两个外膜蛋白OmpA[19]和MspA[16]的细胞外部分是否会被蛋白酶K降解,从而推断其在细胞膜上的定位情况。结果发现:虽然暴露在细胞外面的OmpA的loop区域可以被 OmpA抗体识别[19],但是裸露在细胞外部的loop并不能被蛋白酶K降解 (图1A,“whole”)。同样,对于热稳定性的八聚体外膜蛋白MspA,用蛋白酶K直接处理裂解的细胞,MspA也不会被降解 (图1D,“lysed”);这说明多聚体蛋白本身对蛋白酶K不敏感。而超声波裂解细胞中的OmpA (图1A,“lysed”),内膜蛋白PhoA (图1B,“lysed”) 和胞内蛋白RNAP (图1C,“lysed”) 对蛋白酶K都是敏感的。这说明蛋白酶 K实验并不能作为一个通用方法检测膜蛋白的表面接触性能,需要进一步开发新的鉴定膜蛋白的方法。
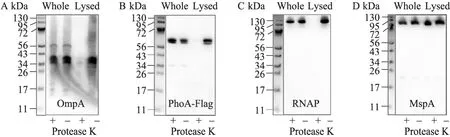
图1 蛋白酶K敏感性试验Fig. 1 Protease K sensitivity assay. M. smegmatis ML1301 (PhoA, OmpA and MspA expressing strain) whole cells (indicated with “whole”) and lysed cells (indicated with “lysed”) were treated with (indicated with “+”) or without (indicated with “-”) protease K. The reactions were terminated by adding protease K inhibitor cocktails. All samples were boiled for 10 min in the presence of protein loading buffer and separated on 10% SDS-PAGE, detected by anti-OmpA, anti-Flag, anti-RNAP and anti-MspA antibodies respectively.
2.2 光激活荧光蛋白 mEos2m和普通绿色荧光蛋白GFP在耻垢分枝杆菌中的表达
mEos2m进行了密码子优化,通过 pSL004 (mEos2m表达载体)、pSL005 (OmpA-mEos2m融合表达载体) 在耻垢分枝杆菌中进行表达。为了更直观地区别 mEos2m和普通的绿色荧光蛋白GFP的不同,我们将pMN406 (GFP) 和pML1429 (OmpA-GFP) 在耻垢分枝杆菌中进行表达。通过荧光体视显微镜 (图2),发现mEos2m及其融合蛋白在分枝杆菌中完全可以实现表达,在光激活之前 (图2,“Before PA”)呈现绿色荧光,在光激活之后呈现红色荧光 (图2,“After PA”)。而普通的GFP及其融合蛋白,在光激活之前呈现绿色荧光;在光激活后荧光不发生转换 (图2)。
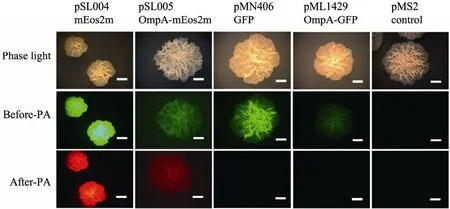
图2 光激活蛋白mEos2m和普通绿色荧光蛋白在耻垢分枝杆菌中的表达Fig. 2 Expression of photoactivatable mEos2m and regular GFP in M. smegmatis. The colonies were visualized under Zeiss fluorescent stereomicroscope before being activated under 405 nm LED light (“Before-PA”) or thereafter (“After-PA). The plasmid and corresponding proteins are indicated on top panel. pMS2, an empty plasmid without any inserts, was used as control. 16-fold magnification was employed under microscope. Scale bar=1 mm.
这说明光激活荧光蛋白 mEos2m及其融合蛋白可以在耻垢分枝杆菌中正确表达且具有光激活现象。这也是进行光激活定位显微镜操作之前的准备工作,即:开启光激活定位显微镜之前,必须先用普通的荧光体视显微镜或者正置荧光显微镜观察目的蛋白是否具有荧光且呈现光激活现象。这是因为光激活定位显微镜捕捉的是单分子信号,只有这些单分子信号达到一定数量之后,才能呈现高分辨率图像,因此需要的成像时间更长。比如:普通荧光显微镜从观察、锁定图像到拍照仅需要几分钟时间,而通过光激活定位显微镜,每个样品从上机调试到锁定图像和拍照至少需要2 h或者更长。因此,如果对样品特性不了解,直接利用PALM调试,无论在时间还是金钱上都是一种浪费。
2.3 光激活荧光蛋白 mEos2m对耻垢分枝杆菌形态的影响
mEos2m与OmpA融合表达后,融合蛋白将会被牵引到细胞膜上,此时细胞的形态可能会发生变化。为了进一步研究光激活荧光蛋白mEos2m对单个细菌形态的影响,我们在正置显微镜下对菌落形态进行了观察 (图3)。由于图2中的 GFP为普通的绿色荧光蛋白,不呈现任何光激活现象,无法进行随后的PALM分析。所以,我们仅采用mEos2m (pSL004) 和OmpA-mEos2m (pSL005) 融合蛋白表达菌株进行荧光和光激活分析。对于仅表达mEos2m的分枝杆菌,表达的mEos2m蛋白聚集于细胞内部;从形态上可以看到表达 OmpA-mEos2m融合蛋白的分枝杆菌(图3B) 比表达mEos2m单一蛋白的分枝杆菌更“胖”(图3A,3B),这一点与预期一致。
普通的荧光显微镜 (包括激光共聚焦) 对荧光信号仅能对荧光进行定性观察,不能进行光子捕捉,其最高分辨率仅能达到200 nm左右,而光激活定位显微镜的分辨率已经超过了20 nm[1]。这也是本研究采用超分辨率 PALM进行膜蛋定位研究的另一目的。
2.4 光激活定位显微镜下的膜蛋白
为了进一步验证 PALM是否可以用于膜蛋白定位研究,以及膜蛋白在分枝杆菌细胞膜上的准确定位,将含有光激活蛋白的分枝杆菌固定于多聚赖氨酸载玻片后,用405 nm激光激发,然后用PALM观察 (图4)。在PALM显微镜下,所有的荧光信号都将以光子的形式释放出来,每一个亮点代表一个单分子信号 (图4)。图像中的彩色斑点代表光子信号距离拍摄镜头的远近。按照七种色彩,红橙黄绿青蓝紫依次代表光子距离拍摄镜头由近到远。然后通过计算机软件计算出光子在物体上的分布。由于OmpA-mEos2m融合蛋白主要定位于细胞膜上,所以通过纵切面 (Y-Z轴,图4D) 可以看到所有光子信号形成一个“桶状”(Barrel-like) 或者“带状”(Belt-like) 结构,而单一的 mEos2m蛋白,由于位于细胞内,在Y-Z纵切面图中将呈现均匀分布 (图 4G)。值得一提的是,PALM显微镜下的X-Y (横切面) 和Y-Z (纵切面) 并不需要对细菌进行物理切割,而是通过软件转换实现三维成像。目前,PALM还处于不断革新和完善之中,对光学配件、计算机硬件和软件系统等都有特殊要求,虽然Zeiss和Nikon等公司已经有商品化样机出售,但是价格是激光共聚焦显微镜的数倍。本实验室使用的PALM 为自行搭建的显微镜平台,软件也是在Metlab的基础上进行自主开发。上述实验表明:PALM可以用于膜蛋白的精准定位研究。
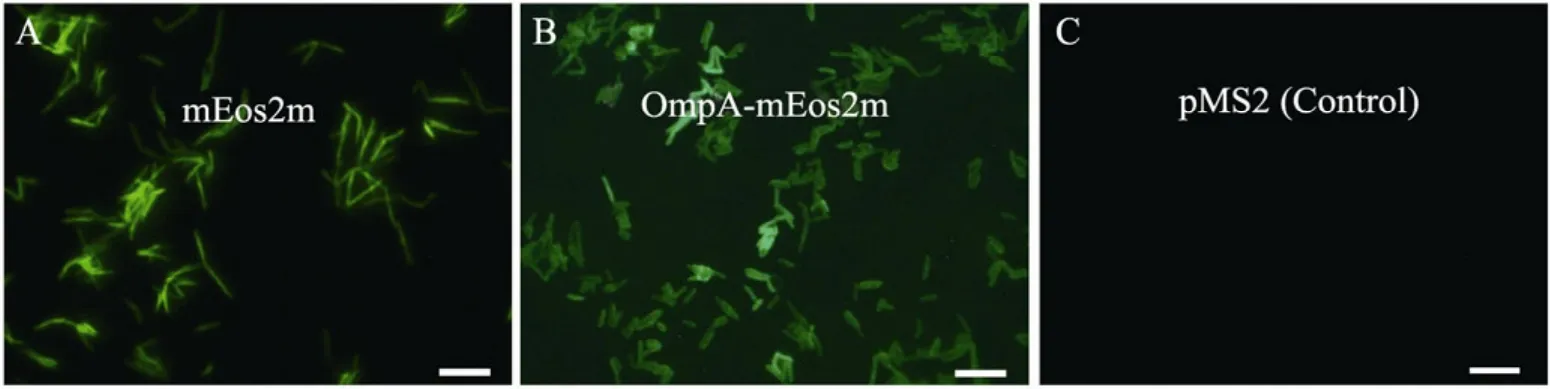
图3 光激活蛋白mEos2m在耻垢分枝杆菌中的表达Fig. 3 Expression of photoactivatable mEos2m in M. smegmatis. The bacterial cells expressing mEos2m (A) and OmpA-mEos2m (B) were fixed on slides and visualized under Zeiss fluorescent research microscope. pMS2, an empty plasmid without any inserts, was used as control (C). Scale bar=5 µm.
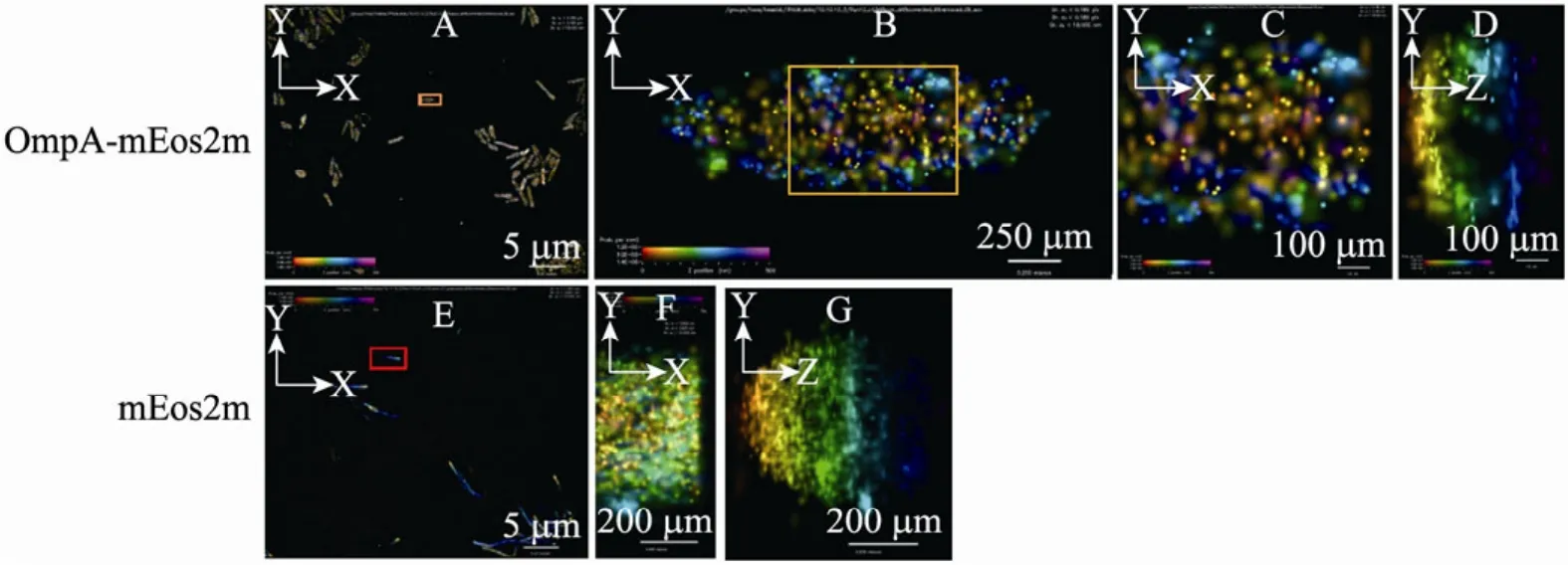
图4 外膜蛋白OmpA在耻垢分枝杆菌细胞膜上的定位Fig. 4 Localization of OmpA in the membrane region of M. smegmatis. The bacterial cells expressing photoactivatable OmpA-mEos2m (A-D) and mEos2m (E-G) were fixed on slides, activated by 405 nm laser, and visualized under a self-built photoactivated localization microscope (PALM).
3 讨论
基于光激活原理的超分辨率 PALM显微成像技术,也称为 STORM (Stochastic optical reconstruction microscopy)[20-22],该技术通过控制荧光基团来实现超分辨率成像,克服了传统光学显微镜中存在的诸如衍射等瓶颈问题[23]。目前在PALM 技术方面走在国际前沿的实验室有哈佛大学Zhuang实验室、美国HHMI Hess实验室、美国 NIH儿童健康和发育研究所的 Lippincott-Schwartz实验室等。本实验室利用PALM技术首次实现了膜蛋白的定位研究,并在耻垢分枝杆菌中成功表达了mEos2m及其融合蛋白,为下一步利用细菌感染宿主细胞,进行细菌追踪、定位和活体成像奠定了基础。
除了mEos2m之外,本实验室还在分枝杆菌中表达了具有光激活特性的 PAmCherry,PATagRFP及其与OmpA的融合蛋白,这些光激活蛋白都可以作为膜蛋白的标记分子,其中mEos2m在单位时间内释放的光子最多,因此我们选用mEos2m进行了详细阐述。
除了OmpA外膜蛋白之外,MspA也是在分枝杆菌中被广泛关注的外膜蛋白。与 OmpA不同,MspA是一种孔道蛋白,可以形成在煮沸条件下也不会失去活性的八聚体蛋白[16],主要负责各种亲水性营养物质[24]、无机盐[15]和小分子药物转运[25],目前正被用作新型纳米通道进行核酸纳米测序研究[7]。我们发现完整的MspA八聚体结构是其功能和定位得以实现的前提,当MspA与mEos2m等任意荧光蛋白融合后,将破坏其八聚体结构的形成,表现为在平板上的菌落形态与mspA基因敲除株类似 (结果未列出),这说明八聚体机构被破坏后,丧失了对各种营养物的摄取能力。这从另一个角度也说明对于自身可以形成多聚体的膜蛋白,与其他蛋白融合表达时将可能失去其原有的空间结构。此外,由于MspA对蛋白酶K不敏感 (图1A),无论利用蛋白酶K实验还是PALM,MspA都不是一个很好的参考蛋白。这也是在膜蛋白研究领域中的一个共识:没有一种方法对所有膜蛋白都适合。
PALM作为一种新的三维成像技术,其优点是“超分辨率”;这种超分辨率必须借助具有光激活特性的荧光蛋白和能够捕捉光子的显微镜才能实现,而普通的绿色荧光蛋白和普通的荧光显微镜和激光共聚焦都无法达到这种效果。但是PALM技术本身也有“缺点”,主要体现在:1) 实际操作时间长。一般对每个样品光信号捕获需要的时间为2~3 h或者更长。2) 价格昂贵。目前在全球也只有少数几个实验室才能拥有,基本上都是自行搭建的平台。但是作为一种新型技术以及科研发展的需求尤其是纳米技术发展的需要,超分辨率成像技术将是未来科学发展的趋势。尤其是近几年Zeiss和Nikon等公司推出商品化的样机以来,相信PALM将很快在国内普及。
综上,本实验室通过光激活蛋白mEos2m成功建立了一种直接用于膜蛋白定位的研究方法,并以非致病性的耻垢分枝杆菌为模型研究致病性的结核分枝杆菌膜蛋白定位和功能,为膜蛋白的研究提供了新的思路。
REFERENCES
[1] Shtengel G, Galbraith JA, Galbraith CG, et al. Interferometric fluorescent super-resolution microscopy resolves 3D cellular ultrastructure. Proc Natl Acad Sci USA, 2009, 106(9): 3125−3130.
[2] Betzig E, Patterson GH, Sougrat R, et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science, 2006, 313(5793): 1642−1645.
[3] Testa I, Parazzoli D, Barozzi S et al. Spatial control of pa-GFP photoactivation in living cells. J Microsc, 2008, 230(Pt 1): 48−60.
[4] Subach FV, Patterson GH, Manley S et al. Photoactivatable mCherry for high-resolution two-color fluorescence microscopy. Nat Methods, 2009, 6(2): 153−159.
[5] Baker SM, Buckheit RW, Falk MM. Green-to-red photoconvertible fluorescent proteins: tracking cell and protein dynamics on standard wide-field mercury arc-based microscopes. BMC Cell Biol, 2010, 11(1): 15.
[6] Subach FV, Patterson GH, Renz M, et al. Bright monomeric photoactivatable red fluorescent protein for two-color super-resolution sptPALM of live cells. J Am Chem Soc, 2010, 132(18): 6481−6491.
[7] Derrington IM, Butler TZ, Collins MD, et al. Nanopore DNA sequencing with MspA. Proc Natl Acad Sci USA, 2010, 107(37): 16060−16065.
[8] Butler TZ, Pavlenok M, Derrington IM, et al. Single-molecule DNA detection with an engineered MspA protein nanopore. Proc Natl Acad Sci USA, 2008, 105(52): 20647−20652.
[9] Svetliíková Z, Škovierová H, Niederweis M, et al. Role of porins in the susceptibility of Mycobacterium smegmatis and Mycobacterium chelonae to aldehyde-based disinfectants and drugs. Antimicrob Agents Chemother, 2009, 53(9): 4015−4018.
[10] Song HH, Sandie R, Wang Y, et al. Identification of outer membrane proteins of Mycobacterium tuberculosis. Tuberculosis, 2008, 88(6): 526−544.
[11] Brok R, van Gelder P, Winterhalter M, et al. The C-terminal domain of the Pseudomonas secretin XcpQ forms oligomeric rings with pore activity. J Mol Biol, 1999, 294(5): 1169−1179.
[12] WHO report 2011 global tuberculosis control[EB/OL]. [2012-04-18]. http://www.who.int/ tb/publications/global_report/en/.
[13] Wolschendorf F, Ackart D, Shrestha TB, et al. Copper resistance is essential for virulence of Mycobacterium tuberculosis. Proc Natl Acad Sci USA, 2011, 108(4): 1621−1626.
[14] Siroy A, Mailaender C, Harder D, et al. Rv1698 of Mycobacterium tuberculosis represents a new class of channel-forming outer membrane proteins. J Biol Chem, 2008, 283(26): 17827−17837.
[15] Wolschendorf F, Mahfoud M, Niederweis M. Porins are required for uptake of phosphates by Mycobacterium smegmatis. J Bacteriol, 2007, 189(6): 2435−2442.
[16] Faller M, Niederweis M, Schulz GE. The structure of a mycobacterial outer-membrane channel. Science, 2004, 303(5661): 1189−1192.
[17] Song HH, Wolschendorf F, Niederweis M. Construction of unmarked deletion mutants in mycobacteria//Parish T, Brown A. Mycobacteria Protocols. Ottawa: Humana Press, 2008: 279−295.
[18] Møller JV, Ning G, Maunsbach AB, et al. Probing of the membrane topology of sarcoplasmic reticulum Ca2+-ATPase with sequence-specific antibodies. Evidence for plasticity of the C-terminal domain. J Biol Chem, 1997, 272(46): 29015−29032.
[19] Song HH, Huff J, Janik K, et al. Expression of the ompATb operon accelerates ammonia secretion and adaptation of Mycobacterium tuberculosis to acidic environments. Mol Microbiol, 2011, 80(4): 900−918.
[20] Huang B, Jones SA, Brandenburg B, et al. Whole-cell 3D STORM reveals interactions between cellular structures with nanometer-scale resolution. Nat Methods, 2008, 5(12): 1047−1052.
[21] Bates M, Huang B, Dempsey GT, et al. Multicolor super-resolution imaging with photo-switchable fluorescent probes. Science, 2007, 317(5845): 1749−1753.
[22] Rust MJ, Bates M, Zhuang XW. Sub-diffractionlimit imaging by stochastic optical reconstruction microscopy (STORM). Nat Methods, 2006, 3(10): 793−795.
[23] Huang B, Babcock H, Zhuang X. Breaking the diffraction barrier: super-resolution imaging of cells. Cell, 2010, 143(7): 1047−1058.
[24] Song HH, Niederweis M. Uptake of sulfate but not phosphate by Mycobacterium tuberculosis is slower than that for Mycobacterium smegmatis. J Bacteriol, 2012, 194(5): 956−964.
[25] Danilchanka O, Pavlenok M, Niederweis M. Role of porins for uptake of antibiotics by Mycobacterium smegmatis. Antimicrob Agents Chemother, 2008, 52(9): 3127−3134.