汉坦病毒M、S基因分型及种系进化分析*
2012-01-24高立冬刘运芝胡世雄刘富强刘佳惠李俊华
蔡 亮,张 红,高立冬,刘运芝,胡世雄,刘富强,曾 舸,刘佳惠,李俊华
汉坦病毒M、S基因分型及种系进化分析*
蔡 亮,张 红,高立冬,刘运芝,胡世雄,刘富强,曾 舸,刘佳惠,李俊华
目的探讨湖南省肾综合征出血热(HFRS)患者及宿主动物黑线姬鼠携带汉坦病毒的基因型别及序列特征。方法应用免疫荧光试验(IFA)对鼠肺标本进行粗筛,提取IFA阳性鼠肺组织及HFRS患者血清总RNA,设计汉坦病毒M、S基因特异性引物,应用RT-PCR技术进行扩增,回收阳性扩增产物并克隆到p MD-18 T载体进行序列测定,将所测序列与国内外HTNV及SEOV病毒株序列进行核苷酸同源性分析,应用MEGA5.0.4软件构建M、S基因种系进化树。结果IFA阳性鼠肺标本荧光显微镜下可见散在的黄绿色针尖大小样颗粒;RT-PCR法扩增汉坦病毒M、S基因,HTNV通用引物可见490 bp与593 bp的阳性条带;阳性产物进一步应用HTNV分型引物进行M、S基因检测,分别扩增出一约242 bp与276 bp大小的条带;SEO型特异性M、S基因片段内侧引物扩增反应均为阴性;编号为Hunan01、Hunan02、Hunan03的M、S基因序列与HTNV型84FLi株、SN7株的同源性最高,与SEOV型Gou3株的同源性最低;种系进化分析表明,Hunan01、Hunan02、Hunan03为汉坦病毒H5亚型。结论湖南省存在HTNV型感染病例与宿主动物,基因分型技术能进一步对病毒亚型进行鉴定。
肾综合征出血热;汉坦病毒;M基因;S基因;分型;种系进化
汉坦病毒(Hantavirus,HV)属于布尼亚病毒科(Bunyaviridae)汉坦病毒属,编码L、M、S 3个基因片段[1],目前至少可以分为30个血清型/基因型,一些新的HV属病毒还在不断的发现中,主要引起肾综合征出血热(HFRS)和汉坦病毒肺综合征(HPS),基因组内极易发生变异,全球约90%以上的 HFRS 病 例 发 生 在 中 国[2-4]。 为 了 解 湖 南 省HFRS患者及宿主动物携带汉坦病毒基因的型别及变异情况,我们应用IFA、RT-PCR、基因测序与种系分子进化等方法对HFRS患者血清及宿主动物鼠肺组织标本进行了检测与分析,现将结果报告如下。
1 材料与方法
1.1 菌株、标本 E.coli DH 5a为湖南省疾病预防控制中心微生物实验室保种;鼠肺组织标本来源于国家级HFRS监测点湖南省宁乡县;病例标本来自全省HFRS患者血清。
1.2 主要试剂与仪器 Trizol@ Regeant(Invitrogen,美 国)、p MD-18 T 载 体、T 4 DNA 连 接 酶、QIAamp viral RNA Mini kit(QIAGENE,德国)、PrimeScript 1st Strand cDNA Synthesis Kit(Ta Ka-Ra)、Agarose Gel DNA Purification Kit Ver.2.0(Ta KaRa),LA PCRTMKit Ver.2.1 (Ta KaRa),2 000 bp DNA Marker、SOC培养基、IPTG、X-Gal等均为Ta KaRa公司产品,PCR仪(PCT-200 Thermal,德国),BigDye Terminator 3.1/1.1 version sequencing kit(ABI,美国),Applied Biosystems 3730 sequencer(ABI,美国),超低温组织冷冻切片机(Leica,德国)。
1.3 引物设计与合成 参照已知汉坦病毒国际标准株76-118株 M、S基因片段核苷酸序列(Gen-Bank Accession No:M14627.1,M14626.1)及相关文献设计特异性的逆转录引物、扩增引物与分型引物,委托宝生物(大连)合成,见表1。
1.4 IFA鼠肺组织标本应用IFA进行粗筛,鼠经断颈处死,3%来苏水浸泡5 min,无菌条件下解剖取肺脏,低温冷冻切片(4~10μm),切片应用FITC标记的HV直接荧光血清(中国疾病预防控制中心病毒病所出血热室提供)于37℃水浴箱孵育30 min,荧光显微镜下进行观察。具体操作参考全国肾综合征出血热诊断标准进行,阳性标本及疑似阳性标本采用巢式PCR进行复核与基因分型。
1.5 总RNA提取 取100mg IFA阳性肺组织于冰上碾磨,参照Trizol@Reagent试剂说明书提取鼠肺组织总RNA,溶于40μL DEPC水。
1.6 RT-PCR 应用 P1引物及 PrimeScript 1st Strand cDNA Synthesis Kit试剂盒合成c DNA,反应体系与反应条件参照试剂盒说明书进行;巢式PCR分两轮进行:首轮应用汉坦病毒M基因通用上下游引物(20μmol/L)与LA PCRTMKit Ver.2.1试剂盒进行扩增,阳性产物应用M基因特异性分型上下游引物(20μmol/L)进行第二轮扩增。对M基因阳性标本,在相同条件下进行S基因的扩增与分型,具体反应体系与反应条件参考试剂盒说明书进行。
1.7 T-A克隆与测序 PCR产物经1.5%琼脂糖凝胶电泳,按照Agarose Gel DNA Purification Kit试剂盒说明书进行切胶纯化回收,回收产物与p MD-18 T载体进行连接,转化到E.coli DH5a感受态细胞,建立T-A克隆[5]。采用蓝白斑筛选和PCR法对阳性克隆进行鉴定,克隆产物通过质粒提取纯化后在ABI3730基因测序仪上参照BigDye Terminator 3.1/1.1 version sequencing kit试剂盒说明书进行序列测定。
1.8 序列分析 运用 ATGC(Gentyx Co.,Tokyo,Japan)生物学软件对所测序列进行校对与拼接,对拼接后的序列进行命名,运行BLAST与DNAStar(Lasergene○Rv8.0)程序与 GenBank报道的汉坦病毒HTNV及SEOV型病毒株M基因、S基因序列进行核苷酸比较,应用BioEdit等生物学软件对所获序列进行基因组成特征的分析。
1.9 进化分析 选择GenBank公布的不同地区有代表意义的汉坦病毒M、S基因序列用于后续的分子进化分析,应用CLUSTAL W 软件(Version 1.83 package.Thompson et al.,1997)进行多重序列比对,Mega5.0.4(Kumar et al.,)生物学软件构建系统进化树。
2 结 果
2.1 IFA 150份鼠肺组织中,检出3份IFA阳性标本,阳性标本于荧光显微镜下可见散在的针尖大小的黄绿色颗粒,呈均匀分布于感染细胞的胞浆内,正常对照组织被染成橙红色,未见特异性的荧光颗粒。见图1。
2.2 RT-PCR 60份 HFRS患者血清经 RTPCR检测汉坦病毒M、S基因,2份标本 M、S基因通用引物可见明显的阳性扩增条带(Hunan H01、Hunan H02),条带大小约490 bp与593 bp,与预期大小一致。3份IFA阳性鼠肺标本经RT-PCR检测汉坦病毒M、S基因,1份为阳性(Hunan H03),产物大小为490 bp与593 bp。3份PCR通用引物阳性标本应用HTNV特异性分型引物进行检测,均扩增出一242 bp与276 bp大小的条带,SEOV型特异性分型引物扩增结果均为阴性,与预期结果一致。见图2。
2.3 测序及同源性分析 对3份M、S基因通用引物PCR 阳性标本(Hunan H01、Hunan H02、Hunan H03)扩增产物进行T-A克隆后应用ABI3730基因分析仪进行序列测定,经序列拼接与校正,大小与预期结果一致。编号为Hunan H01、Hunan H02、Hunan H03株测序结果去掉引物序列信息及引物附近的核苷酸序列信息后与汉坦病毒国际标准株76-118株核苷酸序列 M基因(1 190 nt~1 680 nt)的比较碱基同源性分别为85%、84%和86%,S基因(494 nt~1 086 nt)的比较碱基同源性分别为85%、84%和83%;与其他HTNV型及SEOV型病毒M基因片段(1190 nt~1 680 nt)碱基同源性的比较见表2,S基因片段(494 nt~1 086 nt)碱基同源性比较见表3。
2.4 种系进化分析 M基因部分核苷酸(1 190 nt~1 680 nt)种系进化分析结果见图3,由图可知,2株人源性汉坦病毒(Hunan H01、Hunan H02)株与1株鼠源性汉坦病毒株(Hunan H03)处于同一组内,核苷酸序列显示为高度同源,与分离自HFRS病人的84Fli株(H5)在同一分支上,进化上显示出高度的同源性,与SN7株在同一组上,具有较高的同源性和较近的进化关系。
S基因部分核苷酸(494nt~1086nt)种系进化分析结果见图4,3株汉坦病毒(Hunan H01、Hunan H02、Hunan H03)与SN7株完全聚集在一个组内,与84Fli(H5)、KY、SN7、Chen4等株在同一分支内,亲缘关系较近。

表1 汉坦病毒逆转录与分型引物Tab.1 Primer sets designed for hantavirus reverse transcription and genotyping

图1 IFA法检测鼠肺组织中汉坦病毒抗原A:IFA阳性结果;B:IFA阴性对照Fig.1 Immunofluorescence assay(IFA)of hantavirus in lung tissues of rats A:IFA positive result;B.IFA negative result
3 讨 论
早期HV的实验室诊断与分型多采用免疫荧光染色试验(IFA)、酶联免疫吸附试验(ELISA)、反向被动血凝抑制试验(RPHI)和空斑减少中和试验(PRNT)等经典方法进行[6],随着分子生物学技术的迅猛发展,上述方法均显示出了一定的局限性。目前,基因分型技术在HV感染后的回顾性流行病学调查及流行毒株型别的鉴定中得到了广泛的应用[7]。
本文在应用FITC标记的抗HV病毒单克隆抗体对宿主动物鼠肺组织标本进行IFA粗筛的同时,还利用RT-PCR技术对IFA阳性鼠肺标本及HFRS患者血清标本进行了M基因、S基因相关片段的检测与序列测定,为进一步达到基因分型的目的,根据宋干等报道的我国存在Hantaan(HTNV)和Seoul(SEOV)型病毒的研究结果[8-9],在引物设计方面,参考了国际上公布的HTNV与SEOV相关序列进行综合分析所得。检测结果表明,在150份宿主动物鼠肺组织中通过IFA试验检测到了3份HV病毒特异性抗原的存在,在IFA阳性标本与HFRS患者血清中应用RT-PCR法共检测到3份汉坦病毒 M基因、S基因同时阳性的标本,其中Hunan H01、Hunan H02源自HFRS患者血清,Hunan H03源自宿主动物黑线姬鼠肺组织。进一步应用所设计的分型引物进行扩增,同时扩增出M基因(1190nt~1680nt)、S基因(494nt~1086nt)相应大小的基因片段,表明所建立的PCR方法在汉坦病毒基因检测与分型中具有较好的特异性,可以推广应用。
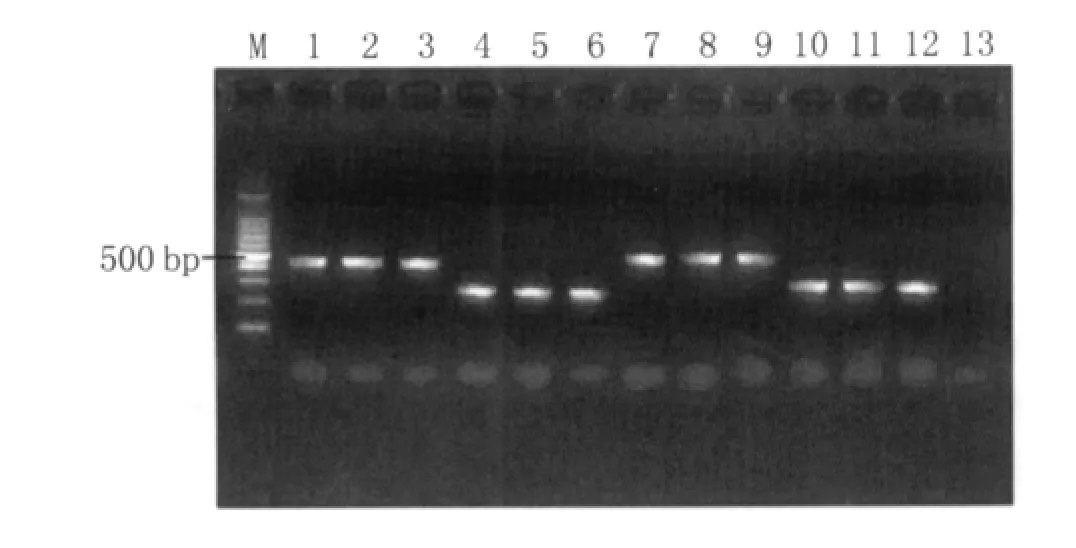
图2 RT-PCR检测汉坦病毒 M、S基因及其分型(1.5%琼脂糖凝胶电泳)M:100bp DNA Marker;1-3:Hunan01,Hunan02,Hunan03汉坦病毒 M基因通用引物;4-6:Hunan01,Hunan02,Hunan03汉坦病毒M基因特异性分型引物;7-9:Hunan01,Hunan02,Hunan03汉坦病毒S基因通用引物;10-12:Hunan01,Hunan02,Hunan03汉坦病毒S基因特异性分型引物;13:PCR阴性对照Fig.2 RT-PCR and genotyping results of hantavirus M and S genes(1.5%agrose gel)M:100bp DNA Marker;1-3:PCR results of Hunan01,Hunan02 and Hunan03 with hantavirus M genes general primers;4-6:PCR results of Hunan01,Hunan02 and Hunan03 with hantavirus M genes special genotyping primers;7-9:PCR results of Hunan01,Hunan02 and Hunan03 with hantavirus S gene general primers;10-12:PCR results of Hunan01,Hunan02 and Hunan03 with hantavirus S gene special genotyping primers;13:PCR negative control.
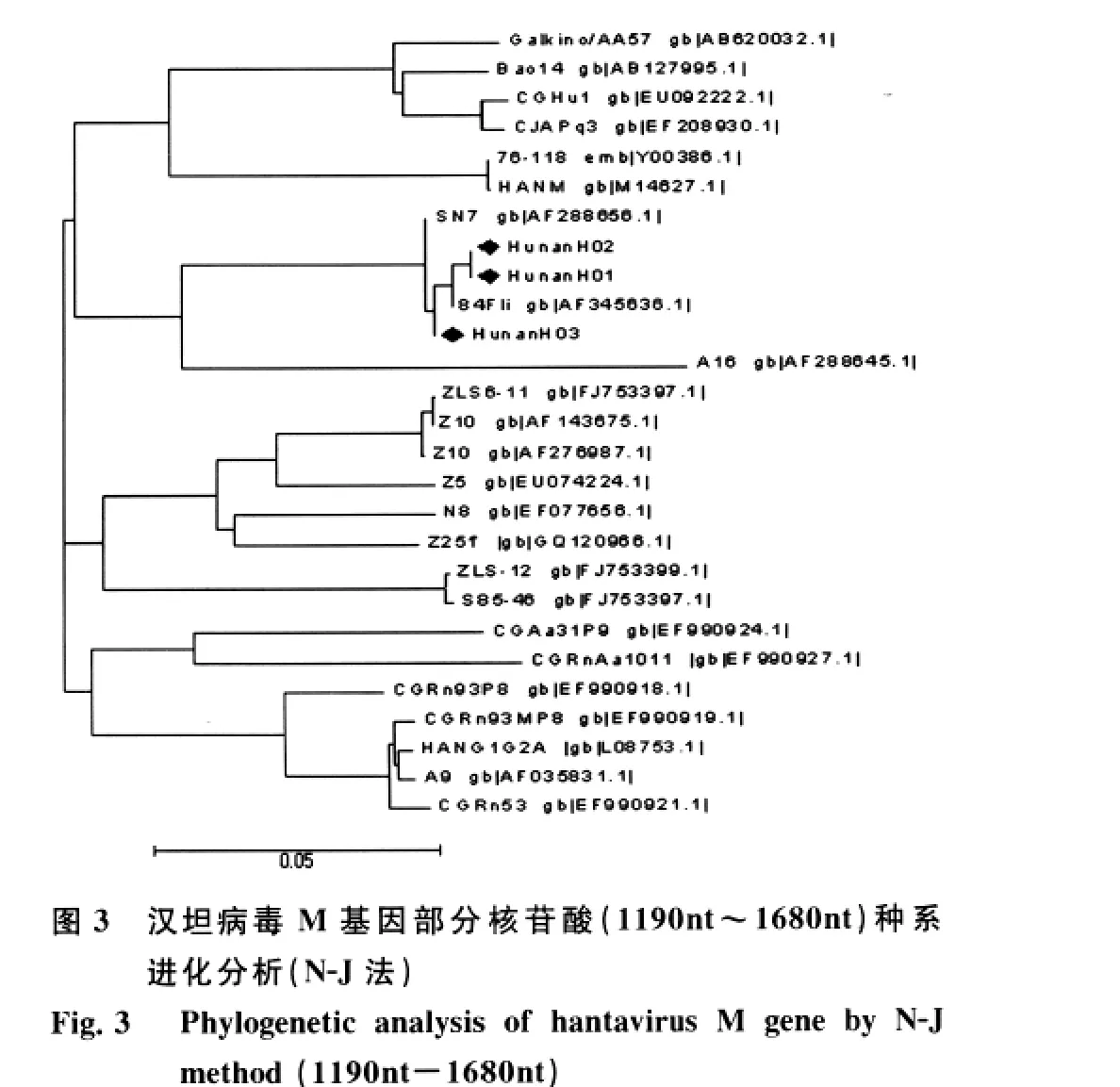
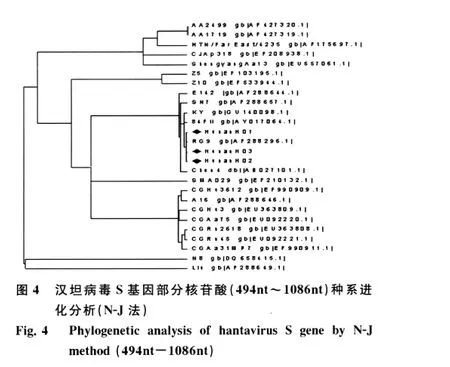

表2 编号Hunan01,Hunan02,Hunan03汉坦病毒M基因部分核苷酸与其他汉坦病毒株同源性比较Tab.2 Homology comparison on partial nucleotide sequence of hantavirus M gene from Hunan01,Hunan02 and Hunan03 to the other strains(%)
对M基因、S基因序列的测定,采用了正反引物同时进行,通过生物信息学方法对序列进行相互比对和核实,确保了测序结果的准确性。对所获3份标本M、S基因核苷酸序列与已公布的汉坦病毒属HTNV型和SEOV型M、S基因进行对比,3份标本M、S基因核苷酸均与84FLi株同源性最高,在85%~99%之间,与SEOV属Gou3株同源性最低,仅为76%~80%。

表3 编号Hunan01,Hunan02,Hunan03汉坦病毒S基因部分核苷酸与其他汉坦病毒株同源性比较Tab.3 Homology comparison on partial nucleotide sequence of hantavirus S gene from Hunan01,Hunan02 and Hunan03 to the other strains(%)
我国流行的HV种系进化研究表明,HTNV可分为9个亚型,而SEOV分为4-6个亚型,表现为相对稳定[10],另有研究表明,汉坦病毒部分核苷酸序列分析可以代表完整的基因序列进行分子进化的分析[11],本研究中,我们对M基因、S基因部分核苷酸进行了种系分子进化分析,Hunan H01、Hunan H02及Hunan H03均与HTNV聚集在一个大分支下,进一步的M、S基因亚型分析结果显示Hunan H01、Hunan H02及Hunan H03与为H5亚型84Fli株在同一组内,提示3份标本均为HTNV型H5亚型,与PCR分型检测结果一致。
[1]Schmaljohn C,Hasty SE,Darymple JM,et al.Antigenic and genetic properties of virus linked to hemorrhagic fever with renal syndrome[J].Science,1985,227:1041-1044.
[2]李家亮,李德新.汉坦病毒病原学研究进展[J].中华实验和临床病毒学杂志,2005,19(2):198-200.
[3]Xiao S Y,Liang M F,Schmaljohn C S,et al.Moleculor and antigentic characterization of HV114,a hantavirus isolated from a patient with hemorrhagic fever with renal syndrome in China[J].Journal of General Virology,1993,74:1657-1659.
[4]郭海强,丁海龙,曲波,等.应用 ARIMA模型对全国2004-2009年肾综合征出血热疫情分析及预测[J].中国人兽共患病学报,2010,26(12):1137-1140.
[5]J.萨姆布鲁克,E.F弗里奇.曼尼阿蒂斯,著,金冬雁,等译.分子克隆实验指南[M].2版,北京:科学出版社,1999:57-60.
[6]宋干,汪诚信,姜克俭,等.流行性出血热防治手册[M].2版,北京:人民卫生出版社,1987,36-48.
[7]雷万里,唐家琪,陆水平,等.肾综合征出血热病毒基因检测及分型研究[J].中国病毒学,1998,13(1):23.
[8]宋干,杭长寿,裘学昭,等.流行性出血热病原学研究I.用非疫区黑线姬鼠分离病毒及其特异性检查[J].中国医学科学院学报,1982,4:75-80.
[9]宋干,杭长寿,裘学昭,等.流行性出血热病原学研究Ⅱ.流行性出血热病毒一般形状的研究[J].中华微生物和免疫学杂志,1983,3:76.
[10]王世文,杭长寿,王华,等.我国汉坦病毒基因型和基因亚型的分布研究[J].病毒学报,2002,18(3):211-216.
[11]丁晓华,杨占秋,肖红,等.汉坦病毒 M片段的核苷酸序列变异和系统发生树分析[J].病毒学报,2003,19(2):169-172.
Detection and genotyping of hantavirus M and S gene and its phylogenetic analysis
CAI Liang,ZHANG Hong,GAO Li-dong,LIU Yun-zhi,HU Shi-xiong,LIU Fu-qiang,ZENG Ge,LIU Jia-hui,LI Jun-hua
(Hunan Provincial Center for Disease Control and Prevention,Key Laboratory of Microbial Molecular Biology of Hunan Province,Changsha 410005,China)
To investigate the genotype and characteristic of hantavirus M and S hemorrhagic fever with renal syndrome(HFRS),lung tissues of rats were screened by immun of luorescent antibody assay (IFA),and the total RNA was extracted from the serum of HFRS patients and IFA positive lung tissues.RT-PCR was applied to amplify the M and S gene of hantavirus after specific primers were designed.PCR positive product was recovered and purified before cloning to the p MD-18 T vector and the sequencing was carried by ABI3730 gene sequencer.The obtained sequencings were compared to the HTNV and SEOV sequences of han tavirus for nucleotide homology analysis and applied MEGA5.0.4 software to construct the phylo genetic tree.The yellow-green needle-like particles were scattered in the positive lung tissues under fluorescence microscope.The PCR products of M and S gene was about 490bp and 593bp with general primers and the further amplification with HTNV typing primers to the M and S gene was respectively 242bp and 276bp,while the amplified results of M and S gene were negative with the primers of SEOV.The sequences of Hunan01,Hunan02 and Hunan03 had a high nucleotide homology to the 84FLi and SN7 strains,which had a lower homology to the Gou3 strain.Hunan01,Hunan02 and Hunan03 were belonged to H5 subtype of HTNV.In conclusion,the technology of genotyping is appropriate for hantavirus diagnosis,and the HTNV patients and animal hosts are exist in Hunan Province.
HFRS;hantavirus;M gene;S gene;genotyping;phylogenetic
湖南省疾病预防控制中心,湖南省微生物分子生物学重点实验室,长沙 410005
R373
A
1002-2694(2012)02-0111-05
*湖南省卫生厅科研基金(No.A2007008)和湖南省科技厅科研基金(No.2010TD2027)联合资助
李俊华,Email:hncdc_ljh@163.com
2011-08-11;
2011-09-26
