退火温度对聚合酶链式反应焓值的影响
2011-12-28周国燕李红卫
周国燕 蓝 浩 李红卫
(上海理工大学生物系统热科学研究所,上海 200093)
退火温度对聚合酶链式反应焓值的影响
周国燕 蓝 浩 李红卫
(上海理工大学生物系统热科学研究所,上海 200093)
在聚合酶链式反应(PCR)热循环中,退火温度的选取至关重要,该因素的稍微变化对PCR扩增会产生很大的影响,导致整个PCR的热现象变化。通过差示扫描量技术(DSC)研究HBV在不同退火温度时焓值随循环数的变化。结果表明:PCR扩增在变性阶段吸热,在退火和延伸阶段放热,每一个PCR循环呈现放热效应,同时退火温度高导致平台期的前移和焓值的降低,53℃退火最佳。
聚合酶链式反应;焓值;差示扫描热量技术;退火温度;循环数
聚合酶链式反应 (polymerase chain reaction,简 称PCR),是一种体外快速扩增特异 NA片段技术[1],由Kary.B.Mullis等于1985年在PE-Cetus公司发明并命名。PCR虽然在设计上十分简单,但实际上是个非常复杂的生化反应过程,目前为止还是一种半经验性的技术,此外PCR的高度敏感性导致任何因素的微小变化都会很大程度影响检测结果,如假阴、假阳性问题和非特异扩增[2]。故探索研究PCR的影响因素及控制方法很有必要。
PCR热循环过程各阶段温度的选取对其扩增结果的影响很大,其中退火温度的高低不仅决定着反应的特异性,也影响着反应的扩增效率。现有的研究[3,4]表明:退火过程首先是引物和模板形成复合物,其次形成引物-模板-酶三倍体复合物,该过程伴随着化学键的不断生成和断裂,引起吸放热现象的产生。Conceicao等[5]用等温滴定法研究了模板引导DNA复制过程中的碱基插入和延伸时热动力学变化,结果表明酶促催化的碱基插入和DNA延伸过程是个放热过程。此外,随着循环数的增加,扩增效率的变化以及引物二聚体等物质的生成,引起的热现象也不同,故可从量热学角度研究退火温度对PCR扩增的影响。
差示扫描量热术(differential scanning calorimetry,简称DSC)指“在程序温度下测量输入到试样和参比物的功率差与温度关系的技术”,是最近几年发展起来的量热技术,对研究DNA精确复制有着十分重要的意义[6-11]。本试验依据PCR反应的温度特性,设置合理的DSC温度扫描程序,在DSC上实现PCR扩增,研究退火温度为55,54,53,52℃时PCR扩增各阶段的热流及其随循环数的变化情况,探讨了不同退火温度的热现象与PCR的假阴性和扩增效率的关系。
1 材料与方法
1.1 材料与仪器
模板:重组质粒 pUC-19,每质粒的拷贝数为1.5×103COPIES/μL,带有HBV完整基因组,吉林大学第二医院中心实验室提供;
大肠杆菌JM109:吉林大学第二医院中心实验室提供;
Taq DNA聚合酶、dNTP:大连宝生物工程有限公司;
质粒DNA小量抽提试剂盒:杭州爱思进生物技术公司;
差示扫描量热仪:DSC-Pyris Diamond,美国 Perkin Elmer公司;
天平:BP-211D,赛多利斯公司;
其他试剂:均为国产分析纯。
1.2 AXYGEN试剂盒提取质粒
培养、收集含有重组质粒pUC-19的大肠杆菌细胞[12],回收和纯化质粒DNA。用紫外分光光度计测出质粒浓度为96.9μg/mL(A260/A280=1.80),计算出每质粒的拷贝数为1.5×1010copies/μL,质粒倍比稀释得到本试验采用的质粒浓度1.5×106copies/μL[13]。
1.3 引物设计
根据GeneID:944568,设计扩增HBV前C基因合适的引物。本试验所用的引物委托上海生工生物工程技术服务有限公司合成并纯化。合成的HBV前C区基因引物序列:5′-TTT GGG GCA TGG ACA TTG AC-3′;5′-GCC TCG TCG TCT AAC AAC AG-3′。
1.4 PCR反应体系
PCR反应体系组成如表1所示。
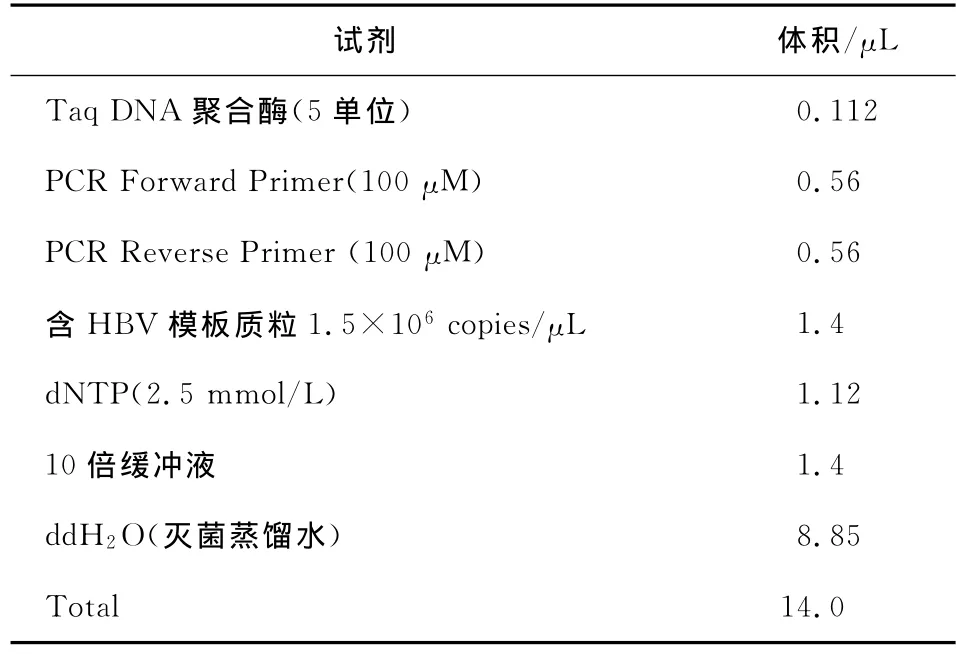
表1 PCR反应体系组成Table 1 Reagent concentration in PCR experiment
1.5 DSC扫描程序
常温下加样,变性温度为94℃,持续时间为0.8min;退火温度依次为55,54,53℃,持续时间为0.8min;延伸温度为72℃,持续时间为1.0min,30个循环,最后一个循环延伸时间增加3min。升降温扫描速率都为200℃/min,冷却采用Perkin-Elmer公司的二级制冷CryoFillTM;样品冲洗气体为高纯度氮气(纯度>99.999%),流量20mL/min保持不变;样品皿为PE不锈钢高压皿。
2 结果与分析
将配制好的14μL PCR反应体系于PE高压皿中,采用上述1.5的DSC温度扫描程序在DSC-Pyris Diamond上依次进行退火温度为55,54,53℃的PCR扩增,得到各个循环的热流随时间变化图。图1为退火温度54℃时的变化图,图中的温度曲线A依次指示,a~b:从72℃以200℃/min的速率升温至94℃,b~c:94℃恒温0.8min,c~d:从94℃以200 ℃/min的速率降温至54 ℃,d~e:54 ℃ 恒温0.8min,e~f:从54℃以200℃/min的速率升温至72℃,f~h:72℃恒温0.8min。由图1可知,b~c高温变性阶段为吸热过程,而d~e低温退火和f~h适温延伸阶段为放热过程,与冉姝等的试验结果一致[14,15]。曲线B﹑C分别为第2个循环和第1个循环的热流随时间变化图,从曲线可以看出PCR体系在b~c﹑d~f﹑f~h恒温段热流明显发生变化,且曲线B比曲线C变化大。用曲线B减去曲线C得到PCR循环数为2和1的热流差值随时间变化曲线D,其中,b~c变性吸热阶段的热焓差值为正,说明第二个循环比第一个循环在变性阶段吸收的热量多,而d~c退火﹑f~h延伸放热阶段的热焓差值为负,说明第二个循环比第一个循环在退火﹑延伸阶段放出的热量多,这是由于第二个循环复制的DNA比第一个循环的多引起的。用DSC自带的Pyris Software 5.0软件采用标准基线得到该循环的焓值为128.761J/g,同样的道理,得到不同退火温度的各个循环的焓值,用Origin 7.0分析这些数据得到不同退火温度时的焓值随循环数变化图,见图2~4。

图1 退火温度为55℃时第一个循环和第二个循环的热流变化图Figure 1 The change of heat flow as cycle number is one when the annealing temperature is 55 ℃

图2 退火温度55℃时热流密度随循环数的变化图Figure 2 The change of heat flow as cycle number raising when the annealing temperature is 55 ℃
退火温度的选取对PCR反应的特异性和扩增效率非常重要,一般来说,较低的退火温度可提高PCR反应的扩增效率但引物与模板间错配几率增大,导致扩增特异性较差,而较高的退火温度则可提高PCR反应的特异性但却降低了其扩增效率[16]。分析不同退火温度的变化图(1~4)可以得出:随着退火温度的降低,PCR反应体系的焓值随循环数的增加呈现不同的变化趋势,其中退火温度为55℃时,前5个循环的焓值变化很大,呈对数型,6~18间有很微弱的变化,但之后的差距变化不是很明显,出现平台效应的前移,说明退火温度较高,引物与模板结合的敏感性较差,导致引物没有和模板很好地结合,降低PCR的扩展效率,甚至检测结果呈假阴性;当退火温度降到54℃时,虽然在0到17个循环,焓值变化随循环数呈现指数扩增效应,而17个循环数后变化不是很大,平台效应没有前移,但平台期的焓值比较小,说明扩增效率比较低;当退火温度为53℃时,PCR体系焓值随循环数的增加呈现指数扩增效应和后期的平台效应,整个扩增过程的焓值都比较大,说明特异性和扩增效率达到了预期的效果,故应选取退火温度为53℃。

图3 退火温度54℃时热流密度随循环数的变化Figure 3 The change of heat flow as cycle number raising when the annealing temperature is 54 ℃

图4 退火温度53℃时热流密度随循环数的变化Figure 4 The change of heat flow as cycle number raising when the annealing temperature is 53 ℃
从图2~4的变化趋势可以看出,不论退火温度多少,PCR扩增都要进入“平台效应”,研究者从酶促扩增动力学的角度进行了相关的研究,可能的原因有:①DNA聚合酶活力的下降:PCR循环中最重要的因素是Tap-DNA聚合酶,虽然Tap-DNA聚合酶耐高温不容易失活,但反复长时间暴露在高温,活力会逐渐降低[17];②PCR扩增产物浓度的增加,抑制了反应的进行[18,19];③ 引物二聚体的形成:随着循环数的增加,引物不仅和模板DNA结合,引物和引物间也相互结合形成引物二聚体,形成的引物二聚体不仅减少引物,同时也和ds-DNA竞争引物和dNTP,导致反应速率的降低[20];④随着循环数的增加,高温下低浓度ds-DNA变性困难和低温下模板再退火及退火延伸时间的延长,也会导致扩增效率的下降。PCR扩增是个复杂过程,受到诸多因素的影响,任何因素的微小变化都会很大程度上改变PCR扩增的忠实性和特异性[21],而开始阶段的反应物浓度,热循环条件微小变化和错配对产物的最对总产量影响极大[22]。
3 结论
本试验利用DSC从量热学方面研究不同退火温度对PCR扩增结果的影响,试验表明:PCR扩增在变性阶段吸热,而在退火和延伸阶段放热,每一个PCR循环呈现放热效应;不同退火温度时的PCR焓值随循环数变化趋势不同,过高或过低都不能达到预期结果,在本试验HBV体系中,53℃ 退火时的结果最佳。
1 Williams J G K,Kubelik A R.DNA polymorphism amplified by arbitary primers are useful as genetic markers [J].Nucl,Acid Res.,1990,18:6 531~6 535.
2 Weihong Liu,David A Saint.Validation of a quantitative method for real time PCR kinetics[J].Biochemical and Biophysical Research Communications,2002,294:347~353.
3 Scott E Whitney,Alugupally Sudhir,R Michael Nelson,et al.Principles of rapid polymerase chain reaction:mathematical modeling and experimental verification[J].Computational Biology and Chemistry,2004,28:195~209.
4 You Y,Tataurov A V,Owczarzy R.Measuring thermodynamic details of DNA hybridization using fluorescence[J].Biopolymers,2011,95(7):472~486.
5 Minetti C A S A,Remeta D P,Miller H,et al.The thermodynamics of template-directed DNA synthesis:base insertion and extension enthalpies[J].Proc.Natl.Acad.Sci.U.S.A.,2003,100:14 719~14 724.
6 Bruylants G,Wouters J,Michaux C.Differential scanning calorimetry in life science:Thermod-ynamics,stability,molecular recognition and application in drug design[J].Current Medicinal Chemistry,2005,12:2 011~2 020.
7 Hui-Ting Lee,Chris M.Olsen,Lela Waters,et al.Thermodynamic contributions of the reactions of DNA intramolecular structures with their complementary strands[J].Biochimie,2008,90:1 052~1 063.
8 Concetta Giancola.A convenient tool for studying the stability of proteins and nucleic acids:differential scanning calorimetry[J].Journal of Thermal Analysis and Calorimetry,2008,91:79~85.
9 Diego Esposito,Pompea Del Vecchio,Guido Barone.A thermodynamic study of herring protamine– DNA complex by differential scanning calorimetry[J].Phys.Chem.Chem.Phys.,2001,3:5 320~5 325.
10 Brooke N Bourdelat-Parks,Roger M Wartell.Thermodynamic stability of DNA tandem mismatches[J].Biochemistry,2004,43:9 918~9 925.
11 Hughesman C B,Turner R F B,Haynes C A .Role of the heat capacity change in understanding and modeling melting thermodynamics of complementary duplexes containing standard and nucleobase-modified LNA[J].Biochemistry,2011,50(23):5 354~5 368.
12 Goergen B,Meyer Zum,Buschenfeld K H,et al.Mutation specific PCR and direct solid phase sequencing assay for the detection of hepatitis B virus pre-C/C mutants in anti-HBe-positive chronic hepatitis[J].Med.Virol.,1994,43(1):97~102.
13 Michael Kleined,Kirsten Schellenberg,Klaus Ritter1.Efficient extraction of viral DNA and viral RNA by the chemagic viral DNA/RNA kit allows sensitive detection of cytomegalo-virus,Hepatitis B Virus,and Hepatitis G Virus by PCR[J].Journal of Clinical Microbiology,2003,11(52):5 273~5 274.
14 冉姝,华泽钊,王景宇.聚合酶链式反应(PCR)量热学的实验研究[J].工程热物理学报,2009,3(30):482~484.
15 周国燕,李红卫,冉姝,等.聚合酶链式反应热流变化的DSC实验研究[J].生物学杂志,2010,27(4):1~4.
16 Rychlik W,Spencer W J,Rhoads R E.Optimization of the annealing temperature for DNA amplificaton in vitro[J].Nucleic acids reaearch,1990,18:6 409~6 412.
17 James T Hsu,Simantini Das,Satish Mohapatra.Polymerase chain reaction engineering[J].Biotechnology and Bioengineering,1997,2(55):359~360.
18 Peter Kainz.The PCR plateau phase-towards an understanding of its limitations[J].Biochimica et Biophysica Acta,2000,1 494:23~27.
19 Christy M Waterfall,Robert Eisenthal,Benjamin D Cobb.Kinetic characterisation of primer mismatches in allele-specific PCR:aquantitative assessment[J].Biochemical and Biophysical Research Communications,2002,299:715~722.
20 Simantini Das,Satish C Mohapatra,James T Hsu.Studies on primer-dimer formation in polymerase chain reaction(PCR)[J].Biotechnology Techniques,1999,13:643~646.
21 Weihong Liu,David A Saint.Validation of a quantitative method for real time PCR kinetics[J].Biochemical and Biophysical Research Communications,2002,294:347~353.
22 D Y Wu,L Ugozzoli,B K Pal,et al.The effect of temperature and oligonucleotide primer length on the specificity and efficiency of amplification by the polymerase chain reaction[J].DNA Cell Biol.,1991,10:233~238.
Research on changes of PCR enthalpy in different annealing temperature with DSC
ZHOU Guo-yan LAN HaoLI Hong-wei
(Institute of Biosystem Thermal Science,Shanghai University of Science and Technology,Shanghai200093,China)
It is very important to select annealing temperature in PCR process,a little change of the factor may lead to huge impact on thermal phenomenon of PCR.In this paper,the HBV as the target gene in different annealing temperature was replicated,and the enthalpy was studied via DSC,the experiment results show:the denaturation phase of the reaction was endothermic process,and the annealing and extension phase were heat emission process,each circulation presents a exothermic effect;simultaneously high annealing temperature caused platform moving ahead and enthalpy reduction,and be best in 53℃.
PCR;enthalpy;DSC;annealing temperature;cycle number
10.3969 /j.issn.1003-5788.2011.06.006
国家自然科学基金青年基金项目(编号:50206013);上海市教委科研创新项目(编号:09YZ230)
周国燕(1970-),女,上海理工大学副教授,博士。E-mail:efly_snow@163.com
2011-08-01
