先天性心脏病相关易感基因的预测性研究*
2011-11-20李秀英莫雪梅孙晗笑
刘 毅, 李秀英, 莫雪梅, 张 光, 孙晗笑
(暨南大学药学院基因组药物研究所,广东 广州 510632)
先天性心脏病相关易感基因的预测性研究*
刘 毅, 李秀英, 莫雪梅, 张 光, 孙晗笑△
(暨南大学药学院基因组药物研究所,广东 广州 510632)
目的: 应用生物信息学预测先天性心脏病相关易感基因,为先天性心脏病的临床诊断和治疗提供理论依据。方法通过OMIM数据库获取已知的先天性心脏病相关易感基因。将已知的疾病相关易感基因作为“种子基因”,利用蛋白质相互作用网络寻找邻近节点中富集了疾病相关易感基因的“种子基因”,并视其邻居基因为候选的疾病相关易感基因。对已知的疾病相关易感基因进行GO、Pathway和Human Phenotype富集分析,并据此注释候选基因;运用文献挖掘法对候选基因进行验证性分析。结果通过OMIM共获取已知的先天性心脏病相关易感基因28个,发现邻近节点中富集了疾病相关易感基因的“种子基因”4个,得到候选的疾病相关易感基因20个,通过功能富集分析从中预测出新的先天性心脏病易感基因7个。文献挖掘发现这7个基因在心脏发育过程中均起着极其重要的作用。结论通过生物信息学分析发现7个候选基因与已知的先天性心脏病相关易感基因关系密切,提示该病的发生是多种基因相互作用的结果,为后续深化该病机制研究提供了有效的指导。
心脏病,先天性; 蛋白质相互作用网络; 疾病易感基因; 功能富集分析
先天性心脏病(congenital heart disease,CHD)是指胚胎时期心血管发育异常对心功能产生了实际或潜在影响的一组先天畸形,在新生儿中发病率约为1%,严重危害婴幼儿健康。随着遗传学研究的深入开展及分子生物技术的广泛应用,许多基因被证实在心脏发育中扮演重要角色,尤其是各种转录因子之间通过相互作用形成精确的调控网络,发挥着中枢性作用,参与心脏发育。研究表明,很多人成年后发生的心律不齐、心肌收缩力下降等心脏疾患也可能是这些基因异常的结果[1]。随着后基因组时代的到来,对基因组功能的研究变得更加广泛而深入,破译基因的生物学功能并且利用它们已成为功能基因组学的一个主要目标。复杂疾病被认为是由许多个基因相互作用导致的疾病。寻找与复杂疾病相关的易感基因并阐述它们在疾病中的作用,对复杂疾病的临床诊断、治疗手段的发展起着巨大的推动作用。本研究立足于蛋白质相互作用网络,并根据功能相似基因可能导致相似疾病表型这一理论依据[2],对先天性心脏病相关易感基因进行了预测性研究,旨在为后续深化该病机制研究提供有效的指导。
材 料 和 方 法
1数据源与软件分析工具
通过OMIM数据库,共获得已知的先天性心脏病相关易感基因28个。经过整合的人类蛋白质相互作用数据来源于HPRD、BIOGRID和BIND数据库。互作数据整理后得到一个包含9 817个蛋白质的互作网络。HPRD互作数据下载地址:http://www.hprd.org/。BIOGRID互作数据下载地址:http://www.thebiogrid.org/。BIND互作数据下载地址:http://bond.unleashedinformatics.com/。功能富集分析工具网络地址:http://toppgene.cchmc.org/。文献挖掘工具Chilibot网络地址:http://www.chilibot.net/。
2蛋白质互作网络分析
将所有已知的疾病易感基因作为“种子基因”在线输入HPRD、BIOGRID和BIND数据库,检索、下载和整合已知的疾病易感基因对应蛋白在数据库中的蛋白质-蛋白质相互作用信息,清除蛋白质相互作用数据中的重复数据。统计“种子基因”和直接与其发生相互作用的基因(亦称邻居基因)所构成的亚网络包含的节点数目,计算以“种子基因”和邻居基因构成的亚网络中所包含的疾病易感基因的比例,并与所有具有蛋白质相互作用信息的“种子基因”及其邻居基因构成的网络中所包含的疾病易感基因的比例比较,判断该“种子基因”邻近节点是否能够富集疾病相关易感基因,并据此将其邻居基因视为候选的疾病相关易感基因。
3功能富集分析
利用功能富集分析工具ToppFun对已知的疾病易感基因从分子功能、生理过程和信号通路、细胞组分以及人体表型特征4个生物学功能角度进行注释。根据疾病致病基因之间相同的生物学功能,筛选候选疾病相关易感基因中与已知易感基因功能一致的基因作为本次研究所预测的新的先天性心脏病相关易感基因。
4文献挖掘分析
将英文关键词(“congenital heart disease” 、 “congenital heart defects” 或 “heart development”)和所预测的结果基因在线输入文献挖掘工具Chilibot。通过阅读Chilibot所列出的相关文献摘要,判断该基因与先天性心脏病、先天性心脏缺损和心脏发育之间的关系。
5统计学处理
结 果
1蛋白质互作网络分析
28个已知的先天性心脏病基因中有24个基因对应的蛋白具有相互作用信息,24个“种子基因”共有邻居基因233个(相互之间无重复),构成了一个由257个蛋白质、993个非自身相互作用构成的蛋白质相互作用网络。每个“种子基因”与其邻居基因构成的亚网络包含的节点数目和所包含的疾病易感基因的比例结果见表1。统计分析发现,24个“种子基因”中,邻近节点富集了疾病易感基因的“种子基因”仅4个,分别为:GATA结合蛋白4(GATA-binding protein 4,GATA4)(6/18,33.3%)、NK2相关转录因子5(NK2 transcription factor related,locus 5,NKX2-5)(4/11,36.4%)、Jumonji样蛋白/AT富集反应性结构域2 (Jumonji/AT-rich interactive domain 2,JARID2)(4/6,66.7%)和T盒转录因子5(T-box transcription factor 5,TBX5)(3/3,100%),明显高于在由257个蛋白质构成的蛋白质相互作用网络中的比例(24/257,9.3%)(分别为:Plt;0.01、Plt;0.05、Plt;0.01和Plt;0.01)。以上分析结果提示,与先天性心脏病相关的易感基因具有更高的概率倾向富集于GATA4、NKX2-5、JARID2和TBX5这4个“种子基因” 邻近节点中。因此,将GATA4、NKX2-5、JARID2和TBX5的邻近节点中尚未被标记为疾病易感基因的基因进行整理,清除重复数据,共得到20个候选的先天性心脏病相关易感基因。

表1 “种子基因”与其邻居基因构成的亚网络包含的节点数目和所包含的疾病易感基因的比例
2功能富集分析
应用功能富集分析工具ToppFun对28个已知的先天性心脏病相关易感基因功能富集分析(显著性水平为0.05)结果如下。
2.1分子功能的富集分析 选择数据库GO-Molecular Function,Plt;0.05,通过富集分析,共有16条分子功能条目具有显著差异,见图1。分析这16条分子功能条目发现:已知的先天性心脏病相关易感基因大部分为转录因子和转录活化因子、转录因子结合蛋白,参与调控基因的正确转录和表达。
2.2生理过程的富集分析 选择数据库GO-Biologic Process,Plt;0.05,通过富集分析,共有98条生理过程条目具有显著差异。分析这98条生理过程条目发现:已知的先天性心脏病相关易感基因大部分为与心血管发育、组织器官形态发生、胚胎发育有着极为密切的联系。
2.3信号通路的富集分析 选择Pathway数据库,Plt;0.05,通过富集分析,共有7条信号通路具有显著差异,见图2。分析这7条信号通路条目发现:所富集的信号通路主要与心肌细胞的生理活动密切相关,同时也从另一方面确证了生理过程的富集分析结果。

Figure 1.Significant molecular function terms for known congenital heart disease-related susceptibility genes.P va-lue was obtained through Fisherps exact probability test,Plt;0.05.
2.4细胞组分的富集分析 选择GO-Cell Component数据库,Plt;0.05,通过富集分析,未发现有显著性细胞成分条目出现。

Figure 2.Significant pathway terms for known congenital heart disease-related susceptibility genes.P value was obtained through Fisherps exact probability test,Plt;0.05.
2.5人体表型特征的富集分析 选择Human Phenotype数据库,Plt;0.05,通过富集分析,共有7条人体表型特征条目具有显著差异,见图3。分析这7条人体表型特征条目发现:所富集的人体表型特征条目反映出已知疾病易感基因突变极易导致心室异常、心中隔异常、室间隔异常、室间隔缺损以及心脏血管异常等病理现象。
2.6功能注释候选基因 以已知疾病相关易感基因所富集的GO、Pathway和Human Phenotype功能条目为背景,对所预测的20个候选的疾病相关易感基因进行注释。结果发现:20个基因全部注释到已知的疾病相关易感基因所富集的GO上,5个基因共享了已知疾病相关易感基因所富集的信号通路,2个基因与已知疾病相关易感基因反映了相同的人体表型特征。为了保证预测结果的准确性,同时考虑到3种数据库所覆盖的基因范围不尽一致,我们选择GO与Pathway或者GO与Human Phenotype共同识别的基因作为本次研究的最终预测结果,共计7个基因,分别为:血清反应因子(serum response factor,SRF)、肌细胞增强子元件因子(myocyte enhancer factor 2C,MEF2C)、E1A结合蛋白p300(E1A binding protein p300,EP300)、促分裂原活化蛋白激酶1(mitogen-activated protein kinase 1,MAPK1)、促分裂原活化蛋白激酶3(mitogen-activated protein kinase 3,MAPK3)、活化T细胞质钙神经素依赖性转录因子4(nuclear factor of activated T-cells,cytoplasmic,calcineurin-dependent 4,NFATC4)和常染色质组蛋白赖氨酸N-甲基转移酶(euchromatic histone-lysineN-methyltransferase 1,EHMT1)。

Figure 3.Significant human phenotype terms for known congenital heart disease-related susceptibility genes.P value was obtained through Fisherps exact probability test,Plt;0.05.
3文献挖掘分析
应用文献挖掘工具Chilibot搜索7个疾病相关易感基因与congenital heart disease、congenital heart defects、heart development等关键词的关联,发现7个基因中有2个基因(MAPK1、MAPK3)与先天性心脏病相关,1个基因(EHMT1)与先天性心脏缺损相关,4个基因(SRF、MEF2C、EP300、NFATC4)与心脏发育相关,见表2。
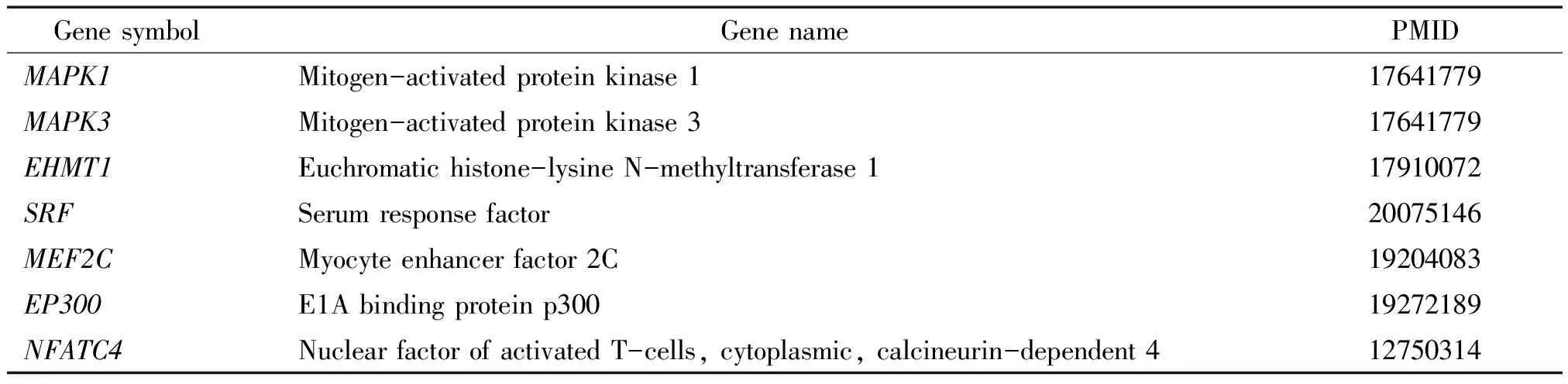
表2 预测所得的7个先天性心脏病易感基因及其参考文献
讨 论
伴随着大量自发突变体的研究,人们已经越来越清楚1个基因控制1个性状的关系是不存在的,所有复杂性状都受控于多个基因。不少复杂疾病被认为是由许多个基因相互作用而导致的疾病。人们研究复杂疾病的一个主要目标是对疾病相关的易感基因进行预测性分析,从为疾病的诊断与防治提供理论参考。
本研究中,我们引入了蛋白质相互作用网络对先天性心脏病相关易感基因进行了预测性分析。从系统生物学角度观察,蛋白质作为生命活动的主要执行者,各种生物学事件都是由蛋白质与别的基因、蛋白质或其它小分子物质相互作用并形成复杂的生物学网络来完成的,基因或蛋白质不能独立发挥生物学作用。而一旦正常的蛋白相互作用发生异常,就可能会导致疾病的发生。因此,研究蛋白质之间如何通过相互作用形成分子间调控网络,具有重要的生物学意义,不仅有助于从系统角度理解各种生物学过程,还能广泛应用于探索复杂性疾病的发生机制,评价临床治疗手段和寻找新的药物靶标。近几年,随着蛋白质相互作用数据的海量增长,与其相关的数据库也与日俱增,利用此类数据信息以研究各种生命活动现象已成为必然。从越来越多关于疾病相关易感基因预测的研究中,我们发现,即使是利用定位候选策略或是基因芯片技术[3],最终都要转化到蛋白质相互作用网络平台来解决问题,如文献[2],部分研究甚至直接利用蛋白质相互作用网络来对疾病相关易感基因进行预测,如文献[4]。本研究正是基于此种考虑,直接利用蛋白质相互作用网络对先天性心脏病相关易感基因进行预测性分析,不失为一种更简便准确的手段。
从生物学角度上观察,基因与其邻居基因往往具有相似的生物学功能,它们彼此之间的相互联系则与某生物表型或者疾病密切相关[5-7]。Goh等[8]研究发现同一疾病蛋白在网络中更倾向于存在相互作用,蛋白网络中与某疾病相关的蛋白,其邻近节点蛋白的功能变化更有可能与该蛋白一样导致相同或相近的疾病[9,10]。Jonsson[11]研究发现,致病基因之间的互作不仅远高于非致病基因之间的互作,而且致病基因比非致病基因在网络中更倾向于成簇存在。在本研究中我们以“种子基因”的邻居基因为筛选对象,其优势在于简单且预测效果准确率高,但是忽略了蛋白质网络的拓扑属性特征与基因功能的关系,可能会造成部分潜在的候选基因被遗漏[12]。通过分析本研究的蛋白质相互作用网络分析结果,我们发现,在由24个“种子基因”和233个邻居基因所构成的蛋白质相互作用网络中,与先天性心脏病相关的易感基因具有更高的概率倾向富集于GATA4、NKX2-5、JARID2和TBX5这4个“种子基因”邻近节点中(Plt;0.05),提示我们:(1)这4个“种子基因”与其邻居基因所构成的亚网络与先天性心脏病的关系较其它“种子基因”更为密切,因此,我们将GATA4、NKX2-5、JARID2和TBX5的邻近节点中尚未被标记为疾病易感基因的基因视为候选的先天性心脏病相关易感基因;(2)以这4个“种子基因”与其邻居基因所构成的亚网络可能与先天性心脏病发病相关的一种亚网络机制。这表明先天性心脏病的发生可能是亚网络中的基因协同作用的结果。而后续功能富集性分析的结果也证实了这一推测,所得到的7个候选易感基因与已知的疾病易感基因相比,有着相似的分子生物学功能,参与了相同的生物信号通路,导致了相关的病理表象。
从研究最终的分析结果来看,所得出的7个候选易感基因,经过文献搜索,发现这7个候选基因与先天性心脏病的发生存在着极为密切的关联。如:Nakamura等[13]的小鼠模型实验证实下调MAPK1/3活性可以有效缓解先天性心脏病病情的发展;而Ghosh等[14]则发现TBX5与MEF2C之间的蛋白质物理相互作用对于早期的心脏发育不可或缺。文献挖掘分析的结果提示,后续对于先天性心脏病机制的研究,可以扩大相关易感基因的研究范围,深入探究这7个候选易感基因与已知的易感基因的互动关系,有利于更清楚地揭示先天性心脏病的发病机制。
[1]Srivastava D.Heart disease: an ongoing genetic battle?[J].Nature,2004,429 (6994): 819-822.
[2]赵 研,陈丽娜,张良才,等.基于功能一致性和网络拓扑属性预测冠心病致病基因[J].生物化学与生物物理进展,2009,36 (6): 781-786.
[3]张 黎,赵仲生,马 杰,等.应用基因芯片筛选胃癌淋巴转移相关基因及TLN1的初步研究[J].中国病理生理杂志,2009,25 (7): 1271-1278.
[4]Ortutay C,Vihinen M.Identification of candidate disease genes by integrating Gene Ontologies and protein-interaction networks: case study of primary immunodeficiencies[J].Nucleic Acids Res,2009,37 (2): 622-628.
[5]Gunsalus KC,Ge H,Schetter AJ,et al.Predictive models of molecular machines involved inCaenorhabditiselegansearly embryogenesis[J].Nature,2005,436 (7052): 861-865.
[6]Gandhi TK,Zhong J,Mathivanan S,et al.Analysis of the human protein interactome and comparison with yeast,worm and fly interaction datasets[J].Nat Genet,2006,38(3): 285-293.
[7]Xia K,Xue H,Dong D,et al.Identification of proliferation/differentiation switch in the cellular network of multicellular organisms[J].PLoS Comput Biol,2006,2(11): e145.
[8]Goh KI,Cusick ME,Valle D,et al.The human disease network[J].Proc Natl Acad Sci USA,2007,104 (21): 8685-8590.
[9]Oti M,Snel B,Huynen MA,et al.Predicting disease genes using protein-protein interactions[J].J Med Genet,2006,43 (8): 691-698.
[10]Karni S,Soreq H,Sharan RA network-based method for predicting disease-causing genes[J].J Comput Biol,2009,16 (2):181-189.
[11]Jonsson PF,Bates PA.Global topological features of cancer proteins in the human interactome[J].Bioinformatics,2006,22 (18): 2291-2297.
[12]Sharan R,Ulitsky I,Shamir R.Network-based prediction of protein function[J].Mol Syst Biol,2007,3: 88.
[13]Nakamura T,Colbert M,Krenz M,et al.Mediating ERK 1/2 signaling rescues congenital heart defects in a mouse model of Noonan syndrome[J].J Clin Invest,2007,117 (8): 2123-2132.
[14]Ghosh TK,Song FF,Packham EA,et al.Physical interaction between TBX5 and MEF2C is required for early heart development[J].Mol Cell Biol,2009,29 (8): 2205-2218.
Predictionofcongenitalheartdisease-relatedsusceptibilitygenes
LIU Yi, LI Xiu-ying, MO Xue-mei, ZHANG Guang, SUN Han-xiao
(InstituteofGenomicMedicine,CollegeofPharmacy,JinanUniversity,Guangzhou510632,China)
AIM: To understand the molecular pathogenesis of congenital heart disease and provide theoretical guideline for clinical diagnosis and treatment,congenital heart disease-related susceptibility genes were predicted.METHODSCongenital heart disease-related susceptibility genes recorded in NCBI OMIM database were considered as “seed genes” and extracted.Protein-protein interaction network was utilized for searching “seed genes”,whose neighborhood enriched disease-related susceptibility genes.Once the target “seed genes” were identified,its neighbor genes were considered as candidate disease-related susceptibility genes.The candidate genes were annotated according to the functional enrichment analysis of GO,Pathway and Human Phenotype for the known disease-related susceptibility genes.Literature-mining method was utilized for validating the predicted genes.RESULTSTwenty-eight congenital heart disease-related susceptibility genes recorded in NCBI OMIM database were extracted.According to the statistical analysis,4 “seed genes”,whose neighborhood enriched disease-related susceptibility genes,were identified.There were 20 candidate disease-related susceptibility genes.Seven genes were predicted to be congenital heart disease-related susceptibility genes and all of them participate in heart development by literature retrieval.CONCLUSIONThere are close relationships between the 7 candidate genes and the known disease-related susceptibility genes.The pathogenesis of congenital heart disease involves multiple genes,and investigation of these genes may provide valuable insights into the mechanism of congenital heart disease.
Heart disease,congenital; Protein-protein interaction network; Disease-related susceptibility genes; Functional enrichment analysis
1000-4718(2011)05-0928-06
R318.04
A
10.3969/j.issn.1000-4718.2011.05.018
2010-12-20
2011-03-30
国家自然科学基金资助项目(No.30872221)
△通讯作者 Tel:020-38375022; E-mail: sunhx718@yahoo.com.cn
