H1N1流感病毒D151G突变株对奥司他韦的抗药性研究及药物筛选
2011-09-18石会娜栗海波
石会娜,栗海波,刘 伟
郑州大学生物工程系郑州450001
(2011-01-14收稿 责任编辑 徐春燕)
2009年3月以来,一种新的H1N1流感病毒的暴发引起了全球越来越多的关注。世界卫生组织推荐奥司他韦为最为有效的抗病毒药物。然而,截止到2010年2月,世界卫生组织宣布已有200多例H1N1病例对奥司他韦产生抗药性[1]。神经氨酸酶(NA)在流感病毒复制、释放和致病过程中起着非常重要的作用。奥司他韦是NA的抑制剂,能够有效阻止流感病毒NA发挥以上功能,从而达到较好的治疗效果。甲型流感病毒NA活性位点的结构在所有类中都是保守的,但是活性位点氨基酸残基的改变有可能使病毒对奥司他韦产生抗药性[2]。作者在GenBank中发现一株新H1N1流感病毒的NA活性位点151位由天冬氨酸突变成了甘氨酸(D151G),并通过分子动力学方法观察了该流感病毒株对奥司他韦的抗药性,再利用分子对接方法筛选出对这一病毒株的NA具有较强抑制作用的候选化合物。
1 材料与方法
1.1 分子动力学模拟
D151G突变株NA氨基酸序列由GenBank获得(序列号:GQ369413.1),其结构是以野生型禽流感H5N1病毒NA和奥司他韦复合物(WT-OTV)的晶体结构(PDB Code:2HU4)为模板通过同源建模得到,两者氨基酸序列相似性为90.4%,然后再将WT-OTV和D151G突变株NA的结构叠加,删除掉WT-OTV的NA后就得到了D151G突变株NA与奥司他韦复合物(MT-OTV)的结构。
分别用WT-OTV和MT-OTV的结构作为初始结构进行分子动力学模拟。蛋白质的力场采用ff99力场[3],小分子的力场采用 gaff力场[4]。利用Gaussian 03软件[5]的 HF/6-31G*方法优化小分子的结构,再应用antechamber程序产生gaff力场中缺失的小分子参数。复合物模型在进行动力学模拟前还需要利用Xleap程序对复合物进行处理,包括增加氢原子、增加抗衡离子(Na+)以使体系中性化和增加8.5Å厚的基于TIP3P模型的八面体水环境。
分子动力学过程优化分为两阶段:首先约束溶质,对溶剂分子进行优化;再无约束优化整个系统。每个阶段均采用最陡下降法优化2 500步,再采用共轭梯度法优化2 500步。优化之后用60 ps时间使系统升温到298 K,在298 K下进行500 ps的无约束的动力学平衡,平衡之后再进行500 ps的分子动力学模拟,用MM-GBSA方法计算结合能。
1.2 药物筛选 从ZINC数据库(http://zinc.docking.org/)里筛选出和奥司他韦相似度为70%的小分子化合物,然后把这些小分子分别和D151G突变株的NA组成复合物,再采用Autodock 4.0程序[6]对这些复合物进行分子对接。对接选用的格点盒子大小为40点×40点×40点,格点间距0.375Å。运用Lamarckian遗传算法[6],对小分子进行100次的独立对接实验,能量极大值设为2 500 000,最大变异代数设为27 000,最后依据复合物的最低结合能来挑选出比MT-OTV结合能更小的小分子。
2 结果与讨论
利用MM-GBSA方法计算出的各项能量值如表1所示,WT-OTV的结合能为 -14.0 kcal/mol(1 kcal=4.184 kJ),而实验值为 -13.0 kcal/mol[7],由此可以看出此种方法得到的结合能跟实验值非常吻合。
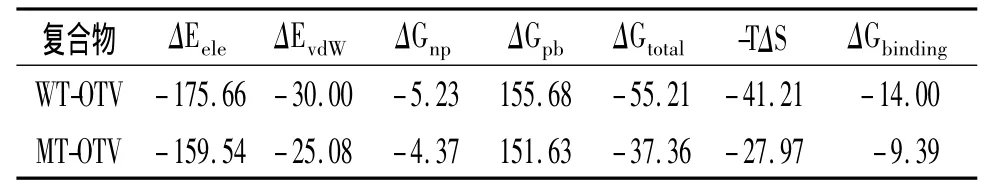
表1 MM-GBSA计算所得到的能量值 kcal/mol
MT-OTV的结合能比 WT-OTV降低了-4.61 kcal/mol,实验[8]已证实 H274Y 突变流感病毒株对奥司他韦的敏感性比野生型流感病毒降低了1 466倍,而H274Y突变株的NA与奥司他韦预测的结合能为 -9.8 kcal/mol[9],由此可以看出 D151G 突变流感病毒株对奥司他韦也将表现出较强的抗药性。
从ZINC数据库里筛选出了21个和奥司他韦相似度为70%的小分子化合物,经Autodock 4.0软件计算,有5个化合物与D151G突变的NA的结合能比与奥司他韦的结合能小。这5个化合物和奥司他韦的结构以及计算结合能如图1所示,其中,4-[1-(1-金刚烷基氧化羧基)乙氨基]-3-氨基-4-氧代-丁酸与 D151G突变株 NA的结合能力最强,对D151G突变流感病毒株具有较强的抑制作用。

图1 5种化合物和奥司他韦的结构式及计算结合能A:奥司他韦;B:3-氨基-4-氧代-4(1-茚酮-2-基)氨基丁酸;C:3-氨基-4-(4-甲基-2-氧代苯并吡喃-7-基)氨基-4-氧代丁酸;D:C的异构体;E:2-[2-氨基-3-(4-羟苯基)-丙醇]氨基-3-甲基-戊酸;F:4-[1-(1-金刚烷基氧化羧基)乙氨基]-3-氨基-4-氧代-丁酸。
总之,D151G突变流感病毒株对奥司他韦具有抗药性;利用分子对接软件从ZINC小分子数据库中筛选出的5个小分子化合物中4-[1-(1-金刚烷基氧化羧基)乙氨基]-3-氨基-4-氧代-丁酸的结合能力最强,是较理想的药物候选化合物。
[1] Update on oseltamivir resistant pandemic A(H1N1)2009 infl uenza virus:January 2010[J].Wkly Epidemiol Rec,2009,85(6):37
[2] Colman P,Varghese J,Laver W.Structure of the catalytic and antigenic sites in influenza virus neuraminidase[J].Nature,1983,303(5912):41
[3] Duan Y,Wu C,Chowdhury S,et al.A point-charge force field for molecular mechanics simulations of proteins based on condensed-phase quantum mechanical calculations[J].J Comput Chem,2003,24(16):1999
[4] Wang J,Wolf R,Caldwell J,et al.Development and tes ting of a general amber force field[J].JComput Chem,2004,25(9):1157
[5] Frisch M,Trucks G,Schlegel H,et al.Gaussian 03,revision B.03[M].Gaussian:Pittsburgh,PA,2003.
[6] Morris G,Goodsell D,Halliday R,et al.Automated docking using a Lamarckian genetic algorithm and an empirical binding free energy function[J].JComput Chem,1998,19(14):1639
[7] Malaisree M,Rungrotmongkol T,Nunthaboot N,et al.Source of oseltamivir resistance in avian influenza H5N1 virus with the H274Y mutation[J].Amino Acids,2009,37(4):725
[8] Hurt A,Ernest J,Deng Y,et al.Emergence and spread of oseltamivir-resistant A(H1N1)influenza viruses in Oceania,South East Asia and South Africa[J].Antiviral Res,2009,83(1):90
[9] Rungrotmongkol T,Malaisree M,Nunthaboot N,et al.Molecular prediction of oseltamivir efficiency against probable influenza A(H1N1-2009)mutants:molecular modeling approach[J].Amino Acids,2009,39(2):393