Cupriavidus necator JMP134细菌人工染色体基因组文库的构建与鉴定
2011-01-31张俊亚宋任涛许政暟朱晨光
张俊亚, 宋任涛, 许政暟, 朱晨光
(上海大学生命科学学院上海市能源作物育种及应用重点实验室,上海200444)
Cupriavidus necator JMP134(C.necator JMP134)又名Alcaligenes eutrophus,Ralstonia eutropha,Wautersia eutropha,最初因其能在含有2,4-二氯苯氧乙酸(2,4-D)的培养基上生长而从土壤中被分离出来.C. necator JMP134是一株典型的能够降解氯代芳香族化合物的菌株,它可以分解2,4-D、卤代苯甲酸、氯酚和硝基苯酚,并可以60多种芳香族化合物作为唯一碳源生长,在环境污染治理上具有很好的应用潜力[1].
C.necator JMP134菌株的基因组已在2005年由美国联合基因组研究所(JGI)完成测序,其大小为7.25 Mb,由2条染色体以及2个大小分别为635和88 kb的大质粒构成,其中与芳香化合物降解功能相关的基因6 616个,包括了12种主要芳香化合物降解途径中的11种,共编码了70多种降解芳香化合物所需的加氧酶[1].
通过比较基因组分析可以发现,C.necator JMP134有2套苯酚降解基因簇,其中长簇大小约为23 kb,短簇约为8 kb.目前关于苯酚降解基因的研究大多是基于其基因簇中单个基因的功能展开的[2-4],在整个基因簇的表达水平上对其进行功能分析的研究尚未见报道.另外,有关2套苯酚降解基因簇在宿主细菌中的协同作用机制的研究,也尚未见有任何报道.因此,以C.necator JMP134菌株为模式材料,对其2套苯酚降解基因簇进行深入的功能分析,将为创造更高效的苯酚降解工程菌提供有效的备选基因和研究方案,具有重要的理论研究意义和实际应用前景.
由于完整的苯酚降解基因簇长度通常在20 kb以上,欲克隆此类基因簇用于表达分析,构建基因组大片段文库必不可少.细菌人工染色体(bacterial artificial chromosome,BAC)载体的复制子来源于单拷贝质粒F因子,与其他基因组DNA大插入片段文库相比,BAC文库除了插入片段较大外,还具有遗传稳定性好、嵌合现象低等优点[5-6].此外,BAC载体以大肠杆菌为宿主,电击转化效率较高,常规方法(碱裂解)即可分离重组BAC质粒,蓝白斑和菌落原位杂交等技术均可用于目的基因的筛选,而且可对克隆在BAC的插入DNA直接测序.目前,BAC已成为应用最广泛的构建大插入片段基因组文库的载体系统.
本研究以 pIndigo-BAC 5为载体,构建了 C.necator JMP134菌株的BAC基因组文库.为了能够迅速、准确地筛选获得目的基因,通过筛选该菌株的2号基因簇苯酚羟化酶大亚基基因phlN2,验证了筛选方法的有效性以及所建文库的覆盖率.该BAC文库的构建以及筛选体系的建立,为克隆降酚基因簇及对其进行酶催化功能分析创造了有利条件.
1 材料和方法
1.1 材料
C.necator JMP134菌株购自德国菌种与细胞保藏中心(DSMZ),菌株号为 DSM 4058;BAC载体pIndigo-BAC 5(HindⅢ)购自Epicentre公司;低熔点琼脂糖胶和大肠杆菌感受态细胞 DH10B购自Invitrogen公司;限制性内切酶HindⅢ,NotⅠ,XbaⅠ购自Fermentas公司;Taq DNA Polymerase和dNTP购自TaKaRa公司;脉冲场凝胶电泳(PFGE)所需的Marker购自NEB公司.
1.2 方法
1.2.1 高分子量基因组DNA的制备
接种C.necator JMP134菌株于LB液体培养基培养至OD=0.8~1.0,加入终质量浓度为180 μg/μL的氯霉素培养 1 h.离心沉淀菌体,用 STE (100 mmol/L NaCl,10 mmol/L Tris-HCl,1 mmol/L EDTA,pH=8.0)洗涤菌体,然后用STE重悬,至终质量浓度约为109个细胞/mL.加入等体积的45℃预热的1%低熔点琼脂糖,混匀后分装入琼脂糖凝胶块模子(BIO-RAD Plug Mold)中,4℃放置10 min使其凝固.凝固后置于 5倍体积 Lysis buffer (10 mmol/L Tris-HCl,50 mmol/L NaCl,100 mmol/L EDTA,1%N-月桂酰肌氨酸钠,0.2%脱氧胆酸钠,1 mg/mL溶菌酶)中,37℃处理1 h.将Lysis buffer更换为ESP(1%N-月桂酰肌氨酸钠,0.5 mol/L EDTA,1 mg/mL蛋白酶K),50℃继续处理16 h[7].用TE(10 mmol/L Tris-HCl,1 mmol/L EDTA,pH= 8.0)洗涤胶块,并用苯甲基磺酰氟(PMSF)终止蛋白酶K的反应,最后置于0.1 mol/L EDTA中保存待用.
1.2.2 文库构建
在正式酶切之前,需预先对酶切条件进行摸索.将一个DNA胶块平均分成8份置于离心管中,加入30倍体积酶切缓冲液(不含BSA),冰上平衡1 h(每0.5 h更换一次缓冲液).吸出上清后,加入5倍体积的酶切缓冲液(1×HindⅢ酶切缓冲液,1/10 BSA)以及所需的酶,冰上平衡1 h,然后于37℃进行酶切反应45 min.设置酶量梯度,每管加入1/8 DNA胶块,用不同量的HindⅢ进行部分酶切.酶切结果经脉冲场凝胶电泳(BIO-RAD CHEF MAPPERTM)检测(1%琼脂糖凝胶,0.5×TBE,6 V/cm电压,40 s脉冲时间,14℃电泳16 h),选取能最大量地产生所需范围片段的酶切条件作为正式酶切条件.酶切反应结束后立即将反应管插入冰中,加入1/10体积的0.5 mol/L EDTA(pH=8.0)终止反应.
正式酶切时,视情况取适量的DNA(4~6个胶块,每块的DNA含量为5~10 μg),以确定的最佳条件进行部分酶切.先经第一轮脉冲场凝胶电泳(1%琼脂糖凝胶,0.5×TBE,6 V/cm电压,90 s脉冲时间,14℃电泳16 h)进行酶切片段的分离,再更换电泳条件进行第二轮电泳(1%琼脂糖凝胶,0.5× TBE,4 V/cm电压,5 s脉冲时间,14℃电泳5 h)压缩目标范围内的DNA,最后对照分子Marker切下含有100~300 kb DNA的凝胶块放入透析袋中,电洗脱回收所需的DNA片段[8].
通过与λDNA标准浓度比较确定得到的大片段DNA的浓度,按载体和插入片段的摩尔浓度比为5∶1的比例在50 μL连接体系中于16℃下连接10 h,后经65℃灭活连接酶,脱盐后进行电击转化[9](BIO-RAD Gene Pulser XcellTM电激仪,0.1 mm BIO-RAD电击杯,1 800 V/cm,200 Ω,25 μF).
1.2.3 文库插入片段大小检测
随机挑取96个克隆接种到含12.5 μg/mL氯霉素的LB培养基中培养过夜.用碱裂解法提取BAC质粒,NotⅠ酶切后,进行脉冲场凝胶电泳(1%琼脂糖凝胶0.5×TBE,6 V/cm电压,0.1~40 s脉冲时间,14℃电泳16 h)以确定插入片段的大小.
1.2.4 BAC文库的筛选
根据NCBI上所获得的C.necator JMP134苯酚羟化酶大亚基基因的phlN2序列,设计引物:jmp2f (5’-ATGGATACCCGCGTTGCCAAGAAGA-3’),jmp2r (5’-ACCGAAGGTGACGGTGCTCATGTCG-3’).随机挑取文库单克隆至96孔板(板内预加了200 μL含12.5 μg/mL氯霉素的LB液体培养基),37℃静置培养文库克隆过夜.将每块板的横列12个样品合并为1个样品,纵列8个样品合并为1个样品,因此每块板共得到20个样品.分别将这20个样品接种至3 mL含12.5 μg/mL氯霉素的LB液体培养基中培养,抽提BAC质粒,以提取的质粒DNA为模板,通过引物jmp2f/jmp2r进行PCR扩增,快速筛选含有目标基因的克隆.
1.2.5 目的条带序列测定
PCR扩增出的阳性DNA片段经PCR产物快速回收试剂盒(杭州Axygen公司)纯化后,与pMD18-T (TaKaRa公司)载体连接,转化大肠杆菌DH5α.挑取正确的转化子,抽提质粒,通过引物M13+(5’-CAGGAAACAGCTATGAC-3’)和M13-(5’-GTTTTCCCAGTCACGAC-3’)进行双向测序(测序仪为MegaBACE 4500 DNA Analysis System,Amersham Biosciences).
2 结果与分析
2.1 文库的构建
高质量BAC文库构建的关键步骤之一就是获得较高纯度的大分子量DNA.本研究采用直接包埋C.necator JMP134细胞的方法制备的大分子量DNA具有较高的DNA得率和质量(见图1),符合BAC文库构建的要求.
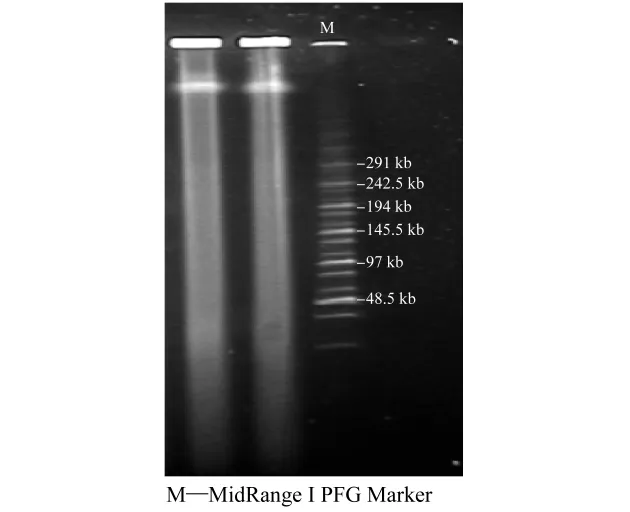
图1 C.necator JMP134基因组大分子量DNAFig.1 High-molecular-weight DNA of C.necator JMP134
为了确定所制备的含有DNA的琼脂糖凝胶块的最佳部分酶切条件,设计使用0,0.05,0.1,0.5,1,10 U不同酶量对1/8个琼脂糖凝胶块进行酶切实验,结果如图2所示(1~6分别为用0,0.05,0.1,0.5,1,10 U酶量对基因组DNA进行酶切).由图可见,1 U HindⅢ酶切产生的片段主要集中在100~300 kb之间,可确定其为最佳酶量.使用1 U酶量对4个胶块进行部分酶切,回收片段与载体连接后,灭活脱盐,电激转化,挑取9 600个阳性克隆,保存于100块96孔板中,形成 C.necator JMP134菌株的BAC文库.
2.2 文库的鉴定
为了鉴定文库平均插入片段的大小,随机挑取96个克隆,经NotⅠ酶切以及脉冲场凝胶电泳检测后发现,NotⅠ酶切后产生的条带较多,这给鉴定文库插入片段的大小带来了不便,其主要原因在于C.necator JMP134菌株的GC含量(64.43%)较高.为了更准确地确定文库插入片段的大小,在对 C.necator JMP134菌株的基因组进行分析后,选用XbaⅠ进行酶切(见图3),并对文库插入片段的大小进行了鉴定.

图2 最佳酶切条件的确定Fig.2 Determination of optimal partial digestion conditions
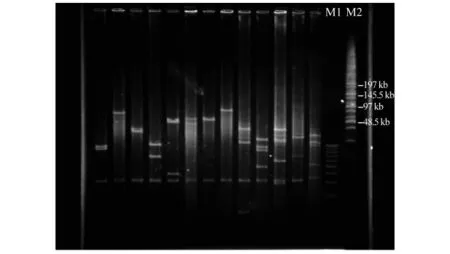
图3 BAC克隆XbaⅠ酶切鉴定Fig.3 Evaluation of insert size digested with XbaⅠ
XbaⅠ的酶切结果表明,99%的克隆含有插入片段,插入片段的大小分布在20~120 kb之间,平均大小约为65 kb.图4为BAC文库的插入片段大小分布图,其中约有61%的克隆插入片段大小大于50 kb.根据平均插入片段(约65 kb)和文库的总容量(约9 600个克隆)计算,此文库覆盖了约6 240 Mb的基因组序列.按照C.necator JMP134菌株基因组7.25 Mb的大小估算,此文库约覆盖了C.necator JMP134菌株基因组的860倍.
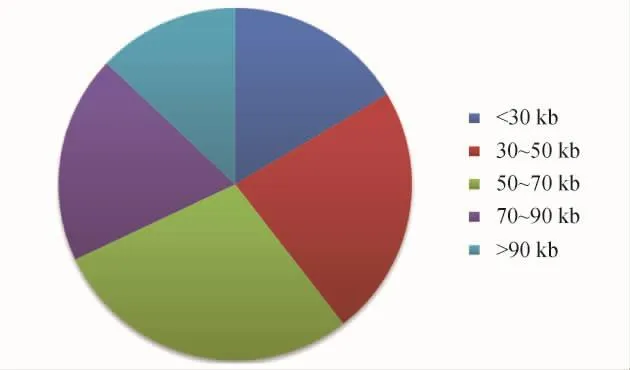
图4 C.necator JMP134 BAC文库插入大小分布Fig.4 Insert size distribution of C.necator JMP134 BAC library
2.3 文库的使用
为了进一步鉴定文库和验证筛选体系,本研究根据C.necator JMP134菌株的2号基因簇苯酚羟化酶大亚基基因 phlN2的序列,合成了特异引物jmp2f/jmp2r,并通过PCR筛选从文库随机的800个单克隆中筛选到了11个阳性克隆(见图5).对这11个阳性克隆的PCR扩增phlN2的产物序列进行了测定,结果显示,所有克隆的PCR扩增序列均与C.necator JMP134菌株基因组上phlN2基因的序列完全一致,表明所构建的文库克隆不存在基因组序列上的突变.用XbaⅠ酶对这11个克隆的插入片段大小进行了鉴定,脉冲场电泳结果表明,11个阳性克隆的平均插入片段大小在50 kb以上,能够满足筛选完整的苯酚降解基因簇片段的需要(见图6).同时也说明该文库能够用于快速、有效地分离大片段功能基因簇序列,验证了该BAC文库的有效性和筛选方法的优越性.
3 讨论
BAC载体克服了酵母人工染色体(yeast artificial chromosome,YAC)载体的不足,具有容量大、遗传稳定性好、重组DNA易分离等优点,近年来己经逐渐成为物理图谱构建、全基因组测序和基因图位克隆等研究中应用最广泛的载体系统之一[10].尽管BAC文库构建技术已经经历了近20年的发展,技术日趋完善,但是构建BAC文库仍然是一件费时费力的工作,在整个文库的构建过程中高分子量插入片段的制备是关键所在.为了尽量提高回收片段中具有酶切粘性末端的DNA片段的比例,首先,要选取合适的酶切条件;其次,由于载体有优先连接较小片段的趋势,因此,为了保持较大的平均插入,在回收的片段中应尽量去除小分子DNA,这需要通过脉冲场电泳来完成.但是由于DNA片段经常发生纠结缠绕,很难通过脉冲场彻底分离,所以不可避免地会在其中混有小片段.此外,过大的片段也不利于连接,会导致连接效率和克隆数的明显下降.C.necator JMP134为革兰氏阴性菌,细胞壁脆弱,将菌体直接包埋后裂解可得到较高浓度的高分子量DNA,为酶切获得高质量的连接片段提供了有力保障.
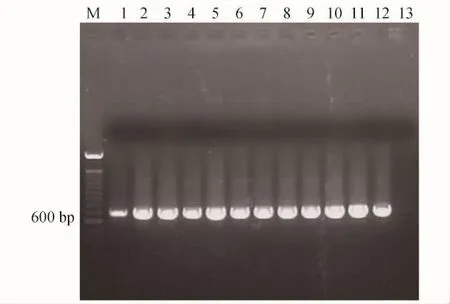
图5 C.necator JMP134 BAC文库中11个phlN2阳性克隆的PCR扩增图谱Fig.5 PCR amplification of the 11 phlN2 positive clones
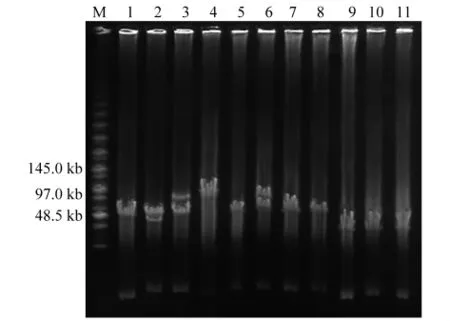
图6 C.necator JMP134的 11个 phlN2阳性克隆XbaⅠ酶切图谱Fig.6 XbaⅠrestriction map of the 11 phlN2 positive clones of C.necator JMP134
许多研究者在构建BAC文库时多采用两次分离回收的方法[11-12].陈凡国等[13]对两种选择方法进行了比较,发现在其他条件相同的情况下,两次选择法得到的DNA连接(以100~150 kb为例)的转化效率较高,转化后插入片段较整齐,空载率低,但是得到的用于连接的大片段DNA浓度较低,浪费严重;而一次选择法虽然克服了两次选择法的缺点,但转化后的平均插入片段较小,空载率也很低.本研究采用一次分离回收的方法,同样取得了较大的外源片段插入,因此,采用适当的分离程序对酶切后的片段进行分离并选取较大的片段范围回收,同样可以提高外源片段的平均插入大小,获得较理想的结果.
转化也是文库构建中很重要的实验步骤,直接决定了最后所得文库的克隆数目.转化用的感受态细胞应尽量避免冻融,因为冻融一次后,感受态的效价往往会大幅降低.在转化操作的整个过程中,动作要快,应在电击后迅速加入恢复培养基,其中从电击到向电击杯中加入培养基的时间非常关键.耿波等[10]研究发现,拖延 1 min将使转化效率降低67%,而拖延10 min将使转化效率降低95%.此外,将连接产物灭活可以提高转化效率,转化条件也可以影响文库插入片段的大小[14].
综上所述,根据实验数据,本研究所构建的BAC文库约覆盖了C.necator JMP134菌株基因组的860倍,因此,整个文库具有很好的基因组覆盖率.本研究成功构建了高质量、高覆盖率的C.necator JMP134菌株的 BAC文库,理论上筛选出任一C.necator JMP134基因的概率为99.99%,适合于单基因的克隆和分离,为进一步对苯酚降解基因簇,以及该菌株其他重要生理代谢途径相关基因簇的克隆和功能分析打下了坚实基础.
[1] PEREZ-PANTOJAD,DELAL R,PIEPERD H,et al.Metabolic reconstruction of aromatic compounds degradation from the genome of the amazing pollutantdegrading bacterium Cupriavidus necator JMP134[J].FEMS Microbiol Rev,2008,32(5):736-794.
[2] FUKUMORIF,HAUSINGERR P.Alcaligenes eutrophus JMP134“2,4-dichlorophenoxyacetat emonooxygenase”is an alpha-ketoglutarate-dependent dioxygenase[J].J Bacteriol,1993,175(7):2083-2086.
[3] FARHANAL,NEW PB.The 2,4-dichlorophenol hydroxylase ofAlcaligeneseutrophusJMP134 is a homotetramer[J].Can J Microbiol,1997,43(2):202-205.
[4] KIMY,HARKERA R.Cloning and characterization of the regulatory genes phlR1 and phlR2 involved in phenol metabolism from Alcaligenes eutrophus JMP134[J].Mol Cells,1997,7(5):620-629.
[5] KIMU J,SHIZUYAH,DEJONGP J,et al.Stable propagation of cosmid sized human DNA inserts in an F factor based vector[J].Nucleic Acids Res,1992,20 (5):1083-1085.
[6] SHIZUYAH,BIRRENB,KIMU J,et al.Cloning and stablemaintenanceof 300-kilobase-pairfragmentof human DNA in Escherchia coli using an F-factor-based vector[J].Proc Natl Acad Sci USA,1992,89(18):8794-8797.
[7] STEIN JL,MARSH T L,WU K Y,etal.Characterization of uncultivated prokaryotes:isolation and analysis of a 40-kilobase-pair genome fragment from a planktonic marine archaeon[J].J Bacteriol,1996,178(3):591-599.
[8] LUOM,WINGR.An improved method for plant BAC library construction[M].Totowa,NJ:Humana Press Inc,2003:3-20.
[9] ZHANGH B,CHOIS,WOOS,et al.Construction and characterization of two rice bacterial artificial chromosome libraries from the parents of a permanent recombinant mapping population[J].Mol Breed,1996,2(1):11-24.
[10] 耿波,关云涛,孙效文.黑龙江野鲤细菌人工染色体基因组文库构建[J].中国水产科学,2009,16(2):165-172.
[11] NAMY W,LEEJ R,SONGK H,et al.Construction of two BAC libraries from cucumber(Cucumis sativus L.) and identification of clones linked to yield component quantitative trait loci[J].Theor Appl Genet,2005,111 (1):150-161.
[12] 孟祥宗,杨晓武,管祯玮,等.盐藻(Dunaliella viridis)细菌人工染色体文库的构建和鉴定[J].上海大学学报:自然科学版,2007,13(1):88-93.
[13] 陈凡国,姜涛,张学勇.BAC文库构建中的几个技术问题探讨[J].生物技术,2002,12(2):28-29.
[14] SHENGY,MANCINOV,BIRRENB.Transformation of Escherichia coli with large DNA molecules by electroporation[J].Nucleic Acids Res,1995,23(11):1990-1996.
