大菱鲆亲子鉴定的微卫星多重PCR技术建立及应用*
2011-01-10苗贵东杨景峰樊廷俊陈松林
苗贵东,杜 民,杨景峰,徐 营,樊廷俊,陈松林
(1.中国海洋大学海洋生命学院,山东青岛266003;2.中国水产科学研究院黄海水产研究所,山东青岛266071;3.上海海洋大学水产与生命学院,上海201306)
大菱鲆(Scophthalm usm axim us)在中国又称“多宝鱼”,是原产于欧洲沿海的一种名贵经济鱼种,隶属于硬骨鱼类(Osteichthyes)、鲽形目(Pleuronectiformes)、鲽亚目(Pleuronectoidei)、鲆科(Bothidae)、菱鲆属(Scophthalm us)。1992年引进我国,1999年突破生产性育苗技术,随后大菱鲆养殖业在我国得到迅速发展,现已成为我国北方沿海重要的鱼类养殖品种。由于累代养殖和近亲交配等因素,良种选育已经成为我国大菱鲆养殖产业健康、可持续发展亟待解决的重要课题之一[1-2]。
家系选育是获得优良品种的重要手段。家系选育过程中,保持完整、准确的系谱信息可
以有效的指导亲本选留,从而避免近交衰退现象。大菱鲆良种培育中,为了实现家系遗传参数的准确评估,需要早期对不同家系进行混养,运用微卫星标记辅助管理与物理标记相比可以显著降低遗传育种中环境效应因素的影响[3-4]。利用微卫星技术进行亲子鉴定和遗传分析已在真鲷(Chrysophrysm ajor)[3]、大西洋鳙鲽(H ippog lossus hippog lossus)[4]、牙鲆(Paralichthys olivaceus)[5]、虹鳟(Oncorhynchusm ykiss)[6]、中国对虾(Penaeus chinensis)[7-8]、塞内加尔鳎(Solea senegalensis)[9]等海洋经济动物中有相关报道。由于多重PCR(M utip lex PCR)技术具有高效、高通量、检测成本低等优点,自1988年Cham bercian等[10]首次建立这一技术起,该技术就已被广泛应用于多病毒、细菌高通量快速检测[11-12]、群体遗传分析[13]、亲子鉴定[14]及数量性状基因座位(Q TL)的定位[15]等研究。
Castro[16]和于非[17]等应用微卫星技术对大菱鲆亲子鉴定及系谱构建进行初步研究,能够完成对大菱鲆的亲子鉴定工作,但现有方法存在检测通量低、检测工作量大、成本高、实现分子标记辅助家系管理难度大等问题,无法在生产中推广应用。目前有关大菱鲆简便、高通量亲子鉴定和系谱认证方法未见报道。本研究建立的微卫星多重PCR技术将微卫星标记技术的稳定性与多重PCR技术的高通量特点结合,建立了大菱鲆的两个微卫星多重PCR体系,与现有方法相比,实现了对大菱鲆家系开展高效、高通量的亲子鉴定目的。同时建立的大菱鲆非荧光标记微卫星多重PCR,克服常用的微卫星多重PCR[13,15]多采用荧光标记微卫星引物,需要专用设备、仪器环境要求高,无法在养殖基地等潮湿环境检测等缺点,能够用于养殖基地现场亲子鉴定和系谱认证,可以用于分子标记辅助良种选育工作。
1 材料与方法
1.1 实验材料
实验用大菱鲆来自山东明波水产有限公司,2009年建立大菱鲆家系7个,家系编号分别为1#、2#、3#、4#、5#、6#、7#,各家系数量大约500尾,随机选取各家系35尾鱼苗提取DNA,其中每个家系取20尾鱼,用于建立亲子鉴定多重PCR体系。另从每个家系中随机取样10尾用于双盲实验,其中3#家系母本缺失,7#家系无父母本信息。
1.2 研究方法
1.2.1 基因组DNA提取 取大菱鲆鳍条组织,利用传统的酚、氯仿法提取大菱鲆基因组DNA[18]。1%琼脂糖凝胶电泳检测基因组DNA完整性,利用RNA/DNA分光光度仪(Pharmacia Biotech L td.)进行基因组DNA浓度的检测并稀释成100 ng/μL终浓度,-20℃保存备用。
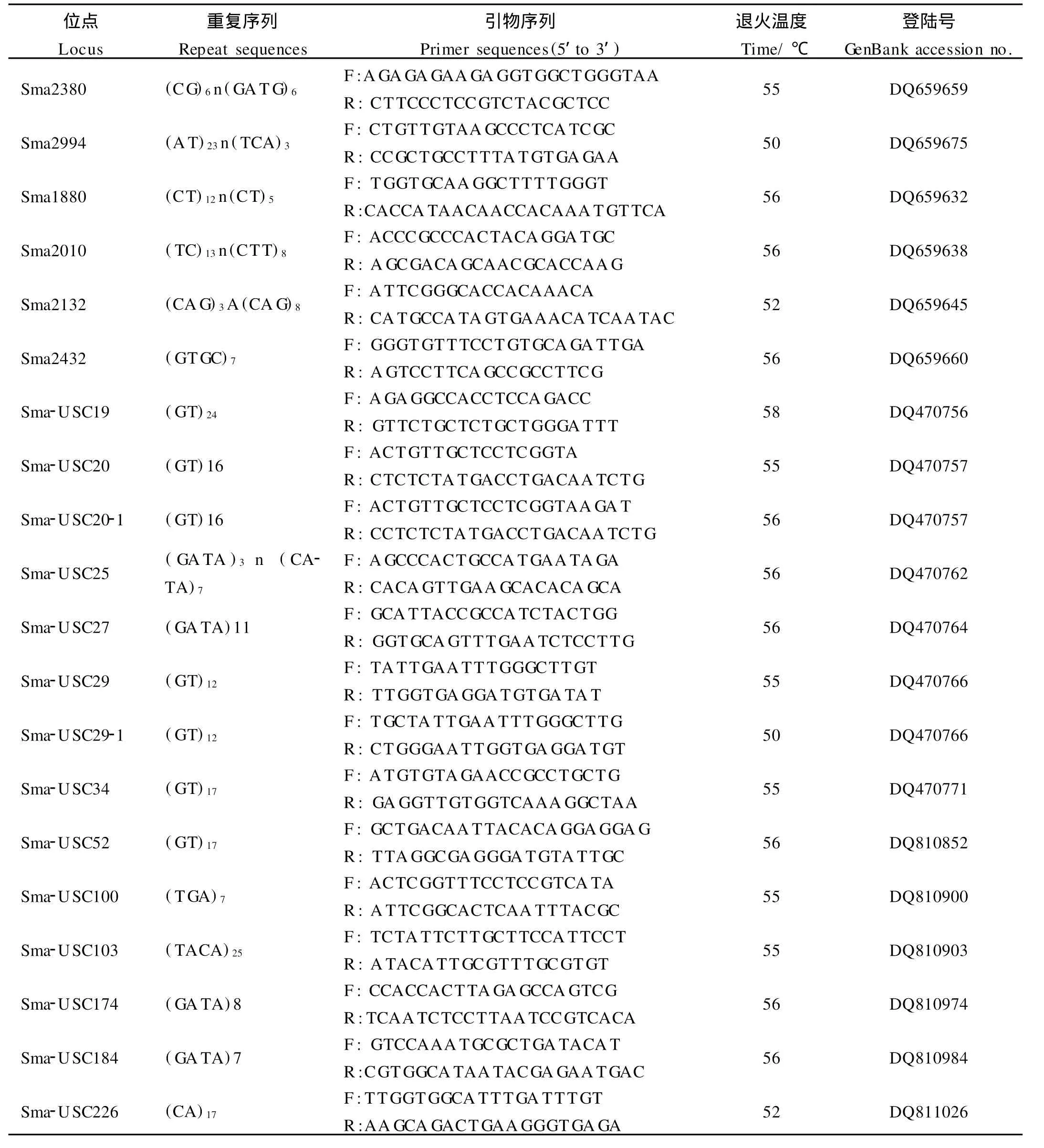
表1 18对大菱鲆微卫星引物序列及其反应条件Table 1 18 p rimer pair sequences and PCR reaction conditionsof S.maxim us
1.2.2 多重PCR设计 选用Sma2380、Sma2994、Sma1880、Sma2010、Sma2132、Sma2432、Sma-USC19、Sma-USC20、Sma-USC25、Sma-USC27、Sma-USC29、Sma-USC34、Sma-USC52、Sma-USC100、Sma-USC103、Sma-USC174、Sma-USC184、Sma-USC226共18对微卫星引物(见表1)在7个家系11个已知亲本中进行PCR扩增,从中筛选出多态性引物13对,这13对引物都能够扩增获得3个及3个以上等位基因的清晰条带。利用Primer Prem ier 5.0软件进行多重PCR引物组合设计,为了满足多重PCR对预期片段大小及反应退火温度的要求,对13个位点中的部分位点依据其序列对引物进行了重新设计,并分别命名为Sma-USC20-1和Sma-USC29-1。13个位点设计的3个多重PCR组合分别为Sma-USC25、Sma-USC20-1、Sma-USC100、Sma-USC103、Sma-USC29-1;Sma-USC27、Sma-USC37、Sma-USC100、Sma-USC184;Sma2380、Sma2994、Sma-USC19、Sma1880、Sma-USC226。经过筛选去除扩增效率低的引物组合及各组合中扩增效率低的引物,多重PCR条件优化主要是指优化各组合中每对引物在体系中的最适宜的反应浓度和获得2个多重PCR组合反应中最佳退火温度和最佳循环次数。
1.2.3 PCR产物的处理 PCR产物通过6%变性聚丙烯酰胺凝胶电泳进行分离,银染,固定。将固定后的聚丙烯酰胺胶晾干后进行基因型统计。
2 结果与分析
2.1 微卫星位点遗传多样性分析
2个多重PCR反应体系中各位点(除Sma-USC100,Sma226外)多态信息含量(PIC)均大于0.5,均为高度多态性微卫星标记,各位点等位基因数也较多,观测杂合度较高,比较适合亲子鉴定分析和群体遗传评。
2.2 多重PCR结果
设计的引物组合经过筛选后获得2个稳定的四重PCR体系。微卫星位点组合分别为Sma-USC25、Sma-USC100、Sma-USC103、Sma-USC29-1和Sma2380、Sma2994、Sma-USC19、Sma226。经条件优化后最终获得PCR反应体系和PCR反应条件如下:
第一组多重PCR的微卫星位点组合为Sma-USC25、Sma-USC100、Sma-USC103、Sma-USC29-1。反应体系为每个PCR反应总体积15μL,包括100 ng基因组DNA,1×PCR缓冲液,Mg2+(1.5 mmol/L),1U Taq酶,dNTPs 0.2 mmol/L。引物组合Sma-USC25、Sma-USC100、Sma-USC103、Sma-USC29-1,上、下游引物分别为0.25μmol/L。PCR反应条件为:95℃变性5 min;95℃45 s,58℃50 s,72℃50 s,10个循环;95℃30 s,55.5℃50 s,72℃50 s,25个循环;95℃30 s,50℃50 s,72℃50 s,10个循环,72℃延伸5 min,10℃保存。
第二组多重PCR的微卫星位点组合为Sma2380、Sma2994、SmaUSC19、Sma226。反应体系为每个PCR反应总体积15μL,包括100 ng基因组DNA,1×PCR缓冲液,Mg2+(1.5 mmol/L),1U Taq酶,dNTPs 0.2 mmol/L。引物组合Sma2380、Sma2994、Sma-USC19、Sma226,上、下游引物0.25μmol/L、0.3μmol/L、0.25μmol/L、0.25μmol/L。PCR反应条件为:95℃变性5 min;95℃45 s,57℃50 s,72℃50 s,10个循环;95℃30 s,55℃50 s,72℃50 s,25个循环;95℃30 s,50℃50 s,72℃50 s,10个循环,72℃延伸5 min,10℃保存。
2.3 多重PCR结果
PCR经电泳后,根据已知亲本及产物片段大小进行基因型统计(见图2),以pBR322/M spⅠM arker(天根生化有限公司)确定等位基因大小(见图1)。

图1 第一组多重PCR体系亲本丙烯酰胺凝胶电泳图(显示其中2个位点)Fig.1 Acrylamide gel electrophoresis of know n Families’parent of No.1 multiplex PCR(Shown two loci)

图2 部分双盲个体丙烯酰胺凝胶电泳图(显示其中2个位点)Fig.2 The partial acrylamide gel electrophoresis of double-blind experiment in 70 trout(Show n two loci)
2.4 聚类分析
利用N TSYS-pc2.1软件,计算7个家系140个子代个体和70个双盲子代个体的遗传距离(GD),分别得到140个大菱鲆个体遗传距离矩阵(140×140)和70个双盲数据的遗传距离矩阵(70×70)。根据个体间的遗传距离,利用N Tsys2.1和M EGA 4.0软件按UPGM A法进行聚类分析。140个已知家系信息个体的聚类分析结果表明:136个个体的聚类结果与已知家系信息相一致,聚类准确率为97.14%;70个双盲个体的聚类分析结果与真实结果比对后有66个个体能够准确确定其父母,4个个体聚类错误,聚类分析准确率为95.71%(见图3,4)。分析后发现聚类错误个体均存在某些位点未扩增出条带现象,从而导致无法获得基因型。

图3 140个体NTsys聚类分析图(显示40个个体)Fig.3 The partial cluster analysis diagram in 140 turbot(Show n 40 individuals)
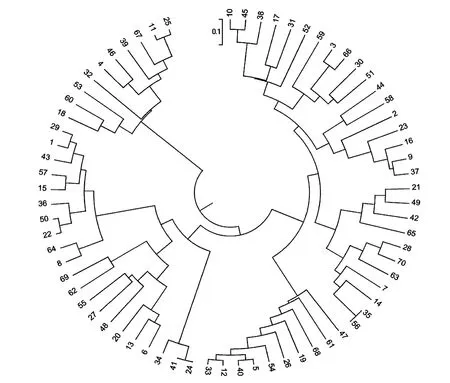
图4 70个双盲个体聚类分析结果(Mega4.0)Fig.4 Cluster analysis of 70 double-blind individuals by Mega4.0 software
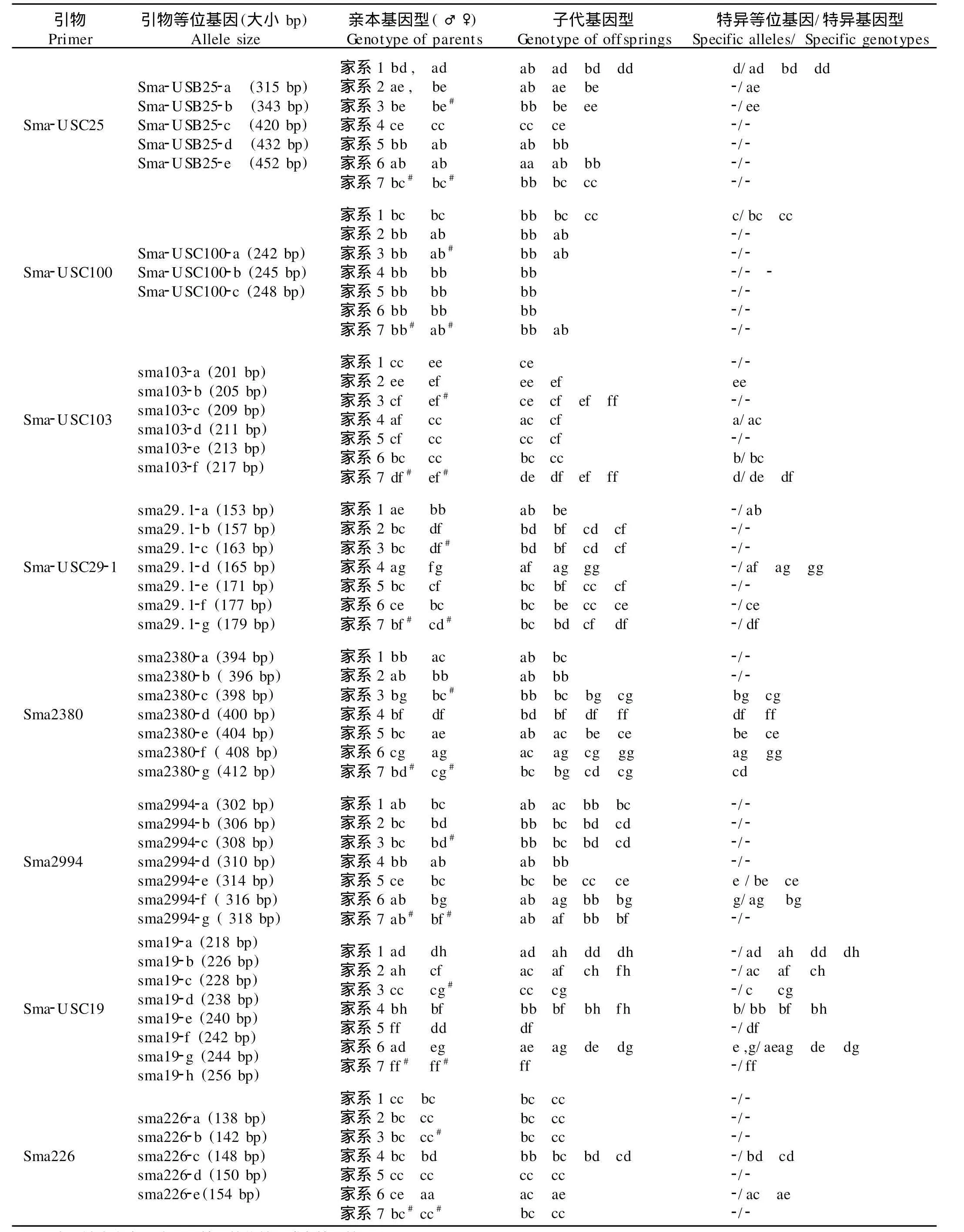
表2 各微卫星引物基因型信息及家系特异等位基因/特异基因型组合Table 2 Genotype of 8 microsatellite loci and specific alleles or specific genotypes in 7 families
2.5 推断缺失亲本的基因型
大菱鲆亲本和家系个体基因型的命名方法依据各引物扩增条带大小。从小到大依次对每个等位基因排序为a,b,c…,如Sma-USC25扩增获得目的片段大小分别为315 bp、343 bp、420 bp、432 bp、452 bp则5个条带分别命名为Sma-USC25-a、Sma-USC25-b、Sma-USC25-c、Sma-USC25-d、Sma-USC25-e,其他引物命名方法与此相同(见表2)。由于微卫星标记符合孟德尔遗传规律,由已知亲本基因型和家系个体基因型可以推断出缺失亲本的基因型(见表2)。
2.6 多重PCR亲子鉴定排除率分析和亲子鉴定准确率分析
运用软件N Tsys分别对140个已知家系信息个体进行系谱认证及70个双盲个体进行系谱认证分析,将140个子代个体和70个双盲子代个体基因型数据转换为软件N Tsys2.1所需的(0,1)矩阵格式,分别得到140个个体遗传距离矩阵(140×140)和70个双盲数据的遗传距离(GD)矩阵(70×70)。根据个体间的遗传距离,分别利用N TSYS-pc2.1和M EGA 4.0软件按UPGMA法进行聚类分析,140个个体和70个双盲个体聚类分析结果表明,分别有97.14%和95.71%与预期的家系信息相一致(见图3,4)。结果表明2个多重PCR可以实现对7个家系个体的系谱认证。通过对7个家系140个个体遗传参数的计算,得到各引物的期望杂合度、观测杂合度、多态信息含量及排除率(表3):在双亲未知的情况下2个多重PCR的8个微卫星位点累积排除概率为96.58%,已知单亲基因型时累积排除概率为99.71%。亲子鉴定的准确率为96.42%。亲子鉴定分析利用Cervus2.0软件,依次进行等位基因的频率分析、模拟分析(Simulation)和亲子分析(Parentage analysis),对随机取样的70个个体双盲进行实验,结果为:在双亲未知时累积排除率为95.72%,已知1个亲本时累积排除率为99.61%。亲子鉴定结果为:92.86%的个体能够准确找到各自父母(见表4)。

表3 微卫星各位点在家系中遗传信息Table 3 Info rmation of themicrosatellite sites
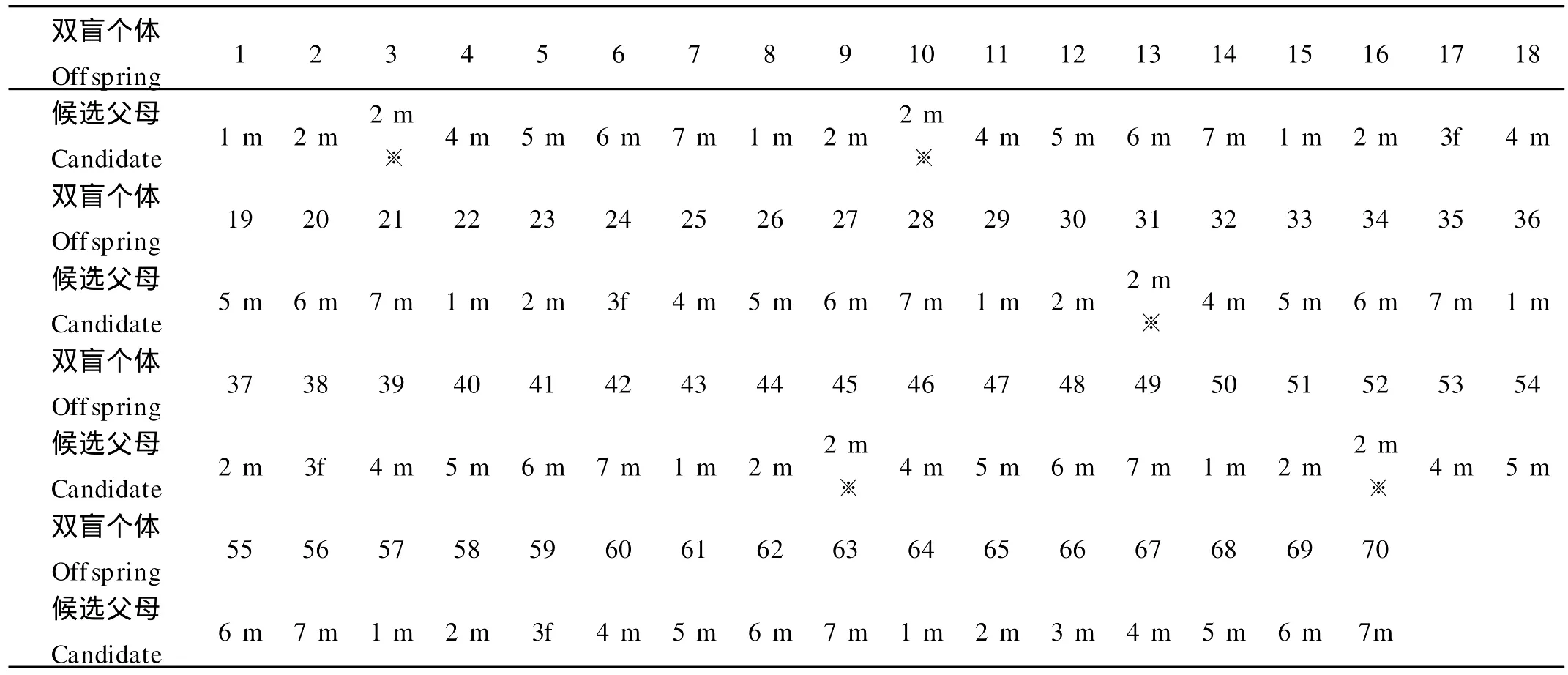
表4 70个双盲个体亲子鉴定结果Table 4 The result of parentage determination in 70 individuals
3 结论与讨论
目前在水产养殖中个体的父母信息不能直接获得,运用分子手段实现个体亲子鉴定和系谱追踪对家系选育和辅助亲本管理提供重要帮助,目前微卫星标记是实现上述目的较为理想的标记。[19]利用微卫星位点的多态性,以排除概率方法进行亲子鉴定和系谱认证,是明确个体间亲缘关系、建立系谱关系的重要手段。Ellegren等[20]研究表明,使用5个微卫星位点组合(每个位点有6个以上的等位基因)时排除率为98%,而使用10个这样的微卫星组合时排除率可高达99.99%。Castro[16]研究结果发现只有一个单亲基因型记录时,要达到95%以上的累积排除概率至少需要8个中的4个多态微卫星位点,而亲本基因型均未知时,则至少需要8个中的7个多态位点。Cervus2.0软件评估了本实验2个多重PCR体系中8个微卫星位点在双亲未知情况下的累积排除概率是96.58%,而已知1个亲本时的累积排除概率达99.71%。这8个微卫星位点平均等位基因数是6.125,其中Sma-USC29-1、Sma2380、Sma19 3个位点超过了6个等位基因,且平均PIC值为0.6558,表明2个微卫星多重PCR体系总体已达到高度多态性水平,其累积排除率达到96.42%。这与上述2个结果基本一致。同时又从7个家系随机选取70尾个体进行双盲实验,利用Cervus2.0软件分析双亲未知时的排除率为95.72%,已知1个亲本时的排除率为99.61%,表明本研究建立的由8个微卫星位点组合成的2个多重PCR体系可以实现比普通PCR方法更高效的亲子鉴定分析。已建立的2个多重PCR体系理论上的排除率与双盲实验结果接近,也与已发表的结果基本一致[4-5,7,17],表明本研究建立的2个多重PCR体系具有较好的稳定性和可靠性。N Tsys2.1和M ega4.0对70尾鱼双盲分析表明,其群体水平的聚类分析准确率为95.71%,4尾鱼的聚类结果出现偏差的原因是,该4个个体均存在1个或几个位点无扩增条带现象,也可能与某些位点等位基因差别较小而产生基因型统计误差、有些引物在某些个体可能存在PCR扩增中引物滑动现象有关。70尾鱼双盲鉴定结果亲子鉴定准确率92.86%,能够满足实际生产对家系管理的需要。Lerceteau-K˚hler[14]使用10个微卫星位点组合成的1个六多重PCR和1个四重PCR用于700尾河鳟(Salmo trutta)养殖群体和野生群体的筛选,只需14块左右96孔板进行PCR扩增,而普通引物方法筛选则需要使用大约70块96孔板进行PCR扩增,上述10个引物组合的2个多重PCR效率约为普通方法的5倍,同时减少多次重复性工作,与本实验2个四重PCR检测效率比现有的简单PCR方法提高2~3倍,检测费用大大减少的结果相一致。
目前研究中采用的多重PCR技术,多采用具有荧光标记的引物进行多重PCR体系的设计和筛选,该方法建立的多重PCR由于不同引物采用不同颜色的荧光标记[14,21-22],只需考虑引物间相容性,各引物扩增片段大小可以交叉重叠,因此常见的多重PCR体系容易建立,但引物需要预先进行荧光标记、检测荧光信号需要专有设备,检测成本较高。水产生物育种和生产中环境湿度大,环境湿度过大使仪器设备维护困难,易造成荧光信号检测误差甚至无法获得检测结果,因此常用的荧光标记多重PCR方法很难在养殖基地推广应用。本研究从生产实际需要出发,采用无荧光标记的普通微卫星引物筛选建立了2个多重PCR体系,利用常规聚丙烯酰胺凝胶电泳设备进行亲子鉴定分析,对环境要求低,应用范围更加广泛,可以实现养殖基地现场快速、高通量的亲子鉴定分析,也可用于家系群体遗传多样性评价、分子辅助家系管理及分子辅助亲本选择。
多重PCR体系建立主要解决的问题是选择合适微卫星位点进行组合和PCR反应条件的
优化,本研究采用以下基本原则进行试验设计:①多重PCR引物组合的各引物间有较好的相容性,要求同一引物内上、下游及各引物之间均不形成二聚体、发夹结构等,若无法避免也要求其稳定性较低。②尽可能避免引物之间的竞争抑制现象,各引物在同一PCR反应体系中扩增效率相近。③尽可可能使用多碱基重复类型微卫星位点组合,如四碱基重复序列的微卫星位点与二碱基重复序列的微卫星位点相比一般不产生或较少出现影子带(Stuttering)[23-24],能够减少基因型统计错误。④尽可能使用完美型或者接近完美型的微卫星标记,因为这样的微卫星位点组合更接近于SMM突变模型,减少由于无效等位基因产生而出现杂合子缺失的现象[25]。由于本研究使用非标记引物建立多重PCR体系,在遵循常规多重PCR设计的基本原则基础上还要考虑到便于基因型统计,从而选取的各位点的扩增产物片段大小需要存在一定间隔。本实验采用4~6个引物组合时各引物预期扩增片段大小分别为大于300 bp、240~300 bp、200~240 bp、160~200 bp、120~160 bp和小于120 bp。
由于限制因素多,本研究中在多重PCR引物的选择上难度比较大,可选引物数量较少,除选用3~4碱基重复序列(Motif)微卫星标记外,同时辅以部分扩增条带清晰的2碱基重复序列的微卫星标记。本研究首先设计4~6个引物的PCR体系进行筛选和条件优化,剔除组合中扩增效率低的引物,最终获得2个较为稳定的四重PCR体系。为了更好的进行基因型统计分析并减少统计误差,消除不同批次凝胶电泳扩增产物片段大小的统计误差,并且pBR322/M spⅠM arker分子量标准并非均匀分布,为保证每批检测样本基因型统计结果的可比性,同时使用已知亲本位于待测样本两侧和pBR322/M spⅠ双重分子量标准辅助进行基因型分析,使基因型统计准确率更高,在本研究中取得了较好的效果。
实验结果显示7个家系中某些位点等位基因数少,遗传多态性较低,家系间遗传距离较小,亲缘关系较近。这可能是因为我国不是大菱鲆的原产地,引进原种亲缘关系较近的原因。由于鱼类后代众多,管理难度大,利用微卫星多重PCR对引进原种进行分析来确定亲本亲缘关系、利用分子标记手段辅助选择苗种亲本以及家系选育中引入分子标记手段辅助家系管理和群体管理,与运用表型性状进行传统选育方法相比,能够有效减少由于近交导致的遗传多样性下降的现象,这将加快选择育种进程,有望缩短培育优良品种(品系)时间,能够有效促进大菱鲆养殖产业的持续健康发展。
总之,本研究建立的2个微卫星多重PCR体系与现有的简单PCR方法相比,效率提高了2~3倍,检测成本大大降低。同时建立的基于非荧光标记引物的2个多重PCR体系与常用荧光标记引物多重PCR相比,无需特殊检测设备,适用范围更广,能够实现在养殖生产基地现场检测。本研究建立的2个多重PCR体系实现了通过已知亲本和子代的基因型来推断未知的3个亲本的基因型;通过群体聚类分析的结果获得家系系谱关系;对70个个体进行双盲实验,亲子鉴定准确率达92.86%,与已知个体信息的鉴定结果(96.42%)相近,表明该方法具有很好的可靠性。以上结果表明建立的2个多重PCR体系能够高效的对大菱鲆进行群体遗传学评价、系谱认证、亲子鉴定,也能够用于分子标记辅助家系管理和分子标记辅助亲本选择。
[1] 雷霁霖,马爱军,陈超,等.大菱鲆(Scophthalmusmaximus)养殖现状与可持续发展[J].中国工程科学,2005,7(5):30-34.
[2] 申雪艳,孔杰,宫庆礼,等.大菱鲆种质资源研究与开发[J].海洋水产研究,2005,26(6):94-100.
[3] Perezenriquez R,Takagi M,Taniguchi N.Genetic variability and pedigree tracing of a hatchery reared stock of red sea bream(Pagrusmajor)used for stock enhancement,based of microsatellite DNA markers[J].Aquaculture,1999,173:413-423.
[4] Jackson T R,Martin Robichaud D J,Reithm E.Application of DNA markers to themanagement of A tlantic halibut(H ippog lossus hippoglossus)broodstock[J].Aquaculture,2003,220:245-259.
[5] Sekino M,Sugaya T,Hara M,et al.Relatedness inferred from m icrosatellite genotypes as a tool for broodstock management of Japanese flounder Paralichthys olivaceus[J].Aquaculture,2004,233:163-172.
[6] Mcdonai D G J,Danzmann R G,Ferguson M M.Relatedness determination in the absence of pedigree information in three cultured strains of rainbow trout(Oncorhynchusm ykiss)[J].Aquaculture,2004,233:65-78.
[7] Dong SR,Kong J,Zhang T S,et al.Parentage determination of Chinese shrimp(Fenneropenaeus chinensis)based on microsatellite DNA markers[J].Aquaculture,2006,258:283-288.
[8] 董世瑞,孔杰,张庆文,等.微卫星分型方法进行中国明对虾家系系谱鉴定[J].海洋学报,2006,28(3):1-5.
[9] Castro J,Pino A,Hermida M,et al.Amicrosatellitemarker tool for parentage analysis in Senegal sole(Solea senegalensis):genotyping errors,null alleles and conformance to theoretical assumptions[J].Aquaculture,2006,261:1194-1203.
[10] Chambercian J S,Gibbs R A,Ranier J E,et a1.Detection screening of the duchennemuscular dystrophy locus via multiplex DNA amplification[J].Nucl Acids Res,1988,16:1141-l156.
[11] Ponce M R,Robles P,Micol J L.High-throughout geneticmapping in Arabidopsos thaliana[J].Mol Gen Genet,1999,261:408-415.
[12] Rosenfield S I,Jaykus L A.Amultiplex reverse transcription polymerase chain reaction method for the detection of food bo rne viruses[J].J Food Prot,1999,62:1210-1214.
[13] Li Yutao,Wongprasert K,Shekhar M,et al.Development of two microsatellite multiplex system s for black tiger shrimp Penaeusmonodon and itsapplication in genetic diversity study for two populations[J].Aquaculture,2007,266:279-288.
[14] Lerceteau-K˚hler E,Weiss S.Development of a multiplex PCR microsatellite assay in brow n trout Salmo trutta,and itspotential application for the genus[J].Aquaculture,2006,258:641-645.
[15] 邹浪萍,杨燕,褚嘉祐,等.多重PCR检测CSF1PO,TPOX和THO1基因座在中国汉族中的多态性[J].遗传学报,1998,25(3):199-204.
[16] Castro J,Bouza C,Presa P,et al.Potential sources of error in parentage assessment of turbot(Scophthalmus maximus)using microsatellite loci[J].Aquaculture,2004,242:119-135.
[17] 于飞,王伟继,孔杰,等.微卫星标记在大菱鲆(Scophthalmus maximus)家系系谱印证中的应用研究[J].海洋学报,2009,31(3):841-847.
[18] Chen S L,M a H Y,Jiang Y.Isolation and characterization of polymorphic microsatellite loci from an EST-library of turbot(Scophthalmus maximus)and cross-species amplification[J].Molecular Ecology Notes,2007,7:848-850.
[19] Goldstein GD,Schlotterer C.Microsatellites:Evolution and app lication[M].New York:Oxfoxd Univ.Press,1999.
[20] Ellegren H.DNA typing of museum birds[J].Nature,1991,354(6349):113.
[21] Visscher P M,Woolliam s J A,Smith D,et al.Estimation of pedigree errors in the U K dairy population using microsatellite makersand the impacton selection[J].Journal of Dairy Science,2002,85:2368-2375.
[22] Po rta J,Porta JM,M art nez-Rodr guez G.Development of am icrosatellite multiplex PCR for Senegalese sole(Solea senegalensis)and its application to broodstock management[J].Aquaculture,2006,256:159-166.
[23] Edwards A,Hammond H A,Jin L,et al.Chakrabo rty R,Genetic variation at five trimeric and tetrameric tandem repeat loci in four human population groups[J].Genomics,1992,12:241-253.
[24] U rquhart A,Oldroyd N J,Kimp ton C P,et al.Highly discriminating heptaplex short tandem repeat PCR system for forensic identification[J].Bio Techniques,1995,18:116-121.
[25] Estoup A,Co rnuet J M.Microsatellite evolution:inferences from population data[M].∥Goldstein D B,Schl˚tterer C.Microsatellites:Evolution and Applications.Oxford:Oxford University Press,1999:49-65.
