温泉环境宏基因组文库中醛脱氢酶基因的克隆及分析
2010-11-22王秋岩魏东芝
谌 容,王秋岩,杨 兵,魏东芝,谢 恬*
(1.杭州师范大学 生物医药与健康研究中心,浙江 杭州 310012;2.生物反应器工程国家重点实验室,华东理工大学 鲁华生物技术研究所,上海 200237)
醛脱氢酶(ALDH)能催化脂肪族或芳香族醛类化合物的氧化,生成相应酸的一类超家族,有宽的底物特异性.甲基丙二酸半醛脱氢酶(Methylmalonate semialdehyde dehydrogenase,EC 1.2.1.27,MMSDH)属于醛脱氢酶超家族,催化丙二酸半醛或甲基丙二酸半醛的不可逆氧化脱羧,同时还原NAD+和合成乙酰基或丙酰-CoA[1].MMSDH是此家族中唯一的,因为此反应中必须需要CoA[2-3].在微生物和高等动物中,MMSDH催化的反应是缬氨酸和嘧啶分解代谢途径的末端部分,许多研究表明,缬氨酸代谢为大环内酯类抗生素和聚醚酸的形成提供脂肪酸前体起到了重要的作用[4].MMSDH由于耦合的醛脱氢酶活力和独特的CoA酯类合成能力吸引了众多研究者的兴趣.从老鼠肝脏中纯化的MMSDH有酯酶的活性,能水解p-硝基苯乙酸[5-6].来自芽孢杆菌[7]、天蓝色链霉菌[8]、绿脓假单胞菌[9]的MMSDH等已经有了较详细的研究.
宏基因组(Metagenome)是由Handelsman等1998年提出的,它包含了比可培养微生物大得多的遗传信息,其定义为生境中全部微小生物遗传物质的总和.宏基因组技术将人们对微生物基因多样性的认识再次扩大了上百倍[10].宏基因组中分离新基因主要通过基于酶活性的筛选和基于序列同源性的筛选两种方法,各有优劣[11].通过免培养-PCR技术成为替代的新酶发现策略[12].因此,为了获得更多微生物的MMSDH信息,从温泉宏基因组中采用免培养-PCR技术结合基因组步移技术从培养微生物宏基因组中进行MMSDH基因的克隆与分析.
1 材料与方法
1.1 材 料
微生物样本采集于广西九曲温泉水及水下的泥土,水温为68 ℃.
1.2 试 剂
E.coliDH5α为该实验室保存;T4 DNA连接酶、Taq DNA聚合酶、Taq酶、克隆载体pMD19-T为TaKaRa公司产品.土壤DNA抽提试剂盒为Biovision公司产品,基因组步移试剂盒为Seegene公司产品.
1.3 方 法
1.3.1 宏基因组DNA的提取
取采集于温泉下的土样及水样混合物,离心收集菌体,按照Power MaxTM Soil DNA Isolation Kit抽提宏基因组DNA.取1-2 μL DNA进行脉冲场电泳分析,采用1%琼脂糖凝胶,0.5*TBE缓冲液,电泳时间为16 h,脉冲时间10~25 s,脉冲角度为120°.
1.3.2 基于序列的醛脱氢酶引物的设计及保守序列的获得
选择450~520个氨基酸长度的醛脱氢酶,包含了Arg25和Gly414之间的区域,这些区域中包含了所有保守的未突变的残基和保守片断[3].根据10个保守模块的氨基酸序列及PCR引物设计原则,最后选择motif 4、motif 6和motif 10三个保守区域的氨基酸序列,结合核苷酸序列的保守性,保持引物的兼并度低于1024原则,最后设计引物,如表1所示.

表1 PCR及步移所用的引物序列
以宏基因组DNA为模板,采用引物分别进行温度梯度PCR,PCR体系为:10×Buffer 2 μL,dNTP 0.2 mmol,rTaq酶1U,模板4 μL,引物对Fm和Rm 2 μL,加去离子水至总反应体积20 μL,PCR反应条件为94 ℃,5 min; 94 ℃,30 s;40 ℃-50 ℃,45 s;72 ℃,40 s;30个循环;最后72 ℃延伸10 min.PCR产物回收后经T载体克隆、测序.根据测序获得的核苷酸序列与NCBI中进行BLASTn和BLASTp分析.
1.3.3 基因组步移获得全长序列
根据获得的保守序列,以及DNA Walking SpeedupTM Premix步移试剂盒的说明书并适当改进,分别设计3条引物,克隆保守序列的5′和3′端序列.PCR产物回收后经T载体克隆、测序.获得的序列与保守序列进行电子拼接.第1次PCR反应体系为:2*SeeAmpTMACPTM Master Mix Ⅱ 25 μL,dNTP,宏基因组DNA 1 μg,10 μM引物对(DW-ACP和TSP1)各1 μL,加去离子水至50 μL.PCR反应条件为:94 ℃,5 min;42 ℃,1 min;72 ℃,2 min;94 ℃,30 s;55 ℃,30 s;72 ℃,100 s;20~30个循环;最后72 ℃延伸10 min.将PCR产物稀释50倍后作为下一轮PCR的模板,引物为DW-ACPN和TSP2.PCR反应条件为:94 ℃,3 min;94 ℃,30 s;55 ℃-60 ℃,30 s;72 ℃,100 s;35个循环;最后72 ℃延伸10 min.PCR产物稀释50倍后作为下一轮PCR产物的模板,引物为Universal primer和TSP3,PCR反应条件同上.将第2、3轮PCR产物进行琼脂糖电泳分析和T载体克隆并测序.
1.3.4 新醛脱氢酶基因编码蛋白质的功能分析
使用Vector NTI 8.0软件分析宏基因组来源的醛脱氢酶,推测其编码蛋白质的氨基酸组成、理论分子量.DNA STAR软件分析其启动子序列.采用Compute pI/MW工具分析其等电点(pI)(http://au.expasy.org/tools/pi_tool.html).BLASTp分析所获得醛脱氢酶与其它醛脱氢酶的氨基酸序列的同源性(http://www.ncbi.nlm.nih.gov/blast)[13].基于SWISS-MODLE进行蛋白质三维建模[13],进行蛋白质功能的初步分析.
2 结果与分析
2.1 宏基因组DNA的提取及检测
采用Power MaxTM土壤DNA提取试剂盒成功提取宏基因组中的DNA,脉冲场电泳分析所获得的宏基因组大小约40 Kb.
2.2 新的醛脱氢酶基因全长的分离
采用温度梯度PCR从宏基因组文库中分离得到约700 bp左右的保守序列.经分析,与NCBI数据库中的MMSDH有同源性.在保守序列的基础上,根据DNA Walking SpeedupTM Premix Kit的原理和操作步骤,经过3次步移,分别获得了5′端和3′端的核苷酸序列.将所获得的序列经序列重叠群软件分析,获得1条完整的电子拼接序列.根据MMSDH的编码区,设计引物,从宏基因DNA文库中再次获得全长序列.
通过ORF finder分析醛脱氢酶的编码序列,起始密码子为GTG,终止密码子为TGA.起始密码子的上游序列有TAGGAG序列,推测为相似的核糖体结合位点(图1).推测编码蛋白含有490个氨基酸,预测其相对分子量为53.9kD,等电点为5.02.将推测的氨基酸序列进行Blastp分析发现,与Genbank库中的来自于BacteriumEllin514的甲基丙二酸半醛脱氢酶序列相似性最高达到73%,序列一致性为53%.与其他微生物的甲基丙二酸半醛脱氢酶或乙醛脱氢酶具有不同程度的相似性.Blastp分析结果表明,笔者成功从宏基因组中分离到1条新的醛脱氢酶基因.
2.3 新醛脱氢酶基因编码蛋白质的分析
将获得的推测的MMSDH氨基酸序列与已鉴定有功能的MMSDH序列进行同源性比对,结果表明,一些重要的氨基酸残基和模块在这些序列中是保守的.醛脱氢酶家族重要的保守模块在序列中均被发现[3].一些保守的Gly被认为是非常重要的核酸结合区域及保守位点.但是没有发现典型的NAD+结合模块(G-X-G-X-X-G).来源于Rattusnorvegicus的MMSDH在N端比微生物来源的MMSDH多一段信号肽序列,这与其定位于线粒体有关(rat liver mmsdh),而微生物来源的MMSDH则没有此段序列(图2).推导的MMSDH的氨基酸序列与芽胞杆菌来源的MMSDH的相似性较高,达到了63%.将推导的氨基酸序列进行SWISS-MODEL基于序列同源性的建模,模板为来自芽孢杆菌的MMSDH,最后获得了推导的MMSDH三维结构图(图3).推导的温泉环境宏基因组来源的MMSDH三维结构与芽孢杆菌的MMSDH三维结构相似.MMSDH的10个保守motif位于蛋白质三维结构的内部.
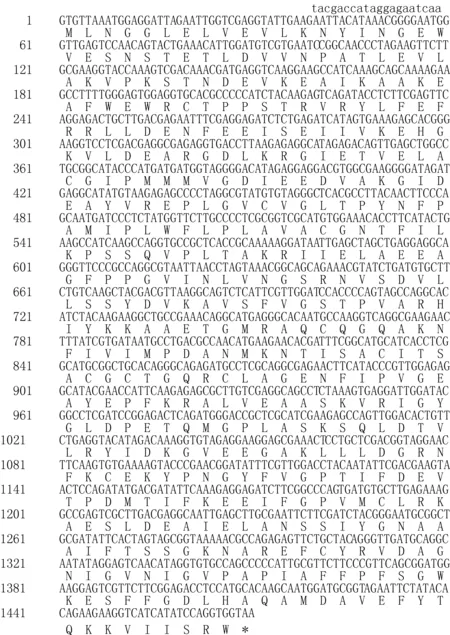
图1 温泉环境宏基因组中分离的MMSDH的核苷酸序列及其推测的氨基酸序列Fig. 1 Nucleotide sequence and deduced amino acid sequence of MMSDH from hot spring metagenomic library


图2 推测的宏基因组来源的MMSDH与其他物种MMSDH同源性比较Fig. 2 Multi-alignment of putative MMSDH and other MMSDHs Bacillus sp. ZP_01169178; Pseudomonas aeruginosa AAA25891; Streptomyces coelicolor AAA99190; Rattus norvegicus: AAA41638.
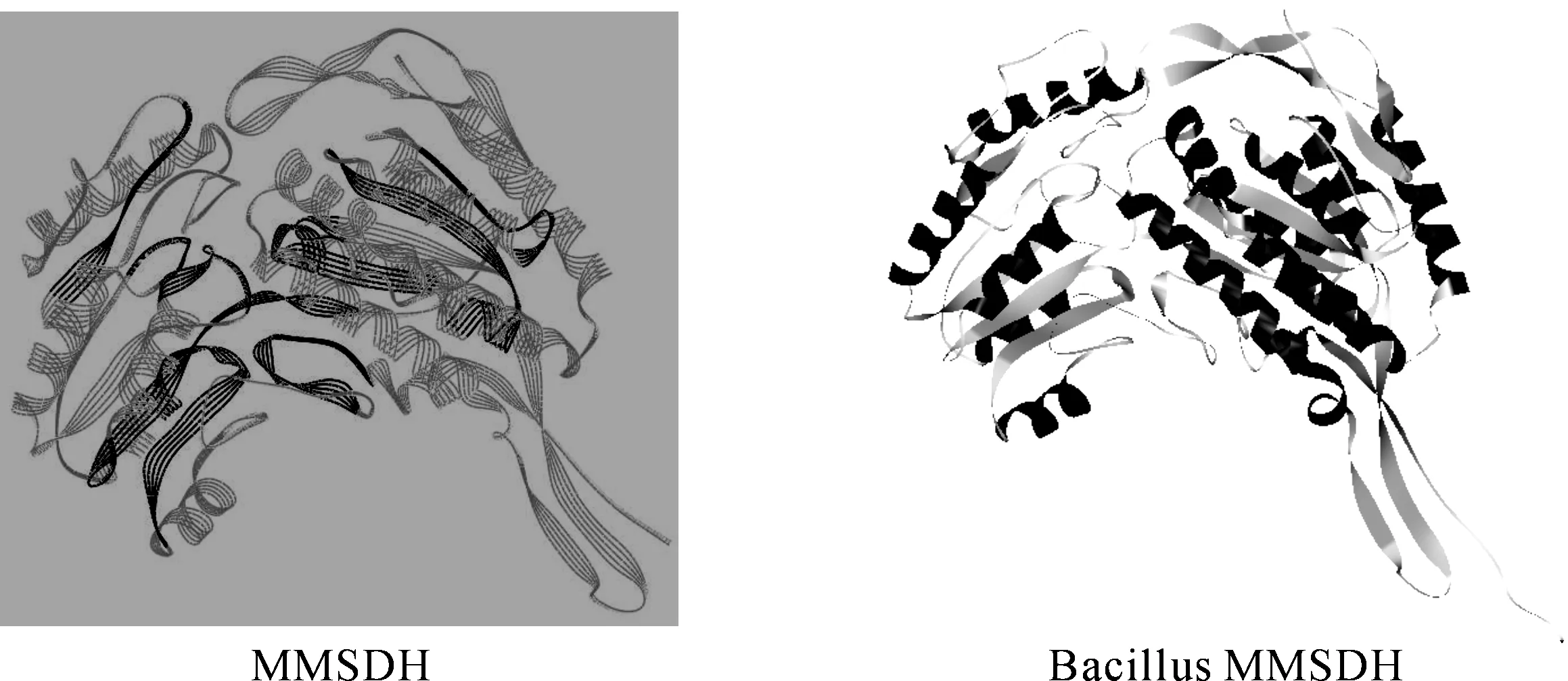
图3 宏基因组来源的MMSDH与bacillus的MMSDH的三维结构对比Fig. 3 Comparison of 3-D structure between bacillus MMSDH and MMSDH from hot spring metagenomic library
3 结论与展望
采用兼并引物进行免培养-PCR技术从真菌DNA中筛选新基因已有相关的报道,如成功获得多条糖基水解酶家族基因[12],Edwards等[14]采用PCR技术直接从森林土壤中分离出了多条纤维素基因部分序列.但是,这些研究并未获得全长基因序列.该研究结合DNA walking技术从宏基因组中成功分离到1条新的醛脱氢酶基因,为采用免培养-PCR技术方便快捷地分离基因提供了依据.该研究证实了免培养-PCR在微生物基因研究中的价值,且新酶的筛选技术得到了发展.获得的MMSDH基因进一步进行其独特的酯类合成能力与酯酶活性分析,为研究微生物来源的MMSDH功能奠定基础.
[1] Bannerjee D, Sanders L E, Sokatch J R. Properties of Purified Methylmalonate Semialdehyde Dehydrogenase of Pseudomonas aeruginosa[J]. Journal of Biological Chemistry,1970,245:1828-1835.
[2] Kedishvili N Y, Popov K M, Rougraff P M,etal. CoA-dependent methylmalonate-semialdehyde dehydrogenase, a unique member of the aldehyde dehydrogenase superfamily. cDNA cloning, evolutionary relationships, and tissue distribution[J]. Journal of Biological Chemistry,1992,267:19724-19729.
[3] Perozich J, Nicholas H, Wang Bicheng,etal. Relationships within the aldehyde dehydrogenase extended family[J]. Protein Science,1999,8(1):137-146.
[4] Omura S, Tsuzuki K, Tanaka Y,etal. Valine as a precursor of the n-butyrate unit in the biosynthesis of macrolide aglycones[J]. Journal of Antibiotics,1983,36:614-616.
[5] Popov K M, Kedishvili N Y, Harris R A. Coenzyme A-and NADH-dependent esterase activity of methylmalonate semialdehyde dehydrogenase[J]. Biochimica Biophysica Acta,1992,1119:69.
[6] Kedishvili N Y, Goodwin G W, Popov K M,etal. Mammalian methylmalonate-semialdehyde dehydrogenase[J]. Methods in Enzymology,2000,324:207-218.
[7] Stines-Chaumeil C, Talfournier F, Branlant G. Mechanistic characterization of the MSDH (methylmalonate semialdehyde dehydrogenase) from Bacillus subtilis[J]. Biochemical Journal,2006,395:107-115.
[8] Zhang Yingxin, Tang Li, Hutchinson C R. Cloning and characterization of a gene (msdA) encoding methylmalonic acid semialdehyde dehydrogenase from streptomyces coelicolor[J]. Journal of Bacteriology,1996,178(2):490-495.
[9] Marilyn I, Steele S, Lorenzj D, Hatter K,etal. Characterization of the mmsAB Operon of Pseudomonas aeruginosa PA0 Encoding Methylmalonate-semialdehyde Dehydrogenase and 3-Hydroxyisobutyrate Dehydrogenase[J]. Journal of Biologicaclh Emistry,1992,267(19):3585-13592.
[10] Tao Junhua, Zhao Lishan. An integrated approach to applying biocatalysis for chemical developmen[C]//Seeberger P H, Blume T. New Avenues to Efficient Chemical Synthesis. Berlin: Sprilinger,2007:117-131.
[11] Schmeisser C, Steele H, Streit W R. Metagenomics, biotechnology with non-culturable microbes[J]. Applied Microbiology and Biotechnology,2007,75(5):955-962.
[12] Jacobsen J, Lydolph M, Lange L. Culture independent PCR: an alternative enzyme discovery strategy[J]. Journal of Microbiological Methods,2005,60(1):63-71.
[13] Arnold K, Bordoli L, Kopp J,etal. The Swiss-model workspace: A web-based environment for protein structure homology modeling[J]. Bioinformatics,2006,22:195-201.
[14] Edwards I P, Upchurch R A, Zak D R. Isolation of fungal cellobiohydrolase I Genes from sporocarps and forest soils by PCR[J]. Applied and environmental microbiology,2008,74(11):3481-3489.
