番茄Mi-1基因的SNP分型
2010-07-09李亚玲李景富康立功董晓慧许向阳
李亚玲,李景富,康立功,张 贺,董晓慧,许向阳*
(1.东北农业大学园艺学院,哈尔滨 150030;2.黑龙江省农业科学院绥化分院,黑龙江 绥化 152052)
单核苷酸多态性(Single nucleotide polymorphism,SNP)是指染色体基因组水平上单个核苷酸的变异引起的DNA序列多态性,而其中最少一种等位基因在群体中的频率不小于1%[1-2],其中包括单碱基的转换(包括C与T转换,在其互补链上则为G与A互换)、颠换(包括C与A、G与T、C与G,A与T互换),以及单碱基的插入/缺失等。通常所说的SNP并不包括后两种情况,转换的发生率也总是明显高于其他几种变异,可能是因为CpG二核苷酸上的胞嘧啶残基是人类基因组中最易发生突变的位点,其中大多数是甲基化的,可自发地脱去氨基而成为胸腺嘧啶[3-4]。SNP被称为继限制性片段长度多态性(Restriction fragment length polymor-phisms,RFLP)以及微卫星多态性(Micro-satellite polymorphisms)等标记之后的第三代分子标记。
SNP检测分析技术目前主要有阵列杂交分析(Array hybridization assays),基因芯片技术(Gene chips),同源杂交(Homogenous hybridization),限制性酶切法,直接测序法(Direct sequencing)。直接测序检测SNP的基本原理是:通对不同个体同一基因或基因片段进行测序和序列比较,以确定所研究的碱基是否变异,其检出率可达100%[5],本试验中的SNP位点即通过直接测序法获得。
SNP分型是SNP研究领域的重要课题。根据不同研究需要和试验条件,已建立了不同的分型方法,包括寡核苷酸连接分析[6-7]、质谱法以及基于PCR的等位基因特异性 PCR(AS-PCR)等[8]。ASPCR法通过特异引物的巧妙设计,PCR对DNA基因组信息的放大和合适的检测方法将不同的SNP基因型分辨开来,具有费用低,程序简单,易于操作等优点,适于中小型研究单位对较小规模SNP检测[9]。目前,改良的AS-PCR方法已有较多报道,如片段长度差异等位基因特异性 PCR(FLDASPCR)、四引物扩增受阻突变体系PCR技术等[10-11]。
本研究试图建立和优化适合于番茄Mi-1基因SNP分型的AS-PCR反应体系,提高这种分型技术在番茄SNP分型中的准确性,为番茄的抗根结线虫Mi基因分子标记连锁作图打下基础。
1 材料与方法
1.1 供试材料
1.1.1 供试番茄材料
抗病亲本08576、感病亲本T431以及08576和T431有性杂交的F2代单株。上述材料均由东北农业大学园艺学院番茄课题组提供。通过对这些种质根结线虫Mi-1基因的序列分析,筛选出候选的SNP位点。
1.1.2 供试试剂
CTAB、EDTA、RNase、TaqDNA聚合酶(均购于IBM公司);Repel、Binding(北京鼎国生物技术公司生产);DNA Marker、dNTP、引物(由上海生工生物工程和大连生物公司合成或生产);其他试剂如琼脂糖、氯仿、异戊醇、异丙醇、无水乙醇、溴化乙锭、TAE缓冲液等化学试剂(由哈尔滨市宝瑞生物试剂公司和哈尔滨市德美试剂公司提供)。
1.2 方法
1.2.1 基因组DNA的提取
取番茄植株幼嫩叶片,用自来水、蒸馏水冲洗两遍,用灭菌后的滤纸吸干后,每个样本称取0.2 g装入1.5 mL离心管中,用液氮速冻后-20℃保存备用;采用CTAB法对番茄抗病亲本品种08576和感病亲本品种T431以及杂交组合的F2代分离群体各单株分别进行DNA提取。
DNA浓度和质量的测定:用0.8%的琼脂糖凝胶电泳检测,λDNA作为Marker,以检查DNA浓度;紫外分光光度计测定样品的纯度,OD260/OD280=1.7~2.0。
1.2.2 等位基因PCR(Allele specific polymerase chain reaction,AS-PCR)
1.2.2.1 引物设计
他们好像不是在分手,为什么这么风轻云淡,没有大吵大闹,没有歇斯底里,没有争吵推诿,或者他们都在等对方先说出来,其实心里早已经对这段感情没有了信心。
试验采用的是原试验数据中一个位于双亲测序片段401 bp的A-T型颠换的SNP突变位点,研究采用了2个平行扩增反应,筛选了3个引物,除都有1个共同上游引物外,另外2个特异下游引物3′末端碱基分别和SNP正常碱基和突变碱基互补,在其3′末端第二或第三碱基分别引入错配碱基增加特异性,引物长度约18~20 bp。用Primer Premier 5软件和Oligo6软件相结合,设计Mi基因特异引物,优化DNA模板浓度梯度、退火温度梯度、引物浓度梯度、dNTP浓度梯度、Mg2+浓度梯度,摸索出扩增效果最佳的PCR程序。
1.2.2.2 PCR扩增
PCR 反应体系为 20 μL,10×buffer 2 μL,引物各 1.5 μL,2 mmol·L-1dNTP 1 μL,25 mmol·L-1Mg2+1.5 μL,DNA 模板 1 μL,5 U·μL-1Taq 酶 0.2 μL。程序为:94℃预变性5 min,(94℃变性1 min,Tm退火1 min,72℃延伸1 min)×35个循环,72℃延伸10 min,4℃保存。
1.2.3 SNP基因型检测
PCR产物通过0.8%琼脂糖凝胶电泳即可分辨,2个纯合等位基因型仅一对引物能扩增出单一谱带,同一样本两对特异引物都能扩增出产物为杂合型。
2 结果与分析
2.1 提高SNP特异性的引物设计与筛选
引物3′末端碱基与模板互补则会在适合的条件下发生延伸,否则不能延伸,但是在实际应用中,3′末端单个碱基错配通常不能阻止扩增延伸。因此,Ye等的研究认为[12],在特异引物3′端1、2位加入错配碱基就可以获得较好的差异产物,并且G/A和C/T错配为强的碱基错配类型;C/A和G/T为弱的碱基错配类型;A/A、C/C、G/G和T/T为中等的碱基错配类型。也就是说:不同嘌呤、不同嘧啶之间的错配分别为强错配,相同嘌呤、相同嘧啶之间的错配分别为中等错配,嘌呤和嘧啶之间的错配为弱错配。在特异引物3′末端错配碱基的搭配方面采取强弱搭配,或者中中搭配,获得理想扩增效果的可能性较大。为了获得较好的特异性条带,根据前人研究经验,本试验针对母本08576和父本T431分别设计了3对反义链引物,这6对引物共用1对正义链引物。
引物S401-1、S401-2、S401-3与母本08576的SNP位点互补匹配,其中,S401-1的3′端第二位引入A/G强碱基错配,S401-2的3′端第三位点引入C/T类型强错配,S401-3为3′端第二位T/G弱碱基错配类型和第三位点采用C/T强错配类型相结合。从PCR效果上看可知,引物S401-3的强弱搭配类型表现出了较好的父母本特异带型(见图1)。

图1 与母本08576的SNP位点匹配的等位基因特异引物的设计与PCR扩增Fig.1 Allele-specific primer designing on the SNP site and PCR results of the parent-08576
引物S401-A、S401-B和S401-C与父本T431的SNP位点互补匹配,其中,S401-A的3'端第二位点引入了A/G强错配类型,S401-B的3'端第三位点引入了C/T的强错配类型,S401-C为第二位点T/G弱配和第三位点C/T强配相结合,但是从PCR效果上看,S401-C在父母本间都扩增出了产物,而仅第二位点引入强错配类型的引物S401-A表现出了良好的特异性(见图2)。

图2 与父本T431的突变SNP位点匹配的等位基因特异引物设计及PCR扩增Fig.2 Allele-specific primer designing on the SNP site and PCR rerults of the parent-T431
2.2 AS-PCR反应体系的优化
等位基因特异PCR非常灵敏,其扩增效果与PCR反应体系中每个组分的用量以及PCR程序都密切相关。其中以引物的退火温度、Mg2+及dNTP浓度的影响较大。在本试验中,先通过引物合成单以及引物设计软件Primer Premier 5推荐的退火温度上下5℃设定梯度,最终确定S401最适温度为48.2℃,同时也对体系中的Mg2+和dNTP浓度进行梯度筛选,得到最佳的Mg2+和dNTP浓度。
本研究使用浓度为25 mmol·L-1的Mg2+设定了5个梯度分别为1~3 μL,以0.5 μL为间隔梯度。结果表明,引物S401-3在1 μL时在两亲本中都没有扩增产物,在2~3 μL时两亲本中都扩增出了产物,但是没有表现出特异性,而1.5 μL时在父母双亲中能扩增出差异的条带,且无杂带;引物S401-A在2~3 μL时都没有表现出引物设计需要的特异性,而在1 μL和1.5 μL时能在亲本中表现出与S403-3相反的特异性,其中1.5 μL时条带更清晰,综合S403-3的扩增情况,选择单次取25 mmol·L-1的Mg2+1.5 μL时浓度为最适 Mg2+浓度,即 1.875 mmol·L-1(见图3、4)。

图3 等位基因引物S401-3在两亲本间的Mg2+浓度筛选Fig.3 Screening of Mg2+concentration of allele-specific primer S401-3 in both parents
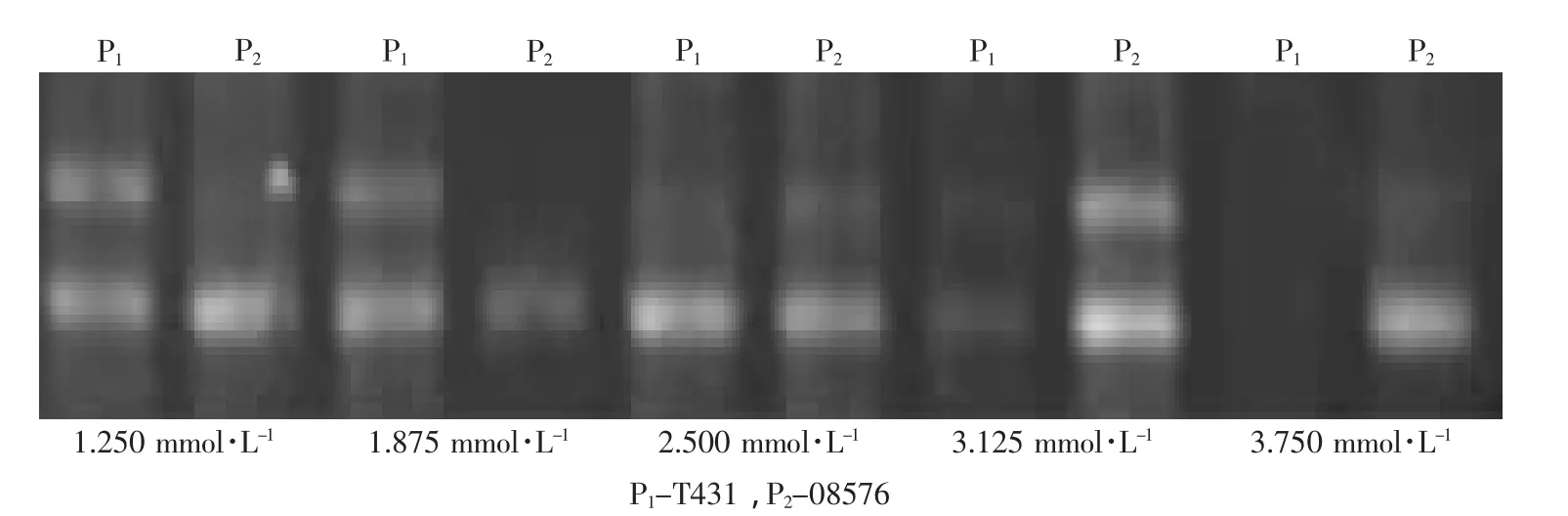
图4 等位基因引物S401-A在两亲本间的Mg2+浓度筛选Fig.4 Screening of Mg2+concentration of allele-specific primer S401-A in both parents
本研究中dNTP浓度也是设定了5个梯度分别为1~3 μL,以 0.5 μL为间隔梯度。结果发现,引物S401-3在1.5~2.5 μL时不能表现出亲本间的差异,而在1~3 μL中都表现出了良好的差异;引物S401-A在1.5~3.0 μL都不能表现出亲本间的差异,而1 μL时能表现出与S401-3相反的特异性。从节约成本的角度出发,选定单次取2 mmol·L-1的dNTP 1 μL为最适浓度,即100 μmol·L-1(见图5、6)。

图5 等位基因引物S401-3在两亲本间的dNTP浓度筛选Fig.5 Screening of dNTP concentration of allele-specific primer S401-3 in both parents

图6 等位基因引物S401-A在两亲本间的dNTP浓度筛选Fig.6 Screening of dNTP concentration of allele-specific primer S401-A in both parents
2.3 F2分离群体的SNP分型
利用前面设计的等位基因特异引物及其互补引物,在F2分离群体中进行PCR,以期获得带型相反的PCR产物,即可以相互验证两对引物的正确性。又可以检测该位点在材料中是哪种纯合基因型还是杂合基因型。S401为A/T突变,在亲本T431中T/T纯合型,在08576中为A/A纯合型,因此在F2群体中,仅在S401-3中获得扩增目的片段的为A/A纯合型,仅在S401-A中获得扩增目的片段的为T/T纯合基因型,同时在S401-3和S401-A中获得目的片段的为A/T杂合SNP基因型。由图7可知,F2分离群体中1、4、9、17为纯合的父本基因型T/T型,3、7、13为纯合的母本基因型A/A型,其余单株为杂合A/T基因型。单株12为不明原因的缺失。图7中所示Marker条带从上到下依次为600、500、400。
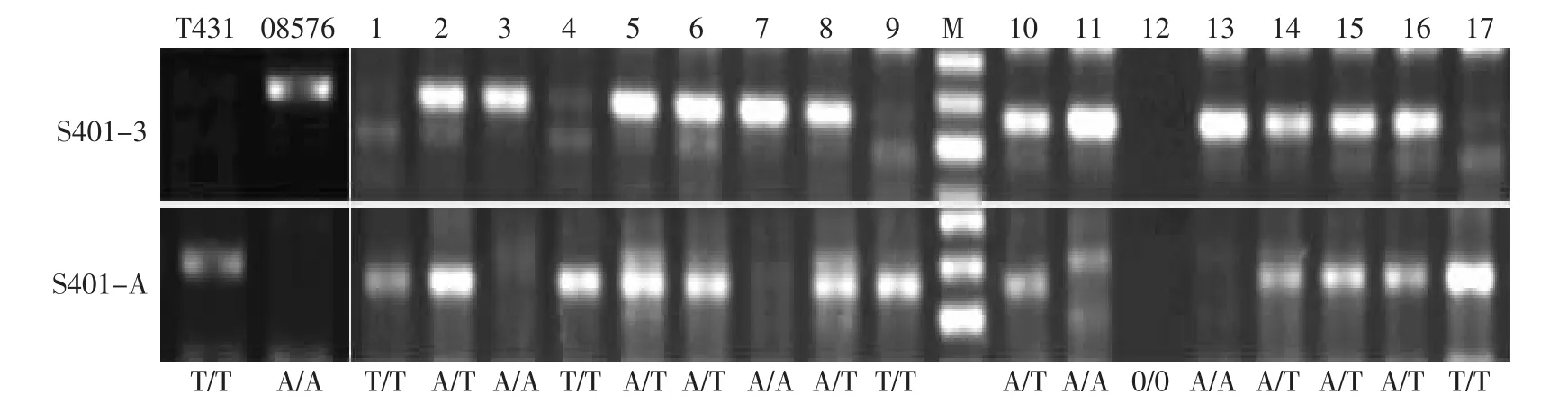
图7 互补引物检测SNP基因型Fig.7 Assaying SNP genotypes with complementary primers
试验对 438个 F2群体进行 SNP分型,其中能检测出的317株单株的基因型分型结果如表1所示。另外有121株表现为不明原因的缺失。

表1 F2群体基因型检测Table 1 Genotype detection of F2group
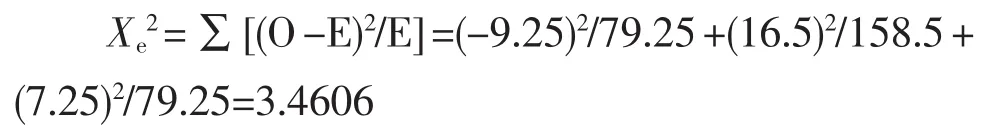
查X2值表,当自由度为2时,χ20.05,2=5.991,因此χe2=3.4606<χ20.05,2,实际检测出的基因分离比与理论分离比1∶2∶1相符合,属于一对等位基因的遗传规律。
3 讨 论
目前国内番茄抗病育种在基因工程方面应用的技术主要有AFLP、RAPD、微卫星技术等第一代、第二代分子标记[13-15]。而SNP密度高,多态性丰富,具有高度遗传稳定性,同时SNP是一种二态的标记,非此即彼,在检测时无需同检测微卫星标记那样对片段的长度做出测量,只需一个“+/-”或“全或无”分析的方式,易于分型,有利于快速、自动化筛选和检测,以及批量、规模筛选和分析[3]。在连锁群体遗传作图方面,具有作为新一代分子标记所特有的价值和意义。
番茄根结线虫病的研究日益重要[16],根结线虫是番茄重要病害之一,在设施番茄栽培中尤为严重,国外研究报道相继指出,番茄根结线虫抗性是受一个显性基因支配,定名为Mi基因,到目前为止,Mi基因的抗性仍然是目前唯一被鉴定和利用的根结线虫抗性,其抗性具有广谱性,能有效抵抗除北方根结线虫以外的其他3种主要根结线虫。本研究中采用的SNP位点是通过对番茄抗根结线虫Mi基因CDS直接测序发现的。
由于AS-PCR比较灵敏,受PCR体系中dNTP、引物、Mg2+等组分浓度以及退火温度影响较大,若浓度过低,扩增弱,易出现假阴性,浓度太高,导致扩增非特异性,出现假阳性[10-12]。且不同方法和不同SNP位点对PCR参数要求也不一致,所以本研究通过调整PCR组分浓度或改变PCR程序优化PCR效果,获得最适浓度和最适退火温度,解决非特异性扩增。
在等位基因特异引物设计方面,结合了前人的研究成果,首选了第二、三位引入强错配碱基,以及第二、三中强错配碱基搭配的方法增加获得较好差异扩增的可能,节约了成本。从这两个方面保证了SNP特异扩增的准确性。不同的SNP类型采用同样的错配方式设计引物得到PCR效果也不尽相同,这可能与引物末端不同错配碱基产生的碱基堆积力、氢键及其立体结构的稳定性有关。还可能与错配碱基对和DNA聚合酶之间的相互作用造成的空间位阻有关[17]。为了达到更好的PCR特异性效果,需要对不同的碱基错配类型的引物都进行PCR反应体系的优化。
SNP分型试验是为番茄SNP分子标记开发应用提供技术保障;在试验中,利用了SNP的二态性和便于分型的特点[18],通过等位基因特异PCR针对S401位点设计的互补的引物对F2群体进行了有效的分型(见图7),避免了多重PCR反应条件复杂的制约,适用于样本量适中的分型检测,其互补引物相互验证,琼脂糖电泳即可检测,分型设计简单,可靠。F2群体的SNP分型中,其中已得基因型分离比例经卡方测验符合1∶2∶1的分离比,验证了试验的可靠性,但是还有121株表现为不明原因的缺失,一部分可能是由于DNA降解,另一部分可能由于此位点没有突变,或是其他不明突变所引起等位基因特异PCR产物无条带。
4 结论
研究表明:引入错配碱基和优化PCR反应体系可以调节PCR扩增效果。本试验针对此位点筛选出了特异性最好的与亲本08576的匹配引物S401-3,与亲本T431的匹配引物S401-A。并针对这两对等位基因特异引物筛选了最适的PCR反应体系为:总体积 20 μL,模板 DNA 1 μL,10×Buffer 2 μL,10 μmmol·L-1引物各 1.5 μL,2 mmol·L-1dNTP 1 μL,25 mmol·L-1Mg2+1.5 μL,5 U·μL-1Taq酶0.2 μL。提高了这种分型技术在番茄SNP分型中的准确性。
试验对438株F2群体进行了SNP分型,其中317株获得了基因型,121株由于不明原因缺失,这317株中,表现为母本纯合基因型的70株,父母本杂合基因型的175株,父本纯合基因型的72株,经卡方测验,符合1∶2∶1分离比例,分型结果可靠,为番茄SNP分子标记开发应用提供技术保障。
[1]杨昭庆,洪坤学.单核苷酸多态性的研究进展[J].国外遗传学分册,2000,23(1):4-8.
[2]Landegren U,Nilsson M,Kwok P Y.Reading bits of genetic information:Methods for single nucleotide polymorphism analysis[J].Genome Res,1998,8(8):769-776.
[3]Wang D G,Fan J B,Siao C J,et al.Large-scale identification,mapping,and genotyping of single nucleotide polymorphism in the human genome[J].Science,1998,280:1077-1082.
[4]王淑新,连林生.单核苷酸多态性作为新一代分子标记的优越性[J].中国牛业科学,2008,34(2):40-43
[5]贾玉艳,陈宏.SNP分子标记研究及应用[J].黄牛杂志,2003,29(1):42-45.
[6]Landegren U,Kaiser R,Sanders J,et al.A ligase-mediated gene detection technique[J].Science,1988,241(4869):1077-1080.
[7]Alves A M,Carr F J.Dot blot detection of point mutations with adjacently hybridising synthe tic oligo-nucleotide probes[J].Nucleic Acids Res,1988,16(17):8723.
[8]Sommer S S,Cassady J D,Sobell J L,et al.A novel method for detecting point mutations or polymorphism and its application to population screening for carriers of phenyl ketonuria[J].Mayo C lin Proc,1989,64:1361-1372.
[9]袁翠平,李英慧,刘章雄,等.一种大豆SNP分型新方法[J].大豆科学,2007,26(4):447-459.
[10]黄代新,杨庆恩,赵贵森.片段长度差异等位基因特异性PCR-一种改良的SNP分型新方法[J].法医学杂志,2005,21(1):11-14.
[11]管峰,艾君涛,杨利国.一种SNP检测新方法:四引物扩增受阻突变体系PCR技术[J].生命的化学,2004,24(6):514-516.
[12]Ye S,Dhillon S,Ke X,et al.An efficient procedure for genotyping single nucleotide polymorphisms.An efficient procedure for genotyping single nucleotide polymorphisms[J].Nucleic Acids Res.2001,29(17):E88-8.
[13]李景富,赵凌侠,许向阳,等.番茄属(Lycopersicon)基因组DNA遗传多样性RAPD分析[J].东北农业大学学报,2004,35(1):11-16.
[14]雷娜,李景富,李烨,等.番茄AFLP技术体系的优化和建立[J].东北农业大学学报,2008,39(3):29-33.
[16]李红双.番茄抗根结线虫病基因的分子标记研究[D].哈尔滨∶东北农业大学,2006.
[17]卫波.小麦抗旱相关基因TaDREB1[D].杨凌∶西北农林科技大学,2006.
[18]Zhang W,Gianibelli M C,Ma W,et al.Identification of SNPs and development of allele-specific PCR markers for gamma-gliadin alleles in Triticum aestivum[J].Theor Appl Genet,2003,107(1):130-138.
