杉木木质素合成酶基因CCR的克隆和序列分析
2010-06-04童再康黄华宏林二培程龙军
陈 喜,童再康,黄华宏,林二培,程龙军
(浙江农林大学 林业与生物技术学院,浙江 临安 311300)
木质素(lignin)是一种由肉桂醇等单体聚合而成的酚类多聚体,是维管植物细胞壁的重要组成成分,在木材植物中含量较高,一般约占干重的15% ~ 35%,为地球上仅次于纤维素的有机组分[1]。木质素的生物合成是在一系列酶催化下使苯丙氨酸或酪氨酸逐步转化为木质素单体,最终聚合成木质素的过程。肉桂酰CoA还原酶(Cinnamyol Co-A Reductase,CCR)是影响木质素合成的一个关键酶,负责将各种羟基肉桂酰—辅酶A(CoA)酯最终还原成各种木质素单体,对木质素的合成与代谢也起关键作用。木质素总含量和单体组成对植物体的最终用途起着决定性作用。CCR基因除了影响木质素合成,还影响木材的微纤夹角(Microfibral Angle,mfa或MFA),进而导致材质密度和硬度的变异[2],因此对CCR基因进行克隆和研究具有重要的实际意义[3~4]。
杉木(Cunninghamia lanceolata)属杉科(Taxodiaceae)杉木属(Cunninghamia),是我国特有的常绿针叶树种之一。杉木生长快,木材纹理直,结构细,耐腐力强,用途广泛,又因其萌芽力强,成林迅速,故成为重要的商品用材和造林树种。建国后,我国加强了杉木科研工作,并取得了重要和丰硕成果[5~8],涉及杉木森林培育、经营、遗传育种、计测、木材加工利用等领域[9~11],其中杉木遗传改良研究最为活跃,研究成果及报道文献最多。
对杉木的CCR基因进行克隆,并与近缘属树种进行对比分析发现CCR基因变异情况,以期为杉木育种、遗传进化及转基因研究提供基础遗传学数据。本文应用RACE(Rapid Amplification of cDNA Ends)等分子生物学方法,获得杉木木质素生物合成酶CCR基因的全长cDNA序列,并进行生物信息学分析,为CCR的进一步研究奠定基础。
1 材料与方法
1.1 材料
1.1.1 实验材料 杉木材料采集于浙江林学院教学实习基地杉木种子园内。样品采集后,直接于液氮速冻,带回实验室,用于mRNA的提取。
1.1.2 试剂 Taq DNA聚合酶、T4 DNA连接酶、DNA凝胶回收试剂盒、RNA逆转录试剂盒、克隆载体均购自TaKaRa公司;RACE试剂盒购自Clontech公司;其他试剂为进口或国产分析纯;宿主菌为E.coli DH5α,为本实验室保存。测序由南京金思特公司完成。
1.2 方法
1.2.1 杉木RNA的提取 参考相关的文献,采用改良的CTAB法进行提取[12~16],具体方法如下:取1 g杉木茎和叶,液氮研磨后用药匙将粉末加入到65℃水浴的700μL提取液中,用手摇动混匀。然后65℃水浴20 min后冷却至室温。加等体积(700μL)的氯仿/异戊醇(24:1),混匀,10 000 r/min,4℃离心10 min。小心吸取上清液,重复上述步骤,直至中间层无白色絮状沉淀。吸取上清液,加1/4体积的10M LiCl混合,4℃沉淀过夜,然后10 000 r/min离心20 min。弃上清液,用500μL SSTE溶解沉淀,加等体积氯仿/异戊醇混匀(24:1),10 000 r/min,4℃离心10 min。离心吸取上清液,加2倍体积的无水乙醇,-70℃沉淀至少30 min,或-20℃下沉淀2 h。12 000 r/min,4℃,离心20 min,去上清液,用75%的乙醇洗2次12 000 r/min,4℃离心5 min,小心弃上清液,吹干沉淀。用40μL DEPC处理的水溶解沉淀,得到RNA样品。
1.2.2 CCR基因cDNA部分序列的克隆 cDNA合成:按照反转录TaKaRa PrimscriptTM RT-PCR Kit试剂盒(TaKaRa)的说明进行总RNA的反转录反应,获取cDNA。
1.2.2.1 RT-PCR扩增 ①引物设计。根据GenBank中各种植物的CCR基因的核酸序列进行同源性比较,找出保守的区段,依据同源性高和简并性低的原则,设计一对简并引物:
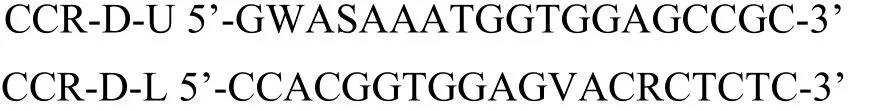
引物由上海生工合成;②进行RT-PCR扩增,扩增体系为20μL体系,其中cDNA 2μL,10×PCR buffer 2 μL,2.5 mM dNTP Mixture 0.8μL,5 U/μL TaKaRa Ex Taq HS 0.1μL,10μM Forward Primer 0.2μL,10μM Reverse Primer 0.2μL,ddH2O14.7μL。扩增条件采用Touchdown PCR进行,反应程序:94℃预变性5 min;94℃变性30 s,54℃ 30 s,72℃延伸1 min,共14个循环,每个循环下降1℃;94℃变性30 s,54℃ 30s,72℃延伸1 min,30个循环;最后72℃延伸10 min。RT-PCR产物经1%琼脂糖凝胶电泳检测,将回收产物进行目的片断的克隆和重组子筛选,并将目的基因片段测序鉴定。
1.2.3 杉木CCR基因cDNA片段3’端和5’端的扩增 RACE采用BD SMARTTM RACE cDNA Amplification Kit(BD Biosciences Clontech),方法按照说明书再稍加改良。①RACE引物设计及扩增。反应利用Primer 5.0软件设计基因特异引物 GSP1:5’>TGCTGGCATTGATGGCAGATGCAG<3’,用于 3’-RACE;GSP2:5’>GGTACGCCGTGTGGTGTTTACATCT < 3’,用于5’-RACE。按Clontech Smart RACE试剂盒说明操作,首先制备RACE-cDNA,然后进行RACE-PCR。用1%琼脂糖凝胶电泳检测PCR结果。②RACE产物的克隆测序和cDNA全长获得。3’RACE产物、5’RACE产物分别用pGEM-T载体连接,经Amp抗性筛选、X-gal/IPTG蓝白斑显色筛选、重组质粒酶切鉴定后,送生物技术公司测序。将3’RACE产物测序结果和5’RACE产物测序结果拼接为杉木CCR基因全长cDNA序列,将此cDNA序列GenBank数据库进行核苷酸同源性比较分析。利用DNA-STAR软件分析测序结果的序列与拼接结果的序列,最终将所得到的序列在NCBI的Blast进行比对,核苷酸序列的相似性比对在NCBI的Blastn(http://www.ncbi.nlm.nih.gov/blastn)中分析,并确定开放阅读框分析(ORF),将其推导相应的氨基酸序列。蛋白质氨基酸相似性比对在NCBI的Blastp中分析;同样,运用Mega3.0软件对推导的氨基酸序列与已知报道植物推导的氨基酸序列进行系统进化分析。
2 结果与分析
2.1 杉木次生木质部总RNA的提取
采用CTAB-LiCl法提取杉木次生木质部总RNA,经琼脂糖凝胶电泳检测,测得结果见图1。从图1中可以看出,提取的RNA质量较好,测定OD260/280为1.87,无DNA污染、无降解,具有清晰的两条带,分别是总RNA的28SrRNA和18SrRNA。说明提取的总RNA纯度较高,可以用于双链cDNA的合成。
2.2 RT-PCR扩增
以杉木次生木质部cDNA为模板,以CCR-D-U、CCR-D-L为引物进行RT-PCR扩增,产物经1%琼脂糖凝胶电泳、溴化乙锭染色后观察(图2),在750 bp处有一条亮带,450 bp处有一条较浅的带,分别将2条带切胶回收后,克隆出长约750 bp的片段。
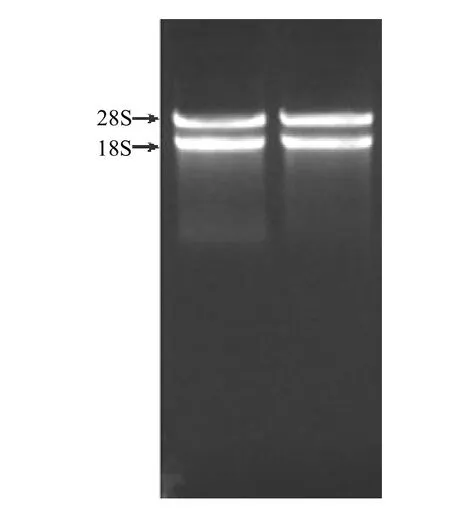
图1 杉木总RNA的琼脂糖凝胶电泳分析Figure l Electrophoretic analysis of total RNA isolated from C. lancoealata
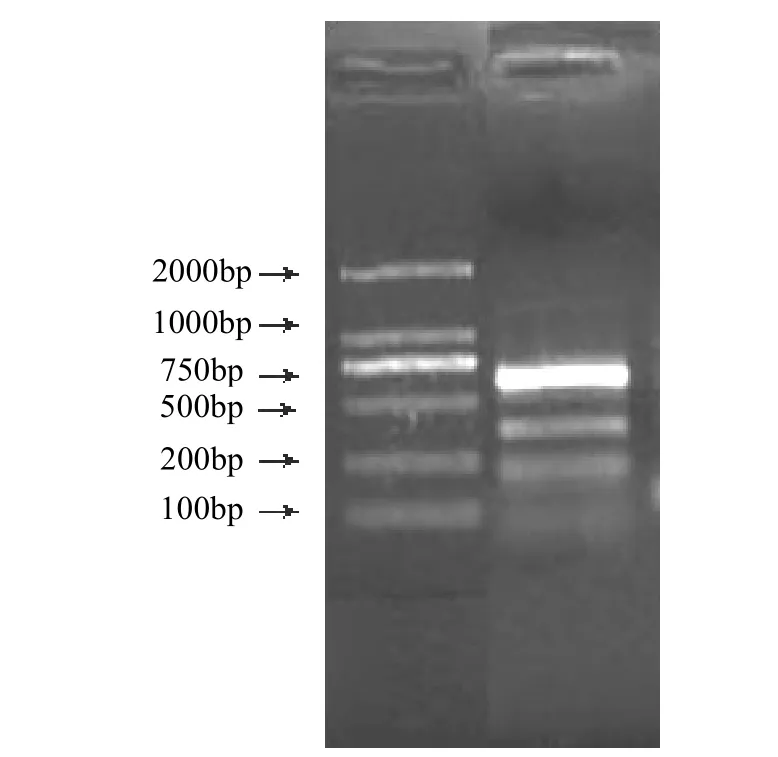
图2 杉木CCR cDNA RT-PCR产物琼脂糖电泳分析鉴定Figure 2 Identification of the RT-PCR product of C. lancoealata CCR cDNA by DNA agar gel electrophoresis
2.3 RACE产物的分析
利用扩增出的杉木CCR部分序列设计5’端RACE特异引物GSP1和3’端RACE特异引物GSP2,分别用于基因5’端和3’端序列的扩增。经5’和3’RACE分别获得大小约为800 bp和1 100 bp的PCR产物(图3),克隆测序发现,5’和3’端序列与已获得的杉木CCR编码序列有重叠,为CCR基因mRNA的5’和3’端序列,3个序列经软件拼接,得到杉木CCR基因完整的cDNA序列,共1 379 bp。然后以拼接得到的CCR全长cDNA设计引物,通过PCR扩增,凝胶电泳分析(图4)、测序,也验证了所获基因的准确性。
2.4 生物信息学分析
2.4.1 杉木CCR基因cDNA全长核苷酸序列的分析 对测序所得到的5’RACE、3’RACE cDNA及中间核苷酸序列进行拼接,得到杉木CCR的全长cDNA,其长度为1 379 bp,该序列的5’端存在57 bp的非翻译区(UTR),其中975 bp的开放阅读框(ORF),3’端非翻译区(UTR)长度为347 bp,其中包括38 bppoly(A)尾巴,该基因编码324个氨基酸的蛋白(图5),分子量约为35.71 KD,等电点为6.164。将此cDNA全长序列在NCBI上用BLAST工具进行序列比对,结果表明:该全长序列与己知的松科植物欧洲云杉(Picea abies, AM260972.1)肉桂酰辅酶A还原酶(CCR)基因有83%的最高相似性,与火炬松(Pinus taeda, AY064169.1)、马尾松(P.massoniana, EU753854.2)等植物的肉桂酰辅酶A还原酶(CCR)基因具有82%的较高相似性,表明了所克隆基因确为杉木的肉桂酰辅酶A还原酶(CCR)基因。
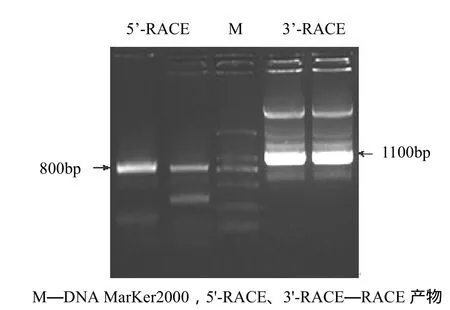
图3 杉木CCR基因cDNA 5’,3’RACE产物的琼脂糖电泳分析鉴定Figure 2 Identification of the 5’,3’ RACE product of C.lancoealata CCR cDNA by DNA agrose gel electrophore
图4 杉木CCR全长cDNA PCR产物琼脂糖电泳分析鉴定Figure 4 Identification of the PCR product of C. lancoealata CCR full-length cDNA by DNA agar gel

图5 杉木CCR基因cDNA全长的核苷酸序列及其推导的氨基酸序列Figure 5 Nucleotide sequence and deduced amino acid sequence of the CLCCR full-length cDNA
2.4.2 杉木CCR基因核苷酸序列与推导的氨基酸序列同源性及系统进化分析 将推测的杉木CCR基因编码的324个氨基酸序列在GenBank中进行Blast结果表明:该CCR氨基酸序列与数据库中其它植物的CCR氨基酸序列有高度相似性,其中与己知松科植物的欧洲云杉(CAK18610.1)、马尾松(ACE76870.3)、火炬松(AAL47684.1)CCR基因氨基酸序列分别具有 87%、84%、84%的相似性;与其他的被子植物如桦木科的光皮桦(Betula luminifera)、杨柳科的杨树(Populus canadensis)、桃金娘科的桉树(Eucalyptus robusta)、茄科的有丝茄(Solanum lycopersicum)等的CCR基因氨基酸序列,也有较高的相似性。这表明所克隆基因为杉木的CCR基因。多序列比对结果也进一步表明(图6),杉木CCR蛋白氨基酸序列与其它高等植物的CCR蛋白氨基酸序列在系统进化上具有高度保守性。
将CCR基因推导的酶蛋白氨基酸序列与其他植物已报道的相应序列用MAGA3.0软件进行多序列比较,并构建系统进化树(图 7)。结果显示:杉木与松科植物欧洲云杉、马尾松、火炬松聚为一类,同源性较高,表明同为裸子植物的杉科植物与松科植物在进化上有较近的亲缘关系;与模式植物十字花科的拟南芥(Arabidopsis thaliana)以及禾本科植物的水稻(Oryza Sativa)、玉米(Zea Mays)、高粱(Sorghum bicolor)、小麦(Triticum aestivum)同源性次之,但不聚为一类;与杨柳科的杨树、桦木科的光皮桦、豆科的大豆、葡萄科的葡萄(Vitis vinifera)同源性相对较低,亲缘关系较远。

图6 杉木CCR氨基酸序列与其他植物的CCR氨基酸序列的多重比较Figure 6 Multiple comparisons of the deduced amino acid sequences of CCR from C. lanceolata and from other plant
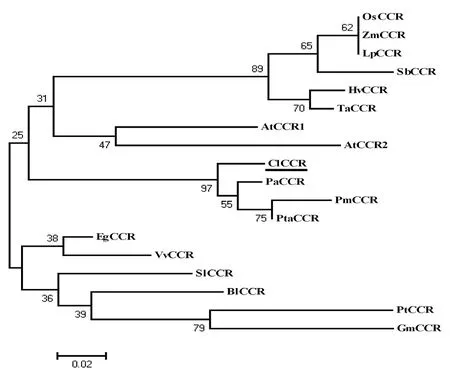
图7 植物CCR的系统进化树Figure 7 Phylogenetic tree based on amino acid sequences of CCR from different plants
3 讨论
(1)高质量RNA的提取是开展分子生物学研究的最基本也是最重要的基础步骤,特别是要分离克隆出完整的全长cDNA,获得完整的、未被降解的大量的RNA显得尤为关键,因为RNA的质量决定着可被反转录为cDNA最大的序列信息。
在RNA提取中防止RNA酶对RNA的降解极为重要。在提取RNA过程中要严格防止RNA酶的污染,所有塑料制品要用DEPC水处理并灭菌,玻璃器皿干热灭菌,所用试剂均要做无RNA酶处理。在剥取植物组织时速度要快,尽量减少直接暴露于空气中的时间,立即投入液氮中研磨。
(2)通过GenBank联机检索,根据已报道的CCR基因序列的高度保守区用Primer Primier 5设计简并引物,再用Oligo6验证。基因的高度保守区在不同物种中个别碱基的差异给简并引物的有效使用带来了困难。虽然在引物设计时考虑到诸多因此,但是究竟能否有效扩增目的片段毕竟还是需要将每一对引物,通过反复优化PCR条件的方法来验证。因此,得到一对有效的引物可以大大地提高工作效率。
(3)木质素由不同的木质素单体聚合而成。近年来,人们利用分子生物学的手段对参与木质素合成的一些关键酶基因进行了研究,对木质素合成过程的关键酶进行了克隆,并进一步通过转基因的实验对这些关键酶对于改变木质素的含量或木质素的组成及化学结构的影响进行了研究。参与木质素合成的酶有许多,CCR作为木质素生物合成的关键酶,对于木质素生物合成具有重要作用。本文从杉木木质部中克隆了木质素合成酶CCR基因,通过对该基因的生物信息学分析证明了我们克隆到的基因就是CCR基因,为后续的CCR结构功能鉴定和研究打下了基础。我们期望通过克隆、鉴定杉木木质素合成基因,能对通过基因工程改变杉木木质素含量以及改良杉木材性方面起到一定的作用。
[1] Zhong R Q,Morrison W H,Himmelsbach D S,et al. Essential role of caffeoyl coenzyme A O-methyltransferase in lignin biosynthesis in woody poplar plants[J]. Plant Physiol,2000(124):563-578.
[2] Hillis W E. Wood quality and growing to meet market requirements[A]. IUFRO. The Future Of Eucalyptus For Wood Products[C]. 2000,Launceston, Tasmania, Australia, 2000. 256-264.
[3] Chen L,Auh C K,Dowling P,et al. Improved forage digestibility of tall fescue (Festuca arundinacea ) by transgenic down-regolation of cinnamyl alcohol dehydrogenase [J]. Plant Biotechnol J,2003,1(6):437-449.
[4] He X,Hall M B,Gallo-Meagher M,et al. Improvement of forage quality by downregulation of maize O-Methyltransferase[J]. Crop Sci,2003,43(6):2 240-2 251.
[5] 阳含熙. 杉木营林的研究[A]. 中国林业科技30年[M]. 北京:中国林业科学院,1979. 164-175.
[6] 俞新妥. 中国杉木研究[J]. 福建林学院学报,1988,8(3):203-220.
[7] 丁蕴一. 中国杉木研究动态[J]. 世界林业研究,1990,3(1):67-74.
[8] 施季森. 杉木文献引读及检索[M]. 上海:上海科学文献出版社,1992.
[9] 吴火和,叶功富,俞新妥. 杉木栽培制度的基本特征及其可持续发展探讨[J]. 福建林业科技,2002,29(1):58-60.
[10] 程朝阳. 杉木人工林无节材培育技术研究[J]. 林业科学研究,2005,18(5):530-534.
[11] 杨玉盛,陈光水,王小国. 皆伐对杉木人工林土壤呼吸的影响[J]. 土壤学报,2005,42(4):584-590.
[12] 宋蓓,赵锦,刘孟军,等. 改良CTAB-LiCl法提取枣总RNA建立[J]. 中国农学通报,2007,23(7):79-83.
[13] 刘晓菊,洪海波,李敏,等. 改良法提取核桃总RNA试验[J]. 山东农业科学,2008(1):97-99.
[14] 李菁芳,黄劭毅,田仁鹏,等. 一种适用于RT-PCR的杉树类植物中总RNA提取的方法[J]. 武汉植物学研究,2004,22(6):551-556.
[15] 李标,王伯初,唐坤,等. 铁皮石RNA提取及RT-PCR检测[J]. 中草药,2006,37(4):585-588.
[16] Jaakola L,Pirttila A M,Halonen M,et al. Isolation of high quality RNA from bilberry fruit[J]. Molecular Biotechnol,2001,19(2):201-203.
