DNA甲基化在非小细胞肺癌诊断和治疗中的应用
2010-01-24王中秋
王中秋, 许 林
肺癌是人类最常见的恶性肿瘤之一,我国肺癌的发病率和病死率一直呈上升趋势。传统观点认为,肿瘤发生、发展的主要机制是致癌因素造成DNA序列变异而导致细胞生长、分化失控。近年来,随着研究的深入,人们发现DNA序列以外的调控机制异常在肿瘤的发生、发展过程中更为普遍[1]。这种不依赖于DNA序列变化的可遗传的调控机制称为表观遗传学机制,主要包括DNA甲基化修饰、组蛋白修饰、染色体修饰、基因组印迹及microRNA调控等方式,其中DNA甲基化是表观遗传学研究最深入的一种机制。DNA甲基化是真核细胞基因组常见的表观遗传学修饰,也是脊椎动物DNA唯一的自然化学修饰方式,在细胞增殖、分化、发育、基因印迹等方面起重要作用,与肿瘤的发生、发展关系密切。目前,肿瘤相关基因DNA甲基化研究获得了大量的研究成果,这些结果不仅对深入理解癌变的分子机制具有重要意义,并且提示DNA甲基化研究对于肿瘤的诊断、治疗也具有巨大的应用潜力[2]。本文对DNA甲基化在非小细胞肺癌诊断和治疗中的研究进展作一综述。
1 DNA甲基化与肿瘤
DNA甲基化是最早发现的基因表观修饰方式之一,可能存在于所有高等生物中。甲基化的主要形式有5-甲基胞嘧啶、N6-甲基腺嘌呤和7-甲基鸟嘌呤。原核生物中CCA/TGG和GATC常被甲基化,而真核生物中甲基化仅发生于胞嘧啶。DNA的甲基化是在DNA甲基化转移酶(DNMTs)的作用下使CpG二核苷酸5′端的胞嘧啶转变为5’-甲基胞嘧啶。这种DNA修饰方式并没有改变基因序列,却可以调控基因的表达[3]。DNA甲基化能关闭某些基因的活性,去甲基化则诱导了基因的重新活化和表达。
甲基化状态的改变是引发肿瘤的一个重要因素。目前认为在肿瘤细胞基因组中发生甲基化模式的改变主要有2种模型:(1) 基因组整体甲基化水平降低导致基因组的不稳定(如染色体的不稳定、可移动遗传因子的激活、原癌基因的表达)[4];(2)CpG岛局部甲基化水平的异常升高,导致基因的不表达。健康人基因组中,CpG岛中的CpG位点通常处于非甲基化状态,而在CpG岛外的CpG位点则通常甲基化。这种甲基化的形式在细胞分裂的过程中能够稳定保留[5]。当肿瘤发生时,抑癌基因CpG岛以外的CpG序列非甲基化程度增加,而CpG岛中的CpG则呈高度甲基化状态,以致染色体螺旋程度增加及抑癌基因表达的丢失。大量报道显示,抑癌基因的沉默与其启动子区域CpG岛的异常甲基化直接相关,如许多细胞生长增殖相关基因与DNA损伤修复有关的基因启动子区域的异常甲基化都与其失活有关。DNA高甲基化不仅影响基因转录,还可导致C→T突变。DNA低甲基化则可导致原癌基因活化,增加染色体的不稳定性,形成突变热点。这些都会引起基因功能异常,进而导致细胞生长失控,最终形成肿瘤。由于CpG岛的局部高度甲基化早于细胞的恶性增生,因此甲基化的诊断可以用于肿瘤发生的早期预测。全基因组的低甲基化可随着肿瘤发生而出现,且随着肿瘤恶性度的增加而显著,可用于肿瘤的分级。因此,甲基化研究为肿瘤的早期诊断、分类、分级及预后评估提供了新的依据。
2 DNA甲基化与非小细胞肺癌早期诊断
目前对非小细胞肺癌的诊断主要依据临床症状、影像学检测和组织病理学检查等,但多数患者临床症状出现较晚,且活体取样困难,在首次诊断时已呈晚期,治疗困难,因此进行无创性早期诊断意义重大。与DNA变异相比,基因甲基化改变常是细胞癌变过程中更为早期的事件,可能在肿瘤早期诊断中具有更大的价值。大多数肿瘤都有多个独立的启动子甲基化事件,因此建立合理的多指标甲基化图谱能为肿瘤风险评估、早期诊断以及肿瘤预后提供有用信息。针对肿瘤组织的DNA甲基化研究为肿瘤无创性诊断奠定了基础。由于实体肿瘤取材困难,体液循环DNA容易获取,因此在体液中寻找更多的肿瘤特异DNA甲基化分子标志物的研究备受关注。
2.1 外周血循环DNA甲基化分析 正常人的外周血循环DNA来源于白细胞,而肿瘤患者的外周血循环DNA含有坏死或凋亡的肿瘤细胞、微转移灶或循环肿瘤细胞的裂解以及增值旺盛的肿瘤细胞所释放的DNA。已证实,在肿瘤患者血循环DNA中可检测到与原发肿瘤细胞相一致的DNA甲基化变化。在非小细胞肺癌患者外周血中已发现有超过25种基因的甲基化异常,部分列举见表1。
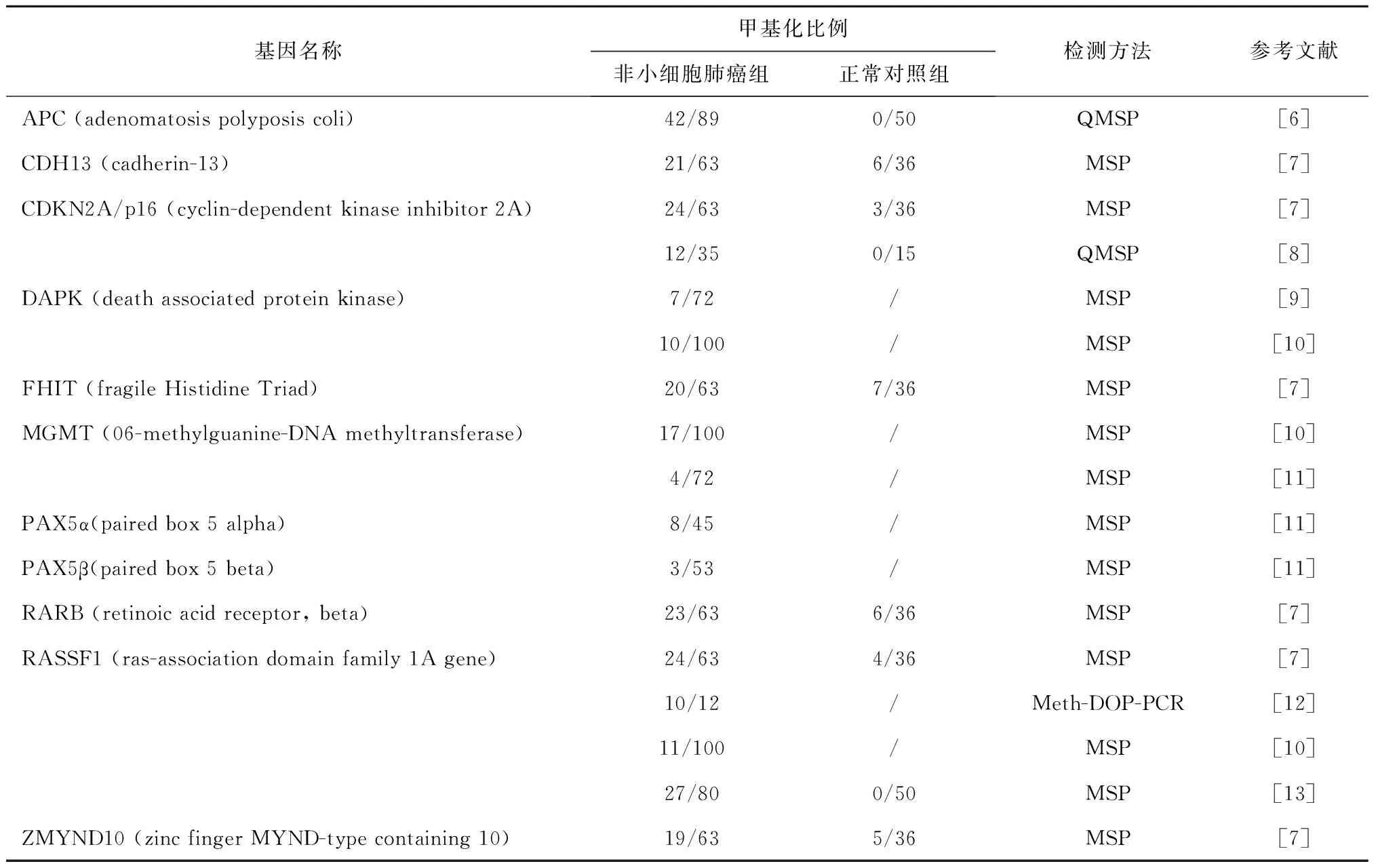
表1 非小细胞肺癌患者外周血中DNA甲基化分析
注:MSP:Methylation sensitivie PCR; QMSP:Qantitative MSP; Meth-DOP-PCR:Methylation-degenerate oligonucleotide primed PCR
如CDH13, CDKN2A/p16, DAPK, MGMT, PAX5α PAX5β, RASSF1A等诊断非小细胞肺癌的敏感性为7%~27%[11]。Fujiwara等[10]发现CDKN2A/P16, DAPK, MGMT, RARB, RASSF1A 5个基因中,任一基因的甲基化水平诊断非小细胞肺癌的敏感性仅约49.5%,特异性为85.0%。而联合检测多个基因可显著提高敏感性。Hsu等[7]联合检测了CDH13, CDKN2A/p16, FHIT, RARB, RASSF1A和ZMYND10,发现其中任何2个基因甲基化即判断为阳性,统计发现诊断敏感性为73%,特异性为82%,较单一指标显著提高。此外,外周血中CDKN2A甲基化检测联合微卫星分析的敏感性增加到62%, CDKN2A 甲基化检测联合循环DNA水平的特异性可增加到80%[8]。这些研究表明,联合检测多种基因甲基化水平或联合其他分子生物学方法在非小细胞肺癌的诊断方面具有重要的价值。然而,外周血的检测也有其局限性,首先从外周血中获得的肿瘤生物学特性无器官特异性,上述在非小细胞肺癌中有诊断意义的基因(如APC)在其他肿瘤中也存在。因此,寻找肺癌特殊的基因谱仍是目前研究的方向。其次,随着对支气管肺泡灌洗液及痰液研究的深入,研究者们逐渐发现,基因甲基化的检出率在后者中显著增高。
2.2 支气管肺泡灌洗液DNA甲基化分析 与外周血相比,支气管肺泡灌洗液更易于获得肺癌细胞并检测出肺癌特异性的分子生物学改变。Ahrendt等[14]首次报道了支气管肺泡灌洗液DNA甲基化分析对肺癌的诊断价值,发现19例p16基因高甲基化肺癌患者中,有16例患者的肺泡灌洗液中可检出相应的p16异常甲基化。而Topaloglu等[15]通过对31例肺癌患者肺泡灌洗液中8个基因(CDH1,APC,MGMT,RASSF1A,GSTP1,p16,RAR-beta2和ARF)的甲基化分析,发现可检出68%的肺癌患者。Belinsky等[16]的研究也显示支气管肺泡灌洗液DNA甲基化分析用于肺癌诊断具有比血清更高的敏感性。
2.3 痰液DNA甲基化分析 咳嗽、咳痰是非小细胞肺癌患者的常见症状之一。痰液作为一种特殊的体腔积液,比血液、支气管灌洗液更易于收集。此外,痰液在吸烟人群中的产生率远远高于非吸烟人群,因此痰液检测所提供的分子生物学信息有利于在高危人群中开展疾病筛查。Palmisano等[17]发现检测痰液CDKN2A/P16和(或)MGMT的启动子甲基化状态改变可比临床最终诊断为非小细胞肺癌早3年。Belinsky等[16]检测了72例非小细胞肺癌Ⅲ期患者痰液中的8个基因(p16, MGMT, RASSF1A, DAPK, PAX5 α, PAX5 β, H-cadherin, GATA5)的甲基化状态,并与各自肿瘤组织和血液中的甲基化状态相比较发现,p16、DAPK、PAX5 β、GATA5等4个基因的阳性预测率为44%~72%,阴性预测率≥70%。上述4个基因联合检测的阳性预测率高达86%。与相应的血清相比,痰液中DNA甲基化分析用于肺癌诊断的敏感性更高。上述研究表明,痰液中DNA甲基化的检测对晚期非小细胞肺癌的诊断,尤其是对不能手术无法获得组织标本的患者,有着重要的意义。
3 DNA甲基化与非小细胞肺癌的治疗
与遗传学改变不同,表观遗传修饰是可逆的,使其成为潜在的疾病治疗靶点[18]。由于在肿瘤形成过程中,DNA 上的多个基因位点发生高甲基化,导致肿瘤抑制因子等重要基因失活。因此,通过DNA 甲基转移酶抑制剂抑制特定位点上的甲基化可以纠正错误的甲基化修饰,进而直接改变基因表达。DNA 甲基转移酶抑制剂可分为核苷类DNA甲基转移酶抑制剂和非核苷类甲基转移酶抑制剂,其中核苷类甲基转移酶抑制剂包括5-氮胞嘧啶及其衍生物,非核苷类甲基转移酶抑制剂包括多酚-表没食子儿茶酚-3-没食子盐(EGCG)和RG108[19]。其中,研究得最多的DNA甲基转移酶抑制剂是5-氮杂-2′-脱氧胞苷酸(5-aza-dC, decitabine,地西他滨),在进入DNA后,与DNMT1 形成永久共价键,从而导致DNMT1的降低,DNA复制中DNMT1的缺少最终引起甲基化程度降低,低剂量持续给药可以获得癌细胞去甲基化的最大效果[20]。其主要副作用是骨髓抑制。地西他滨可以将沉默的肿瘤抑制因子激活,组蛋白脱乙酰基酶抑制剂 LAQ824(LAQ)可以将细胞周期停止的基因激活,两者联合使用可以协同产生更大的抗肿瘤效果。该结果已经在乳腺癌和骨髓恶性病变中证实[21],并正在开展肺癌的Ⅰ/Ⅱ期临床试验。然而,Stresemann 等[22]分析比较了6种常见的DNA 甲基化抑制剂,发现这些DNA 甲基化抑制剂具有很强的去甲基化效应,但其伴有较强的细胞毒性,在作用位点选择上也缺乏特异性。因此,DNA甲基化转移酶抑制剂的临床应用仍需进一步研究。
4 DNA甲基化的检测方法
近15年来,人们越来越认识到DNA甲基化研究的重要性,开发出一系列检测DNA的方法。根据研究目的将这些方法分为:基因组整体水平的甲基化检测、特异位点的甲基化检测和新甲基化位点的寻找。根据研究所用的处理方法不同分为:基于PCR的甲基化分析方法、基于限制性内切酶的甲基化分析方法、基于重亚硫酸盐的甲基化分析方法和柱层法等。
常用的DNA甲基化检测方法:(1)亚硫酸氢钠-测序法,是反映基因组甲基化状态的最直接、最可靠的方法。测序法以CpG岛两侧不含CpG点的一段序列为引物配对区,能够同时扩增出甲基化和未甲基化序列。缺点在于费时且耗资过多,并至少要测序10个以上的克隆才能获得可靠数据。(2)亚硫酸氢钠-限制性酶切法(combined bisulfite restriction analysis recovery assay,COBRA),引物设计与测序法相同, ssPCR 扩增经亚硫酸氢钠修饰的DNA样品中特定序列,用限制性内切酶识别序列中包含CpG的酶切位点。COBRA在检测完全甲基化和完全不甲基化的样品最常用。(3)MSP方法,原理是首先用亚硫酸氢钠修饰处理基因组DNA,所有未甲基化的C都被转化为U,而甲基化的胞嘧啶则不变。然后设计针对甲基化和非甲基化序列的引物并进行PCR扩增。MSP是目前最敏感的甲基化检测技术,能发现0.1%甲基化DNA (50 pg)。(4)实时荧光PCR法(QMSP),是基于甲基化特异性探针的Taq酶方法检测甲基化。(5)甲基化敏感的单一核苷酸引物延长法(Ms-SNuPE),用于定量分析CpG点的甲基化差异。
研究甲基化的方法之多,一方面说明了甲基化研究难度大,另一方面说明这些方法都存在着一定缺陷。面对具体问题,选择最合适的解决方法就显得尤为重要。首先,根据研究目的选择合适方法:是研究整体水平的甲基化还是特定位点的甲基化,或是要发现全基因组中新的甲基化位点;其次,根据客观条件筛选方法,如:目标的序列是否已知,是定量研究还是定性研究,样本来源及数量如何,是否需要高通量的样本检测方法;最后,全面分析,选取敏感、可靠、经济、简便的方法,以达到理想的效果。
5 问题与展望
表观遗传学改变在肿瘤形成过程中起着重要的作用,可以导致相关的多个基因失活和功能障碍。研究已经充分证明在非小细胞肺癌发生和发展的过程中存在着一系列甲基化水平的改变,因此可以通过多种甲基化标志物检测进行肺癌的早期诊断。利用DNA甲基化特异性PCR、RT-PCR、核酸定量分析和基因芯片等技术,对基因组范围内的甲基化进行检测,可能是非小细胞肺癌和其他肿瘤分子检测的发展方向。此外,由于基因启动子等区域的高甲基化是可逆的,通过去甲基化药物处理可以使其恢复表达,有可能为肺癌的治疗提供新的思路。
尽管过去数年利用DNA甲基化检测进行非小细胞肺癌的诊治已取得令人瞩目的进步,但这些研究仍限于实验室中。目前对DNA甲基化调节基因表达的复杂性仍然知之甚少。DNA异常甲基化在肿瘤形成机制中的确切作用尚不清楚,进一步探讨抑癌基因甲基化与肺癌的早期发生、发展的关系,了解DNA甲基化如何建立和维持的机制等,有助于阐明肺癌形成的原因。针对DNA甲基化的靶向治疗药物的研究,将为非小细胞肺癌的治疗提供新的希望。因此,对非小细胞肺癌相关抑癌基因异常的甲基化研究有助于肿瘤的早期诊断并推动病因、发病机制、转归、治疗及预后的研究。
[1] Buysschaert I, Schmidt T, Roncal C, et al. Genetics, epigenetics and pharmaco-(epi) genomics in angiogenesis[J]. J Cell Mol Med, 2008, 12(6B): 2533-2551.
[2] Humeniuk R, Mishra PJ, Bertino JR, et al. Molecular targets for epigenetic therapy of cancer[J]. Curr Pharm Biotechnol, 2009, 10(2): 161-165.
[3] Herman JG, Baylin SB. Gene silencing in cancer in association with promoter hypermethylation[J]. N Engl J Med, 2003, 349(21): 2042-2054.
[4] Feinberg AP, Tycko B. The history of cancer epigenetics[J]. Nat Rev Cancer, 2004, 4(2):143-153.
[5] Laird PW. The power and the promise of DNA methylation markers[J]. Nat Rev Cancer, 2003, 3(4): 253-266.
[6] Usadel H, Brabender J, Danenberg KD, et al. Quantitative adenomatous polyposis coli promoter methylation analysis in tumor tissue, serum, and plasma DNA of patients with lung cancer[J]. Cancer Res, 2002, 62(2): 371-375.
[7] Hsu HS, Chen TP, Hung CH, et al. Characterization of a multiple epigenetic marker panel for lung cancer detection and risk assessment in plasma[J]. Cancer, 2007, 110(9): 2019-2026.
[8] Bearzatto A, Conte D, Frattini M, et al. p16(INK4A) Hypermethylation detected by fluorescent methylation-specific PCR in plasmas from non-small cell lung cancer[J]. Clin Cancer Res, 2002, 8(12): 3782-3787.
[9] Laird PW, Jaenisch R. The role of DNA methylation in cancer genetic and epigenetics[J]. Annu Rev Genet, 1996, 30: 441-464.
[10] Fujiwara K, Fujimoto N, Tabata M, et al. Identification of epigenetic aberrant promoter methylation in serum DNA is useful for early detection of lung cancer[J]. Clin Cancer Res, 2005, 11(3): 1219-1225.
[11] Belinsky SA, Grimes MJ, Casas E, et al. Predicting gene promoter methylation in non-small-cell lung cancer by evaluating sputum and serum[J]. Br J Cancer, 2007, 96(8):1278-1283.
[12] Di Vinci A, Gelvi I, Banelli B, et al. Meth-DOP-PCR: an assay for the methylation profiling of trace amounts of DNA extracted from bodily fluids[J]. Lab Invest, 2006, 86(3):297-303.
[13] Wang Y, Yu Z, Wang T, et al. Identification of epigenetic aberrant promoter methylation of RASSF1A in serum DNA and its clinicopathological significance in lung cancer[J]. Lung Cancer, 2007, 56(2): 289-294.
[14] Ahrendt SA, Chow JT, Xu LH, et al. Molecular detection of tumor cells in bronchoalveolar lavage fluid from patients with early stage lung cancer[J]. J Natl Cancer Inst, 1999, 91(4): 332-339.
[15] Topaloglu O, Hoque MO, Tokumaru Y, et al. Detection of promoter hypermethylation of multiple genes in the tumor and bronchoalveolar lavage of patients with lung cancer[J]. Clin Cancer Res, 2004, 10(7): 2284-2288.
[16] Belinsky SA, Grimes MJ, Casas E, et al. Predicting gene promoter methylation in non-small cell lung cancer by evaluating sputum and serum[J]. Br J Cancer, 2007, 96(8): 1278-1283.
[17] Palmisano WA, Divine KK, Saccomanno G, et al. Predicting lung cancer by detecting aberrant promoter methylation in sputum[J]. Cancer Res, 2000, 60(21): 5954-5958.
[18] Egger G, Liang G, Aparicio A, et al. Epigenetics in human disease and prospects for epigenetic therapy[J]. Nature, 2004, 429(6990): 457-463.
[19] Lyko F, Brown R. DNA methyltransferase inhibitors and the development of epigenetic cancer therapies[J]. J Natl Cancer Inst, 2005, 97(20):1498-1506.
[20] Yang AS, Doshi KD, Choi SW, et al. DNA methylation changes after 5-aza-2′-deoxycytidine therapy in patients with leukemia[J]. Cancer Res, 2006, 66(10): 5495-5503.
[21] Gore SD, Baylin S, Sugar E, et al. Combined DNA methyltransferase and histone deacetylase inhibition in the treatment of myeloid neoplasms[J]. Cancer Res, 2006, 66(12): 6361-6369.
[22] Stresemann C, Brueckner B, Musch T, et al. Functional diversity of DNA methyltransferase inhibitors in human cancer cell lines[J]. Cancer Res, 2006, 66(5): 2794-2800.
