甜玉米基因Sugary1(Su1)序列的变异分析
2024-12-31杨彩霞顾炜关媛瞿静涛党冬冬吴鹏昊郑洪建



摘 要:【目的】研究甜玉米基因Sugary1(Su1)序列,分析核苷酸多态性及检验,研究不同来源甜玉米中的Su1的等位变异位点,为甜玉米的品种改良和提高品质提供理论基础。
【方法】以B73的Su1基因组序列为模板,对170份甜玉米自交系的Su1基因进行测序,研究玉米Su1基因序列的多态性。
【结果】共有97个核苷酸位点发生变异,包含85个SNP位点,12个InDel位点。其中,位于编码区的SNP位点12个,InDel位点0个;位于非编码区的SNP位点85个,InDel位点12个。编码区中SNP位点同义突变有5个,非同义突变7个。自交系材料分为76个单倍型,该基因编码区序列将自交系材料分为47种单倍型。
【结论】甜玉米Su1基因经历了明显的人工选择。
关键词:甜玉米;Sugary1;等位变异;中性检验;单倍型
中图分类号:S513;S188"" 文献标志码:A"" 文章编号:1001-4330(2024)07-1605-10
0 引 言
【研究意义】甜玉米(Zea mays L.saccharata Sturt)含有丰富的维生素、氨基酸、矿物质和微量元素,以及谷胱甘肽、亚油酸等营养物质,是一种高品质、高营养的果蔬[1-4]。普通甜玉米可以加工成不同的食品和饮料,如玉米面粉、玉米糖浆、玉米油等[5-6]。甜玉米是受一个或多个隐性基因控制的胚乳突变体,是一种特用型玉米。根据基因型和胚乳特性不同可分为普通甜玉米Sugary1,(Su1),超甜玉米(Shrunken2,Brittleendosperm1),加强甜玉米(Suguary enchancer1)[1-5]。【前人研究进展】Su1基因突变影响普通甜玉米的糖分含量,su1突变体在淀粉合成途径中失去了功能[6-8],编码淀粉脱分支酶,该酶通过切断α-1,6-糖苷键去除了淀粉中部分分枝[9-13],蔗糖积累变少。普通甜玉米在胚乳乳熟期的总糖量达到了15.6%,外型呈现透明角质状,有细小褶皱[14-16]。在su1主要的等位基因中,su1-ref、su1-nc和su1-sw3个等位基因编码酶蛋白序列中包含单氨基酸替换使蛋白失活[12]。【本研究切入点】目前针对甜玉米Su1基因序列变异多态性鲜见报道。需分析不同来源甜玉米中Su1的等位变异位点和单倍型分型。【拟解决的关键问题】分析170份甜玉米自交系Su1基因进行序列变异,检测甜玉米自然群体中Su1的变异位点,研究群体材料的单倍型,以辅助甜玉米品种改良。
1 材料与方法
1.1 材 料
选用170份甜玉米自交系作为材料(上海市农业科学院作物育种栽培研究所玉米中心提供)。种植于2022年3月在上海市农业科学院庄行综合试验站的大棚内。
1.2 方 法
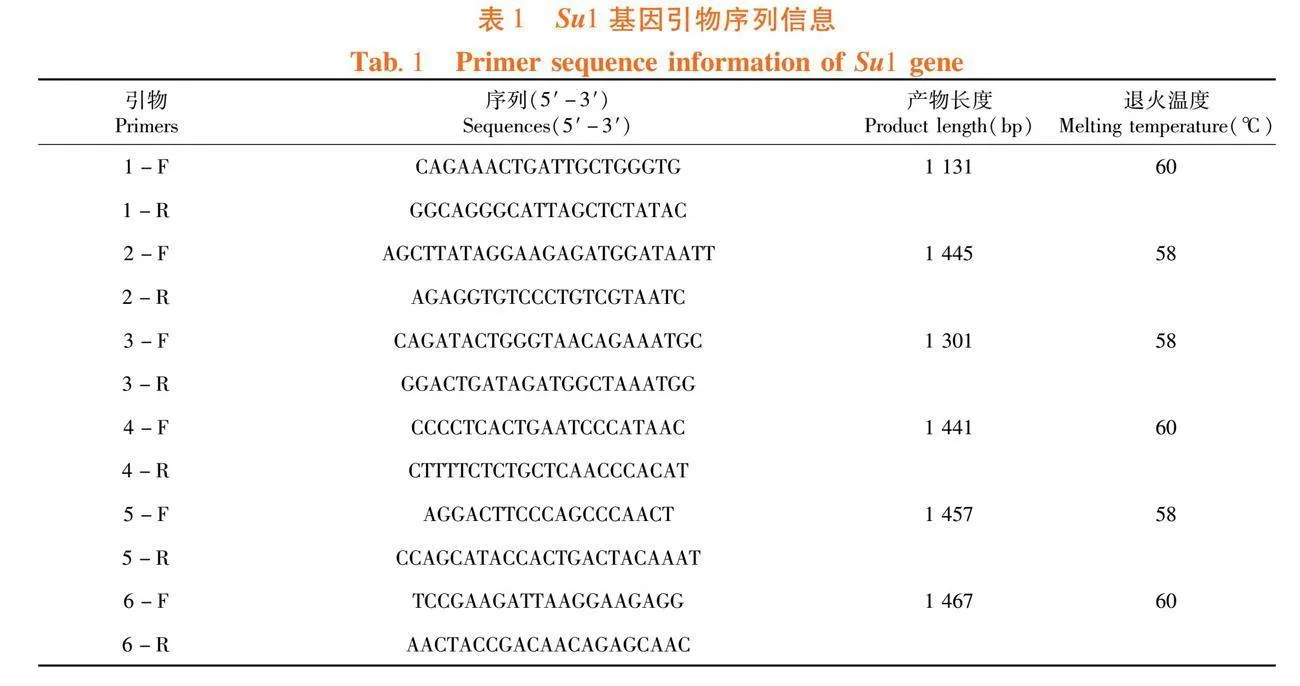
供试玉米幼苗3叶1心时剪取嫩叶,采用改良的CTAB[17]法提取基因组DNA。并用分光光度计检测DNA浓度、利用琼脂糖凝胶电泳方法检测DNA质量。在玉米数据库(https://www.maizegdb.org/),以B73参考基因组的Su1 (Zm00001eb174590)序列为模板,分别获得各材料的Su1基因序列信息,使用Primer 5.0软件根据基因序列设计引物序列,并在NCBI数据库(https://www.ncbi.nlm.nih.gov/)验证引物的特异性。分段扩增各材料的Su1基因,并进行一代测序。表1
1.3 数据处理
采用ContigExpress软件和MEGA 7.0进行人工校对和序列变异分析。中性检验采用DNAsp软件[18]进行,Tajima’ D[19]检验通常被广泛用于检测目标DNA序列是否符合中性进化模型,以确定其在进化过程中是否存在非随机性。θ和π[20]是核苷酸多态性分析过程中的常用参数,其中π被用来衡量同一位点不同序列两两之间的差异;而群体突变率则是用θ来衡量。
2 "结果与分析
2.1 Su1基因目的片段扩增
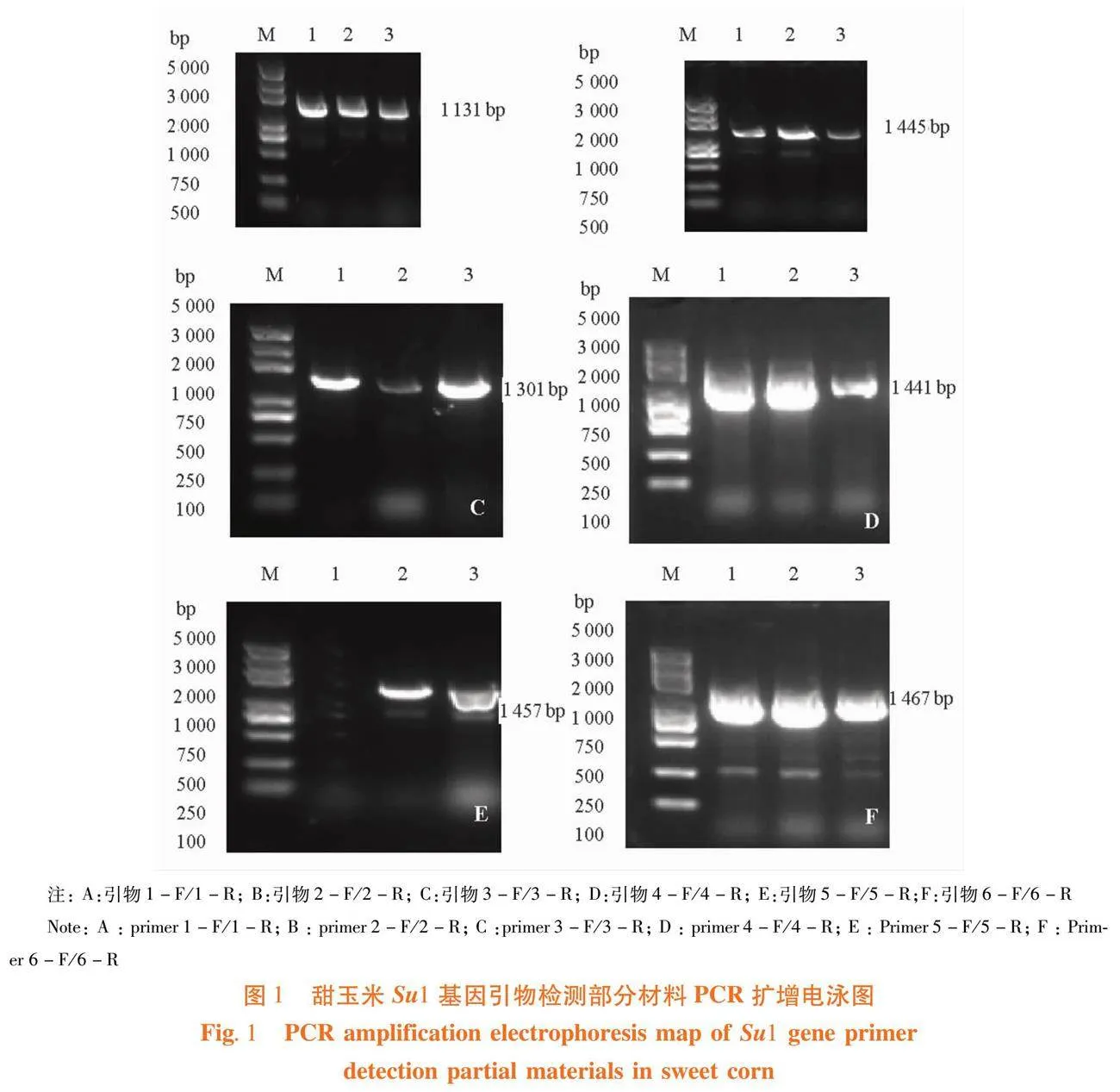
研究表明,6对引物覆盖甜玉米Su1基因的不同区域。通过普通PCR方法,以B73的基因Su1 (Zm00001eb174590)序列为模板,利用6对引物1-F/1-R,2-F/2-R,3-F/3-R,4-F/4-R,5-F/5-R,6-F/6-R,分别对170份自交系材料的Su1基因进行分段扩增。图1
2.2 甜玉米Su1基因的序列变异
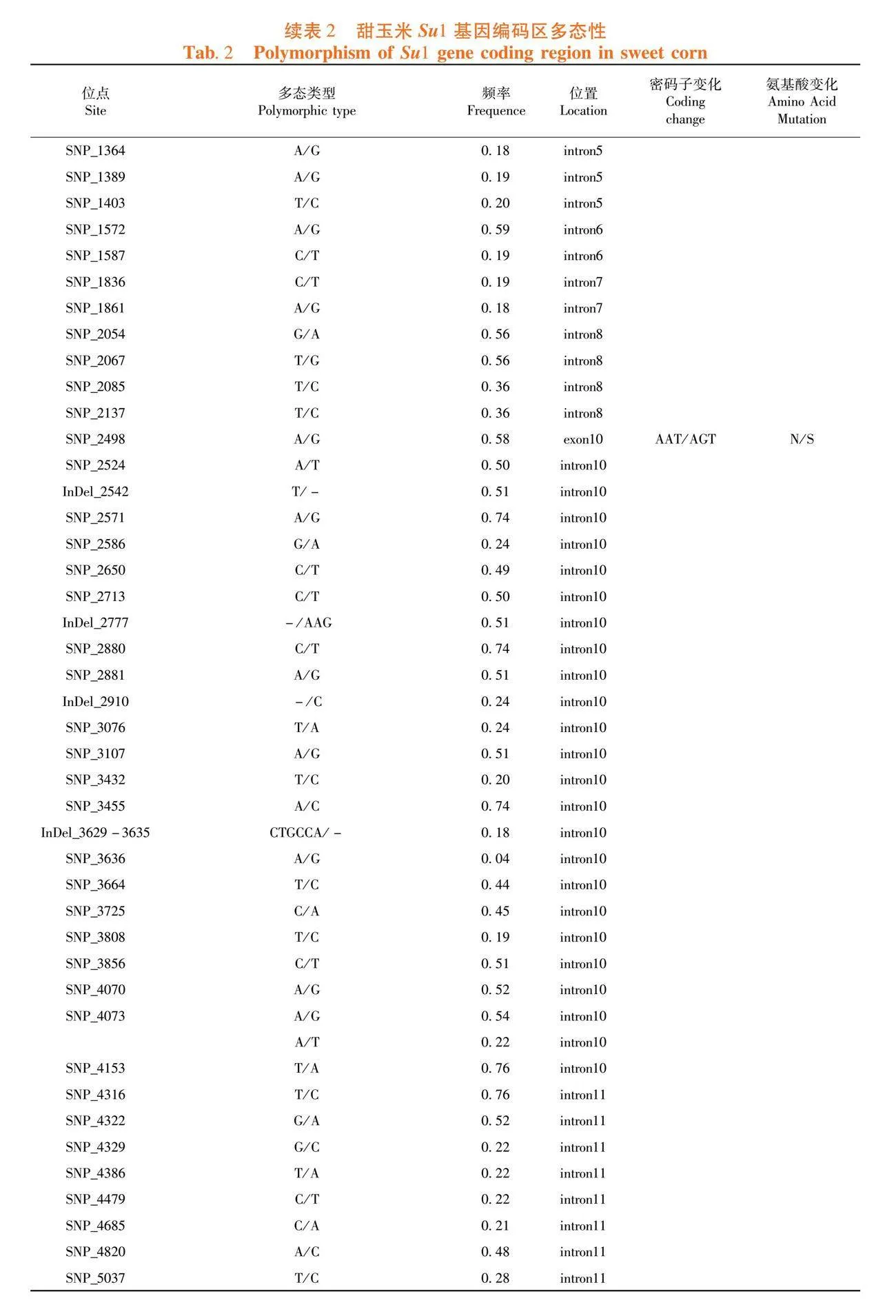
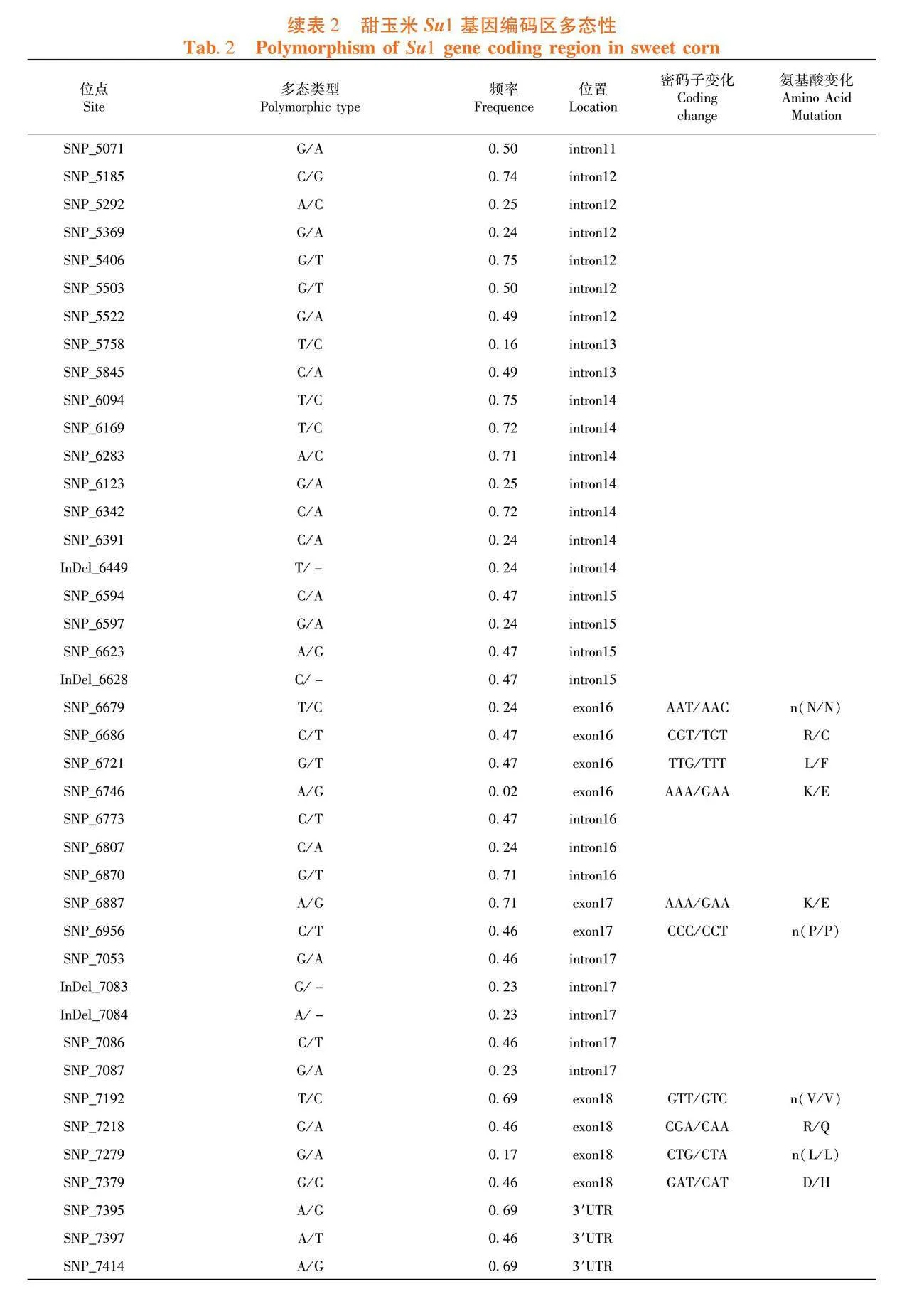
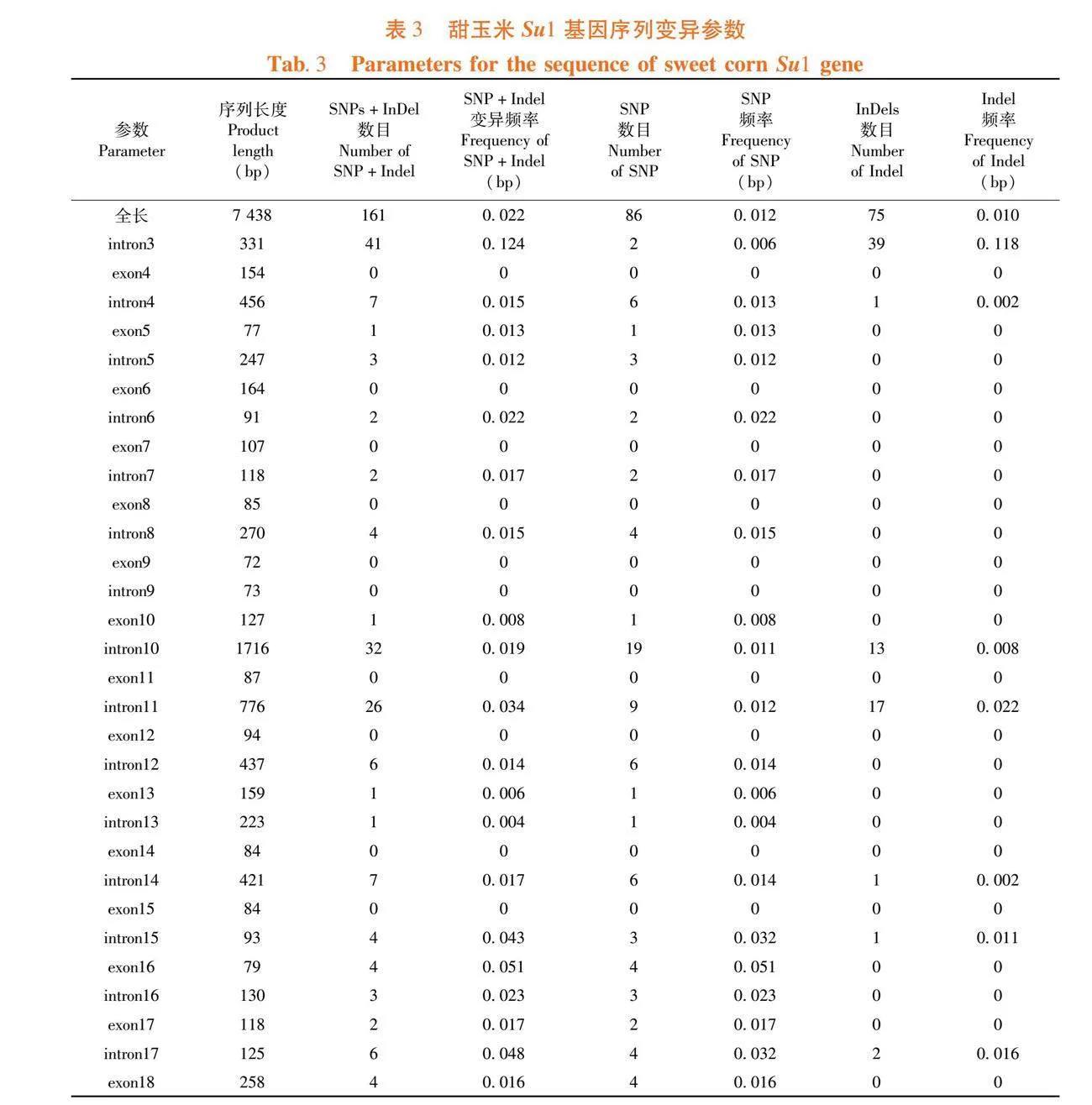
研究表明,以玉米自交系B73的Su1基因为参考,检测170个玉米自交系中Su1的等位变异位点,共得到Su1基因的全长序列7 429 bp,包括5 609 bp的非编码区和1 820 bp的编码区。非编码区的变异位点显示高于编码区,非编码区是主要的遗传变异来源。共有97个核苷酸位点发生变异,其中包含85个SNP位点和12个InDel位点。其中,位于编码区的SNP位点有12个,InDel位点0个,位于非编码区的SNP位点有85个,InDel位点有12个。变异频率最高的第16外显子区段,为0.51,在10个区段未发现变异位点。表2,表3
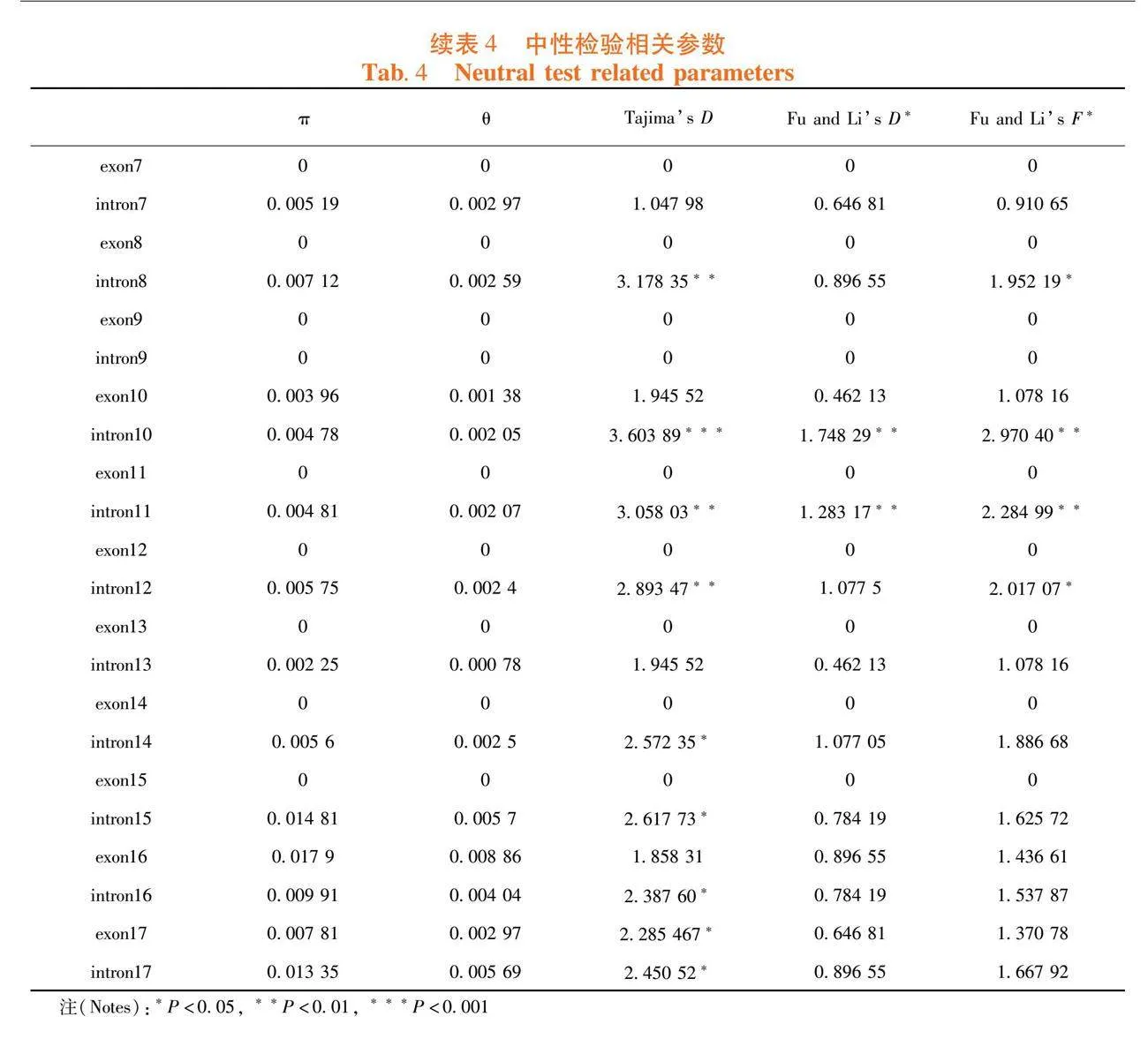
所有内含子,外显子的Tajiama’ D值均为正值,内含子3、4、14、15、16和17和外显子17、18达到了显著水平(Plt;0.05),内含子8、10、11、12的Tajiama’ D的值均为正值且达到了极显著水平(Plt;0.01),这些内含子区和外显子区序列中大多变异位点具有低等频率的等位差异存在。Fu与Li的D*测验和F*测验结果与Tajim′s D测验结果基本相吻合。内含子9、外显子4、6、7、8、9、11、12、14和15未发生碱基变化,所以Tajim’s D值为0。在供试群体中,Su1基因经历了明显的人工选择。表4
2.3 Su1基因的单倍型
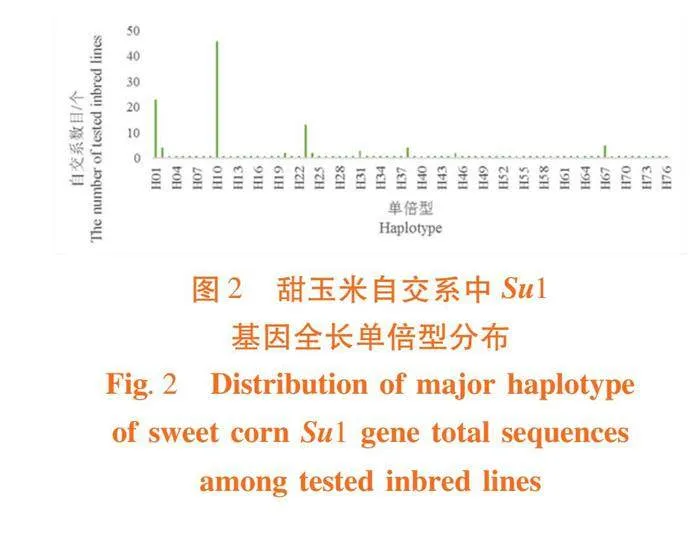
研究表明,从170份材料中共检测出76个单倍型,携带单倍型Hap_10的自交系数目最多,为46个。有10种单倍型包含两个及以上的自交系,有160个单倍型仅包含一个自交系。图2
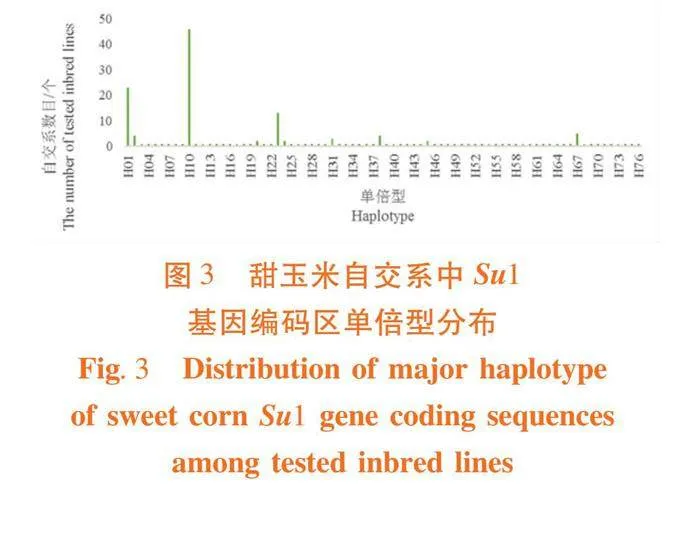
将供试材料划分成47种单倍型,自交系在这些单倍型中分布不均匀,其中,HC09包含的单倍型个数最多,有55个供试自交系,占供试材料的32.4%,是该基因编码区主要单倍型。携带两个以上自交系的编码区单倍型有14个,仅含有一个供试自交系的单倍型有33个。图3
在编码区的12个SNP变异位点中,有5个为同义突变位点,不能引起编码蛋白质序列的改变;其余7个为非同义突变位点,可以引起编码蛋白质序列的改变。自然群体中Su1基因编码21种SU1蛋白质,长度为606或605或604 aa。序列长度的差异是由于编码区一个6 bp的InDel引起的。在47种单倍型中,只有11种单倍型包含2个以上的自交系,其余的只包含1个自交系。第9种单倍型包含55个甜玉米自交系,所占比率超过32.4%。此外,HC01、HC07和HC18也是高频单倍型。图4
3 讨 论
3.1
研究结果表明,所有内含子,外显子的Tajiama’ D值均为正值,内含子3、4、14、15、16、17和外显子17、18达到了显著水平(Plt;0.05),内含子8、10、11、12的Tajiama’ D的值均为正值且达到了极显著水平(Plt;0.01),这些内含子区和外显子区序列中大多变异位点具有低等频率的等位差异存在,对于编码区的选择强于其他各个区段。Fu与Li的D*测验和F*测验结果与Tajim′s D测验结果基本相吻合,表明在供试群体中,Su1基因经历了明显的自然和人工双重选择。
3.2
杨泽峰等[21]对104个玉米自交系的Dbf1基因进行研究,结果发现,在编码区平均120 bp有1个SNP。Ching等[22]使用序列变异分析对36个玉米自交系进行了研究,发现在非编码区,每31 bp会出现一个SNP;而在编码区,每124 bp会出现一个SNP。研究发现在编码区1 749 bp有12个SNP,平均每146 bp中有1个SNP。与前人研究相比编码区的SNP变异频率略高,可能因为试验研究的群体较杨泽锋等[22]的群体大。
4 结 论
甜玉米Su1基因的全长序列在170个甜玉米自交系中共发现85个SNP位点和12个InDel位点。该基因的编码区含有12个SNP,其中7个为非同义突变位点,并编码6种Su1基因蛋白质。在供试的玉米群体中,该基因经历了明显的人工选择。
参考文献(References)
[1]
陈骁熠, 瞿宏杰. 甜玉米的品质改良技术[J]. 农村经济与科技, 2001, 12(8): 15.
CHEN Xiaoyi, QU Hongjie. Quality improvement technology of sweet corn [J]. Rural Economy and Science-Technology, 2001, 12(8): 15.
[2] 曹忠良, 张俊江. 甜玉米的类型及关键性技术[J]. 民营科技, 2013, (5): 212.
CAO Zhongliang, ZHANG Junjiang. Types of sweet corn and key technologies [J]. NonState Running Science Technology Enterprises, 2013, (5): 212.
[3] 蒙云飞, 冯云敢, 贺囡囡, 等. 甜玉米的起源和分布及遗传基因研究进展[J]. 现代农业科技, 2017, (5): 4-5.
MENG Yunfei, FENG Yungan, HE Nannan, et al. Research progress on origin distribution and genetic genes of sweet corn[J]. Modern Agricultural Science and Technology, 2017, (5): 4-5.
[4] 邵振中. 甜玉米的研究现状与发展[J]. 襄樊职业技术学院学报, 2004, 3(4): 23-25.
SHAO Zhenzhong. The current situation of research and development of sweet corn[J]. Journal of Xiangfan Vocational and Technical College, 2004, 3(4): 23-25.
[5] Zhang X, Mogel K J H V, Lor V S, et al. Maizesugary enhancer1 (se1) is a gene affecting endosperm starch metabolism[J]. Proceedings of the National Academy of Sciences of the United States of America," 2019, 116(41): 20776-20785.
[6] Ferguson J E, Dickinson D B, Rhodes A M. Analysis of endosperm sugars in a sweet corn inbred (illinois 677a) which contains the sugary enhancer (se) gene and comparison of se with other corn genotypes[J]. Plant Physiology, 1979, 63(3): 416-420.
[7] Chhabra R, Muthusamy V, Gain N, et al. Allelic variation in sugary1 gene affecting kernel sweetness among diverse-mutant and-wild-type maize inbreds[J]. Molecular Genetics and Genomics: MGG," 2021, 296(5): 1085-1102.
[8]"""" La Bonte D R, Juvik J A. Characterization ofsugary-1 (su-1) sugary enhancer (se) Kernels in Segregating Sweet Corn Populations[J]. Journal of the American Society for Horticultural Science, 1990, 115(1): 153-157.
[9] Schultz J A, Juvik J A. Current models for starch synthesis and the sugary enhancer1 (se1) mutation inZea mays[J]. Plant Physiology and Biochemistry: PPB, 2004, 42(6): 457-464.
[10] Yu J K, Moon Y S. Corn starch: quality and quantity improvement for industrial uses[J]. Plants, 2021, 11(1): 92.
[11] Pan D, Nelson O E. A debranching enzyme deficiency in endosperms of the sugary-1 mutants of maize[J]. Plant Physiology, 1984, 74(2): 324-328.
[12] Trimble L, Shuler S, Tracy W F. Characterization of five naturally occurring alleles at the Sugary1 locus for seed composition, seedling emergence, and Isoamylase1 activity[J]. Crop Science, 2016, 56(4): 1927-1939.
[13] Rahman A, et al. Characterization of SU1 isoamylase, a determinant of storage starch structure in maize[J]. Plant Physiology, 1998, 117(2): 425-435.
[14]""" Revilla P, Anibas C M, Tracy W F. Sweet Corn Research around the World 2015–2020[J]. Agronomy, 2021, 11(3): 534.
[15] Szke L, Moloi M J, Kovács G E, et al. The application of phytohormones as biostimulants in corn smut infected Hungarian sweet and fodder corn hybrids[J]. Plants, 2021, 10(9): 1822.
[16] Chourey P S, Nelson O E. The enzymatic deficiency conditioned by the shrunken-1 mutations in maize[J]. Biochemical Genetics, 1976, 14(11/12): 1041-1055.
[17]""" 顾炜. 玉米籽粒突变基因Mn219和Dek39的图位克隆与基因功能分析[D]. 北京:中国农业大学. 2018.
GU Wei. The map-based cloning and functional analysis of MN219 and Dek39 in maize[D]. Beijing: China Agricultural University, 2018.
[18] Rozas J, Ferrer-Mata A, Sánchez-DelBarrio J C, et al. DnaSP 6: DNA sequence polymorphism analysis of large data sets[J]. Molecular Biology and Evolution," 2017, 34(12): 3299-3302.
[19] Fu Y X, Li W H. Statistical tests of neutrality of mutations[J]. Genetics, 1993, 133(3): 693-709.
[20] Tajima F. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism[J]. Genetics, 1989, 123(3): 585-595.
[21] 杨泽峰, 张恩盈, 蒋莹, 等. 玉米耐旱相关基因dbf1的序列变异分析[J]. 玉米科学, 2014, 22(5): 20-28.
YANG Zefeng, ZHANG Enying, JIANG Ying, et al. Nucleotide diversity of drought tolerance related maize gene dbf1[J]. Journal of Maize Sciences, 2014, 22(5): 20-28.
[22] Ching A, Caldwell K S, Jung M, et al. SNP frequency, haplotype structure and linkage disequilibrium in elite maize inbred lines[J]. BMC Genetics, 2002, 3: 19.
Variation analysis of sweetness gene Sugary1 (Su1)
sequence in Sweet corn
YANG Caixia1,2, GU Wei2,3, GUAN Yuan2,3, QU Jingtao2,3,
DANG Dongdong2,3,WU Penghao1, ZHENG Hongjian2,3
(1." College of Agronomy, Xinjiang Agricultural University, Urumqi, 830052, China; 2 .Institute of Crop Breeding and Cultivation, Shanghai Academy of Agricultural Sciences/CIMMYT-China Special Maize Research Center, Shanghai 201403, China; 3.Shanghai Special Maize Engineering Technology Research Center, Shanghai 201403, China)
Abstract:【Objective】 The Sugary1 (Su1) gene of sweet corn was analyzed by nucleotide polymorphism analysis and neutral test, and the allelic variation sites of Su1 in sweet corn from different sources were analyzed, which provided a theoretical basis for variety improvement and quality improvement of sweet corn.
【Methods】 In order to illustrate the sequence polymorphism of maize gene Su1, the Su1gene in 170 sweet corn inbred lines was sequenced, using the genomic sequence of B73 as the reference.
【Results】" Analysis showed that 97 nucleotide sites were mutated, including 85 SNPs sites and 12 InDels locus. Among them, there were 12 SNPs loci and 0 InDel in the coding region, 85 SNPs loci and 12 InDels in the non-coding region. There were 5 synonymous mutations at SNP site and 7 non-synonymous mutations in the coding region. The inbred lines were divided into 76 haplotypes according to the full-length sequence of Su1 gene, and 47 haplotypes were divided into 47 haplotypes according to the coding region of the gene.
【Conclusion】" The Su1 gene had undergone obvious artificial selection.
Key words:sweet corn; Sugary1; allelic variation; neutral test; haplotype
Fund projects: Science and Technology Plan Project of Shanghai Municipal Commission of Science and Technology"Nutritional and Healthy Functional Germplasm Innovation and Breeding of New Varieties of Fresh"(20392000400); Shanghai Science and Technology Agricultural Technology Innovation Project"Creation of haploid-inducing lines in glutinous maize and application to engineering breeding"(2022-02-08-00-F01190)
Correspondence author: WU Penghao(1984-), male, from Xinjiang,professor,research direction: maize genetic breeding,(E-mail)craie788@126.com
ZHENH Hongjian(1973-), male, from Shandong, researcher, research direction: maize genetic breeding,(E-mail)hjzh6188@163.com
收稿日期(Received):
2023-12-17
基金项目:
上海市科学技术委员会科技计划项目“鲜食玉米营养、健康功能种质创新与新品种选育”(20392000400); 上海市科技兴农项目“糯玉米单倍体诱导系的创制及工程化育种应用”(2022-02-08-00-F01190)
作者简介:
杨彩霞(1997-),女,重庆人,硕士研究生,研究方向为玉米遗传育种,(E-mail)yangcx4760@163.com
通讯作者:
吴鹏昊(1984-),男,新疆人,教授,研究方向为玉米遗传育种,(E-mail)craie788@126.com
郑洪建(1973-),男,山东人,研究员,研究方向为玉米遗传育种,(E-mail)hjzh6188@163.com
