基于线粒体D-loop序列的江苏克氏原螯虾养殖群体遗传多样性分析
2024-07-03鲁苏皖徐宇李佳佳高志宝严维辉许志强王兴强李旭光
鲁苏皖 徐宇 李佳佳 高志宝 严维辉 许志强 王兴强 李旭光
doi:10.15889/j.issn.1002-1302.2024.10.026
摘要:江苏是我国克氏原螯虾的主产区,近年来繁养模式不断变化,多代繁养后的遗传多样性变化情况尚未可知,以及江苏地区克氏原螯虾养殖种群的群体遗传学特征数据尚不清楚。为研究当前江苏主产区克氏原螯虾养殖群体的遗传多样性特征,对来自江苏南京、徐州、连云港、扬州、淮安、无锡、泰州兴化和宿迁泗洪的8个克氏原螯虾养殖群体样本的线粒体控制区D-loop基因片段进行PCR扩增和测序分析,分析了不同养殖群体的遗传结构特点。共测序获得了8个群体223个样本的线粒体D-loop序列,比对长度包括863个位点,其中,变异位点95个,简约信息位点25个。基因序列中AT含量(80.01%)高于CG含量(19.99%),表现出较为明显的碱基组成偏倚性。223个样本共定义了37种单倍型,其中,单倍型Hap 1为8个群体的共有单倍型。泗洪群体独有的单倍型最多,泰州兴化群体独有的单倍型最少。本研究所涉及的养殖群体总体单倍型多样性指数(Hd)为0.39,总体核苷酸多样性指数(Pi)为0.003 37,遗传多样性处于较低水平。其中,宿迁泗洪群体的遗传多样性指数最高(Hd为0.632),兴化群体的遗传多样性指数最低(Hd为0.228)。不同群体间的遗传距离总体较小,其中,南京群体与其他群体间的遗传距离相对较大(0.005),提示南京群体与其他群体间的基因交流较少。8个养殖群体间的遗传分化系数为-0.219 6~0.317 33,徐州、扬州、无锡及连云港群体间的遗传分化系数为负值,表明这些群体间存在较为频繁的基因交流。分子方差分析(A-MOVA)结果显示,95.35%的遗传变异来自于群体内,群体间的遗传变异较少(4.65%)。最小跨度网络图和系统发育分析均未显示出单倍型与不同群体的对应关系,提示当前江苏克氏原螯虾养殖群体间的基因交流较为频繁。
关键词:克氏原螯虾(Procambarus clarkii);江苏地区;遗传多样性;遗传结构
中图分类号:S968.22 文献标志码:A
文章编号:1002-1302(2024)10-0192-05
收稿日期:2023-07-21
基金项目:江苏省种业振兴“揭榜挂帅”项目(编号:JBGS[2021]032);江苏现代农业(克氏原螯虾)产业技术体系建设项目(编号:JATS[2022]411);江苏省农业重大新品种创制项目(编号:PZCZ201746)。
作者简介:鲁苏皖(1997— ),男,江苏宿迁人,硕士研究生,主要从事甲壳动物增养殖研究。E-mail:136502596@qq.com。
通信作者:李旭光,博士,研究员,主要从事水生甲壳类繁育和生态养殖研究。E-mail:xuguangli1981@163.com。
克氏原螯虾(Procambarus clarkii)俗称小龙虾,属于节肢动物门甲壳亚目软甲纲十足目螯虾科,原产于美洲,20世纪30年代从日本引入我国南京,2021年,我国克氏原螯虾养殖面积173万hm2、产量263.36万t,现已成为我国最重要的水产经济物种之一[1]。长期以来,国内克氏原螯虾养殖过程中普遍采用繁养一体的繁育方式、捕大留小的捕捞手段,捕捞剩下的小个体成虾留塘自繁自养,繁养过程中的“逆向选择”问题较为严重。水产养殖群体的遗传多样性评估对良种选育和资源保护等方面具有重要意义。王长忠等对长江中下游地区野生的克氏原螯虾的遗传多样性进行了本底调查,研究表明4个克氏原螯虾群体的遗传多样性处于中等水平[2]。曹玲亮使用了微卫星标记分析了安徽3个水系中野生克氏原螯虾群体的遗传多样性和遗传结构,发现调查水域中克氏原螯虾群体的遗传多样性处于较高水平,但多个群体存在瓶颈效应[3]。彭刚对安徽地区的克氏原螯虾养殖和野生群体进行了遗传多样性分析,发现安徽地区克氏原螯虾人工养殖群体具有较高的遗传多样性[4]。邢智珺对江苏地区克氏原螯虾野生群体进行了群体遗传学特征研究,发现江苏克氏原螯虾野生群体遗传多样性较高,不同群体间存在中度遗传分化[5]。而有关江苏地区克氏原螯虾养殖群体的遗传多样性鲜有报道。
线粒体D-loop区缺乏编码的压力,是线粒体基因中变异速率最快的区域,常被用来研究种群间及种群内的遗传分化。江苏是我国克氏原螯虾产业的发源地,养殖时间长,养殖业发展规模大,同样存在繁养过程中“逆向选择”的问题,目前产生的种质资源退化问题与遗传多样性之间的关系尚不明确。本研究以江苏克氏原螯虾主要养殖地区群体作为研究对象,基于线粒体D-loop区序列变异分析研究了多个养殖群体的群体遗传学特征,以期为克氏原螯虾良种选育及合理选择繁养模式提供参考依据。
1 材料与方法
1.1 样本采集
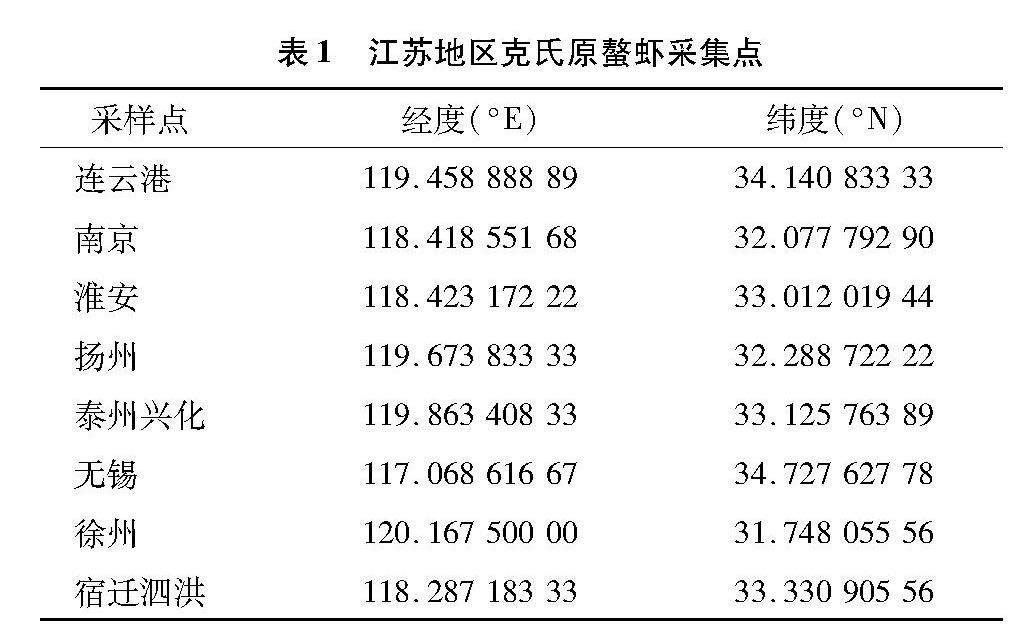
克氏原螯虾样本于2021年5—9月收集于无锡、南京、扬州、淮安、泰州兴化、宿迁泗洪、连云港、徐州8个地区的养殖群体(表1),采样地为江苏地区克氏原螯虾养殖主要产区。每个地区样品采集量30尾以上,共计260尾,所有样品均活体运输至实验室后,取尾部肌肉保存于-20 ℃备用。
1.2 基因组DNA提取
采用TIANGEN公司的基因组DNA提取试剂盒(DP304)提取不同样本的基因组DNA,使用核酸蛋白仪(TGem Pro 分光光度计)测定DNA浓度,使用1%琼脂糖凝胶电泳检测DNA质量。
1.3 PCR扩增、测序
参照克氏原螯虾线粒体序列信息(JAIWQB 000000000.1)设计引物用于扩增D-loop区序列,引物序列为:Crayfish DLOOP-F(5′-TCGCTGTAAAGTTGAAGAAGTT-3′)和Crayfish DLOOP-R(5′-TTAATCTCTTCATATCTTTAATTAC-3′)引物由上海生工生物公司合成。PCR反应体系为50 μL,其中DNA模版6 μL,正反引物(10 μmoL/L)各2 μL,mix 25 μL(诺唯赞,南京),ddH2O 15 μL。PCR反应程序为:98 ℃预变性30 s;98 ℃变性10 s,48.5 ℃退火5 s,72 ℃延伸1 s,32个循环;72 ℃延伸1 min。PCR产物经1%琼脂糖凝胶电泳检测,条带单一且明亮,送南京生工生物有限公司进行Sanger双向测序。
1.4 数据分析
测序所得线粒体D-loop区序列,使用SeqMan软件进行手动拼接。汇总后使用MEGA 7软件对齐后手动校对。计算序列碱基组成,变异点位数及群体间遗传距离等信息,并基于Neis遗传距离构建UPGMA进化树。使用DnaSP软件计算序列的单倍型数(H)、群体的单倍型多样性(Hd)、核苷酸多样性(Pi)、平均核苷酸差异数(K)、中性测试Tajimas D。使用Arlequin 3.5软件计算群体间遗传分化指数(Fst),并进行分子方差分析(AMOVA)。
2 结果与分析
2.1 序列特征与遗传多样性
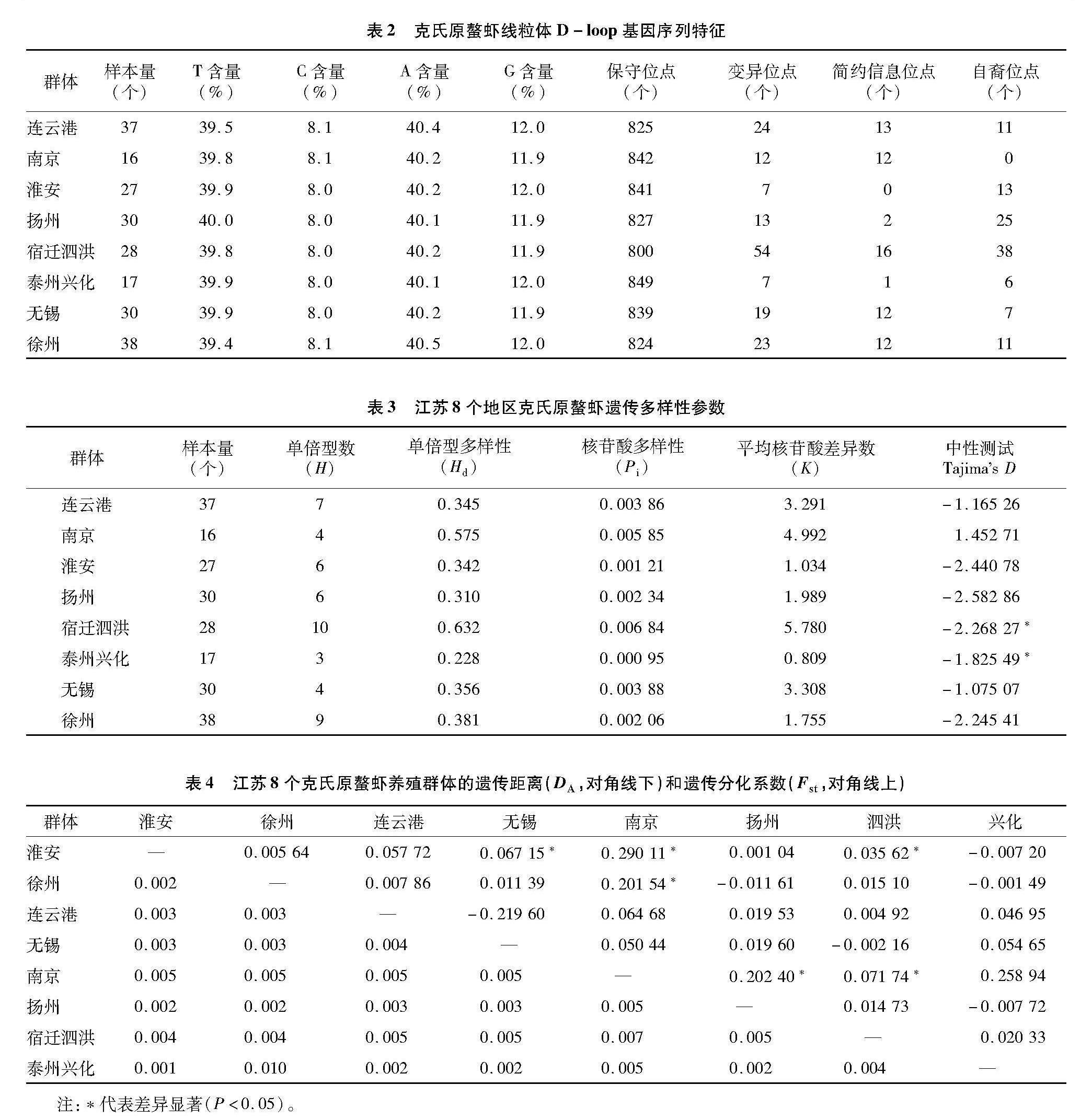
由表2可知,8个群体共260尾的克氏原螯虾经过基因组DNA提取、PCR扩增、测序后共获得223条线粒体D-loop基因序列,经过拼接、排序、校对后,获得全长为863 bp的序列片段,CG含量20.1%。863个核苷酸位点中共有保守位点763个(占88.41%)、突变位点95个、简约信息位点25个、自裔位点70个。
223条序列中共检出37种单倍型。其中,Hap 1为8个群体共有单倍型。宿迁泗洪群体独有的单倍型最多,徐州群体其次,泰州兴化独有的单倍型最少。单倍型Hap 1被173个个体共享,包含个体最多;其次Hap 12,被11个个体共享。
基于克氏原螯虾线粒体D-loop区序列,在8个养殖群体中共定义了37个单倍型。由表3可知,单倍型Hap1分布最广,在8个地区中均有分布,宿迁泗洪地区存在的单倍型数最多,单倍型多样性最高(Hd=0.632),核苷酸多样性也最高(Pi=0.00 684)。泰州兴化地区存在的单倍型数最少(H=3),单倍型多样性(Hd=0.228)最低。本研究中涉及到的8个养殖群体的总体单倍型多样性指数为0.39,江苏克氏原螯虾养殖群体遗传多样性整体处于较低水平。
2.2 种群遗传结果
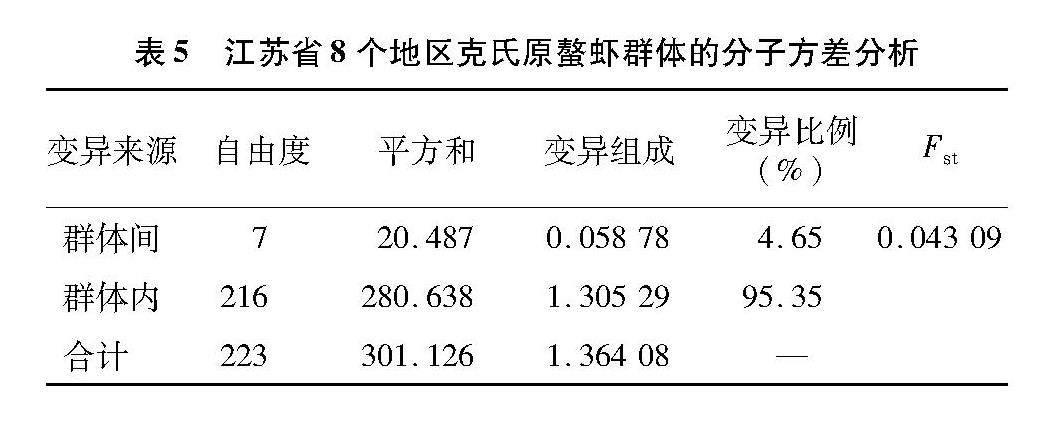
由表4可知,江苏地区8个克氏原螯虾群体间的遗传分化系数Fst为-0.219 60~0.317 33,其中,在无锡地区与连云港地区和宿迁泗洪地区、扬州地区与徐州地区和泰州兴化地区、泰州兴化地区和淮安地区、徐州地区和扬州地区这几个地区间遗传分化系数为负值,表明这几个地区间并未出现显著的遗传分化。南京地区的遗传分化系数显著高于其他地区。扬州地区的遗传分化系数相对低于其他地区。不同群体间的遗传距离总体较小,其中,南京群体与其他群体间的遗传距离相对较大(DA=0.005),表明南京群体与其他群体间的基因交流较少。
由AMOVA分析结果(表5)可知,总Fst为0.043 09,变异不显著,其中,95.35%遗传变异来自于群体内,仅有4.65%来自于群体间。
2.3 单倍型系统进化树与网络图
由图1可知,基于Neis遗传距离对江苏地区克氏原螯虾养殖群体构建UPGMA进化树,8个群体可分为4组,分别为:淮安地区、泰州兴化地区和无锡地区为一组,连云港地区、徐州地区和扬州地区为一组;宿迁泗洪地区和南京地区各为一组。
由使用Network软件生成的单倍型网络图(图2)可知,37种单倍型间关系中,Hap1单倍型位于网络关系图的中心位置,为8个地区共有的单倍型,可预测这一单倍型相对较为原始,周围的单倍型由其衍生而来。不同的单倍型在各地区中均有分布,无明显的谱系分化。
3 讨论
遗传多样性是物种对变化的环境不断适应的结果,物种为适应环境变化会不断进化自身,从而产生丰富的基因型[6-7]。本研究中江苏地区克氏原螯虾养殖群体的单倍型多样性平均值为0.396,核苷酸多样性平均值为0.003 37。与同样基于线粒体D-loop区序列分析的其他甲壳类动物相比,江苏克氏原螯虾养殖群体的遗传多样性低于三疣梭子蟹种群(Hd=0.927,Pi=0.024 3),低于日本沼虾种群(Hd=0.816,Pi=0.019 15)[8-9],研究发现,江苏各克氏原螯虾养殖群体的遗传多样性无明显差异,遗传多样性整体处于较低水平。与山东克氏原螯虾野生群体相比(Hd=0.204,Pi=0.003 6),江苏养殖群体的单倍型多样性较高[10]。与安徽滁州地区克氏原螯虾养殖群体的遗传多样性水平(Hd=0.373 14,Pi=0.001 72)[11]相近。本研究中,江苏地区克氏原螯虾遗传多样性整体不高,造成原因诸多。首先,采样地区的克氏原螯虾苗种多来自池塘自繁,池塘内亲虾存在繁殖期内1个雌体和多个雄体交配的现象,会导致种质资源的退化[12]。其次,江苏地区作为克氏原螯虾的主要养殖地区,因捕捞压力逐年增加,导致稀有位点丢失也可能是江苏地区的克氏原螯虾整体遗传多样性水平不高的原因之一[13]。泰州兴化群体的单倍型多样性(Hd=0.228)和核苷酸多样性(Pi=0.000 95)均最低。泰州兴化地区多采用虾蟹混养模式养殖克氏原螯虾,在每年4月初向河蟹养殖塘中投放约10尾的抱籽亲虾用于苗种繁殖,5月下旬至6月中旬将虾蟹混养塘中的克氏原螯虾尽可能捕尽,尽量减少对河蟹生长影响[14],这种繁养模式可能导致了兴化群体较低的遗传多样性。研究发现,宿迁泗洪地区的单倍型多样性水平和核苷酸多样性水平最高。宿迁泗洪地区临近洪泽湖,可能由于养殖群体与洪泽湖内野生群体交流频繁,从而导致了宿迁泗洪群体较高的遗传多样性。本研究中宿迁泗洪地区遗传多样性较高与高扬等的研究结果[15]相一致。遗传距离是判断群体之间亲缘关系的关键指标,遗传距离越大表明群体间亲缘关系越远[16]。遗传距离较近和遗传分化系数的负值,表明江苏地区的克氏原螯虾存在近亲繁殖、群体间的分化程度较低等问题。江苏省不同地区间盲目引种,是导致遗传距离较近的原因之一。遗传结构是判断新物种适应栖息地能力的主要判断标准[17]。本研究克氏原螯虾8个群体中共发现37种单倍型,其中,单倍型Hap1在各群体中占比最高,在网络图中也处于中心位置。从而推断单倍型Hap1是克氏原螯虾群体中稳定的优势单倍型。从单倍型网络图中发现,以单倍型Hap1为中心呈放射状散开,各群体间并未产生明显遗传分化,各群体间的遗传分化水平整体较低。本研究AMOVA结果显示,有95.35%的变异发生在群体内,仅有4.65%的变异发生在群体间,这与刑智珺等研究中克氏原螯虾的变异多发生在群体内,仅有极少数发生在群体间的结果[5,18-19]相一致。
江苏地区克氏原螯虾养殖群体的遗传多样性整体处于较低水平,建议及时对江苏克氏原螯虾养殖群体进行遗传改良,以提高克氏原螯虾养殖群体的遗传多样性,同时尽快改变自繁自育的苗种繁育和养殖模式。
参考文献:
[1]王亚民,曹文宣. 中国水生外来入侵物种对策研究[J]. 农业环境科学学报,2006,25(1):7-13.
[2]王长忠,李 忠,梁宏伟,等. 长江下游地区4个克氏原螯虾群体的遗传多样性分析[J]. 生物多样性,2009,17(5):518-523.
[3]曹玲亮,周立志,张保卫. 安徽三大水系入侵物种克氏原螯虾的种群遗传格局[J]. 生物多样性,2010,18(4):398-407.
[4]彭 刚,刘伟杰,李佳佳,等. 长江流域3个克氏原螯虾野生群体遗传结构的微卫星分析[J]. 江苏农业学报,2010,26(5):1115-1117.
[5]邢智珺,姜虎成,陆 伟,等. 江苏8个克氏原螯虾群体遗传多样性微卫星分析[J]. 上海海洋大学学报,2014,23(5):656-662.
[6]Vrijenhoek R C. Genetic diversity and fitness in small populations[M]//Conservation genetics. Basel:Birkhuser,1994:37-53.
[7]李大命,李 康,张彤晴,等. 洪泽湖大银鱼(Protosalanx hyalocranius)cytb和COⅠ基因序列多态性分析[J]. 渔业科学进展,2017,38(6):25-31.
[8]董志国,李晓英,王普力,等. 基于线粒体D-loop基因的中国海三疣梭子蟹遗传多样性与遗传分化研究[J]. 水产学报,2013,37(9):1304-1312.
[9]董新培,武小斌,万海付,等. 河北3个日本沼虾野生群体线粒体DNA D-Loop基因序列变异及种群遗传结构分析[J]. 水产学报,2017,41(2):182-188.
[10]杨 玲,李 宁,朱树人,等. 山东省3个克氏原螯虾地理群体线粒体COⅠ基因的序列差异分析[J]. 安徽农业科学,2015,43(20):41-44.
[11]余红喜,刘 帆,朱国美,等. 基于线粒体COⅠ基因滁州地区克氏原螯虾养殖群体遗传多样性分析[J]. 安徽农业科学,2022,50(21):111-113,125.
[12]Villanelli F,Gherardi F. Breeding in the crayfish,Austropotamobius pallipes:mating patterns,mate choice and intermale competition[J]. Freshwater Biology,1998,40(2):305-315.
[13]夏爱军,唐建清. 克氏原螯虾现状分析与研究思路[J]. 水产养殖,2006,27(6):9-11.
[14]张凤翔,陈 杰,徐美香,等. 虾蟹混养模式的创新试验与思考[J]. 科学养鱼,2020(2):32-34.
[15]高 杨,田 灿,姜京京,等. 基于微卫星标记的克氏原螯虾种群遗传多样性和遗传结构分析[J]. 江苏农业科学,2023,51(5):191-199.
[16]张鹤千,杨子拓,李桂峰,等. 珠江流域野生黄颡鱼Pelteobagrus fulvidraco的Cytb基因序列分析[J]. 中山大学学报(自然科学版),2015,54(5):102-108.
[17]Bazin E,Glémin S,Galtier N. Population size does not influence mitochondrial genetic diversity in animals[J]. Science,2006,312(5773):570-572.
[18]崔文涛,邹宇凡,白志毅,等. 安徽地区克氏原螯虾群体的遗传多样性和遗传结构分析[J]. 水产学报,2022,10(28):1-10.
[19]Yi S K,Li Y H,Shi L L,et al. Characterization of population genetic structure of red swamp crayfish,Procambarus clarkii,in China[J]. Scientific Reports,2018,8:5586.
