基于GEO数据库结合网络药理学及分子对接逆向挖掘干预肝癌的中药
2024-06-26魏泽坤杨雨洁刘双王燕董红敬刘春梅
魏泽坤 杨雨洁 刘双 王燕 董红敬 刘春梅


DOI:10.3976/j.issn.1002-4026.20230082
收稿日期:2023-05-12
基金项目:济南市新高校二十条(202228020)
作者简介:魏泽坤(1999—),女,研究方向为中西医结合周围血管病学。E-mail:weizekun333@163.com
*通信作者,刘春梅(1971—),女,博士,副主任医师,研究方向为中西医结合周围血管病学。E-mail:lcmsyw@163.com
摘要:基于GEO(gene expression omnibus)数据库结合网络药理学及分子对接,从分子水平逆向挖掘具有抗肝癌活性的中药。通过GEO、GeneCards、OMIM和TTD等疾病靶点数据库获取肝癌的重要靶点。利用STRING平台获取核心靶点,结合TCMIP(integrative pharmacology-based research platform of traditonal Chinese medicine)和TCMID(traditional Chinese medicine integraive database)数据库筛选核心成分,采用TCMSP(traditonal Chinese medicine system pharmacology database and analysis platform)数据库筛选核心中药,通过分子对接和细胞实验验证筛选结果。从疾病靶点数据库得到398个肝癌的重要靶点,进一步筛选出8个核心靶点、11个核心成分和1味核心中药葛根;分子对接结果表明葛根的3个核心成分(槲皮素、黄岑素、豆甾醇)可以与部分核心靶点(CDK1、CDC20)自发地结合;细胞实验结果表明葛根提取物可以有效抑制肝癌细胞HepG2的增殖。该结果可以为葛根的研究与开发提供参考,为葛根抗肝癌活性成分的挖掘提供理论依据。
关键词:葛根;抗肝癌活性成分;网络药理学;分子对接;GEO数据库
中图分类号:R285 文献标志码:A 文章编号:1002-4026(2024)03-0039-09
开放科学(资源服务)标志码(OSID):
Reverse mining of Chinese medicine for intervention of liver cancer
based on GEO database combined with network pharmacology
and molecular docking technology
WEI Zekun1,2, YANG Yujie1,2, LIU Shuang3, WANG Yan3, DONG Hongjing3, LIU Chunmei1,2*
(1. School of Traditional Chinese Medicine, Shandong University of Traditional Chinese Medicine, Jinan 250014, China;
2. Affiliated Hospital of Shandong University of Traditional Chinese Medicine, Jinan 250014, China; 3. Shandong Analysis
and Test Center, Qilu University of Technology (Shandong Academy of Science), Jinan 250014, China)
Abstract∶Based on the gene expression omnibus(GEO) database, combined with network pharmacology and molecular docking technology, we aimed to conduct reverse network pharmacology research from a molecular level to identify Chinese medicine with anti-hepatocellular carcinoma (HCC) activity. Relevant targets of HCC were acquired from databases including GEO, GeneCards, Online Mendelian Inheritance in Man, and Therapeutic Target Database. The core targets were identified using the String platform, and the core constituents were screened from the TCMIP(integrative pharmacology-based research platform of traditonal
Chinese medicine) and TCMID(traditional Chinese medicine
integraive database) databases. The core traditional Chinese medicine (TCM) was ultimately selected through the traditional Chinese medicine systems pharmacology database and analysis platform(TCMSP). Molecular docking technology and cellular experiments were employed to identify relevant screening results. A total of 398 important targets for HCC were found from the disease target databases, from which 8 core targets, 11 core constituents, and 1 core TCM (Puerariae Lobatae Radix) were further screened. Molecular docking results showed that three core constituents (quercetin, genistein, and coumestrol) from kudzu root could spontaneously bind with some core targets (CDK1 and CDC20), and cell experiments demonstrated that the extract from Puerariae Lobatae Radix could effectively inhibit the proliferation of HepG2 liver cancer cells. This study may provide a reference for the research and development of Puerariae Lobatae Radix and offer a theoretical basis for the discovery of its anti-HCC active ingredients.
Key words∶Puerariae Lobatae Radix; antiliver cancer activity; network pharmacology; molecular docking; gene expression omnibus
肝癌是最常见的肿瘤之一,常常伴随着不良预后和高死亡率,严重威胁患者的生命健康[1]。现有的治疗方法包括手术、化疗和放疗等,已取得了一定的进展,但治疗效果仍然不尽人意[2]。中药作为一种重要的中医药治疗手段,近年来受到越来越多的关注,大多数中药的副作用低、效果好,具有增强免疫力、改善患者生活质量和延长患者生存期的优势,已成为我国综合治疗肝癌方案的组成部分之一[3]。
网络药理学是一种以生物学理论知识为背景,结合多种计算机辅助分析技术,进而构建成分-靶点以及靶点-靶点的相互作用关系网络,实现成分直接或间接作用于靶点的可视化技术。分子对接是一种广泛用于研究配体和受体分子间相互作用的分子模拟方法[4]。网络药理学结合分子对接技术,为中药的潜在作用机制以及核心成分的挖掘研究提供了新工具[5-6]。GEO(gene expression omnibus)数据库是目前全球最大的基因芯片数据公共数据库之一,收集了来自全球的数十万份基因芯片数据,其中包含大量的肝癌相关数据[7]。
本研究旨在利用GEO数据库的肝癌数据,结合网络药理学及分子对接技术,逆向挖掘对肝癌具有干预作用的中药(研究思路见OSID科学数据与内容附图1),以期为抗肝癌活性新药的发现与研究提供参考,为中药新用途的研究与开发提供思路。
1 材料与方法
1.1 实验试剂与仪器
精密称取20 g葛根饮片于250 mL圆底烧瓶中,分别加入100 mL纯水和100 mL乙醇溶液(75%),加热回流提取2 h后,将提取液离心、浓缩、冻干,分别得到葛根水提取物和葛根醇提取物。DMEM 培养基(Gibco公司,批号8122638),PBS缓冲液(Solarbio公司,批号GP21100051207),胰蛋白酶(Solarbio公司,批号20220917),胎牛血清(奥普赛生物科技有限公司,批号20230208)。
HF90气套式二氧化碳培养箱(上海力康公司),Spark多功能微孔板检测仪(瑞士TECAN公司)。
1.2 肝癌疾病靶点库的建立
通过3个疾病靶点数据库,包括GeneCards数据库(https://www.genecards.org/)、OMIM数据库(https://omim.org/)及TTD数据库(http://db.idrblab.net/ttd/),根据检索词“liver cancer”,检索肝癌的疾病靶点,检索结果整合于Excel表中,建立肝癌相关的潜在疾病靶点数据集。基于关键词“liver cancer”检索GEO数据库(https://www.ncbi.nlm.nih.gov/geo/)获取正常组和肝癌组之间的表达矩阵(GSE36376、GSE60502、GSE84005、GSE121248),通过Excel软件进行分析筛选,以对数倍数差异(logarithm of fold change, |log FC|)>2且Padj<0.05为条件筛选肝癌的差异表达基因。基于Venny工具(https://bioinfogp.cnb.csic.es/tools/venny/index2.0.2.html),计算肝癌的潜在靶点数据集及肝癌的差异表达基因的重复靶点,建立肝癌的重要靶点集。
1.3 建立并分析蛋白质-蛋白质相互作用的可视化拓扑网络
基于STRING在线分析数据库(https://cn.string-db.org/),以肝癌的重要靶点为分析数据集,并选择Homo sapiens(人)为分析物种,最小互作分数为highest confidence(0.900)为分析条件进行蛋白质-蛋白质相互作用(protein-protein interaction,PPI)网络分析,隐藏孤立的蛋白名称,下载PPI网络数据并保存为TSV格式文件,随后导入Cytoscape 3.7.2软件,分析该PPI拓扑网络并计算网络指标,包括degree、betweenness centrality(BC)和closeness centrality(CC)。根据网络指标,进一步筛选肝癌的核心靶点。
1.4 核心靶点的富集分析及核心靶点-器官组织拓扑网络的构建
以核心靶点为分析数据集,采用DAVID在线分析平台(https://david.ncifcrf.gov),以标识符为“OFFICIAL GENE SYMBOL”、物种为“Homo sapiens”为分析条件,进行基因本体(gene ontology,GO)功能(生物过程、细胞成分、分子功能)富集分析和京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)通路富集分析。设定P<0.05,筛选出排名前10位的生物学过程和信号通路。
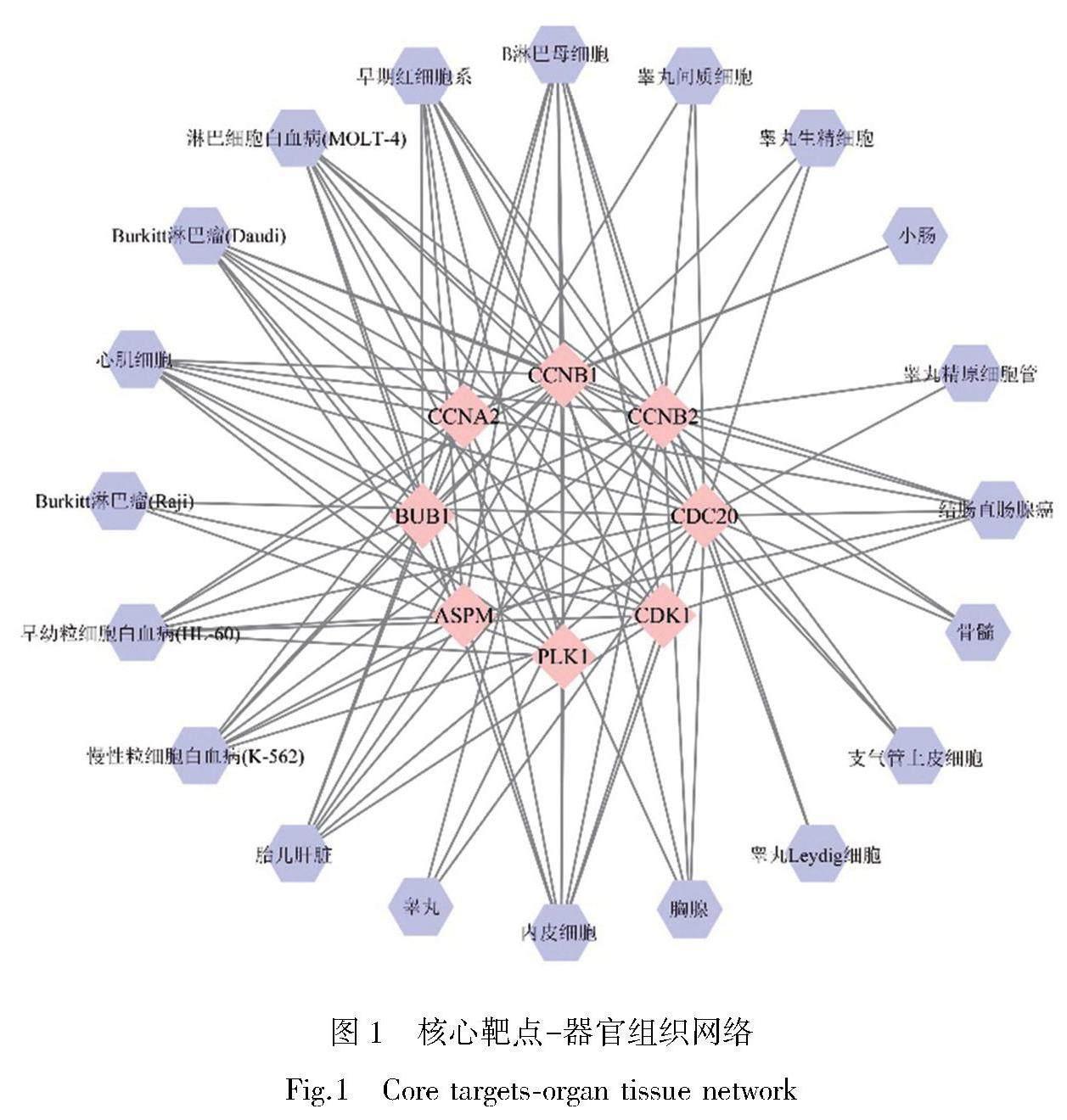
将核心靶点导入BioGPS数据库(http://biogps.org/),分析核心靶点在脏器组织中的表达,将结果导出至Excel中,筛选高于平均表达的器官组织,利用建立的靶点和器官组织之间的互作关系数据,将其导入Cytoscape 3.7.2软件中,实现核心靶点-器官组织拓扑网络的可视化展示。
1.5 核心成分的筛选
基于中医药整合数据库(traditional Chinese medicine integrative database,TCMID)(http://47.100.169.139/tcmid/)和中医药整合药理学研究平台(integrative pharmacology-based research platform of traditional Chinese medicine, TCMIP)(http://www.tcmip.cn/TCMIP),以核心靶点名称为关键词,获取与核心靶点发生相互作用的化学成分,采用Excel记录并规范检索结果。利用建立的核心靶点与化学成分之间的互作关系数据,将其导入Cytoscape 3.7.2软件中,实现核心靶点-化学成分拓扑网络的可视化展示,分析拓扑网络并根据degree值筛选核心成分。
1.6 核心中药的筛选
基于中药系统药理学数据库与分析平台(traditional Chinese medicine systems pharmacology database and analysis platform,TCMSP)(https://tcmsp-e.com/tcmsp.php),以核心成分名称为关键词,获取含有核心成分的中药,采用Excel记录并规范检索结果。利用建立的核心成分与中药之间的互作关系数据,将其导入Cytoscape 3.7.2软件中,实现核心成分-中药拓扑网络的可视化展示,分析拓扑网络并根据degree值筛选核心中药。
1.7 分子对接
通过分子对接技术,计算核心中药的成分与肝癌的核心靶点的自主结合能力,进一步验证筛选结果的可信性。下载RSCB数据库(https://www.rcsb.org/)中核心靶点CDK1(PDB编号:6GU3)和CDC20(PDB编号:4GGA)的结构,以PDB格式文件保存,借助Pymol软件对核心靶点进行预处理,主要包括去水和配体。下载PubChem数据库(https://pubchem.ncbi.nlm.nih.gov/)中核心成分的结构,以SDF格式文件保存,进而采用OpenBable 软件对其进行处理,以mol2格式文件保存;利用AutoDock Vina软件进行分子对接,使用Pymol软件对对接结果进行可视化处理。
1.8 肝癌细胞增殖抑制实验
在-80 ℃的冰箱中取出冻存的HepG2肝癌细胞(购自中国医学科学院基础医学研究所),于37 ℃水浴锅中解冻后转移至离心管,加入DMEM(Dulbuccos modified eagle medium)高糖培养基混匀、离心后,去除上清液。向离心管中加入7 mL含10%FBS(fetal bovine serum)的DMEM高糖培养基,吹打均匀后转移至培养瓶中,于5% CO2、37 ℃培养箱中培养。
取HCT116细胞,加入胰蛋白酶消化后,吹打成单细胞悬液,将细胞浓度调整为1×105 mL-1,以每孔100 μL接种于96孔板中,于培养箱中培养24 h。分别加入基于DMEM高糖培养基配制的葛根水提物组(50、100、200、400、600、800 μg/mL)、葛根醇提物组(50、100、200、400、600、800 μg/mL)和对照组(药物浓度为0),每组接种3孔,于培养箱中培养24 h。随后,每孔加入50 μL MTT(噻唑蓝)溶液(1.0 mg/mL),4 h后吸出上清液,每孔加入100 μL DMSO(二甲基甲砜)溶液,采用酶标仪测定490 nm波长下的光密度(optical density,OD)值。肝癌细胞增殖抑制率的计算公式如下:
增殖抑制率=1-y药物组y对照组×100%,(1)
式中,y表示490 nm波长下的光密度值。
1.9 统计学分析
采用GraphPad Prism软件进行统计分析,采用t检验比较两组数据间的显著性差异,P<0.05表示差异显著,P<0.01表示差异极显著。
2 结果
2.1 肝癌疾病靶点库的建立
基于3个疾病靶点数据库,获得的肝癌疾病相关靶点的数量分别为:GeneCards数据库16 799个、OMIM数据库183个以及TTD数据库63个。整合、去除重复值后,共得到16 886个肝癌的潜在靶点。从4个表达矩阵(GSE36376、GSE60502、GSE84005、GSE121248)中共得到114 920个基因,根据条件筛选后,从4个基因芯片GSE36376、GSE60502、GSE84005、GSE121248的表达矩阵中分别得到73、386、205、176个差异表达基因(DEGs),整合去重后,共得到447个DEGs。将肝癌的潜在靶点与DEGs导入Venny平台,共得到398个共有靶点,认为是治疗肝癌的重要靶点(OSID科学数据与内容附图2)。
2.2 建立和分析PPI的可视化拓扑网络
基于STRING数据库,以398个肝癌的重要靶点为分析基因集,获得PPI数据,采用Cytoscape 3.7.2软件实现PPI拓扑网络的可视化展示。拓扑网络包含217个节点和1 242条边(OSID科学数据与内容附图3)。以degree值≥44,BC值≥0.025,CC值≥0.60为条件筛选出8个核心靶点,包括CDK1(细胞周期蛋白依赖性激酶1),CCNB2(细胞周期蛋白B2),CDC20(细胞分裂周期20),CCNA2(细胞周期蛋白A2),BUB1 (有丝分裂丝氨酸/苏氨酸检查点激酶),CCNB1(细胞周期蛋白B1),PLK1 (Polo样激酶1),ASPM(异常纺锤样小头相关蛋白)。其中,CDK1的degree值(degree值>63)最高,表明CDK1处于PPI网络的核心,可以通过相互作用以调节其他靶点,实现治疗作用。
2.3 核心靶点的功能富集分析及核心靶点-器官组织网络的构建
GO功能富集分析结果显示,8个核心靶点显著富集于25个生物过程条目、12个细胞成分条目和7个分子功能条目,选取排名前10位的条目进行展示(OSID科学数据与内容附图4)。KEGG通路富集分析结果显示,8个核心靶点显著富集于6条信号通路,主要包括p53信号通路、FoxO信号通路等(OSID科学数据与内容附图5)。
将核心靶点导入BioGPS数据库,利用Cytoscape 3.7.2软件构建核心靶点-器官组织网络(图1),网络共有28个节点和103条边,紫色节点代表器官组织,粉色节点代表核心靶点。结果表明,核心靶点在多个器官组织中表达。核心靶点CDK1和CCNB2主要于内皮细胞中表达,核心靶点CCNA2主要于早期红细胞中表达,核心靶点CDC20、BUB1、CCNB1、PLK1和ASPM主要于B淋巴细胞中表达。BioGPS核心靶点信息定位虽未涉及肝脏,但却在内皮细胞、早期红细胞、B淋巴细胞等免疫细胞中表达。此外,核心靶点CDC20涉及的器官组织最多(degree值>20),表明该靶点的分布和影响范围广。
2.4 核心成分的筛选
将核心靶点分别导入TCMIP平台和TCMID平台,检索与核心靶点相关的化学成分,共得到562个化学成分,其中,11个化学成分与ASPM相关,385个化学成分与CCNA2相关,1个化学成分与BUB1相关,72个化学成分与CCNB1相关,14个化学成分与CCNB2相关,10个化学成分与CDC20相关,42个化学成分与CDK1相关,27个化学成分与PLK1相关,根据建立的化学成分与核心靶点的互作关系数据,导入Cytoscape 3.7.2软件,实现核心靶点-化学成分拓扑网络的可视化,分析拓扑网络的degree值,degree值越高表示化学成分可以作用的核心靶点越多。11个化学成分(degree值≥3)被认为是核心成分,包括黄岑素、丁酸、甘波辛、异黄烷酚、二硫代葡萄糖醇、黄烷霉素、白藜芦醇、豆甾醇、苯并芘、β-雌二醇、槲皮素。核心成分的筛选过程如图2所示。
2.5 核心中药的筛选
将11个核心成分分别导入TCMSP平台,检索含有核心成分的中药,共得到262味中药。根据核心成分与中药的互作关系数据,导入Cytoscape 3.7.2软件,实现核心成分-中药拓扑网络的可视化。分析拓扑网络得到的degree值,degree值表示中药中所含核心成分的数量。葛根(degree值≥3)被筛选作为核心中药。核心中药的筛选过程见OSID科学数据与内容附图6。
2.6 分子对接
因为CDK1处于PPI网络的核心,可以通过PPI网络调节其他靶点,而CDC20在器官组织中分布最广泛,因此选取葛根中含有的3个核心成分(槲皮素、黄岑素、豆甾醇)与两个核心靶点(CDK1、CDC20)进行分子对接。3个核心成分与2个核心靶点分子对接的结合能均为负数,表明对接后会放出能量,对接过程可以自发进行(表1)。核心成分可以通过氢键与核心靶点的多个残基相互作用(图3)。
2.7 肝癌细胞增殖抑制实验
如图4所示,葛根水提取物和葛根醇提取物均能对HepG2肝癌细胞增殖产生抑制作用,且抑制作用均随质量浓度升高而增强,呈剂量依赖性;当质量浓度为50 μg/mL时,葛根水提取物和葛根醇提取物的抑制活性无显著性差异(P>0.05);当质量浓度逐渐升高时,相同浓度下的葛根醇提取物较葛根水提取物的抑制作用更强,表现出显著差异性,这可能是由于葛根中槲皮素和黄岑素等2个核心成分在乙醇中的溶解度较高。
注:#表示P>0.05,*表示P<0.05,**表示P<0.01。
3 讨论
GEO数据库是一个储存芯片、二代测序以及其他高通量测序数据的数据库,通过GEO数据库可以得到基因表达谱分析,进而筛选出与肝癌有关的差异基因[7]。GeneCards数据库、OMIM数据库和TTD数据库是常用的疾病靶点数据库,通过以上疾病数据库,可以得到已知的与肝癌发生、发展、治疗或预后等方面相关的所有靶点。将差异基因与靶点数据取交集,将有助于去除肝癌疾病靶点的冗余信息,进一步筛选与肝癌相关性更高的靶点。
基于PPI网络,筛选出8个核心靶点,包括CDK1、CCNB2、CCNA2、CDC20、BUB1、CCNB1、PLK1、ASPM。研究表明,CDK1在肝癌S期会过度表达[8];CCNB1和CCNB2是细胞周期蛋白家族的主要组成部分,具有调节与G2/M检测位点相关的重要细胞周期的功能[9];CCNA2在S期到有丝分裂早期高度表达[10];CDC20是控制染色体分离和有丝分裂结束的重要成分[11];BUB1可激活SMAD2磷酸化,促进肝癌细胞增殖[12];PLK1是肝癌中的一种癌基因,对调节M期有丝分裂纺锤体形成检查点具有重要作用,被认为是肝癌研究的靶点[13-14];ASPM对胚胎成神经细胞正常的有丝分裂纺锤体功能至关重要[15]。综上所述,8个核心靶点均与肝癌密切相关,可作为肝癌的治疗靶点。其中,CDK1的degree值最高,处于PPI网络的核心,表明可以通过CDK1间接地调节其他靶点。
GO富集分析结果显示,首先,核心靶点主要参与有丝分裂核分裂、细胞分裂和有丝分裂细胞周期的泛素蛋白连接酶活性调控等生物过程,其中,有丝分裂核分裂和细胞分裂是肝癌细胞增殖的基础,而泛素蛋白连接酶活性的调控是肝癌细胞有丝分裂期和后期的调控关键,其异常可能引发肝癌细胞的不正常增殖;其次,核心靶点涉及浓缩核染色体外着丝点、中心体和核浆等细胞组分,其中,浓缩核染色体外着丝点异常可能导致癌细胞有丝分裂异常,中心体异常扩散可能与肝癌细胞的快速增殖有关,核浆中突变基因和蛋白质可改变代谢通路影响癌细胞增殖和转化;最后,核心靶点参与周期蛋白依赖蛋白丝氨酸/苏氨酸激酶活性、组蛋白激酶活性和后期促进复合体结合,这三种生物功能都参与了肝癌细胞的分裂和增殖,如周期蛋白依赖蛋白激酶促进细胞周期,组蛋白激酶调节染色体结构,后期促进复合体则参与有丝分裂。这些过程的异常调控可能导致肝癌细胞的不正常生长。
基于核心成分-器官组织网络,核心靶点在内皮细胞、早期红细胞、B淋巴细胞等免疫细胞中表达,虽未直接涉及肝癌,但推测肝癌可能通过影响免疫细胞的功能逃避免疫监视,并促进肿瘤的发生和发展。其中,核心靶点CDC20涉及的器官组织最多,表明CDC20在该网络中具有重要的调控作用。
通过核心靶点-成分拓扑网络和核心成分-中药拓扑网络,筛选出3个核心成分(槲皮素、黄岑素、豆甾醇)和1个核心中药葛根。其中,黄岑素能促进大量ROS的产生并引发细胞死亡,提高人肝癌细胞株HepG2的胞质细胞色素c、裂解半胱氨酸蛋白酶-3和裂解半胱氨酸蛋白酶-9的表达并降低Bcl-2(B淋巴细胞瘤-2)的表达[16]。研究表明,豆甾醇可以与MMP-2(基质金属蛋白酶-2)和MMP-9(基质金属蛋白酶-9)结合,抑制其表达,进而对癌细胞的迁移和侵袭产生抑制作用[17]。槲皮素可以通过增加E-cadherin(钙黏蛋白E)的表达,降低N-cadherin(钙黏蛋白N)和Vimentin(波形蛋白)的表达来抑制TCF-β1(转化生长因子-β1)诱导的肝细胞EMT的增殖,同时,槲皮素可以诱导人肝癌细胞株Hep3B和Huh7的凋亡[18-19]。此外,研究表明,核心中药葛根可以抑制人肝癌细胞HepG2的增殖[20]。
分子对接结果显示,槲皮素可以通过氢键与CDK1的多个残基结合,包括ARG-216、MET-260和TYR-261;豆甾醇可以通过氢键与CDK1的多个残基结合,包括VAL-227、LEU-220和ALA-219;黄岑素可以通过氢键与CDC20的多个残基结合,包括GLN-401、SER-400和PHE-420。然而,槲皮素和豆甾醇中文与CDC20不能进行对接,黄岑素与CDK1不能进行对接,体现了中药多成分调节多靶点的作用特点。
肝癌细胞增殖抑制实验结果显示,葛根水提取物和葛根醇提取物均能抑制HepG2肝癌细胞增殖,且呈剂量依赖性;葛根醇提取物的抑制活性较葛根水提取物的抑制活性强,这可能是因为葛根中的2个核心成分(槲皮素和黄岑素)在醇中的溶解度高[21-22];肝癌细胞增殖抑制活性进一步验证了核心中药挖掘结果的可靠性。
本研究基于GEO数据库结合网络药理学方法,根据“搜集靶点、找寻成分、挖掘中药”的研究思路,逆向挖掘出具有抗肝癌活性的中药葛根,采用分子对接技术将葛根的核心成分与肝癌的核心靶点进行对接,结果显示3个核心成分可以自发地与其特有的核心靶点结合并释放出能量,这充分展示了中药具有多成分调节多靶点的作用特征。同时,采用体外HepG2肝癌细胞增殖的抑制试验,进一步验证了核心中药葛根的水提取物和醇提取物的抗肝癌增殖活性。本研究为葛根的抗肝癌活性研究提供了理论基础,为葛根中抗肝癌活性成分的挖掘与研究提供了参考。同时,在大量数据中筛选得到抗肝癌活性的中药并得到验证,可以节省研究的经济和时间成本。本研究仍存在不足之处,采用葛根粗提物进行细胞实验验证,但葛根中的核心成分对核心靶点的调控未进行研究,我们将在后续的研究中进一步验证。
参考文献:
[1]BRUIX J, HAN K H, GORES G, et al. Liver cancer: approaching a personalized care[J]. Journal of Hepatology, 2015, 62(S1): S144-S156. DOI: 10.1016/j.jhep.2015.02.007.
[2]ZHANG H B, YAO W P, ZHANG M J, et al. TTK inhibitor promotes radiosensitivity of liver cancer cells through p21[J]. Biochemical and Biophysical Research Communications, 2021, 550: 84-91. DOI: 10.1016/j.bbrc.2021.01.089.
[3]郭太明, 张海, 王砚强, 等. 中医药对恶性胶质瘤患者生活质量和生存期的影响[J]. 辽宁中医杂志, 2008, 35(10): 1456-1458. DOI: 10.13192/j.ljtcm.2008.10.20.guotm.005.
[4]王俊琳, 张亚, 刘双清, 等. 计算机辅助药物设计在新农用化合物开发中的应用[J]. 农药, 2022, 61(4): 242-249. DOI: 10.16820/j.cnki.1006-0413.2022.04.005.
[5]周珊珊, 李伟男, 艾中柱, 等. 基于网络药理学和分子对接探讨清肺达原颗粒治疗新型冠状病毒肺炎(COVID-19)的作用机制[J]. 中草药, 2020, 51(7): 1804-1813. DOI: 10.7501/j.issn.0253-2670.2020.07.014.
[6]HAO J, LI S J. Recent advances in network pharmacology applications in Chinese herbal medicine [J]. Traditional Medical Research, 2018, 3(6): 260-272.
[7]EDGAR R, DOMRACHEV M, LASH A E. Gene expression omnibus: NCBI gene expression and hybridization array data repository[J]. Nucleic Acids Research, 2002, 30(1): 207-210. DOI: 10.1093/nar/30.1.207.
[8]GARNIER D, LOYER P, RIBAULT C, et al. Cyclin-dependent kinase 1 plays a critical role in DNA replication control during rat liver regeneration[J]. Hepatology, 2009, 50(6): 1946-1956. DOI: 10.1002/hep.23225.
[9]LIU S, YAO X X, ZHANG D, et al. Analysis of transcription factor-related regulatory networks based on bioinformatics analysis and validation in hepatocellular carcinoma[J]. BioMed Research International, 2018, 2018: 1431396. DOI: 10.1155/2018/1431396.
[10]杨结,刘树业,刘运德. CCNA2基因在肝细胞癌中的表达、信号通路和预后关系生物信息分析及验证[J]. 世界华人消化杂志,2019,27(24):1940-1501. DOI:10.11569/wcjd.v27.i24.1490.
[11]KAPANIDOU M, CURTIS N L, BOLANOS-GARCIA V M. Cdc20: At the crossroads between chromosome segregation and mitotic exit[J]. Trends in Biochemical Sciences, 2017, 42(3): 193-205. DOI: 10.1016/j.tibs.2016.12.001.
[12]ZHU L J, PANY, CHENX Y, et al. BUB1 promotes proliferation of liver cancer cells by activating SMAD2 phosphorylation[J]. Oncology Letters, 2020, 19(5): 3506-3512. DOI: 10.3892/ol.2020.11445.
[13]WANG D Z, CHANG R N, WANG G, et al. Polo-like kinase 1-targeting chitosan nanoparticles suppress the progression of hepatocellular carcinoma[J]. Anti-Cancer Agents in Medicinal Chemistry, 2017, 17(7): 948-954. DOI: 10.2174/1871520616666160926111911.
[14]BOUHLAL H, OULED-HADDOU H, DEBUYSSCHER V, et al. RB/PLK1-dependent induced pathway by SLAMF3 expression inhibits mitosis and control hepatocarcinoma cell proliferation[J]. Oncotarget, 2016, 7(9): 9832-9843. DOI: 10.18632/oncotarget.6954.
[15]XUE J M, LIU Y, WAN L H, et al. Comprehensive analysis of differential gene expression to identify common gene signatures in multiple cancers[J]. Medical Science Monitor: International Medical Journal of Experimental and Clinical Research, 2020, 26: e919953. DOI: 10.12659/MSM.919953.
[16]ZHANG Q A, BAO J A, YANG J H. Genistein-triggered anticancer activity against liver cancer cell line HepG2 involves ROS generation, mitochondrial apoptosis, G2/M cell cycle arrest and inhibition of cell migrationand inhibition of cell migration[J]. Archives of Medical Science, 2019, 15(4): 1001-1009. DOI: 10.5114/aoms.2018.78742.
[17]KUANG W W, HU W L, REN H, et al. Plant derived coumestrol phytochemical targets human skin carcinoma cells by inducing mitochondrial-mediated apoptosis, cell cycle arrest, inhibition of cell migration and invasion and modulation of m-TOR/PI3K/AKT signalling pathway[J]. Saudi Journal of Biological Sciences, 2021, 28(5): 2739-2746. DOI: 10.1016/j.sjbs.2021.03.043.
[18]郑岩松, 雷文俤, 陈明流, 等. 槲皮素抑制TGF-β1诱导SMMC-7721人肝癌细胞发生EMT的效果研究[J]. 毒理学杂志, 2019, 33(6): 465-468. DOI: 10.16421/j.cnki.1002-3127.2019.06.005.
[19]陈兆勋, 高彦宇, 李冀. 不同浓度槲皮素对人肝癌细胞株Hep3B、Huh7细胞凋亡的影响[J]. 中医杂志, 2015, 56(12): 1051-1053. DOI: 10.13288/j.11-2166/r.2015.12.017.
[20]唐亚男, 陈朝霞, 刘阿龙, 等. 荷叶、葛根和鱼腥草提取物体外抗肝癌及抗HBV活性研究[J]. 中华中医药学刊, 2018, 36(4): 811-814. DOI: 10.13193/j.issn.1673-7717.2018.04.011.
[21]杨宏志, 丁洁, 李志江, 等. 响应面化优化豆酱中染料木素的乙醇-超声波法提取工艺[J]. 食品科学, 2013, 34(10): 112-116. DOI: 10.7506/spkx1002-6630-201310024.
[22]赵永明, 王书华, 刘泽华, 等. 正交实验法优化款冬花中槲皮素的提取工艺[J]. 医药导报, 2014, 33(6): 800-802. DOI: 10.3870/yydb.2014.06.031.
