基于CRISPR-Cas9系统构建LEU1基因缺失酿酒酵母用于酿造低醉酒度米酒
2024-05-14王泽翔何娇娇梁思宇周世水
王泽翔,何娇娇,梁思宇,周世水*
(1.华南理工大学 生物科学与工程学院,广东 广州 510006;2.广东石湾酒厂集团有限公司,广东 佛山 528031)
米酒是大米经过毛霉(Mucorsp.)、根霉(Rhizopussp.)以及酵母发酵后得到的产品,具有历史悠久、种类繁多等特点。米酒在发酵过程中通过糖代谢途径(Harris途径)和氨基酸代谢途径(Ehrlich途径)生成多种高级醇,包括正丙醇、异丁醇、异戊醇等[1-2],是形成米酒风味的重要化合物,但因高级醇具有较强的致醉性,饮用后会出现呕吐和“上头”等不适症状,对人体健康有危害[3-6]。不同高级醇比例导致醉酒度不同[7-8],其对神经刺激程度随分子质量的增大而加剧,其中对醉酒度影响最大的物质是异戊醇,尤其是异戊醇与异丁醇的比值越高,米酒的醉酒度越高[9]。因此,降低异戊醇/异丁醇的比值,能够降低米酒的醉酒度,进而改善米酒的品质,减少对人体健康的危害。
在Ehrlich途径中,LEU1基因编码的异丙基苹果酸合成酶催化由丙酮酸转化而来的柠康酸生成β-苹果酸甲酯,随后转化成α-酮丁酸,α-酮丁酸进一步转化成异丁醇的前体物质α-酮异戊酸和异戊醇的前体物质α-酮异己酸[10]。目前LEU1基因对不同酒类影响均有较多研究[11-12],但对米酒醉酒度影响的研究较少。
成簇规则间隔的短回文重复序列及其相关蛋白9(clustered regularly interspaced short palindromic repeat/CRISPRassociated protein 9,CRISPR-Cas9)基因编辑技术利用向导核糖核酸(guide ribonucleic acid,gRNA)识别目标序列,依靠Cas9蛋白切割靶位点序列,使得细胞内的脱氧核糖核酸(deoxyribonucleic acid,DNA)双链断裂,以此来激发非同源末端连接修复机制或同源重组修复机制,达到基因组高效定点敲除、定点敲入和基因修饰的目的[13-18]。与传统的基因改造技术相比,CRISPR-Cas9系统具有精准、高效的优势,避免了在基因改造过程中对酿酒酵母基因组的污染,使得改良酵母应用于酿造酒更加安全[19-20]。本研究以酿酒酵母(Saccharomycescerevisiae)XF0为出发菌株,通过构建重组质粒p414-Cas9- BleoR和p426-gRNA.LEU1-kanMX,建立通过博来霉素和G418抗生素来筛选酿酒酵母的基因敲除系统,以此获得非营养缺陷型LEU1基因缺失的重组菌,探究LEU1基因缺失对高级醇生成的影响,通过优化高级醇之间的比例酿造低醉酒度米酒,为提高米酒品质提供参考。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株与质粒
本研究所用的菌株和质粒载体见表1。

表1 本研究所用菌株和质粒Table 1 Strains and plasmids used in this study
1.1.2 引物
从美国国家生物技术信息中心(nationalcenterforbiotechnology information,NCBI)获得酿酒酵母S288c的LEU1基因组序列(序列号:NC-001139),采用SnapGene 5.2.4软件设计引物,并由上海生物工程技术有限公司负责合成。本研究所用引物见表2。

表2 本研究所用引物序列Table 2 Primer sequences used in this study
1.1.3 试剂
G418硫酸盐:北京普博欣生物科技有限责任公司;博来霉素TM筛选试剂:赛默飞世尔科技公司;氨苄青霉素、D-山梨醇、乙酸丁酯、醋酸锂:上海阿拉丁生化科技股份有限公司;质粒小量快速抽提试剂盒、琼脂糖凝胶DNA回收试剂盒:上海迈跟生物科技有限公司;Apex HF HS DNA聚合酶预混液-FS:湖南艾科瑞生物科技有限公司;Clon ExpressRII One Step Cloning Kit试剂盒:南京诺唯赞生物科技股份有限公司;2×GSTaq聚合酶链式反应(polymerase chain reaction,PCR)mix:北京金沙生物科技有限公司;酵母总核糖核酸(ribonucleic acid,RNA)快速抽提试剂盒、一步法反转录荧光定量试剂盒:上海生工生物工程股份有限公司;无酵母米酒酒曲:本实验室。其他试剂均为国产分析纯。
1.1.4 培养基
Luria-Bertani(LB)液体培养基[9]:酵母提取粉5 g/L、氯化钠5 g/L、蛋白胨10 g/L。
酵母浸出粉胨葡萄糖(yeast extract peptone dextrose,YPD)液体培养基[21]:酵母提取粉10 g/L、蛋白胨20 g/L、无水葡萄糖20 g/L。
模拟发酵培养基[12]:麦芽粉130 g/L、白砂糖100 g/L。
以上对应固体培养基需添加2%琼脂,以上培养基均在115 ℃高压蒸汽灭菌30 min,备用。
1.2 仪器与设备
Gel Doc 2000凝胶成像分析系统、MicroPulser电穿孔仪:伯乐生命医学产品有限公司(bio-rad);EPS300电泳仪:上海天能生命科学有限公司;TC1000-G PCR基因扩增仪:大龙兴创实验仪器(北京)股份公司;GC8100气相色谱(gas chromatography,GC)仪:滕州市经纬分析仪器有限责任公司。
1.3 方法
1.3.1 CRISPR-Cas9系统的构建
CRISPR-Cas9系统包括Cas9蛋白和gRNA,Cas9蛋白通过与gRNA结合实现基因编辑[22]。Cas9质粒的构建需要含有Cas9基因和博来霉素抗性基因。以质粒p414-Cas9为模板,Cas9-F和Cas9-R为引物,经PCR扩增得到含有Cas9基因的片段(8 330 bp);以pPICZ(alpha)质粒为模板,BleoR-F和BleoR-R为引物,经PCR扩增得到两端与含有Cas9基因片段末端20 bp序列同源的BleoR抗性基因片段(1 324 bp);将两个片段混合后,用同源重组试剂盒实现DNA的体外环化。直接转化感受态大肠杆菌DH5α,取100 μL转化液涂布于含有100 mg/mL氨苄青霉素的LB平板,筛选得到Cas9表达质粒p414-Cas9-BleoR。
gRNA质粒的构建需要将LEU1基因设置为靶位点,并含有G418抗性基因。以质粒p426-gRNA为模板,gRNA-F和gRNA-R为引物,进行PCR扩增得到含有SNR52启动子、gRNA scaffold及SUP4终止子的片段(4 953 bp);以pUG6质粒为模板,kanMX-F和kanMX-R为引物进行PCR扩增,得到两端与含有gRNA基因片段末端20 bp序列同源的G418抗性基因片段(1 482 bp);将两个片段混合后,用同源重组试剂盒实现DNA的体外环化。直接转化感受态大肠杆菌DH5α中,取100 μL转化液涂布于含有100 mg/mL氨苄青霉素的LB平板,筛选得到gRNA表达质粒p426-gRNA-kanMX。
参照NCBI中酿酒酵母S288c基因组序列(序列号:NC-001139)信息,选取LEU1基因为目标基因,利用SnapGene软件找到间隔序列前体旁基序位点(5-NGG或NAG结构)。通过融合PCR技术得到含有20 ntLEU1靶点序列的片段(60 bp),以质粒p426-gRNA-kanMX为模板,LEU1r-F和LEU1r-R为引物进行PCR扩增,得到与含有LEU1靶点序列片段末端20 bp序列同源的片段(6 375 bp);将两个片段混合后,用同源重组试剂盒实现DNA的体外环化。直接转化感受态大肠杆菌DH5α,取100μL转化液涂布于含有100mg/mL氨苄青霉素的LB平板,筛选得到能够识别LEU1靶点的gRNA表达质粒p426-gRNA.LEU1-kanMX。
以酿酒酵母S288c基因组为模板,在LEU1靶点序列的上游和下游,分别用引物LT-F1和LT-R1、LT-F2和LT-R2进行PCR扩增得到上下游两段同源臂,再通过融合PCR技术即可得到donor DNA。
1.3.2 酿酒酵母电转化和阳性转化子筛选
将1 μL纯化后的p414-Cas9-BleoR质粒加到100 μL酿酒酵母XF0感受态进行电转化,30 ℃温育30 min后涂布于含有200 μg/mL博来霉素的YPD平板,30 ℃倒置培养2~3 d,挑取阳性菌落进行PCR验证。在此酵母的基础上制备感受态,加入1 μL纯化后的p426-gRNA.LEU1-kanMX质粒和10 μL donor DNA进行电转化,30 ℃温育30 min,将温育的转化子涂布于含有100 μg/mL G418抗生素的YPD平板,30 ℃倒置培养2~3 d,挑取阳性菌落进行PCR验证[23]。
1.3.3 基因敲除菌的筛选验证
将菌落振荡裂解,采用引物LEU1-A/LEU1-D进行菌落PCR验证,确认donor DNA通过酵母的同源重组修复机制正确地整合到基因组上。用酵母总RNA快速抽提试剂盒提取阳性菌株总RNA,以其为模板再用一步法反转录荧光定量试剂盒进行实时荧光定量PCR。
1.3.4 质粒丢失
将验证阳性的菌株划线至YPD平板,挑取单菌落接种于10 mL液体培养基,30 ℃、200 r/min条件下24 h,连续传代培养12代以上,用含200 μg/mL博来霉素平板和100 μg/mL G418抗生素平板挑选质粒丢失、没有抗性的重组酵母菌XF0-L[9]。
1.3.5 生长曲线的绘制
取3 mL酵母种子液接种到100 mL YPD液体培养基,30 ℃、200 r/min摇床培养,每2 h取2 mL菌液测定OD600nm值。
1.3.6 模拟发酵酒的制备
将斜面保存的酵母菌接种于YPD液体培养基,30 ℃、180 r/min摇床培养24 h得到种子液。取3 mL种子液接种到100mL模拟发酵培养基,30℃静置发酵4d,即得模拟发酵酒。
1.3.7 米酒的制备
将80 g米煮饭冷却后置于500 mL烧杯中,加20%无酵母米酒酒曲和100 mL水,接种12%酵母种子液,33 ℃静置发酵10 d,即得米酒。
1.3.8 分析方法
将模拟发酵酒液过滤后测定CO2产生量、pH值、还原糖含量、酒精度及高级醇;将米酒蒸馏出100 mL酒液,测定高级醇。
CO2产生量:称量法测定[24];还原糖含量:3,5-二硝基水杨酸(3,5-dinitrosalicylic acid,DNS)比色法测定[25];pH值:pH计测定;酒精度:酒精计测定;高级醇:气相色谱内标法测定[26-27]。
1.3.9 数据处理
每个试验重复3次,结果以“平均值±标准差”表示,通过SPSS 29.0软件进行显著性分析,使用Origin pro 2021软件绘制柱状图、生长曲线图。
2 结果与分析
2.1 CRISPR-Cas9系统的构建
2.1.1 重组质粒p414-Cas9-BleoR的构建
重组质粒p414-Cas9-BleoR的构建结果见图1。将重组质粒委托上海生工生物工程股份有限公司进行测序。测序结果显示,质粒序列正确,证明重组质粒构建成功。
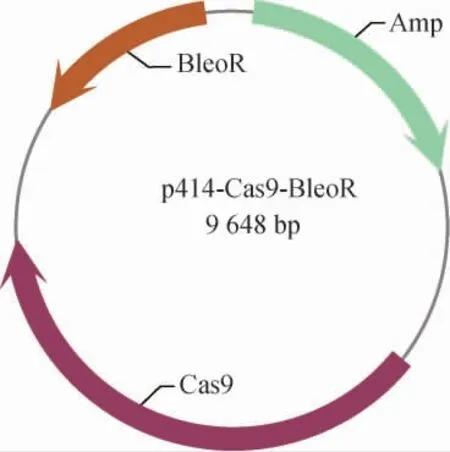
图1 重组质粒p414-Cas9-BleoR的构建Fig. 1 Construction of recombinant plasmid p414-Cas9-BleoR
2.1.2LEU1基因gRNA打靶质粒的构建
重组质粒p426-gRNA.LEU1-kanMX的构建结果见图2。将重组质粒委托上海生工生物工程股份有限公司进行测序。测序结果显示,质粒序列正确,证明重组质粒构建成功。
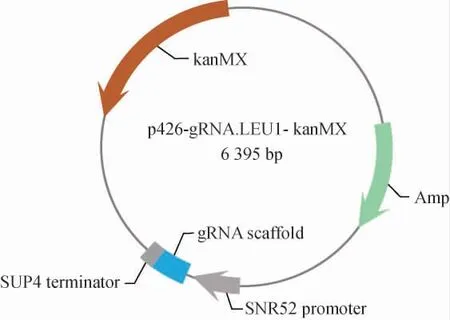
图2 重组质粒p426-gRNA.LEU1-kanMX的构建Fig. 2 Construction of recombinant plasmid p426-gRNA.LEU1-kanMX
2.2 LEU1靶点基因敲除的结果
电转化后筛选出重组菌株XF0-L,以其基因组DNA为模板,出发菌株XF0基因组DNA为阴性对照,采用引物LEU1-A和LEU1-D进行PCR扩增,扩增产物经1%琼脂糖凝胶电泳检测,结果见图3。由图3可知,以重组菌株XF0-L和出发菌株XF0基因组DNA为模板分别扩增出1 279 bp(敲除510 bp)和1 789 bp片段,结果与预期一致,证明LEU1基因已成功被敲除。
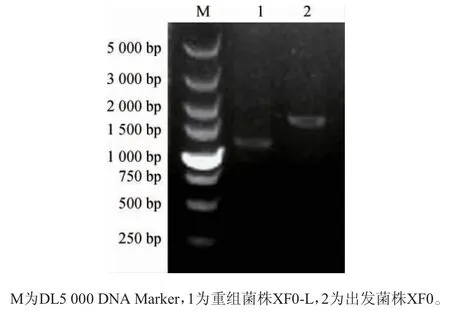
图3 重组菌株XF0-L的PCR验证结果Fig. 3 PCR validation results of recombinant strain XF0-L
为进一步验证LEU1基因是否被敲除,提取重组菌株XF0-L和出发菌株XF0的总RNA并反转录成互补DNA(complementary DNA,cDNA),以其为模板进行实时荧光定量PCR,分析LEU1基因的表达量,结果见图4。由图4可知,重组菌株XF0-L的LEU1基因表达量极低,这说明LEU1基因已成功被敲除。
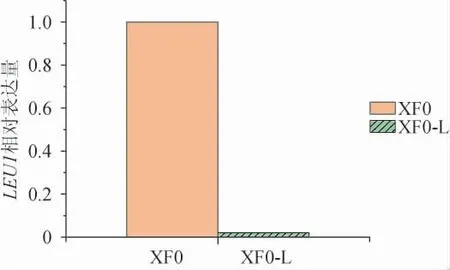
图4 出发菌株XF0和重组菌株XF0-L中LEU1基因表达量的测定结果Fig. 4 Determination results of LEU1 gene expression in original strain XF0 and recombinant strain XF0-L
2.3 质粒丢失验证
通过影印平板法筛选传代丢失质粒的重组菌株XF0-L,结果见图5。由图5可知,重组菌株XF0-L在不含G418抗生素的平板上正常生长,在含G418抗生素的平板上不会生长或生长缓慢。这说明重组菌株XF0-L中质粒p426-gRNA.LEU1-kanMX已成功被去除。

图5 重组菌株XF0-L质粒丢失验证结果Fig. 5 Verification results of plasmid loss of recombinant strain xf0-l
2.4 LEU1基因敲除对菌株生长性能的影响
出发菌株XF0和重组菌株XF0-L的生长曲线见图6。由图6可知,LEU1基因缺失重组菌株XF0-L的生长速率与出发菌株XF0无明显区别,说明LEU1基因缺失不影响酵母菌的生长性能。
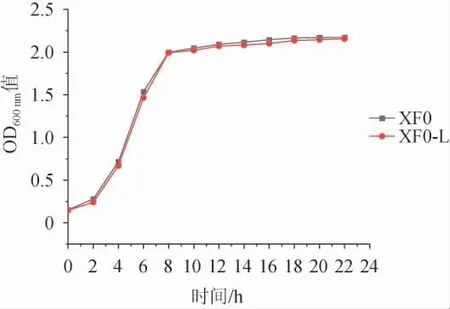
图6 出发菌株XF0和重组菌株XF0-L的生长曲线Fig. 6 Growth curves of original strain XF0 and recombinant strain XF0-L
2.5 LEU1基因敲除对菌株发酵性能的影响
出发菌株XF0和重组菌株XF0-L模拟发酵酒的理化指标见表3。由表3可知,重组菌株XF0-L与出发菌株XF0模拟发酵后的CO2产生量、还原糖含量、pH值和酒精度均无显著差异(P>0.05),说明LEU1基因缺失对酵母发酵性能没有影响。

表3 出发菌株XF0和重组菌株XF0-L模拟发酵酒的理化指标Table 3 Physicochemical indexes of simulated fermented wine with original strain XF0 and recombinant strain XF0-L
2.6 LEU1基因敲除对菌株生成高级醇的影响
采用出发菌株XF0与重组菌株XF0-L进行发酵,模拟发酵酒和米酒中正丙醇、异丁醇、异戊醇和总高级醇的含量检测结果见图7。
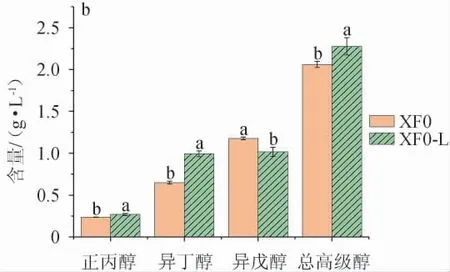
图7 出发菌株XF0和重组菌株XF0-L模拟发酵酒(a)及发酵米酒(b)中的高级醇含量Fig. 7 Higher alcohols contents in simulated fermented wine (a)and fermented rice wine (b) with original strain XF0 and recombinant strain XF0-L
由图7a可知,重组菌株XF0-L模拟发酵酒中正丙醇、异丁醇、总高级醇含量分别为26.27mg/L、88.64mg/L、256.72mg/L,比出发菌株XF0分别提高21.96%、85.25%、8.18%;异戊醇含量为141.82 mg/L,比出发菌株XF0降低15.53%。由图7b可知,重组菌株XF0-L发酵米酒中正丙醇、异丁醇、总高级醇含量分别为268.67 mg/L、992.33 mg/L、2 277.33 mg/L,比出发菌株XF0分别提高14.17%、52.90%、10.53%,异戊醇含量为1 016.33 mg/L,比出发菌株XF0降低13.58%。重组菌株XF0-L模拟发酵酒和发酵米酒中的异戊醇/正丙醇分别为5.39和3.78,比出发菌株XF0分别降低30.99%、24.40%;异戊醇/异丁醇分别为1.60和1.02,比出发菌株XF0分别降低54.42%、43.65%。
结果表明,利用LEU1基因敲除重组菌株XF0-L发酵酒能够提高正丙醇和异丁醇含量,降低异戊醇含量。这与LI W等[11-12]敲除酿酒酵母LEU1基因使得正丙醇、异丁醇提高,异戊醇降低的结论类似。这证明LEU1基因的缺失影响了苏氨酸及高丝氨酸合成α-酮丁酸的代谢流,导致正丙醇升高;LEU1基因的缺失阻碍了α-酮异己酸的合成,直接导致异戊醇生成量降低,而下游代谢途径受阻导致α-酮异戊酸的积累,造成异丁醇生成量提高。
3 结论
本研究基于CRISPR-Cas9基因编辑技术构建重组质粒p414-Cas9-BleoR和p426-gRNA.LEU1-kanMX,建立了通过博来霉素和G418抗生素来筛选酿酒酵母的基因敲除系统,并通过电转化成功获得LEU1基因敲除的重组菌XF0-L。结果表明,菌株XF0-L与XF0模拟发酵酒的理化指标无显著差异(P>0.05),说明LEU1基因的缺失对酿酒酵母发酵性能无影响。相较于出发菌株XF0,重组菌株XF0-L模拟发酵酒中正丙醇(26.27 mg/L)和异丁醇含量(88.64 mg/L)分别提高21.96%和85.25%,异戊醇含量(141.82 mg/L)、异戊醇/正丙醇(5.39)、异戊醇/异丁醇(1.60)分别降低15.53%、30.99%、54.42%;相较于出发菌株XF0,重组菌株XF0-L发酵米酒中正丙醇(268.67 mg/L)和异丁醇含量(992.33 mg/L)分别提高14.17%、52.90%,异戊醇含量(1 016.33 mg/L)、异戊醇/正丙醇(3.78)、异戊醇/异丁醇(1.02)分别降低13.58%、24.40%、43.65%,说明重组菌株XF0-L能够显著降低发酵酒中的异戊醇/正丙醇、异戊醇/异丁醇从而降低醉酒度(P<0.05),这为酿造低醉酒度高品质米酒提供了新的技术思路。综上所述,利用CRISPR-Cas9技术改良酿酒酵母菌是优化酿造酒中不同高级醇比例实现降低醉酒度和提高口感品质的可行酿酒技术。
