3种方法对1株临床疑难菌的鉴定及结果比较
2024-05-14许一丹谢成彬王频佳
许一丹,谢成彬,苏 喆,王频佳△
1.成都医学院检验医学院临床微生物学教研室,四川成都 610500;2.四川省妇幼保健院/成都医学院附属妇女儿童医院检验科,四川成都 610045
细菌的准确鉴定是细菌学中最重要、最具挑战性的问题之一,也是抗感染治疗的基础。细菌鉴定的方法有很多种,常用的包括形态学鉴定、生化鉴定和分子生物学鉴定等。由于基于生化鉴定原理的自动微生物鉴定仪早在1985年就进入我国临床实验室,并从20世纪90年代至今广泛用于临床标本的检测,因此细菌生化鉴定是目前临床最常用的细菌鉴定方法[1-3]。但由于细菌的种类繁多,存在多样性和复杂性,选择的生化鉴定特征可能难以代表全部被鉴定菌株,特别是临床少见菌、罕见菌和未明确分类的细菌,生化鉴定往往难以发挥作用。本课题组前期从临床标本中分离出1株细菌,经生化鉴定疑似为盐单胞菌(Halomonas),但无法准确鉴定其菌种。针对这株疑难菌,本研究将利用基质辅助激光解吸电离飞行时间质谱(MALDI-TOF MS)、16S rRNA序列分析和基于全基因组测序(WGS)的基因组序列比对3种方法进行菌种鉴定,并对鉴定结果进行比较。现将结果报道如下。
1 材料与方法
1.1菌株来源 2018年7月从四川省妇保健院新生儿重症监护室的1例患儿血液标本中分离出1株革兰阴性菌,生化鉴定结果显示该菌疑似为盐单胞菌属的细菌,菌株编号为18071143。
1.2主要试剂和仪器 哥伦比亚血琼脂平板(郑州博赛生物技术股份有限公司),Autof ms1000全自动微生物质谱检测系统(郑州安图生物工程股份有限公司)。
1.3方法
1.3.1菌株培养 将受试菌株18071143接种于哥伦比亚血琼脂平板,35 ℃培养24~48 h,再次转种于哥伦比亚血琼脂平板,35 ℃培养18~24 h,挑取纯菌,按照实验要求进行后续的MALDI-TOF MS检测、16S rRNA测序分析以及基因序列比对。
1.3.2MALDI-TOF MS检测 按照仪器说明书操作[4]步骤进行操作。
1.3.316S rRNA 序列分析 16S rRNA基因测序由北京睿博兴科生物技术有限公司完成。将测序所得的16S rRNA 序列输入EZBioCloud细菌16S鉴定数据库 (https://www.ezbiocloud.net/identify)[5-6],进行序列相似性分析。物种水平鉴定以99%核苷酸相似度为标准[7]。
1.3.4基于WGS的基因组序列比对 WGS由北京百迈客生物科技有限公司完成。将与菌株18071143的16S rRNA 基因序列相似性>99%的模式菌株作为平均核苷酸一致性(ANI)分析的对比菌株。利用美吉生物云工具(https://cloud.majorbio.com/page/tools/)进行ANI分析,使用ANIm和ANIb 2种方法计算ANI值。此外,将菌株18071143的基因组序列与基因组分类数据库(GTDB)[8]进行比对,以获得菌种信息。
1.3.5系统发育分析 基于WGS中的16S rRNA序列和看家基因(dnaG,frr,infC,nusA,pgk,pyrG,rplA,rplB,rplC,rplD,rplE,rplF,rplK,rplL,rplM,rplN,rplP,rplS,rplT,rpmA,rpoB,rpsB,rpsC,rpsE,rpsI,rpsJ,rpsK,rpsM,rpsS,smpB,tsf),选择在GTDB中种属水平上与菌株18071143最接近的20株菌,通过MEGA7.0软件采用近邻相接法构建系统进化树。
1.3.6基因组聚类分析 通过Spearman相关性分析,在COG和KEGG功能上对基因组进行聚类分析,评估菌株18071143和与其在16S rRNA基因序列相似性>99%的模式菌株之间基因组水平上的亲缘关系。相关系数越接近于1,表明样本间相关性越好。
2 结 果
2.1MALDI-TOF MS检测 MALDI-TOF MS对菌株18071143的鉴定未出现低分辨和无法鉴定的情况,鉴定结果为Halomonas hamiltonii。
2.216S rRNA序列分析 菌株18071143的16S rRNA 基因序列的GenBank登录号是MZ097522.1。该菌株的16S rRNA序列经与细菌16S鉴定数据库比对后,得到相似度较高的细菌,相似性排名前5的细菌见表1,其中菌株18071143与Halomonas hamiltonii、Halomonas stevensii、Halomonas johnsoniae的相似度均>99%。

表1 菌株18071143的16S rRNA序列比对结果
2.3基因组序列比对 菌株18071143的全基因组序列的GenBank登录号是CP078120.1。通过ANIm和ANIb 2方法分别计算菌株18071143与Halomonas hamiltonii、Halomonas stevensii和Halomonas johnsoniae 3种盐单胞菌的ANI值。2种算法均显示菌株18071143与Halomonas stevensii的亲缘关系最近(ANIb= 0.975 1,ANIm= 0.978 6)。将菌株18071143与GTDB数据库进行比对后发现,与菌株18071143最为相似的细菌是Halomonas stevensii,见表2。故基于WGS的序列比对分析,菌株18071143被鉴定为Halomonas stevensii。

表2 菌株18071143与GTDB数据库对比结果
2.4系统发育分析 菌株18071143基因组中预测到6个16S rRNA和31个看家基因。基于16S rRNA和看家基因分别构建系统进化树,见图1和图2。16S rRNA和看家基因的进化树均显示,菌株18071143与Halomonas stevensii的亲缘关系最为接近。

图1 菌株18071143与20株盐单胞菌的16S rRNA系统的进化树

图2 菌株18071143与20株盐单胞菌的看家基因系统的进化树
2.5基因组聚类分析 基于的COG和KEGG功能对基因组进行聚类分析,菌株18071143与Halomonas stevensii的相关系数分别是0.993 5和0.703 7,相关性最高,因此菌株18071143与Halomonas stevensii的亲缘关系最为接近。见图3。
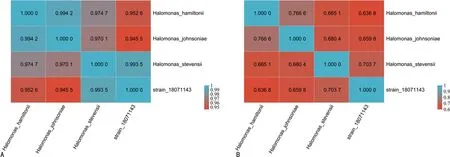
注:A是基于COG功能分类的结果;B是基于KEGG功能注释的结果。
3 讨 论
作为常规细菌鉴定方法的生化试验虽操作简单,但需要一定的培养时间,且对于一些细菌只能达到区分菌属的程度。MALDI-TOF MS技术是近年来快速发展的一种用于鉴定多肽、蛋白质的新型软电离质谱技术,由于其具有快速、准确、高通量、易于操作和重复性好等优点,该技术越来越广泛地取代常规方法成为细菌鉴定的一线工具[9-11]。16S rRNA基因测序被认为是一种成熟的分类学研究方法[12],特别适用于MALDI-TOF MS数据库中无数据细菌的鉴定[12-15]。另外,需要进行生物信息学分析,且成本较高的基于WGS的菌种鉴定因其方法的高分辨率,使之成为传染病诊断和感染源追踪的一项非常有前景的技术[16]。在基因组水平上,ANI分析被认为是菌种鉴定的神器。普遍认为亲缘关系较近的种群间ANI值应为70%~75%,而定义一个种的ANI值需要达到95%~96%[17-18]。ANI值一般可以通过2种运算方法得出:一种是以BLASTn方法为基础(ANIb),另一种以MUMmer运算法则为基础(ANIm)。ANI具有方便、快速、分辨率高的优点,近年来被广泛用于细菌鉴定。ZHOU等[19]通过高通量测序技术和ANI识别一个名为AV208的革兰阳性球菌菌株。JIN等[20]利用ANIb和全基因组测序的系统发育分析,纠正奈瑟菌属中错误的菌种分类,并指出GenBank中的Neisseria mucosa(n=13)和Neisseria sicca(n=16)应使用ANIb和高分辨率系统发育分析重新进行分类。
本研究结果发现,MALDI-TOF MS、16S rRNA基因测序和WGS对菌株18071143在菌属层面的鉴定一致性达到100%,但在菌种水平的鉴定上却存在差异。MALDI-TOF MS的鉴定结果为Halomonas hamiltonii。16S rRNA基因序列分析最为相似的细菌也是Halomonas hamiltonii,但菌株18071143与3种盐单胞菌的序列相似度均>99%,且不同种之间的分辨率低于0.8%。根据美国临床和实验室标准协会(CLSI) 指南[21]要求,本次16S rRNA基因序列分析无法在菌种水平上鉴定细菌。基于WGS的ANI分析发现,菌株18071143更倾向于是Halomonas stevensii,这一点也从该菌的基因组序列与GTDB数据库的比对结果、与相似菌的系统发育以及基因组的聚类分析得到证实。以往有学者指出,16S rRNA序列分析的主要问题是无法准确确定物种[22-23]。尽管16S rRNA基因序列被广泛用于分类学和系统发育学的研究,但在区分密切相关的菌株和物种时有一定的局限性,尤其是没有基于与公共数据库中序列的相似性来明确识别物种的普遍认可的阈值[24]。MALDI-TOF MS也存在同样的问题。MALDI-TOF MS方法操作简便,非常适用于临床工作,但仪器数据库的完备性也会影响到结果的可靠性[25]。细菌基因组包含其全部的遗传信息,确定基因组序列成为了解细菌生物学特性与功能特征的基础和前提。细菌全基因组序列信息能够对细菌的鉴定提供最高的分辨率。
本研究利用3种方法对临床常规方法无法鉴定到种的盐单胞菌进行了菌种鉴定,结果显示基于WGS的鉴定结果更加准确。MALDI-TOF MS方法简单、快速,适用于临床常规工作;16S rRNA序列分析分辨率较差,对于某些疑难菌难以准确鉴定;WGS分辨率高,适用于具有相似遗传学特征的菌种之间的鉴别。因此,在临床工作中若分离到罕见菌株和疑难菌株,建议使用WGS技术获取细菌全基因组序列信息,以全面了解该菌的生物学特性与功能特征。