东北春播区糜子核心种质及其DNA分子身份证构建
2024-04-28丁艺冰辛旭霞冯智尊DipakSANTRA王瑞云陈喜明
丁艺冰 辛旭霞 冯智尊 曹 越 郭 娟 Dipak K SANTRA 王瑞云,* 陈喜明
东北春播区糜子核心种质及其DNA分子身份证构建
丁艺冰1,2辛旭霞2冯智尊2曹 越2郭 娟2Dipak K SANTRA3王瑞云2,*陈喜明1,4,*
1山西农业大学玉米研究所, 中国山西忻州 034000;2山西农业大学农学院, 中国山西太谷 030801;3内布拉斯加大学林肯分校农艺系小宗粮豆研究与推广中心, 美国内布拉斯加州斯克茨布拉夫 69361;4山西农业大学杂粮种质创新与分子育种山西省重点实验室, 中国山西太原 030000
本研究以500份东北春播区糜子资源为材料, 利用169个SSR标记, 采用UPGMA聚类分组, 进行分层抽样, 构建核心种质, 同时应用ID Analysis 4.0软件构建分子身份证。利用等位基因数(Na)等遗传多样性衡量指标评估核心种质的遗传差异, 并利用PCOA分析核心种质。结果表明, 对169对SSR引物进行筛选, 发现30对多态性好, 利用30对SSR引物构建的糜子核心种质包含190份材料, 占全部种质的38%, 全部种质与核心种质的均检测出91个等位变异, 保留了100%等位基因; 有效等位基因数为2.2977~2.9975和2.2872~3.0173, 平均值分别为2.8198和2.8297; Shannon多样性指数为0.9532~1.0990和0.9535~1.1162, 平均值为1.0645和1.0667; 观测杂合度为0.3434~0.8037和0.3162~0.7849, 平均值为0.5399和0.5359; 期望杂合度为0.5654~0.6672和0.5645~0.6707, 平均值为0.6448和0.6473; Nei’s基因多样性指数为0.5648~0.6664和0.5628~0.6686, 平均值为0.6441和0.6452; 多态性信息含量为0.6657~0.8356和0.6493~0.8340, 平均值为0.7974和0.7944。全部种质与核心种质的分子标记的相关指标进行检验, 结果无显著性差异, 且PCOA分析表明核心种质与全部种质具有相似的遗传多样性和群体结构, 同时发现8个SSR标记(RYW5、RYW8、RYW16、RYW28、RYW40、RYW53、RYW62和RYW67)可区分190份核心种质, 构建了东北糜子核心种质的分子身份证。
糜子; 东北春播区; SSR; 核心种质; DNA分子身份证
糜子(L.)是起源于我国最古老的作物之一, 栽培历史超过10,300年[1-2]。具有生育期短、耐旱、耐贫瘠等特点, 是旱作地区最主要的粮食之一[3-5]。糜子种植广泛, 划分为7个糜子栽培生态区[6-8]。其中, 东北春糜子区包括黑龙江、吉林、辽宁(朝阳地区除外)三省及内蒙古兴安盟和通辽市。在中国, 糜子可能至少有2个独立的驯化区域, 就是黄土高原和东北[9]。东北糜子的播种季节主要是春季, 品种主要为糯性, 穗型主要为散穗, 籽粒形态较小, 颜色以黄色黑色为主[10]。糜子是中国古代北方地区的主要粮食作物, 辽宁是糜子的文化遗址之一[6]。糜子富含蛋白质、矿物质、维生素和微量元素, 包括铜、锌、铁、锰, 脱壳后称黄米, 可制作粘糕、酿制黄酒[11-12]。随着新资源不断收集与引入, 对种质资源整理与保存, 鉴定与评价及研究与利用都提出了重大挑战, 种质保存比较分散, 存在重复引进、同种异名和同名异种的现象, 种质之间亲缘关系不明确, 极大地影响了种质的高效利用[6,8,13-14]。
SSR是基于DNA长度多态性的分子标记, 常用于分子作图和标记辅助选择[1]。SSR较好的共显性、多等位性和高重复性等优势, 在基因调控和基因组进化等遗传研究中发挥重要作用[15-16]。SSR标记具有易于检测、信息量大、稳定、成本低的优点, 已成为品种鉴定的有力工具, 标记的确定在指纹库的构建方面成为关键环节[17]。
糜子种质资源丰富, 核心种质的构建能最大限度地保护糜子品种的遗传多样性[18]。澳大利亚的Frankel[19]提出核心种质的概念, Brown等[20]对其进一步的完善和论述。水稻(L)[21]、大麦(L.)[22]、花生(L.)[23]、大豆()[24]和蚕豆(L)[25]等作物均已构建核心种质。就糜子而言, 种质资源遗传多样性的研究较多, 源于开发了一批糜子特异性SSR标记, 同时也构建了一批DNA分子身份证[26]。基于SSR构建糜子核心种质的研究不多, 大多数研究仅限于糜子的生理、生化、栽培及营养品质等方面。迄今, 构建核心种质主要基于表型特征(包括农艺性状等)和基因型(SSR等标记), 其中可能有一个或者两者兼而有之[27]。陈伊航等[28]对1091份甘薯((L.) Lam.)种质的20个表型性状数据和10个SSR标记进行分析, 构建了289份材料的核心种质。汪磊等[29]对422份向日葵(L.)的11个农艺性状连续2年观察, 构建了84份材料的核心种质。刘松等[30]对342个中国板栗(Bl.)品种的21个SSR荧光标记和19个表型性状进行分析, 构建了85份材料的核心种质。孙永强等[31]对201份西伯利亚杏()的45个表型性状进行分析, 研究构建核心种质的最佳策略, 构建了44份材料的核心种质。
近年来, 糜子[32]、小麦(L.)[33]、大豆()[34]、苹果(Mill.)[35]、茶树()[36]等植物都基于SSR技术构建了分子身份证。王宇晴等[37]利用22对SSR引物分析111份甜菜(L.), 发现6对引物组合可区分全部材料。高源等[38]用16对TP-M13-SSR引物对131份苹果(Mill.)进行指纹图谱构建, 最终用3对引物区分了全部材料。高运来等[39]以黑龙江的83份大豆()品种为材料, 用43对大豆SSR引物进行检测, 发现仅用9对核心引物即可区分所有材料。马琳等[40]利用33对甘蓝型油菜()特异性SSR引物分析来自于国内外130份甘蓝型油菜, 最终仅用7对引物构建了所有供试材料的分子身份证。王宇卓等[26]发现20对SSR核心引物可区分272份糜子材料, 构建糜子分子身份证。陈小红等[32]利用30对SSR核心引物对130份糜子进行分析, 发现17对SSR可区分全部材料, 即可构建出糜子分子身份证。薛亚鹏等[41]利用7对荧光SSR区分了235份中国糜子核心种质来构建糜子分子身份证。利用分子标记技术, 建立DNA分子身份证可以解决发生同名异物的现象, 促进作物物种溯源、品种精准鉴定、遗传多样性评估等研究, 以确保良种的商品化和可靠性、相关品种在市场流通中保持纯正性[41]。
本研究以500份东北区糜子种质资源为材料, 用30对多态性较好的SSR标记对其进行遗传多样性分析, 构建糜子核心种质, 评价材料的代表性, 旨在为糜子种质资源的保护、优异种质挖掘及品种的遗传改良提供理论依据。同时以东北区糜子核心种质为材料, 构建东北区糜子核心种质的分子身份证, 实现对东北春糜子区核心种质的数字化管理。
1 材料与方法
1.1 材料
供试材料为500份东北区糜子资源(附表1), 包括黑龙江省290份、吉林省143份、辽宁省54份、内蒙古部分地区13份。169对SSR引物为课题组前期开发, 由生工生物工程(上海)股份有限公司合成。
1.2 DNA提取
采集糜子三叶期的叶片, 采用改良CTAB法[42]提取样本的基因组DNA。用1%琼脂糖凝胶电泳检测DNA完整性。用核酸检测仪检测DNA的浓度和纯度。
1.3 SSR引物的筛选
以地理来源差异大的12份糜子种质为材料, 利用169对引物进行PCR扩增, 用8%聚丙烯酰胺凝胶电泳检测扩增产物, 筛选出条带清晰、多态性较好且扩增稳定的引物, 方法同王宇卓等[26]。
1.4 数据分析
通过人工读带的方法统计稳定且易于区分的目的条带, 同一位置上有条带记为“1”, 没有条带记为“0”, 构建0、1二元数据矩阵。利用POPGEN1.32[43]计算遗传多样性衡量参数(包括等位基因数、有效等位基因数、Nei’s遗传多样性指数、Shannon’s多样性指数、观测杂合度、期望杂合度)。用Powermarker3.25[44]计算多态性信息含量, 利用MEGA11.0.10[45]进行UPGMA聚类分析, 基于UPGMA聚类进行分层抽样。用Bioladder在线云平台进行PCOA (坐标轴分析)验证核心种质与全部种质的遗传多样性和群体结构。
1.5 分子身份证的构建
利用软件ID Analysis 4.0[46]区分核心种质得到最优的引物组合。用NTSYS 2.11[47]进行主成分分析, 基于不同SSR标记读带结果依次排序构建字符串DNA分子身份证。利用在线条形码生成工具(http://barcode.cnaidc.com/app/html/bcgcode128.php)将对应字符串生成可扫描的条形码DNA分子身份证。利用二维码在线技术(https://cli.im/)将材料的基本信息(包括名称、统一编号、来源地、类别、字符串)转化为可扫描的二维码DNA分子身份证。
2 结果与分析
2.1 SSR引物的筛选
用169对SSR引物扩增12份糜子种质(表1)发现, 30对引物(表2)的多态性较好, 条带清晰。利用30对SSR引物对500份糜子进行PCR扩增 (图1)构建核心种质, 同时发现利用ID Analysis 4.0软件分析电泳结果, 表明8对SSR引物可区分核心种质。
2.2 核心种质的构建
基于SSR标记对全部种质进行UPGMA聚类分析(图2), 在遗传距离为0.09处将全部种质分为4组, 样品数量分别为290、143、54和13。根据分组结果进行分层抽样, 得到190份核心种质, 占全部种质的38%。

表1 用于筛选引物的12份材料
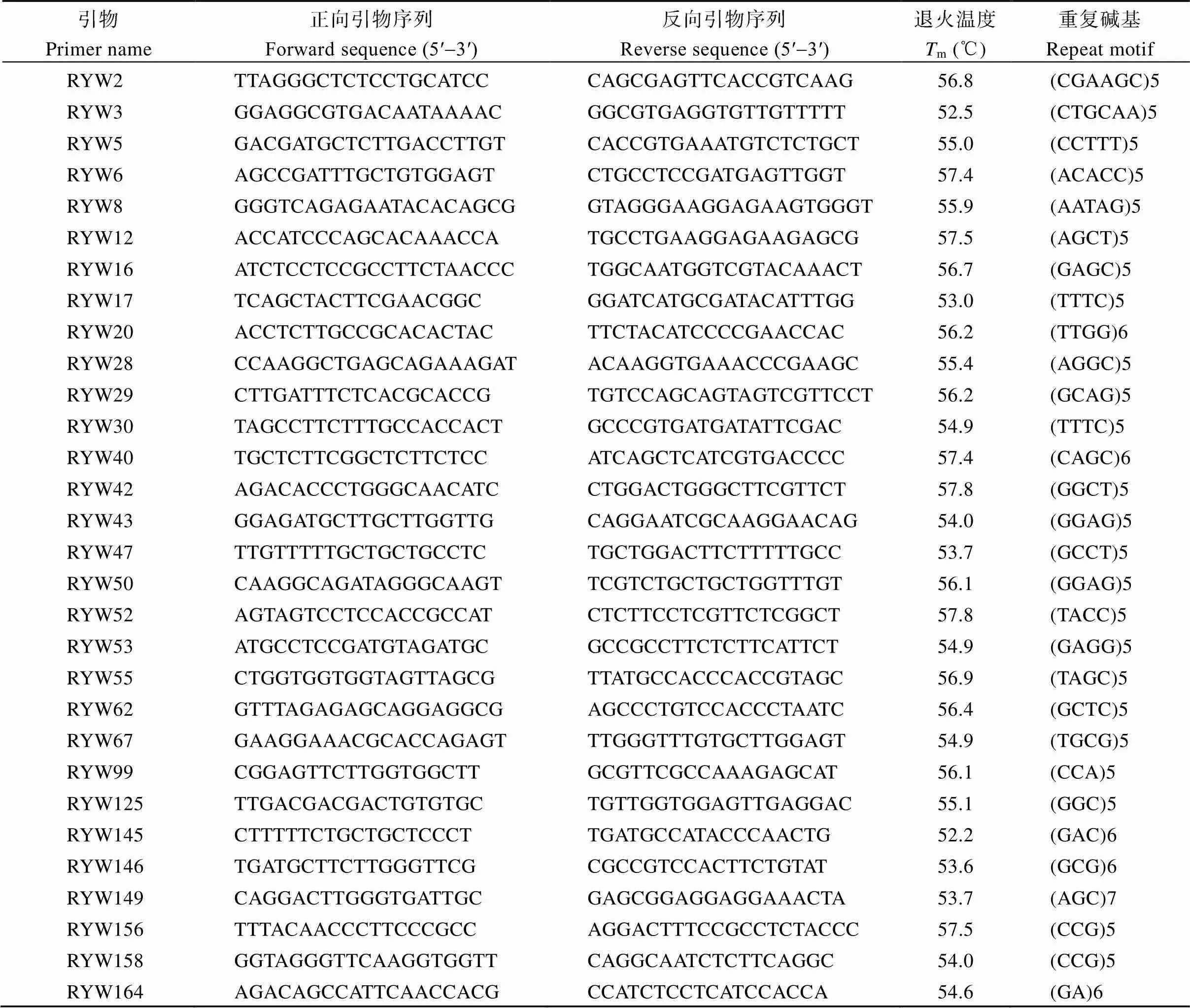
表2 构建核心种质的30对SSR引物

图1 RYW40引物对部分材料扩增的聚丙烯凝胶电泳图
1~20为黑龙江材料, 21~35为吉林材料, 36~45为辽宁材料, 46~50为内蒙古材料。
1-20 are Heilongjiang materials; 21-35 are Jilin materials; 36-45 are Liaoning materials; 46-50 are Inner Mongolia materials.
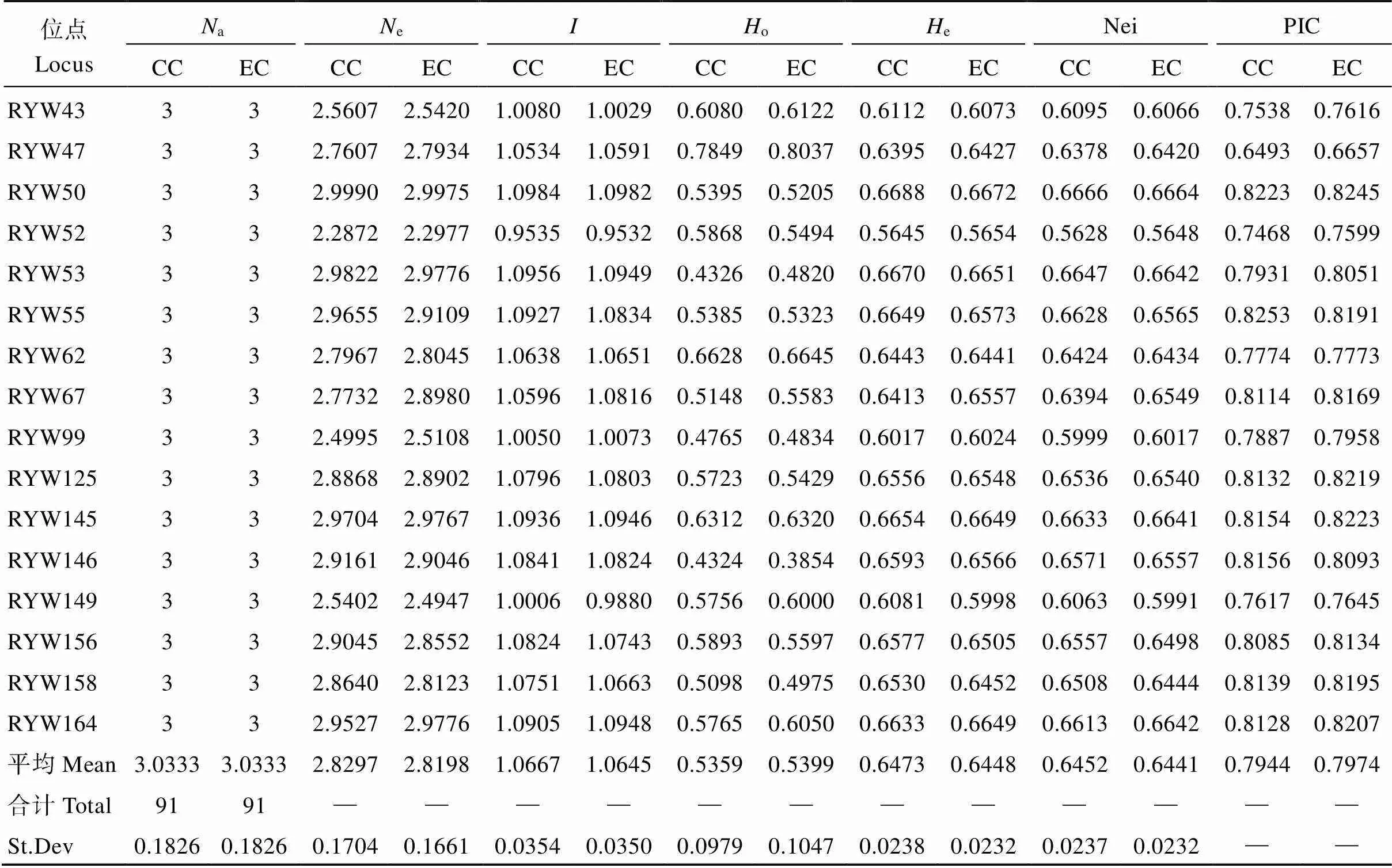
用上述的30对SSR引物扩增500份种质资源, 分别处理500份种质资源和根据分组进行分层抽样得到的190份核心种质的遗传多样性参数, 通过对比全部种质和核心种质SSR分子标记的相关指标, 全部种质与核心种质的均检测出91个等位变异, 保留了100%等位基因; 有效等位基因数平均值分别为2.8198和2.8297; Shannon多样性指数平均值为1.0645和1.0667; 观测杂合度为平均值为0.5399和0.5359, 其他数据见表3, 对全部种质与核心种质的遗传多样性参数进行检验结果表明(表4)差异不显著; 分别对4个省份的全部种质和核心种质的各个遗传多样性参数进行对比(表5)。内蒙古的材料较少没有代表性, 不计入分析结果中。其他三省结果表明,检验结果表明(表4), 遗传多样性均差异不显著。
最后用主坐标轴分析进行确认, 从全部种质和核心种质的主坐标轴分布图可知, 样本分布均匀分散, 发现核心种质的样本在全部种质样本中呈均匀分布(图3)。因此, 核心种质很好地保留了全部种质的遗传多样性和群体结构, 确保了核心种质的有效性和代表性。
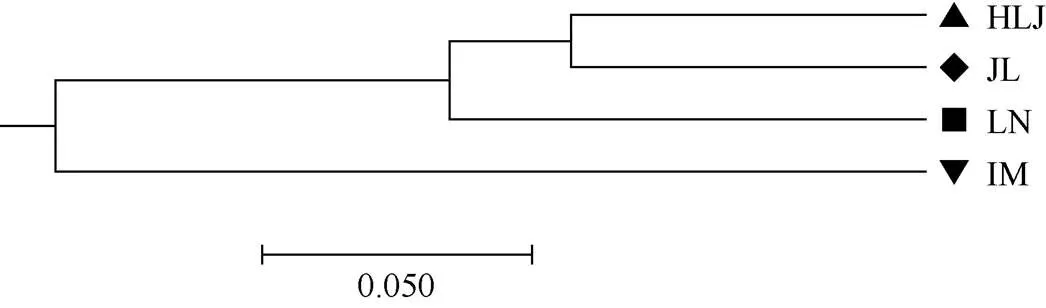
图2 基于30对SSR数据对4个糜子种质资源群体的聚类分析图
HLJ: 黑龙江; JL: 吉林; LN: 辽宁; IM: 内蒙古。
HLJ: Heilongjiang; JL: Jilin; LN: Liaoning; IM: Inner Mongolia.

表3 种质资源与核心种质遗传参数的对比
(续表3)
a: 等位基因数;e: 有效等位基因数;: Shannon多样性指数;o: 观测杂合度;e: 期望杂合度; Nei: Nei’s基因多样性指数; PIC: 多态性信息含量; EC: 种质资源; CC: 核心种质。
a: the number of alleles;e: the number of effective alleles;: Shannon’s diversity index;o: the observed heterozygosity;e: the expected heterozygosity; Nei: Nei’s gene diversity index; PIC: polymorphism information content; EC: the entire collection; CC: core collection.
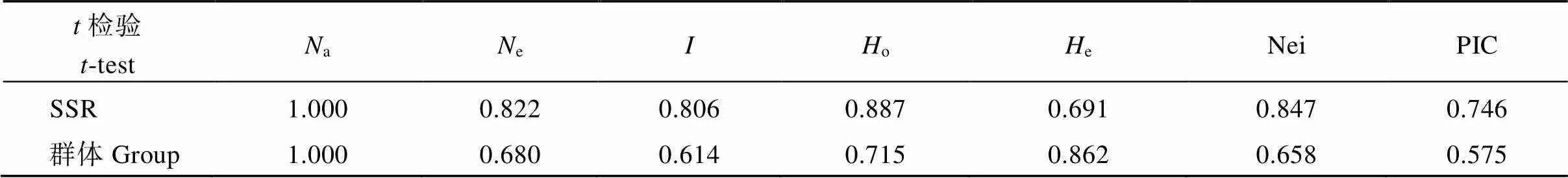
表4 种质资源与核心种质之间SSR和群体遗传参数的t检验
缩写同表3。
Abbreviations are the same as those given in Table 3.
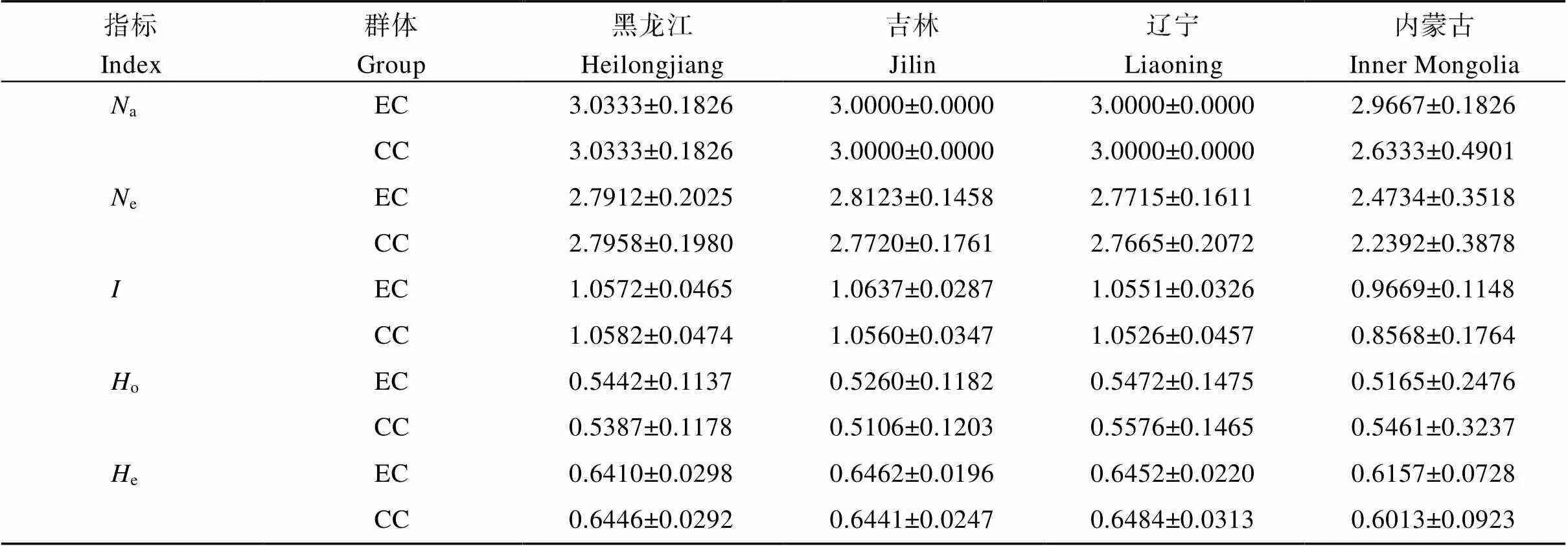
表5 不同来源糜子的遗传多样性参数
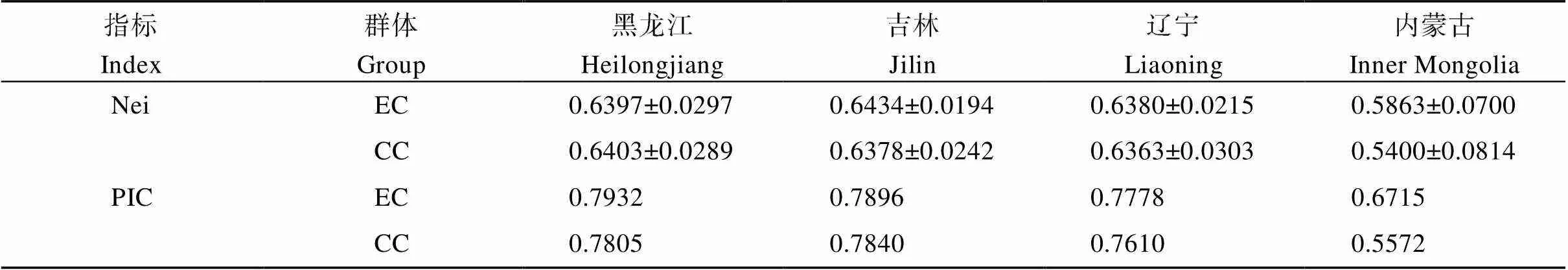
(续表5)
缩写同表3。Abbreviations are the same as those given in Table 3.
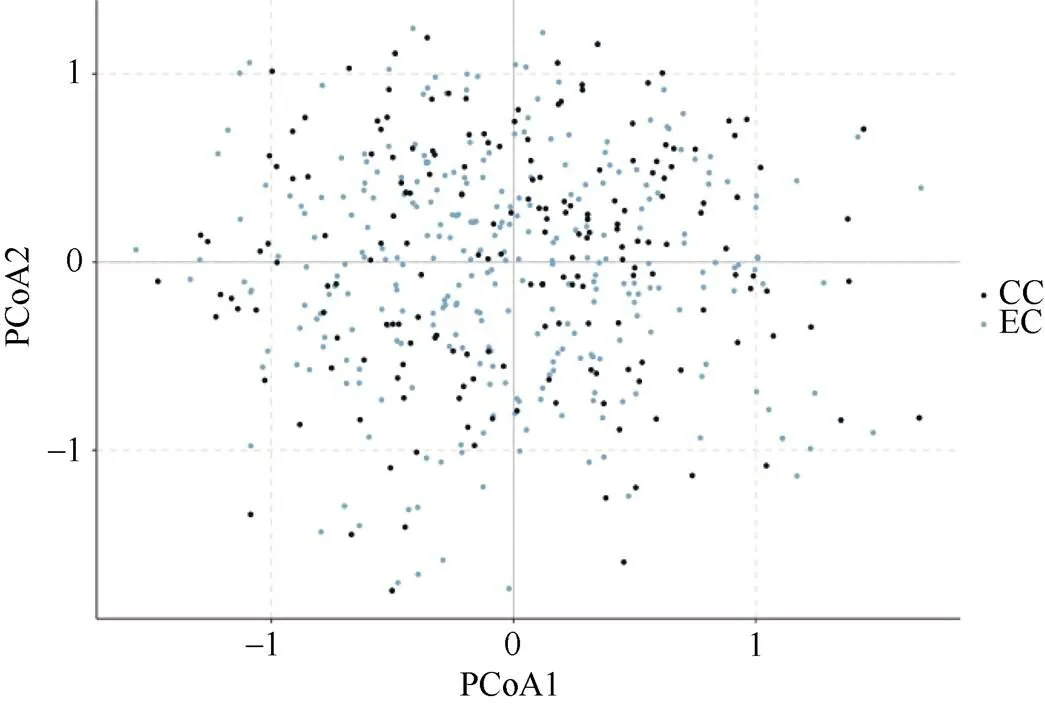
图3 核心种质与原种质的主坐标图
EC: 种质资源; CC: 核心种质。
EC: the entire collection; CC: core collection.
2.3 糜子核心种质分子身份证的构建
用上述8对引物分析190份核心种质的遗传多样性, 全部材料在8个位点共检测到24个等位基因(a), 平均每个位点检测出3个; 检测到有效等位变异(e)平均为2.8213; 检测到Shannon多样性指数()平均值为1.0654; 多态性信息含量(PIC), 平均为0.7925, 其他结果见表6。
对190份核心种质进行主成分分析(PCA)发现, 前3个主成分PC1、PC2、PC3分别解释总变异的49.81%、10.73%、9.86%, 总变异为70.40%。190份材料划归4个类群, 类群1全部来自黑龙江, 类群2全部来自吉林, 类群3全部来自辽宁, 类群4全部来自内蒙古(图4)。PCA聚类结果与资源的地理来源一致。
将190份糜子品种的扩增条带结果按照RYW67、RYW53、RYW62、RYW5、RYW40、RYW28、RYW8、RYW16先后顺序进行排列, 获得每个品种唯一的24位字符串, 即每个品种的字符串分子身份证, 见附图1, 以图5为例。采用线上条形码及二维码生成技术, 在相应字符串上生成直观条形码及可扫描二维码分子身份证, 并结合糜子品种DNA指纹数据及品种基本情况, 扫描二维码能够更方便的获得糜子品种的相关信息。
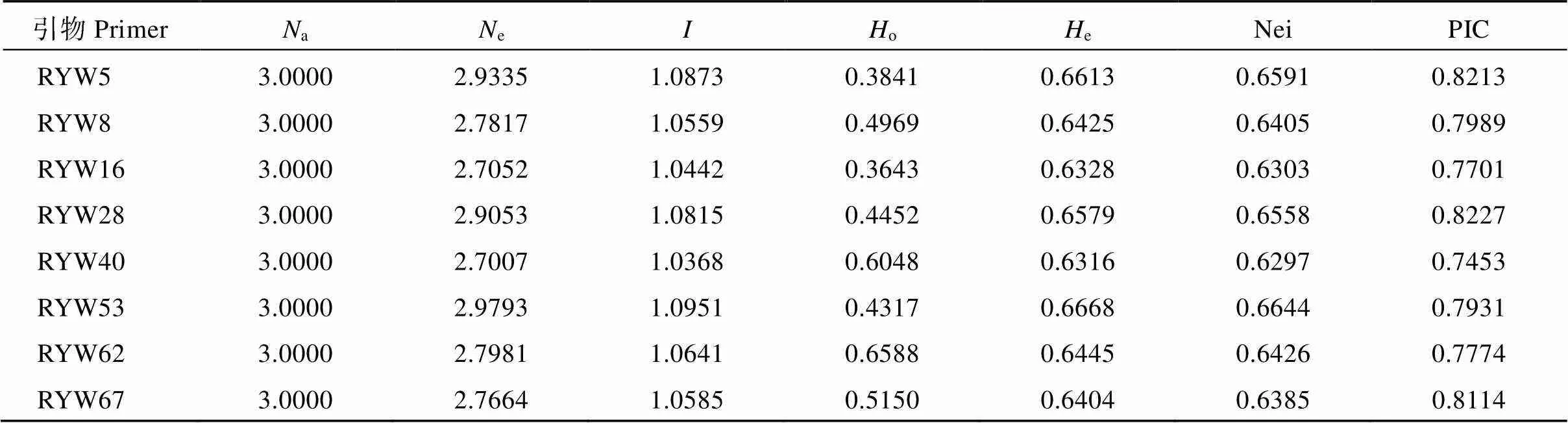
表6 8对SSR引物的遗传多样性参数
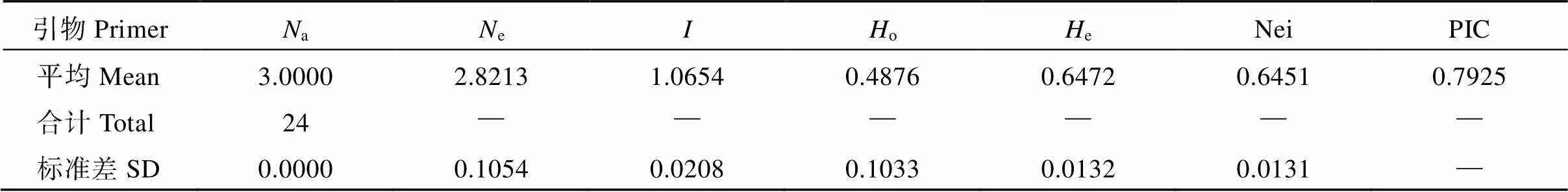
(续表6)
缩写同表3。Abbreviations are the same as those given in Table 3.
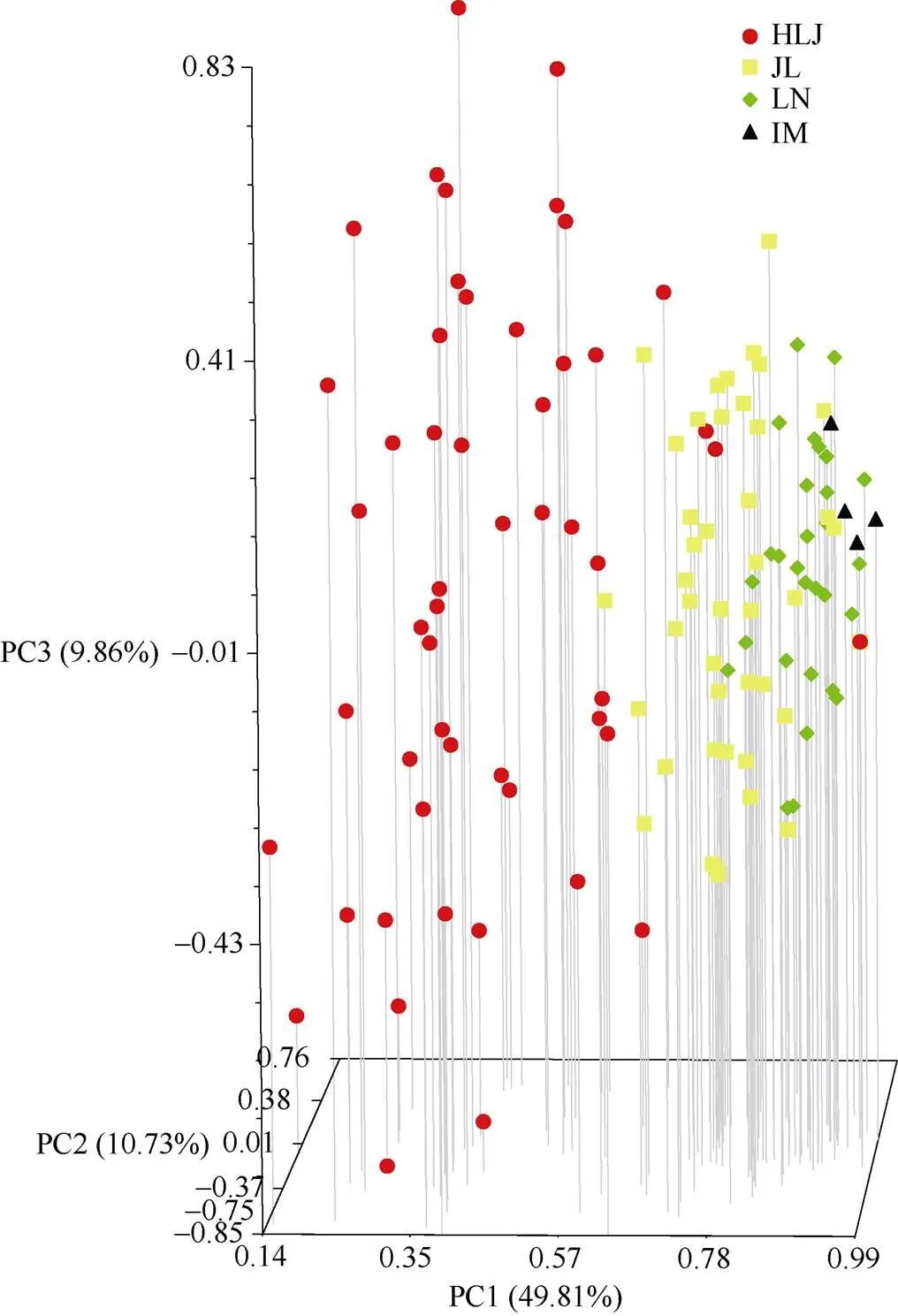
图4 190份糜子核心种质的主成分分析
HLJ: 黑龙江; JL: 吉林; LN: 辽宁; IM: 内蒙古。
HLJ: Heilongjiang; JL: Jilin; LN: Liaoning; IM: Inner Mongolia.

图5 糜子材料1品种条形码(A)和二维码(B)DNA分子身份证
3 讨论
Shannon多样性指数()和多态信息含量(PIC)能够全面的体现群体遗传多样性水平[48]。王宇卓等[26]用20对引物对272份山西糜子进行遗传多样性分析, 得到值和PIC值分别为1.0552和0.6921。王倩等[16]利用52对引物对6个生态区进行遗传多样性分析, 得到值和PIC值平均值分别为0.7277和0.5104。陈小红等[32]利用30对引物对130份糜子进行遗传多样性分析, 得到值和PIC值分别为1.0472和0.6966。何杰丽等[1]利用80对引物分析黄土高原春夏糜子区和北方春糜子区糜子的遗传多样性, 发现值和PIC值分别为(0.8506±0.1534)、0.4203和(0.8604±0.1576)、0.4536。本研究利用30对引物对500份东北区糜子进行遗传多样性分析, 得到值和PIC值分别为1.0645和0.7974, 均高于前人的研究, 说明不同生态区遗传多样性的不同可能与材料来源、引物的多态性有关。
核心种质用最少的种质必须尽可能多的代替种质资源的遗传多样性, 以往构建核心种质的研究多数基于表型性状。表型性状受多种因素影响, 且糜子完成生命周期时间需要3~4个月, 而分子标记生长至三叶期即可, 具有高效、快速、稳定的特点, 是评价植物遗传多样性和构建核心种质的有效手段[49]。张馨方等[50]利用21对SSR标记通过聚类抽样分析, 比较使用3种遗传相似系数和2种取样方法相组合确定的不同样本群遗传多样性参数, 最终获得46份板栗核心种质, 占原始161份种质的28.57%。黄雨芹等[51]利用7个SSR位点采用随机取样策略、位点优先取样策略、等位基因数目最大化策略、遗传距离最大化策略4种取样方法构建闽楠核心种质, 并将各遗传多样性指标进行分析, 筛选出59份核心种质材料能够较好地代表闽楠种质资源的遗传多样性。艾叶等[52]利用16对荧光SSR引物进行扩增226个建兰品种, 基于等位基因最大法按照不同压缩比例逐步聚类, 形成备选种质, 确定压缩比例32.30%为构建核心种质的最佳比例, 包含73个品种。本研究利用30对SSR基于UPGMA聚类结果、对比遗传多样性参数、主坐标轴进行分析, 获得190份核心种质, 占原始500份种质的38%, 符合前人关于核心种质取样比例在5%~40%的结论[50]。
DNA分子身份证有利于糜子资源的合理开发和高效利用。本实验室构建糜子核心种质是基于前期开发的SSR标记。陈小红等[32]发现17对SSR 标记(RYW35、RYW40、RYW37、RYW18、RYW30、RYW16、RYW20、RYW19、RYW8、RYW5、RYW3、RYW7、RYW1、RYW14、RYW9、RYW6和RYW10)可区分4个生态区130份黍稷资源,值为1.0228、PIC值0.6209。王宇卓等[26]发现20对引物组合(RYW67、RYW53、RYW37、RYW65、RYW62、RYW77、RYW5、RYW49、RYW84、RYW19、RYW11、RYW40、RYW54、RYW28、RYW31、RYW7、RYW16、RYW8、RYW9和RYW18)可区分山西省272份材料,值为1.0552, PIC值为0.6921。本研究利用8对引物组合RYW67、RYW53、RYW62、RYW5、RYW40、RYW28、RYW8、RYW16区分了190份东北春糜子区核心种质, 同时发现了RYW5、RYW8、RYW16引物与陈小红等[32]一致, 说明东北春糜子区核心与陈小红等[32]构建的4个生态区的糜子分子身份证所用的SSR标记可能具有通用性; 8对SSR均与王宇卓等[24]一致, 说明东北春糜子区核心种质与山西省核心种质的分子身份证所用的SSR标记可能具有通用性。
4 结论
本研究构建了东北春播区糜子的核心种质190份, 用8个SSR构建上述材料DNA分子身份证。
附表和附图 请见网络版: 1) 本刊网站http://zwxb. chinacrops.org/; 2) 中国知网http://www.cnki.net/; 3) 万方数据http://c.wanfangdata.com.cn/Periodical- zuowxb.aspx。
[1] 何杰丽, 石甜甜, 陈凌, 王海岗, 高志军, 杨美红, 王瑞云, 乔治军. 糜子EST-SSR分子标记的开发及种质资源遗传多样性分析. 植物学报, 2019, 54: 723–732. He J L, Shi T T, Chen L, Wang H G, Gao Z J, Yang M H, Wang R Y, Qiao Z J. The genetic diversity of common millet () germplasm resources based on the EST-SSR markers., 2019, 54: 723–732 (in Chinese with English abstract).
[2] 薛延桃, 陆平, 史梦莎, 孙昊月, 刘敏轩, 王瑞云. 新疆、甘肃黍稷资源的遗传多样性与群体遗传结构研究. 作物学报, 2019, 45: 1511–1521. Xue Y T, Lu P, Shi M S, Sun H Y, Liu M X, Wang R Y. Genetic diversity and population genetic structure of broomcorn millet accessions in Xinjiang and Gansu., 2019, 45: 1511–1521 (in Chinese with English abstract).
[3] 王瑞云, 季煦, 陆平, 刘敏轩, 许月, 王纶, 王海岗, 乔治军. 利用荧光SSR分析中国糜子遗传多样性. 作物学报, 2017, 43: 530–548. Wang R Y, Ji X, Lu P, Liu M X, Xu Y, Wang L, Wang H G, Qiao Z J. Analysis of genetic diversity in common millet () using fluorescent SSR in China., 2017, 43: 530–548 (in Chinese with English abstract).
[4] 邵欢欢, 陆平, 史梦莎, 王璐琳, 刘敏轩, 王瑞云. 黍稷芽、苗期抗旱性评价及抗旱资源鉴定. 分子植物育种, 2021, 19: 1284–1296. Shao H H, Lu P, Shi M S, Wang L L, Liu M X, Wang R Y. Evaluation and identification of broomcorn millet resources for drought resistance at germination and seedling stages., 2021, 19: 1284–1296 (in Chinese with English abstract).
[5] 薛延桃, 陆平, 乔治军, 刘敏轩, 王瑞云. 基于SSR标记的黍稷种质资源遗传多样性及亲缘关系研究. 中国农业科学, 2018, 51: 2846–2859. Xue Y T, Lu P, Qiao Z J, Liu M X, Wang R Y. Genetic diversity and genetic relationship of broomcorn millet (L.) germplasm based on SSR markers., 2018, 51: 2846–2859 (in Chinese with English abstract).
[6] 王瑞云. 糜子遗传多样性及进化研究进展. 北京: 中国农业出版社, 2017. pp 20–92. Wang R Y. Research Progress on Genetic Diversity and Evolution of Proso Millet. Beijing: China Agriculture Press, 2017. pp 20–92 (in Chinese).
[7] 柴岩. 糜子. 北京: 中国农业出版社, 1999. pp 225–229. Chai Y. Broomcorn Millet. Beijing: China Agriculture Press, 1999. pp 225–229 (in Chinese).
[8] Wang R Y, Hunt H V, Qiao Z J, Wang L, Han Y H. Diversity and cultivation of broomcorn millet (L.) in China: a review., 2016, 70: 332–342.
[9] Xu Y, Liu M X, Li C X, Sun F J, Lu P, Meng F S, Zhao X Y, He M Y, Wang F Z, Zhu X Y, Zhao X, Zhou H. Domestication and spread of broomcorn millet (L.) revealed by phylogeography of cultivated and weedy populations., 2019, 9: 835.
[10] 孟繁霜. 糜子遗传多样性与栽培起源研究. 吉林大学硕士学位论文, 吉林长春, 2018. Meng F S. Research on the Influence of Electroplating Process on Studies on Population Genetics and Domestication ofMS Thesis of Jilin University, Changchun, Jilin, China, 2018 (in Chinese with English abstract).
[11] Boukail S, Macharia M, Miculan M, Masoni A, Calamai A, Palchetti E, Dell’Acqua M. Genome wide association study of agronomic and seed traits in a world collection of proso millet (L.)., 2021, 21: 330.
[12] Kalinova J, Moudry J. Content and quality of protein in proso millet (L.) varieties., 2006, 61: 43–47.
[13] Li K, Zhang T Z, Narayanamoorthy S, Jin C, Sui Z, Li Z, Li S, Wu K, Liu G, Corke H. Diversity analysis of starch physicochemical properties in 95 proso millet (L.) accessions., 2020, 324: 126863.
[14] 陈昌文, 曹珂, 王力荣, 朱更瑞, 方伟超. 中国桃主要品种资源及其野生近缘种的分子身份证构建. 中国农业科学, 2011, 44: 2081–2093. Chen C W, Cao K, Wang L R, Zhu G R, Fang W C. Molecular ID establishment of main China peach varieties and peach related species., 2011, 44: 2081–2093 (in Chinese with English abstract).
[15] 连帅, 陆平, 乔治军, 张琦, 张茜, 刘敏轩, 王瑞云. 利用SSR分子标记研究国内外黍稷地方品种和野生资源的遗传多样性. 中国农业科学, 2016, 49: 3264–3275. Lian S, Lu P, Qiao Z J, Zhang Q, Zhang Q, Liu M X, Wang R Y. Genetic diversity in broomcorn millet (L.) from China and abroad by using SSR markers., 2016, 49: 3264–3275 (in Chinese with English abstract).
[16] 王倩, 张立媛, 许月, 李海, 刘少雄, 薛亚鹏, 陆平, 王瑞云, 刘敏轩. 黍稷高基元EST-SSR标记开发及200份核心种质资源遗传多样性分析. 作物学报, 2023, 49: 2308–2318. Wang Q, Zhang L Y, Xu Y, Li H, Liu S X, Xue Y P, Lu P, Wang R Y, Liu M X. High motif EST-SSR markers development and genetic diversity evaluation for 200 core germplasms in proso millet., 2023, 49: 2308–2318 (in Chinese with English abstract).
[17] 赵久然, 王凤格, 郭景伦, 陈刚, 廖琴, 孙世贤, 陈如明, 刘龙洲. 中国玉米新品种DNA指纹库建立系列研究: II. 适于玉米自交系和杂交种指纹图谱绘制的SSR核心引物的确定. 玉米科学, 2003, 11(2): 3–5. Zhao J R, Wang F G, Guo J L, Chen G, Liao Q, Sun S X, Chen R M, Liu L Z. Series of research on establishing DNA fingerprinting pool of Chinese new maize cultivars: II. Confirmation of a set of SSR core primer pairs., 2003, 11(2): 3–5 (in Chinese with English abstract).
[18] 董文堂, 邓昌蓉, 侯全刚, 李江, 邵登魁. 辣椒种质资源遗传多样性分析及核心种质构建研究进展. 青海农林科技, 2023, 53(3): 46–49. Dong W T, Deng C R, Hou Q G, Li J, Shao D K. Research progress on genetic diversity analysis and core collection construction of pepper germplasm resources., 2023, 53(3): 46–49 (in Chinese with English abstract).
[19] Frankel O H. Genetic Manipulation: Impact on Man and Society. Cambridge University Press, 1984. pp 161–170.
[20] Brown A H D. Core collections: a practical approach to genetic resource management., 1989, 31: 818–824.
[21] Zhang H L, Zhang D L, Wang M X, Sun J L, Qi Y W, Li J J, Wei X H, Han L Z, Qiu Z E, Tang S X, Li Z C. A core collection and mini core collection ofL. in China., 2011, 122: 49–61.
[22] Hintum T J L. Comparison of marker systems and construction of a core collection in a pedigree of European spring barley., 1995, 89: 991–997.
[23] Upadhyaya H D, Otiz R, Singh S. Development of a groundnut core collection using taxonomical, geographical and morphological descriptors., 2003, 50: 139–148.
[24] Wang L X, Guan Y, Guan R X, Li Y H, Ma Y S, Dong Z M, Liu X, Zhang H Y, Zhang Y Q, Liu Z X, Chang R Z, Xu H M, Li L H, Lin F Y, Luan W J, Yan Z, Ning X C, Zhu L, Cui Y H, Piao R H, Liu Y, Chen P Y, Qiu L J. Establishment of Chinese soybean () core collections with agronomic traits and SSR markers., 2006, 151: 215–223.
[25] 姜俊烨, 杨涛, 王芳, 方俐, 仲伟文, 关建平, 宗绪晓. 国内外蚕豆核心种质SSR遗传多样性对比微核心种质构建. 作物学报, 2014, 40: 1311–1319. Jiang J Y, Yang T, Wang F, Fang L, Zhong W W, Guan J P, Zong X X. Genetic diversity analysis of germplasm resources and construction of mini-core collections forLat home and abroad., 2014, 40: 1311–1319 (in Chinese with English abstract).
[26] 王宇卓, 林元香, 薛亚鹏, 段政勇, 王晓丹, 陈凌, 曹晓宁, 王瑞云, 乔治军. 山西糜子核心种质分子身份证构建. 植物学报, 2023, 58: 159–168. Wang Y Z, Lin Y X, Xue Y P, Duan Z Y, Wang X D, Chen L, Cao X N, Wang R Y, Qiao Z J. Construction of molecular ID card of core germplasm of hog millet () in Shanxi., 2023, 58: 159–168 (in Chinese with English abstract).
[27] 王建成, 胡晋, 黄歆贤, 徐盛春. 植物遗传资源核心种质新概念与应用进展. 种子, 2008, 27(5): 47–50. Wang J C, Hu J, Huang X X, Xu S C. New concept and application of plant genetic resources., 2008, 27(5): 47–50 (in Chinese with English abstract).
[28] 陈伊航, 唐朝臣, 张雄坚, 姚祝芳, 江炳志, 王章英. 基于表型性状和SSR分子标记构建甘薯核心种质. 作物学报, 2023, 49: 1249–1261. Chen Y H, Tang Z C, Zhang X J, Yao Z F, Jiang B Z, Wang Z Y. Construction of core collection of sweet potato based on phenotypic traits and SSR markers., 2023, 49: 1249–1261 (in Chinese with English abstract).
[29] 汪磊, 王姣梅, 汪魏, 王玲, 王力军, 严兴初, 谭美莲. 基于表型多样性构建向日葵核心种质. 中国油料作物学报, 2021, 43: 1052–1060. Wang L, Wang J M, Wang W, Wang L, Wang L J, Yan X C, Tan M L. Development of a core collection in sunflower (L.) germplasm using phenotypic diversity., 2021, 43: 1052–1060 (in Chinese with English abstract).
[30] 刘松, 聂兴华, 李伊然, 刘海涛, 张卿, 王雪峰, 田寿乐, 曹庆芹, 秦岭, 邢宇. 基于SSR荧光标记构建板栗品种(系)核心种质群. 果树学报, 2023, 40: 230–241. Liu S, Nie X H, Li Y R, Liu H T, Zhang Q, Wang X F, Tian S L, Cao Q Q, Qin L, Xing Y. Construction of core germplasm collection of Chinese chestnut cultivars (lines) based on SSR fluorescence markers., 2023, 40: 230–241 (in Chinese with English abstract).
[31] 孙永强, 陈建华, 张剑, 董胜君, 刘权钢, 刘青柏. 基于表型性状的西伯利亚杏核心种质构建. 沈阳农业大学学报, 2022, 53(1): 43–54. Sun Y Q, Chen J H, Zhang J, Dong S J, Liu Q G, Liu Q B. Construction a core collection ofbased on phenotypic traits., 2022, 53(1): 43–54 (in Chinese with English abstract).
[32] 陈小红, 林元香, 王倩, 丁敏, 王海岗, 陈凌, 高志军, 王瑞云,乔治军. 基于高基元SSR构建黍稷种质资源的分子身份证. 作物学报, 2022, 48: 908–919. Chen X H, Lin Y X, Wang Q, Ding M, Wang H G, Chen L, Gao Z J, Wang R Y, Qiao Z J. Development of DNA molecular ID card in hog millet germplasm based on high motif SSR., 2022, 48: 908–919 (in Chinese with English abstract).
[33] 秦瑞英, 许学, 张立平, 李莉, 方钰, 汪秀峰, 倪金龙, 陆徐忠,杨剑波. 小麦SSR指纹图谱及品种身份证的构建: 基于毛细管电泳分析. 中国农学通报, 2017, 33(34): 46–55. Qin R Y, Xu X, Zhang L P, Li L, Fang Y, Wang X F, Ni J L, Lu X Z, Yang J B. Construction of wheat variety SSR fingerprint and ID: based on capillary electrophoresis., 2017, 33(34): 46–55 (in Chinese with English abstract).
[34] 李清, 罗永坚, 吴柔贤, 贾俊婷, 张文虎, 宋松泉, 刘军. 广东省大豆种质资源遗传多样性分析及DNA分子身份证构建. 广东农业科学, 2020, 47(12): 221–228. Li Q, Luo Y J, Wu R X, Jia J T, Zhang W H, Song S Q, Liu J. Analysis on genetic diversity and construction of DNA molecular identity card of soybean germplasm resources in Guangdong province., 2020, 47(12): 221–228 (in Chinese with English abstract).
[35] 侯丽媛, 董艳辉, 邓舒, 肖蓉, 张春芬, 赵菁, 曹秋芬. 部分苹果属种质遗传多样性分析及分子身份证构建. 山西农业科学, 2020, 48: 1171–1179. Hou L Y, Dong Y H, Deng S, Xiao R, Zhang C F, Zhao J, Cao Q F. Study on genetic diversity and construction of molecular identity card for someMill. germplasm resource., 2020, 48: 1171–1179 (in Chinese with English abstract).
[36] 刘冠群, 吴祠平, 谭礼强, 谭杰, 杨婉君, 唐茜. 利用SSR分子标记构建名山茶树基因身份证. 四川农业大学学报, 2019, 37: 469–474. Liu G Q, Wu C P, Tan L Q, Tan J, Yang W J, Tang Q. Construction of SSR-based molecular IDs for tea planted in Mingshan., 2019, 37: 469–474 (in Chinese with English abstract).
[37] 王宇晴, 李乔乔, 阚文亮, 邳植, 吴则东. 利用SSR分子标记构建甜菜登记品种的分子身份证. 江苏农业科学, 2022, 50(18): 289–294. Wang Y Q, Li Q Q, Kan W L, Pi Z, Wu Z D. Molecular identity cards of sugar beet registration varieties were constructed using SSR molecular markers., 2022, 50(18): 289–294 (in Chinese with English abstract).
[38] 高源, 刘凤之, 王昆, 王大江, 龚欣, 刘立军. 苹果部分种质资源分子身份证的构建. 中国农业科学, 2015, 48: 3887–3898. Gao Y, Liu F Z, Wang K, Wang D J, Gong X, Liu L J. Establishment of molecular ID for some apple germplasm resources., 2015, 48: 3887–3898 (in Chinese with English abstract).
[39] 高运来, 朱荣胜, 刘春燕, 李文福, 蒋洪蔚, 李灿东, 姚丙晨, 胡国华, 陈庆山. 黑龙江部分大豆品种分子ID的构建. 作物学报, 2009, 35: 211–218. Gao Y L, Zhu R S, Liu C Y, Li W F, Jiang H W, Li C D, Yao B C, Hu G H, Chen Q S. Establishment of molecular ID in soybean varieties in Heilongjiang, China., 2009, 35: 211–218 (in Chinese with English abstract).
[40] 马琳, 刘海珍, 陆徐忠, 倪金龙, 张晓娟, 杨剑波. 130份甘蓝型油菜种质分子身份证的构建. 中国油料作物学报, 2013, 35: 231–239. Ma L, Liu H Z, Lu X Z, Ni J L, Zhang X J, Yang J B. Molecular identity of 130varieties., 2013, 35: 231–239 (in Chinese with English abstract).
[41] 薛亚鹏, 丁艺冰, 王宇卓, 王晓丹, 曹晓宁, Santra Dipak K, 陈凌, 乔治军, 王瑞云. 基于荧光SSR构建中国糜子核心种质DNA分子身份证. 中国农业科学, 2023, 56: 2249–2261. Xue Y P, Ding Y B, Wang Y Z, Wang X D, Cao X N, Santra Dipak K, Chen L, Qiao Z J, Wang R Y. Construction of DNA molecular identity card of core germplasm of broomcorn millet in China based on fluorescence SSR., 2023, 56: 2249–2261 (in Chinese with English abstract).
[42] Murray M G, Thompson W F. Rapid isolation of high molecular weight plant DNA., 1980, 8: 4321–4325.
[43] Yeh F C, Boyle T J. Population genetic analysis of co-dominant and dominant markers and quantitative traits., 1997, 129: 157.
[44] Liu K, Muse S V. PowerMarker: an integrated analysis environment for genetic marker data., 2005, 21: 2128–2129.
[45] Tamura K, Stecher G, Kumar S. MEGA11: mevolutionary genetics analysis version 11., 2021, 38: 3022–3027.
[46] 胡振帮, 高运来, 齐照明, 蒋洪蔚, 刘春燕, 辛大伟, 胡国华, 潘校成, 陈庆山. 作物分子身份证构建软件ID analysis的编制.中国农业科学, 2016, 49: 2255–2266.Hu Z B, Gao Y L, Qi Z M, Jiang H W, Liu C Y, Xin D W, Hu G H, Pan X C, Chen Q S. Software development of ID analysis for crop molecular identity construction., 2016, 49: 2255–2266 (in Chinese with English abstract).
[47] Essid A, Aljane F, Ferchichi A, Hormaza J I. Analysis of genetic diversity of Tunisian caprifig (L.) accession using simple sequence repeat (SSR) markers., 2015, 152: 1.
[48] 何瑞超. 绿豆遗传多样性研究及种子萌发期耐盐性评价. 内蒙古农业大学硕士学位论文, 内蒙古呼和浩特, 2021.He R C. Study on Genetic Diversity of Mung Bean and Evaluation of Salt Tolerance During Seed Germination. MS Thesis of Inner Mongolia Agricultural University, Hohhot, Inner Mongolia, China, 2021 (in Chinese with English abstract).
[49] 马庆国, 宋晓波, 贺君星, 周晔, 黄勇, 张俊佩, 裴东. 基于SSR分子标记的核桃种质资源分子身份证构建. 植物资源与环境学报, 2023, 32(2): 1–9. Ma Q G, Song X B, He J X, Zhou Y, Huang Y, Zhang J P, Pei D. Establishment of molecular identity cards of walnut (spp.) germplasm resources based on SSR molecular marker., 2023, 32(2): 1–9 (in Chinese with English abstract).
[50] 张馨方, 张树航, 李颖, 郭燕, 王广鹏. 基于SSR标记构建燕山板栗核心种质. 华北农学报, 2021, 36(增刊1): 31–38. Zhang X F, Zhang S H, Li Y, Guo Y, Wang G P. Construction of core collection of Yanshan chestnut germplasm based on SSR markers., 2021, 36(S1): 31–38 (in Chinese with English abstract).
[51] 黄雨芹, 尹光天, 杨锦昌, 余纽, 邹文涛, 李荣生. 基于SSR分子标记的闽楠核心种质的构建. 分子植物育种, 2020, 18: 2641–2648. Huang Y Q, Yin G T, Yang J C, Yu N, Zou W T, Li R S. Developing a mini core germplasm ofbased on SSR molecular marker., 2020, 18: 2641–2648 (in Chinese with English abstract).
[52] 艾叶, 陈璐, 谢泰祥, 陈娟, 兰思仁, 彭东辉. 基于SSR荧光标记构建建兰品种核心种质. 园艺学报, 2019, 46: 1999–2008. Ai Y, Chen L, Xie T X, Chen J, Lan S R, Peng D H. Construction of core collection ofcultivars based on SSR fluorescent markers., 2019, 46: 1999–2008 (in Chinese with English abstract).
Core germplasm and DNA molecular identity card of proso millet in Northeast Spring sowing region in China
DING Yi-Bing1,2, XIN Xu-Xia2, FENG Zhi-Zun2, CAO Yue2, GUO Juan2, Dipak K SANTRA3, WANG Rui-Yun2,*, and CHEN Xi-Ming1,4,*
1Corn Research Institute, Shanxi Agricultural University, Xinzhou 034000, Shanxi, China;2College of Agronomy, Shanxi Agricultural University, Taigu 030801, Shanxi, China;3Panhandle Research and Extension Center, Department of Agronomy and Horticulture, University of Nebraska- Lincoln, Scottsbluff 69361, Nebraska, USA;4Shanxi Key Laboratory of Minor Crops Germplasm Innovation and Molecular Breeding, Shanxi Agricultural University, Taiyuan 030000, Shanxi, China
In this study, 500 proso millet resources in Northeast Spring Sowing Region were used as the experimental materials, 169 SSR markers, UPGMA clustering, and stratified sampling were used to construct core germplasm, and ID Analysis 4.0 software was used to construct molecular identity card. The genetic diversity of the core collection was evaluated by genetic diversity metrics such as allele number (a), and the core collection was analyzed by PCOA. The results showed that 169 pairs of SSR primers were screened, and 30 pairs of SSR primers were found to have good polymorphism. The core collection of proso millet constructed by 30 pairs of SSR primers contained 190 materials, accounting for 38% of all germplasm. Ninety-one alleles were detected in all germplasm and core collection, and 100% alleles were retained. The number of effective alleles was 2.2977–2.9975 and 2.2872–3.0173, with an average of 2.8198 and 2.8297, respectively. The Shannon diversity index was 0.9532–1.0990 and 0.9535–1.1162, with an average of 1.0645 and 1.0667. The observed heterozygosity was 0.3434–0.8037 and 0.3162–0.7849, with an average of 0.5399 and 0.5359. The expected heterozygosity was 0.5654–0.6672 and 0.5645–0.6707, with an average of 0.6448 and 0.6473. Nei’s gene diversity index was 0.5648–0.6664 and 0.5628–0.6686, with an average of 0.6441 and 0.6452. The polymorphism information content was 0.6657–0.8356 and 0.6493–0.8340, with an average of 0.7974 and 0.7944. The results of-test showed that there was no significant difference in the related indexes of molecular markers between all germplasm and core germplasm, and PCOA analysis showed that the core germplasm and all germplasm had similar genetic diversity and population structure. At the same time, 8 SSR markers (RYW5, RYW8, RYW16, RYW28, RYW40, RYW53, RYW62, and RYW67) were found to distinguish 190 core germplasms, and the molecular identity card of the core germplasms of Northeast proso millet was constructed, which providing a scientific basis for the efficient utilization and rapid traceability of proso millet germplasm.
; the Northeast Spring sowing region; SSR; core collection; DNA molecular ID card
10.3724/SP.J.1006.2024.34153
本研究由山西农业大学杂粮种质创新与分子育种国家实验室(筹) (202204010910001)项目, 国家自然科学基金项目(31271791), 财政部和农业农村部国家现代农业产业技术体系建设专项(CARS-06-14.5-A16), 山西省现代农业产业技术体系建设专项资金项目(2023CYJSTX03-12), 山西省重点研发项目(2022ZDYF110), 山西农业大学杂粮种质创新与分子育种山西省重点实验室项目(K462202040), 山西农业大学生物育种工程项目(YZGC069), 山西农业大学农学院研究生教育改革项目(2023YJG05)和山西省科技成果转化引导专项(201904D131056)资助。
This study was supported by the National Laboratory of Minor Crops Germplasm Innovation and Molecular Breeding (in Prepare, 202204010910001), the National Natural Science Foundation of China (31271791), the China Agriculture Research System of MOF and MARA (CARS-06-14.5- A16), the Special Fund for Construction of Modern Agricultural Industrial Technology System of Shanxi Province (2023CYJSTX03-12), the Key Research & Development Project of Shanxi Province (2022ZDYF110), the Shanxi Key Laboratory of Innovation and Molecular Breeding, Shanxi Agricultural University (K462202040), the Biological Breeding Project of Shanxi Agricultural University (YZGC069) , and the Graduate Education Reform Project of Agronomy College in Shanxi Agricultural University(2023YJG05), and the Shanxi Province Scientific and Technological Achievements Transformation and Guidance Project (201904D131056) .
陈喜明, E-mail: 516834898@qq.com; 王瑞云, E-mail: wry925@126.com
E-mail: 2771284358@qq.com
2023-09-12;
2024-01-12;
2024-01-31.
URL: https://link.cnki.net/urlid/11.1809.S.20240130.1132.002
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).
