玉米多叶矮化突变体lyd1的鉴定与基因克隆
2024-04-28刘孝伟牛群凯时子文侯雨微冯开洁荣廷昭曹墨菊
苏 帅 刘孝伟 牛群凯 时子文 侯雨微 冯开洁 荣廷昭 曹墨菊
玉米多叶矮化突变体的鉴定与基因克隆
苏 帅 刘孝伟 牛群凯 时子文 侯雨微 冯开洁 荣廷昭 曹墨菊*
四川农业大学玉米研究所 / 农业部西南玉米生物学与遗传育种重点实验室, 四川成都 611130
玉米株高降低通常是由节间数目减少、节间长度变短或二者共同作用所致。而本研究在基因编辑后代中发现的玉米多叶矮化突变体却表现为节间数目显著增加, 株高显著降低。株高仅为93.10 cm, 与野生型KN5585的株高159.95 cm相比, 降低了41.79%, 差异达到极显著水平。然而叶片数平均达到27.8片, 相较野生型平均17.8片叶, 增加56.18%, 差异达到极显著水平。遗传分析表明,的突变表型由1对隐性核基因控制, 通过图位克隆将控制多叶矮化性状的基因定位于玉米3号染色体标记Indel10和Indel11之间, 物理距离0.74 Mb。对定位区间内13个基因(不包含假基因)的序列进行测序, 发现仅在第4外显子出现1个A碱基的替换, 其他基因无差异。编码一个RNA结合蛋白, 氨基酸的替换发生在第3个RNA结合结构域内(RRM3), 导致天冬氨酸转变为缬氨酸。突变体的突变位点与已报道的、、、不同,的发现为进一步解析玉米叶片和节间发育平衡的遗传机制提供了宝贵的材料。
玉米; 叶片数量; 节间长度; 基因定位
玉米(L.)是我国粮食和饲料作物, 在保障国家粮食安全方面发挥重要作用。现阶段玉米产量的增长主要通过提升种植密度, 适当降低株高以增强抗倒伏能力是提高种植密度的关键[1-2]。玉米节间数量(叶片数量)和节间长度是决定株高的关键因素, 因此对于玉米叶片数量和节间长度以及协调二者平衡相关基因的挖掘具有重要的理论和实践意义。
玉米节间长度由节间居间分生组织细胞的分裂、分化和伸长决定。植物激素作为重要的信号分子, 参与玉米节间长度的调控。[3]、[4-5][6]编码赤霉素合成途径的中间产物,[7-9]、[9]编码DELLA蛋白作为GA信号转导的负调控转录因子, 通过调控赤霉素的生物合成或信号转导影响玉米节间长度。生长素以极性运输的方式对节间长度产生影响,编码生长素极性运输蛋白PGP, 通过促进节间生长素的积累抑制节间的伸长, 且对穗下部节间长度影响较大[10-11]。[12]、[13]位于油菜素内酯生物合成的上游,[14]位于油菜素内酯生物合成的下游,参与油菜素内酯信号转导途径[15], 上述基因突变后均会对玉米节间长度产生影响。编码乙烯生物合成中的关键限速酶ACC合成酶, 通过增强蛋白的稳定性, 促进乙烯合成, 抑制节间伸长[16]。玉米叶片由茎顶端分生组织的周缘区以一定的时间间隔和空间间隔分化形成[17], 但具体的分子机制尚未解析。迄今已经报道的叶片数量相关突变体主要与生长素和细胞分裂素有关。玉米基因编码IAA生物合成途径中的色氨酸转移酶,突变体展示出叶片数目显著减少而导致的矮化表型[18]。编码细胞分裂素诱导的A型反应调节因子, 玉米突变体叶片数增多, 但对株高没有显著影响。在突变体中, 玉米IAA外排载体在初始叶原基的表达量大幅下降, 其生长素水平也明显降低[19]。
RNA结合蛋白是含有一个或多个RNA结合结构域, 通过结合RNA并改变结合RNA的命运或功能的蛋白质[20-21]。RNA结合蛋白的研究主要在哺乳动物和细菌系统中进行, 植物RNA结合蛋白研究较少。是在酵母中发现的一个编码RNA结合蛋白的基因, 含有3个RNA结合结构域, 促进酵母细胞减数分裂S期DNA的合成和减数第一次分裂的有序进行[22]。水稻基因编码Mei2-like蛋白, 该基因突变后植株矮小, 叶片数增加, 植株早熟[23]。是在玉米中的同源基因, 该基因在叶原基起始处表达量最低, 其突变体株高降低, 叶片数目增多, 叶脉紊乱, 植株雄穗缺失, 原雄穗发育处展示出雌性化的特征[33-34]。尽管在植物中很多基因已经被发现, 且均与酵母基因类似, 含有3个RNA结合结构域, 但在植物中未找到酵母下游基因的同源物, 表明该基因在酵母和植物中的功能和作用机制并不相同[24]。在植物中基因下游结合的RNA的挖掘, 以及3个RNA结合结构域在发挥功能过程中的作用仍需要进一步深入研究。
本研究以多叶矮化突变体()为研究对象, 对突变体的叶片数和节间长度等表型特征进行了系统调查, 采用分子标记技术对突变基因进行了定位, 并对候选基因进行预测和分析, 确定了关键候选基因, 该基因编码RNA结合蛋白。本研究为进一步阐明玉米叶片数量和节间长度的平衡关系以及RNA结合蛋白对玉米生长发育的影响提供了宝贵材料。
1 材料与方法
1.1 试验材料
玉米多叶矮化突变体、玉米自交系B73、Mo17和KN5585。突变体选自基因()的CRISPR/Cas9敲除后代, 对突变体中的进行测序, 结果显示突变体中未发生编辑, 即该突变体的突变表型与无关。
1.2 突变体lyd1的表型鉴定和遗传分析
2020年6月在四川农业大学温江试验基地发现该多叶矮化突变体, 将其自交保种。2020年12月在四川农业大学云南试验基地种植、Mo17和B73, 并将分别与Mo17和B73杂交得到F1。2021年3月在四川农业大学温江试验基地种植F1, 自交获得F2种子。2022年3月在四川农业大学温江试验基地种植F2群体。成熟期统计F2分离群体中正常和多叶矮化植株的分离比并进行卡平方测验。
2021年3月将野生型KN5585和突变体种植于四川农业大学温江试验基地, 野生型KN5585二叶期后, 每间隔3 d, 统计野生型KN5585和突变体各30株完全展开叶数量。在植株成熟期, 田间随机选取野生型KN5585和各15株, 考察株高、穗位、叶片长度、叶片宽度、雄穗分支数、地上部分各节间长度和散粉期等重要农艺性状。使用Microsoft Excel 2016进行数据处理和绘图。
1.3 茎秆细胞学观察
植株散粉后, 选取野生型KN5585和地上部分最长节间(均为雌穗下方第2节, 野生型KN5585为第11节, 突变晰完体为第19节)的中部, 纵切并将其固定于FAA固定液中。将样品依次进行软化、洗涤、脱水、透明、包埋、切片、烤片、染色, 在显微镜下进行节间细胞观察, 野生型KN5585和突变体分别选取细胞形态清整的3张切片, 共10个视野进行照相。通过ImageJ软件进行细胞长度测量, 并根据实际测量得出的最长节间平均长度进行节间细胞数目的计算, 使用Microsoft Excel 2016进行数据处理和绘图。
1.4 基因定位
利用CTAB法[25]提取玉米自交系Mo17、KN5585、突变体以及[×Mo17] F2分离群体内多叶矮化单株的基因组DNA。利用均匀分布在玉米10条染色体上1050对Indel和SSR标记, 对Mo17和进行多态性分析。在[×Mo17] F2群体中, 选取10株多叶矮化单株提取叶片的DNA, 稀释成相同浓度并等体积混合, 每5株混合为一个池, 构建2个突变池。同时, 另外选取10株表型正常的单株提取叶片的DNA, 稀释成相同浓度并等体积混合为一个正常池。采用BSA (bulk segregant analysis)方法[26]筛选在正常池和突变池之间具有稳定性差异的多态性标记, 并利用上述标记对F2群体内多叶矮化单株进行基因分型, 筛选与突变表型连锁的多态性标记, 对目标基因进行初步定位。利用NCBI在线引物设计网站根据初步定位区段内序列信息, 设计产物片段600 bp左右的引物, 通过PCR扩增出亲本基因片段后进行测序, 利用DNAMAN软件对所扩增的双亲基因序列进行比对分析, 找出双亲间的序列差异位点, 根据差异位点开发片段大小为150~300 bp的Indel标记。利用新开发的标记, 对扩大的定位群体内多叶矮化单株进行基因分型, 缩小定位区间, 对目的基因进行精细定位。
1.5 候选基因的预测与分析
在MaizeGDB和NCBI网站上, 获取精细定位区间内基因的注释信息。根据精细定位区间内基因序列和注释, 设计基因特异引物, 以野生型和基因组DNA或全长CDS为模板, 利用KOD高保真DNA聚合酶进行PCR扩增。扩增体系为30 µL: 2×PCR buffer for KOD FX 15 µL、正向和反向引物(10 µmol L–1)各2 µL、KOD FX 0.6 µL、dNTPs (0.002 mol L–1) 6 µL、ddH2O 3.4 µL、模板3 µL, PCR程序为: 95℃预变性5 min; 98℃变性10 s, 58℃退火30 s, 68℃延伸(1 kb min–1), 循环数为35次; 68℃ 10 min。PCR扩增产物送公司进行测序并利用DNAMAN软件进行序列比对, 确定变异位点。
以定位群体内关键交换单株DNA为模板, 根据变异位点附近序列设计特异性引物, 对变异位点所在的基因片段进行扩增, 长度约600 bp, 变异位点位于扩增片段中间, 并将PCR扩增产物送公司进行测序, 利用DNAMAN软件进行序列比对, 进行变异位点群体内共分离验证。
1.6 关键候选基因的表达及其蛋白的亚细胞定位
为了检测在不同时期不同组织的表达模式, 利用TRIzol试剂盒提取野生型KN5585在V3、V7、V11、V17时期的根、茎、叶、雌穗、雄穗的RNA; 同时为了检测突变体中基因表达水平变化, 利用TRIzol试剂盒提取野生型KN5585和的V3和V7时期茎尖的RNA, 电泳检测RNA的完整性, 并将质量符合标准的RNA反转录得到cDNA, 根据玉米公共数据库MaizeGDB中提供的候选基因cDNA信息, 使用Primer 3在线设计特异性引物, 引物扩增片段长度在100~200 bp左右。cDNA稀释至100 ng μL–1作为模板。以作为内参。qRT-PCR反应在CFX 96 Real-Time System上进行。qRT-PCR反应体系为10 μL: Diluted cDNA 1 μL, 2×SYBR Green PCR Mix 5 μL, 上游引物(10 μmol L–1) 0.4 μL, 下游引物(10 μmol L–1) 0.4 μL, ddH2O 3.2 μL。反应程序为: 95℃预变性3 min; 95℃变性10 s, 58℃退火30 s, 72℃延伸15 s, 40个循环, 3次生物重复, 3次技术重复。扩增结束后产生的数据导入Microsoft Excel中, 采用2–DDCt方法进行基因相对表达量的分析。
为了检测蛋白在细胞内的定位模式, 设计带有PCAMBIA2300-eGFP载体同源臂的引物扩增全长转录本。利用同源重组的方法, 将pCAMBIA2300-35S-eGFP载体和目的片段连接, 并转入大肠杆菌感受态, 挑选阳性克隆检测并测序比对, 挑选正确序列的单克隆扩繁, 提取质粒。将质粒转化GV3101农杆菌, 200转 min–128℃过夜扩繁至OD600为0.6左右, 然后5000转 min–1离心5 min收集菌体, 弃上清, 加入适量的注射液。用已灭菌的1 mL注射器将菌体悬浮液注射至4~8周左右的烟草叶背部表皮细胞层中, 然后在正常条件下培养48 h, 用荧光显微观察并采集图像。
2 结果与分析
2.1 突变体的表型鉴定及遗传分析
为了明确突变体的遗传规律, 将自交后代于每年的春季在四川、秋季在云南进行多年种植观察,的突变表型不受环境影响, 可以稳定遗传。将分别与Mo17和B73杂交, F1代植株均表现正常表型, 将F1自交获得F2, 在5380株[×Mo17] F2群体中, 正常植株数量为4083株, 多叶矮化株数量为1297株, 经卡平方检测, 正常植株与多叶矮化株分离比符合3∶1 (c2C=2.24, 小于c20.05,1=3.84); 在286株[×B73] F2群体中, 正常植株数量为220株,多叶矮化株数量为66株, 经卡平方检测, 正常植株与多叶矮化株分离比符合3∶1 (c2C=0.46, 小于c20.05,1=3.84)。不同遗传背景下的F2群体, 正常植株与多叶矮化植株分离比均符合3∶1, 表明该突变表型是由单个隐性核基因控制。
对突变体的农艺性状进行调查发现,株高93.10 cm、穗位高43.48 cm, 仅为野生型株高159.95 cm、穗位高64.37 cm的58.21%和67.55% (图1-A~C), 进一步分析表明穗上部和穗下部节间缩短是造成株高降低的原因(图2-A)。生育期内完全叶数量平均为27.80±1.18片, 野生型生育期内完全叶数量平均为(17.80±0.95)片(图2-B), 差异达到极显著水平。野生型从播种至群体内50%植株散粉需要(89.03±2.10) d,从播种至群体内50%植株散粉需要(88.64±1.79) d, 二者无显著差异(表1)。综合叶片数量及生育期的比较结果可以发现,相邻2片叶片完全展开的时间间隔为2.31 d, 野生型相邻2片叶片完全展开的时间间隔为3.50 d, 两者差异达到显著水平,相邻叶原基起始所用时间间隔缩短, 叶片起始速率加快。此外叶片长度和宽度较野生型显著缩小(图1-D), 穗长、穗粗、行粒数、穗粒重、百粒重均显著下降(图1-F, G), 雄穗分支数显著减少, 雄穗基部由未完全伸展的叶片包裹(图1-E和表1), 这可能也暗示了雄穗基部节间伸长受阻。

图1 野生型KN5585和突变体lyd1的性状比较
A、B、C: 分别表示五叶期、十叶期和成熟期的野生型KN5585和植株; D: 野生型KN5585和成熟期的叶片表型; E: 野生型KN5585和突变体的雄穗表型; F、G: 野生型KN5585和的果穗和籽粒的表型。标尺: A 15 cm; B、C、D 30 cm; E 5 cm; F 2 cm。
A, B, and C: represent the 5th leaf stage, 10th leaf stage, and mature stage of wild type KN5585 and mutant, respectively. D: leaves phenotypes of wild type KN5585 and mutantat maturity. E: the tassel phenotype of wild type KN5585 and mutant. F, G: phenotypes of ear and kernel of wild types KN5585 and mutant. Bar: A 15 cm; B, C, D 30 cm; E 5 cm; F 2 cm.

图2 野生型KN5585和突变体lyd1的节间、叶片发育的比较
A: 野生型KN5585和地上部分节间长度的比较, 0表示雌穗所在节间、–1表示雌穗下方第一节、+1表示雌穗上方第一节, 依此类推; B: 野生型KN5585和叶片发育过程比较。纵坐标表示调查群体内植株叶片的平均数, 横坐标表示调查日期。
A: the comparison of internodes length between the wild type KN5585 and mutant, 0 represents the internode of the ear, –1 represents the first internode below the ear, +1 represents the first internode above the ear, and so on. B: the comparison of leaves development between wild type KN5585 and mutant. The vertical axis shows the average number of leaves in the surveyed population. The horizontal axis indicates the survey date.

表1 突变体lyd1和野生型KN5585的农艺性状数据分析
表中数据以“平均值±标准差”形式呈现;**表示< 0.01差异极显著,*表示< 0.05差异显著。
Data are present “means ± SDs” in the table;**:< 0.01;*:< 0.05.
对和野生型最长节间进行石蜡切片观察(图3-A, B)。结果表明,最长节间的10个细胞纵向长度为17.70 μm, 与野生型KN5585最长节间的10个细胞纵向长度22.13 μm相比, 减少了20.02%, 差异达到极显著水平;最长节间细胞数目36,003.50个, 野生型KN5585最长节间细胞数目44,984.00个, 差异达到极显著水平(图3-C, D)。因此,节间缩短是由于节间细胞数目减少和细胞纵向长度缩短共同导致。

图3 野生型KN5585和突变体lyd1散粉后雌穗穗位下第二节节间的石蜡切片观察
A: 散粉后野生型KN5585最长节间(第11叶所在节间)细胞观察; B: 散粉后突变体最长节间(第19叶所在节间)细胞观察; C, D: 野生型KN5585最长节间和最长节间细胞长度和数量的比较。标尺: 5 μm;**表示< 0.01差异极显著。
A: the observation of longest intersegmental (the internode where the 11th leaf is located.) cells of wild type KN5585 after loose powder; B: the observation of longest intersegmental (the internode where the 19th leaf is located.) cells of mutantafter loose powder; C, D: the comparison of longest intersegmental cell length and number between wild type KN5585 and mutant. Bar: 5 μm;**:< 0.01
2.2 突变基因的定位
将[×Mo17] F2作为定位群体, 利用均匀分布在玉米10条染色体上的SSR和Indel标记对、Mo17进行多态性分析, 从中共筛选到172对具有双亲多态性的标记。将筛选得到的多态性标记对正常基因池和突变基因池进行BSA混池分析, 结果显示位于3号染色体上3对多态性标记Bnlg1019、Indel1、Indel9 (表2)在正常基因池和突变基因池之间存在差异。进一步利用[×Mo17] F2中的93株多叶矮化单株进行基因分型, 发现标记Bnlg1019、Indel1和Indel9的交换单株分别为35、34和9个, 因此将候选基因定位于玉米3号染色体标记Indel1和Indel9之间。在Indel1和Indel9之间开发出8对新的多态性Indel标记, 利用8对Indel标记对[×Mo17] F2中93个多叶矮化单株进行基因分型, 发现Indel2、Indel3、Indel4、Indel5、Indel6、Indel7、Indel10、Indel8 (表2)的交换单株数分别为30、15、10、7、6、2、0和8个, 将定位区段锁定在标记Indel7和Indel8之间, 物理距离10.94 Mb (图4-A)。
为了精细定位, 在标记Indel7和Indel8之间新开发出5对有多态性的标记。同时扩大定位群体, 利用新开发的5对多态性标记对[×Mo17] F2群体中的剩余的1204株多叶矮化单株进行基因分型, Indel7、Indel10、SNP1、Indel11、Indel12、Indel13和Indel8 (表2)的交换单株数分别为35、6、0、11、11、39和89个, 最终将候选基因定位在Indel10和Indel11标记之间, 物理距离约0.74 Mb (图4-B, C)。
2.3 候选基因分析
根据B73_V5版本的基因注释信息, 0.74 Mb定位区间中共有14个候选基因, 其中存在1个假基因(表3)。根据候选基因的基因组和CDS序列的长度及扩增的难易程度, 对基因组序列较短且较容易扩增的基因扩增其基因组序列, 否则扩增其CDS序列, 并将扩增产物进行测序。结果显示KN5585和之间在基因组上、、、、、和均无序列差异, 在全长CDS上、、、、均无序列差异。而()在基因组上存在1个碱基的替换, 为了验证该碱基替换的真实性, 对全长CDS序列进行扩增, 发现该位置的碱基替换在的基因组和CDS序列中稳定存在(图5-A)。全长4132 bp, 只有1个转录本, 包含6个外显子和5个内含子, 编码664个氨基酸, 具有3个典型的RNA结合结构域(RRM), 编码一个RNA结合蛋白。中碱基替换发生在第4外显子上, 位于全长CDS第1478碱基由A碱基转换为T碱基, 导致第492个氨基酸由天冬氨酸转变为缬氨酸, 该突变的氨基酸残基位于RRM3结构域内。RRM3在玉米、水稻、拟南芥、小麦、高粱、大豆、藜麦等植物中保守性较高, 并且突变的天冬氨酸也具有较高保守性(图5-B, C)。

图4 突变体lyd1的精细定位
A: 候选基因初步定位在3号染色体Indel7和Indel8分子标记之间; B: 候选基因精细定位在3号染色体Indel10和Indel11分子标记之间; C: 精细定位区间内包含14个候选基因。横线上方为分子标记, 横线下方为重组单株数。
A: the candidate gene was preliminarily located between the Indel7 and Indel8 molecular markers on chromosome 3; B: the candidate gene was finely positioned between the Indel10 and Indel11 molecular markers on chromosome 3; C: there are 14 candidate genes in the fine localization interval. Above the horizontal line is the molecular marker, below the horizontal line is the number of recombinant individual plants.

表2 基因定位相关标记信息
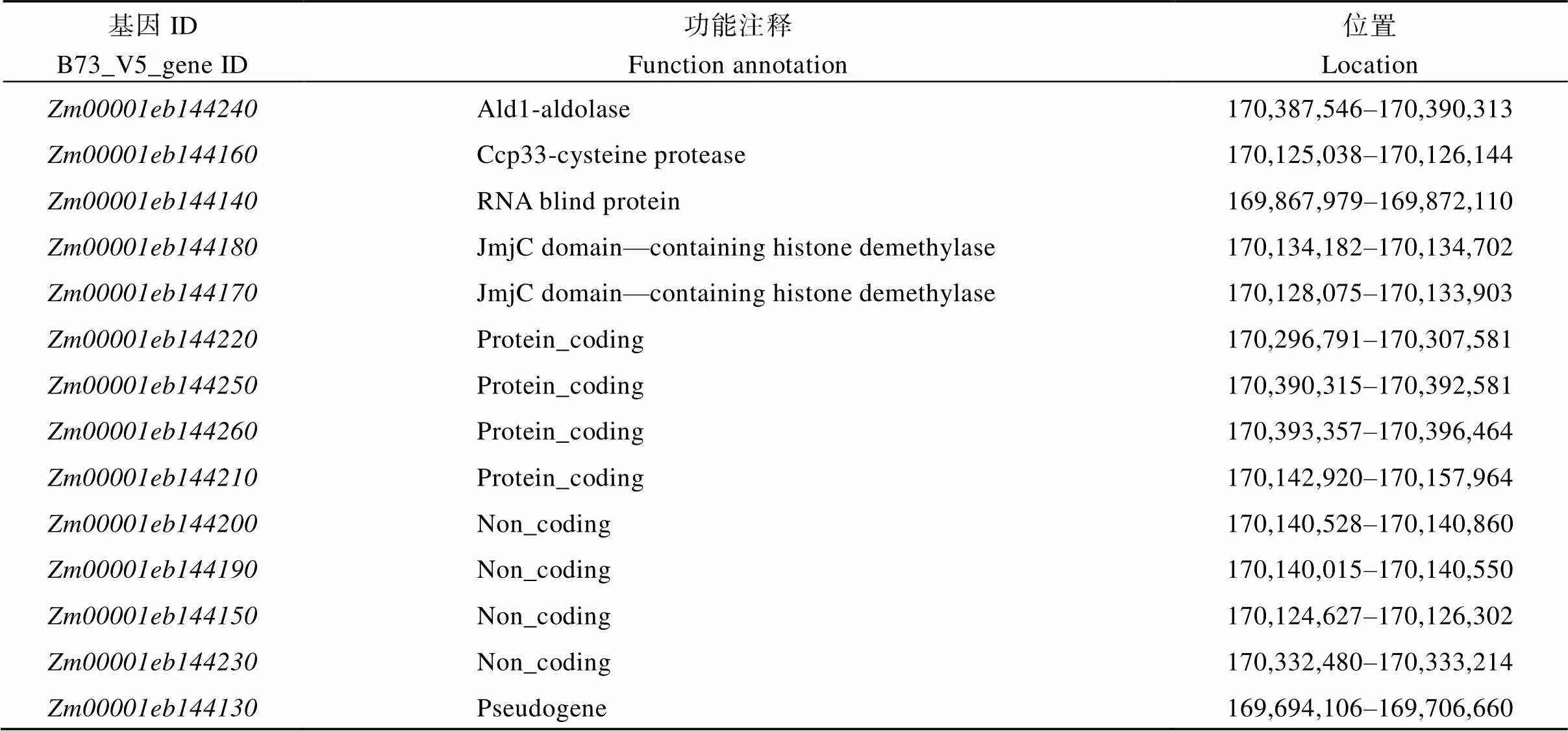
表3 定位区间内候选基因功能注释
根据在和野生型差异位点设计特异性SNP引物(SNP1), 以标记Indel10和Indel11在[×Mo17] F2定位群体中检测到的17个关键交换单株的基因组DNA为模板, 进行扩增测序, 测序结果显示17个单株在该位点都显示的T碱基(图6), 以上结果表明该位置发生的碱基替换与多叶矮化突变表型共分离。
2.4 ZmTE1的表达分析
利用qRT-PCR分析了在野生型的表达特征, 结果显示在茎尖和幼嫩的雌穗中表达量较高, 在叶片、茎、雄穗中表达量很低, 在根中几乎不表达(图7-A)。同时分析了V3和V7时期在野生型与茎尖中的表达情况, 结果显示2个时期在突变体和野生型之间的表达量均未达显著差异(图7-B), 说明A碱基到T碱基的替换可能不影响该基因在茎尖的表达。
为进一步了解蛋白的定位情况, 对蛋白进行亚细胞定位分析, 结果显示GFP-的GFP信号与核、膜Marker的信号很好地重叠(图8-A), 表明蛋白定位在细胞核和细胞膜上, 这与RNA结合蛋白在转录水平对细胞核内的mRNA进行加工的功能一致。
3 讨论
玉米的株高通常由节间的数目和节间长度决定, 一般来说节间数目(叶片数目)越多, 节间越长植株越高。目前已报道的玉米矮化突变体, 有些叶片数目不变, 株高降低由节间长度缩短导致, 如[10-11]、[27]、[7-9]、[28]过表达株系[29]等, 有些为叶片数目减少和节间长度缩短, 但叶片数减少是导致株高降低的主要原因, 如[18]、过表达株系[30]等。此外还有些玉米矮化突变体, 表现为节间数量的增加, 其株高降低是由节间长度缩短导致, 如突变体叶片数较野生型增加3.2片[31];突变体叶片数较野生型增加1.6片[32];突变体叶片数较野生型增加1.75片[16]。而本研究发现的多叶矮化突变体叶片数较野生型KN5585增加10片叶, 增加了56.18%, 节间严重缩短, 为研究节间数目和节间长度的平衡提供了宝贵材料。

图5 lyd1突变位点及突变位点保守性分析
A: 突变体与野生型KN5585的CDS差异序列比较; B, C:蛋白序列突变氨基酸残基保守性分析
A: the comparison of CDS difference sequence ofin mutantand wild type KN5585; B, C: the conservative analysis of amino acid residues mutated inprotein sequence.

图6 野生型KN5585和突变体lyd1的序列差异位点群体验证

图7 ZmTE1组织表达分析
A:在野生型KN5585不同组织中的表达; B:在野生型KN5585和突变体中的表达比较。V3、V7、V11、V17分别表示玉米植株分别有3、7、11、17片完全展开叶所处的时期。
A: the relative expression level ofgenes in different tissues of wild type KN5585; B: the comparison ofexpression in wild type KN5585 and mutant. V3, V7, V11, and V17 represent the stage when the maize has 3, 7, 11, and 17 fully unfolded leaves, respectively.

图8 ZmTE1的亚细胞定位
PC2300-eGFP-与NLS (细胞核标记)和mCherry (细胞膜标记)共定位, 标尺为50 μm。
PC2300-eGFP-are colocalized with NLS (nuclear marker) and mCherry (cell membrane marker). Bar: 50 μm.
控制突变表型的基因被定位在玉米3号染色体, 位于标记Indel10和Indel11之间, 物理距离0.74 Mb, 将确定为的关键候选基因。与已报道的、、突变体的突变位点不同,和突变体中转座子插入在第3外显子,突变体中转座子插入在第1内含子[33],突变体中第1外显子单碱基的替换导致蛋白翻译提前终止[34], 上述已报道的突变体植株生活力较弱, 难以完成正常生长发育, 给后续研究带来困难, 并且由于突变位点的不同导致株高降低的程度存在差异, 突变体仅在第4外显子上发生1处碱基替换, 植株矮化程度较轻, 且并未出现雌穗和雄穗发育缺失, 能够正常生长。RNA结合蛋白通过在转录水平上改变与之结合RNA的命运或功能, 调控植物的生长发育[35]。RRM是特征最明确的RNA结合基序, 其拓扑结构为βαββαβ, 与RNA的特异性识别通常发生在β-sheet的表面[36]。编码含有3个RRM结构域的RNA结合蛋白, 但该基因下游的靶标RNA尚未找到。突变体中发生突变的天冬氨酸残基具有较高的保守性, 且出现在保守性较高的RRM3结构域内, 位于与RNA的特异性识别相关的β-sheet的表面, 因此我们推测中的点突变可能通过影响与下游RNA的结合能力进而影响其他基因的表达。由此可见,突变体的发现对于从RRM3结构域出发探索下游靶标RNA, 以及解析3个RRM在多叶矮化表型产生过程中所扮演的角色具有重要研究意义。
4 结论
本研究发现了一个玉米多叶矮化突变体, 其株高较野生型KN5585显著下降41.79%, 叶片数较野生型KN5585显著增加56.18%。该突变表型受一对隐性核基因控制, 被定位在3号染色体分子标记Indel10和Indel11之间, 物理距离0.74 Mb。进一步对定位区间内候选基因测序, 发现第4外显子出现1个碱基替换, 其他基因无差异, 因此, 将确定为突变表型的关键候选基因, 该基因编码一个含有3个RNA结合结构域的RNA结合蛋白, 突变位点位于第3个RNA结合结构域。突变体的发现为探究玉米叶片数目(节间数目)与节间长度的平衡以及二者对株型发育的影响提供了宝贵的试验材料。
[1] Tester M, Langridge P. Breeding technologies to increase crop production in a changing world., 2010, 327: 818–822.
[2] Haarhoff S J, Swanepoel P A. Plant population and maize grain yield: a global systematic review of rainfed trials., 2018, 5: 1819–1829.
[3] Bensen R J, Johal G S. Cloning and characterization of the maizegene., 1995, 7: 75–84.
[4] Chen Y, Hou M M, Liu L J, Wu S, Shen Y, Ishiyama K, Kobaya, Shi M, McCarty D R, Tan B C. The maizeencodes a gibberellin 3-oxidase and is dual localized to the nucleus and cytosol., 2014, 166: 2028–2039.
[5] Teng F, Zhai L H, Liu R X, Bai W, Wang L Q, Huo D G, Tao Y S, Zheng Y L, Zhang Z X., a candidate gene for a major QTL,, for plant height in maize., 2013, 73: 405–416.
[6] Winkler R G, Helentjaris T. The maizegene encodes a cytochrome P450-mediated early step in gibberellin biosynthesis., 1995, 7: 1307–1317.
[7] Cassani E, Bertolini E, Cerino Badone F, Landoni M, Gavina D, Sirizzotti A, Pilu R. Characterization of the first dominant dwarf maize mutant carrying a single amino acid insertion in the VHYNP domain of thegene., 2009, 24: 375–385.
[8] Harberd N P, Freeling M. Genetics of dominant gibberellin- insensitive dwarfism in maize., 1989, 121: 827–838.
[9] Lawit S J, Wych H M, Xu D, Kundu S, Tomes D T. Maize DELLA proteinsandas modulators of plant development., 2010, 51: 1854–1868.
[10] Multani D S, Briggs S P, Chamberlin M A, Blakeslee J J, Murphy A S, Johal G S. Loss of an MDR transporter in compact stalks of maizeand sorghummutants., 2003, 302: 81–84.
[11] Zhang X, Hou X, Liu Y, Zheng L, Yi Q, Zhang H, Huang X, Zhang J, Hu Y, Yu G, Liu H, Li Y, Huang H, Zhan F, Chen L, Tang J, Huang Y. Maize brachytic2 () suppresses the elongation of lower internodes for excessive auxin accumulation in the intercalary meristem region., 2019, 19: 589.
[12] Hartwig T, Chuck G S, Fujioka S, Klempien A, Weizbauer R, Potluri D P V, Choe S, Johal G S, Schulz B. Brassinosteroid control of sex determination in maize., 2011, 108: 19814–19819.
[13] Best N B, Hartwig T, Budka J, Fujioka S, Johal G, Schulz B, Dilkes B P.encodes a maize ortholog of thebrassinosteroid biosynthesis gened, identifying developmental interactions between brassinosteroids and gibberellins., 2016, 171: 2633–2647.
[14] Makarevitch I, Thompson A, Muehlbauer G J, Springer N M.gene in maize encodes a brassinosteroid C-6 oxidase., 2012, 7: e30798.
[15] Kir G, Ye H, Nelissen H, Neelakandan A K, Kusnandar A S, Luo A, Inzé D, Sylvester A W, Yin Y, Becraft P W. RNA interference knock down ofin maize reveals novel functions for brassinosteroid signaling in controlling plant architecture., 2015, 169: 826–839.
[16] Li H, Wang L, Liu M, Dong Z, Li Q, Fei S, Xiang H, Liu B, Jin W. Maize plant architecture is regulated by the ethylene biosynthetic gene., 2020, 183: 1184–1199.
[17] Schaller G E, Bishopp A, Kieber J J. The Yin-Yang of hormones: cytokinin and auxin interactions in plant development., 2015, 27: 44–63.
[18] Phillips K A, Skirpan A L, Liu X, Christensen A, Slewinski T L, Hudson C, Barazesh S, Cohen J D, Malcomber S, Mcsteen P.encodes a grass-specific tryptophan aminotransferase required for vegetative and reproductive development in maize., 2011, 23: 550–566.
[19] Lee B H, Johnston R, Yang Y, Gallavotti A, Kojima M, Travençolo B A, Costa Lda F, Sakakibara H, Jackson D. Studies ofmutants of maize indicate complex interactions between auxin and cytokinin signaling in the shoot apical meristem., 2009, 150: 205–216.
[20] 张在宝, 李婉杰, 李九丽, 张弛, 胡梦辉, 程琳, 袁红雨. 植物RNA结合蛋白研究进展. 中国农业科学, 2018, 51: 4007–4019. Zhang Z B, Li W J, Li J L, Zhang C, Hu M H, Cheng L, Yuan H Y. The research progress of plant RNA binding proteins., 2018, 51: 4007–4019 (in Chinese with English abstract).
[21] Cho H, Cho H S, Hwang I. Emerging roles of RNA-binding proteins in plant development., 2019, 51: 51–57.
[22] Jeffares D C, Phillips M J, Moore S, Veit B. A description of the Mei2-like protein family; structure, phylogenetic distribution and biological context., 2004, 214: 149–158.
[23] Kawakatsu T, Itoh J, Miyoshi K, Kurata N, Alvarez N, Veit B, Nagato Y.regulates leaf initiation and maturation in rice., 2006, 18: 612–625.
[24] Anderson G H, Alvarez N D, Gilman C, Jeffares D C, Trainor V C, Hanson M R, Veit B. Diversification of genes encoding mei2–like RNA binding proteins in plants.,2004, 54: 653–670.
[25] 王关林, 方宏筠. 植物基因工程(第2版). 北京: 科学出版社, 2002. pp 742–744. Wang G L, Fang H Y. Plant Gene Engineering, 2nd edn. Beijing: Science Press, 2002. pp 742–744 (in Chinese).
[26] Michelmore R W, Paran I, Kesseli R V. Identification of markers linked to disease-resistance genes by bulked segregant analysis: a rapid method to detect markers in specific genomic regions by using segregating populations., 1991, 88: 9828–9832.
[27] Avila L M, Cerrudo D, Swanton C, Lukens L., a putative inositol polyphosphate 5-phosphatase, is required for internode elongation in maize., 2016, 67: 1577–1588.
[28] Zhang D, Sun W, Singh R, Zheng Y, Cao Z, Li M, Lunde C, Hake S, Zhang Z.regulates shoot architecture and meristem determinacy in maize., 2018, 30: 360–374.
[29] Li W, Ge F, Qiang Z, Zhu L, Zhang S, Chen L, Wang X, Li J, Fu Y. Maizeencodes a microtubule-associated protein that controls plant and ear height., 2020, 18: 1345–1347.
[30] Heuer S, Hansen S, Bantin J, Brettschneider R, Kranz E, Lörz H, Dresselhaus T. The maize MADS box geneaffects node number and spikelet development and is co-expressed withduring flower development, in egg cells, and early embryogenesis., 2001, 127: 33–45.
[31] Lyu H K, Zheng J, Wang T Y, Fu J J, Huai J L, Min H W, Zhang X, Tian B H, Shi Y S, Wang G Y. The maize, a novel allele of, is required for maize internode elongation., 2014, 84: 243–257.
[32] Bommert P, Je B I, Goldshmidt A, Jackson D. The maize Gα genefunctions in clavata signalling to control shoot meristem size., 2013, 502: 555–558.
[33] Veit B, Briggs S P. Regulation of leaf initiation by thegene of maize., 1998, 393: 166–168.
[34] Wang F, Yu Z, Zhang M, Wang M, Lu X, Liu X, Li Y, Zhang X, Tan B C, Li C, Ding Z.promotes plant height by regulating intercalary meristem formation and internode cell elongation in maize., 2022, 20: 526–537.
[35] Hentze M W, Castello A, Schwarzl T, Preiss T. A brave new world of RNA-binding proteins., 2018, 19: 327–341.
[36] 唐蜻. 植物RNA结合蛋白的研究进展. 安徽农业科学, 2010, 38(1): 38–41. Tang Q. The research progress of plant RNA binding proteins., 2010, 38(1): 38–41 (in Chinese with English abstract).
Identification and gene cloning of leafy dwarf mutantin maize
SU Shuai, LIU Xiao-Wei, NIU Qun-Kai, SHI Zi-Wen, HOU Yu-Wei, FENG Kai-Jie, RONG Ting-Zhao, and CAO Mo-Ju*
Maize Research Institute, Sichuan Agricultural University / Key Laboratory of Biology and Genetic Improvement of Maize in Southwest Region, Ministry of Agriculture and Rural Affairs, Chengdu 611130, Sichuan, China
The decrease of plant height in maize is usually caused by the decrease in the number of internodes, the shortening of internodes or the combination of both. However, in this study, the mutant() found in the progeny of gene editing, exhibited more leaves and shorter stature. Quantitative measurements indicated the plant height of mutantwas only 93.10 cm, the plant height of wild-type KN5585 was 159.95 cm. The plant height was significantly reduced by 41.79% in mutantcompared with the wild type KN5585. The wild type KN5585 produced an average of 17.8 leaves at maturity stage, whereas mutantsproduced 27.8 leaves. The number of leaves were significantly increased by 56.18% in mutantcompared with the wild type. Genetic analysis showed that the mutation phenotype ofwas controlled by a pair of recessive nuclear genes. We applied a map-based cloning strategy to identify the gene responsible for thephenotype. The gene was located between Indel10 and Indel11 on maize chromosome 3, and the physical distance was 0.74 Mb. Gene sequencing analysis of 13 genes (excluding pseudogenes) within the interval revealed that one base A was substituted in the fourth exon of, and there was no significant difference in other genes.encoded an RNA-binding protein. The amino acid substitution was in the third RNA binding domain (RRM3), resulting in the conversion of aspartic acid to valine. The mutation sites of the mutantwere different from,,, andin previously reported. The discovery ofprovides valuable materials for further analysis of the genetic mechanism of the balance between leaves and internodes development in maize.
maize; the number of leaves; the length of internodes; gene mapping
10.3724/SP.J.1006.2024.33044
本研究由四川省科技计划项目(2021YFYZ0011, 2021YFYZ0017, MZGC20230108)和四川农业大学学科建设专项研究支持计划项目资助。
This study was supported by the Sichuan Science and Technology Program (2021YFYZ0011, 2021YFYZ0017, MZGC20230108) and the Specific Research Supporting Program for Discipline Construction at Sichuan Agricultural University.
曹墨菊, E-mail: caomj@sicau.edu.cn
E-mail: 1018714902@qq.com
2023-07-31;
2024-01-12;
2024-02-08.
URL: https://link.cnki.net/urlid/11.1809.S.20240206.1114.002
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).
