基于复杂网络理论的酵母菌PPI网络中关键蛋白质与核心蛋白质组的识别
2024-04-23武子惠彭雨萱李子正丁彦蕊
万 杰, 武子惠, 彭雨萱, 李 羚, 李子正, 丁彦蕊
(江南大学 理学院信息与计算科学系,江苏, 无锡 214122)
识别关键蛋白质对疾病治疗、药物设计等领域有重要作用。生物实验方法确定关键蛋白质具有实验周期长、成本高和研究成功率低等弊端。借助计算机技术识别关键蛋白质越来越成为生物研究方法的有效辅助,尤其体现在从复杂的蛋白质-蛋白质相互作用(protein protein interaction, PPI)中确定关键蛋白质方面。结合复杂网络理论来分析PPI网络[1],通过PPI网络的拓扑特征识别关键蛋白质是研究蛋白质功能的有效方法。
在PPI网络中,每个蛋白质是网络的1个节点,蛋白质间的相互作用是网络的边。为了识别关键蛋白质节点,Jeong等人[2]提出了中心性-致死性法则,指出蛋白质节点在网络中的中心性越高,成为关键蛋白质的可能性越大。基于该法则,较多学者将复杂网络中的一系列节点重要性排序算法[3-5]用于研究蛋白质的重要性,例如,Hahn等人通过度中心性(degree centrality, DC)[6]发现3种真核蛋白质相互作用网络中的关键蛋白质;Joy等人将介数中心性 (betweenness centrality, BC)[7]应用到酵母菌蛋白质相互作用网络中;Manimaran等人将度中心性、介数中心性和接近中心性 (closeness centrality, CC)[8]用于分析基因网络中的关键节点。不同的节点重要性指标分别从不同角度衡量节点的重要性。度中心性是基于节点邻居的排序方法,介数中心性和接近中心性是基于最短路径的排序方法,特征向量中心性( eigenvector centrality, EC)是基于特征向量的排序方法[9],k-壳分解法(k-shell)是基于节点位置的排序方法[10-14]。由于采用不同的衡量指标可以获得互补的信息,因此,综合考虑以上5种指标是有必要的。本文以Nature期刊报道的Gavin、Babu、Krogan(LCMS)和Krogan(MALDI) 4种酵母菌PPI网络为研究对象[15-17],采取度中心性、介数中心性、接近中心性、特征向量中心性以及k-壳分解法这5种节点重要性评估指标,对PPI网络的节点进行重要性排序,挖掘关键蛋白质。
通过中心性指标,本文筛选出了4种网络中的关键蛋白质,并构建了关键蛋白质构成的子网。为了进一步筛选出子网中紧密关联的关键蛋白质对,引入杰卡德相似度指标(Jaccard index)[15],计算分析子网中的关键蛋白质之间的相似性,得到拓扑结构相似的关键蛋白质对,并获得核心子网以发现不同的酵母菌 PPI 网络中蛋白质相互作用的异同。
1 材料与方法
1.1 数据集
本文使用了4个高质量的酵母菌PPI网络作为研究对象[15-17],分别为Gavin、Babu、Krogan(LCMS)和Krogan(MALDI)酵母菌PPI网络。Gavin网络是第1个全基因组水平上的PPI网络,包含有膜蛋白质的相互作用。Babu网络是膜蛋白相互作用网络,包含有1 726个膜蛋白质的相互作用。LCMS和MALDI网络来自Krogan等的研究,MALDI网络采用MALDI-TOF质谱方法测定,共有2 708个蛋白质的7 123个相互作用。LCMS通过LC-MS/MS方法测定,网络中含有更多的小蛋白质。
1.2 酵母菌蛋白质-蛋白质相互作用网络关键节点的挖掘
针对上述4种酵母菌PPI网络,通过度中心性、介数中心性、接近中心性、特征向量中心性和k-壳分解法这5个指标对关键蛋白质进行识别。

介数中心性是用最短路径的思想对图进行评价的一种度量。对于酵母菌PPI网络中的每一对节点,在节点之间至少存在一条最短路径,使得路径通过的边数(未加权图)或者边权重的和(加权图)最小。酵母菌PPI网络节点的介数中心性即为经过该节点的最短路径的数量。介数中心性反映了某节点在图中的关键程度,某节点的介数中心性越大,意味着该节点在整个网络中越重要。对于一个节点i,其介数中心性计算式为:

接近中心性用于发现可通过图高效传播信息的节点。对于每个酵母菌PPI网络的蛋白质节点,在计算所有节点对之间的最短路径的基础上,再计算它到其他各节点的最短路径之和,然后对得到的和求倒数,得到节点的接近中心性数值。节点的接近中心性计算公式为:
其中:u为待计算接近中心性的节点,v为图中除u外的任意一个节点,d(u,v)表示节点u和节点v的最短路径。
特征向量中心性是基于网络邻接矩阵的特征向量和特征值的概念衡量重要性的方法。它表明1个节点的重要性既取决于其邻居节点的数量,也取决于其邻居节点的重要性。与之相连的邻居节点越重要,则该节点就越重要。特征向量中心性的计算公式为:
其中,c为比例常数,xi表示网络中节点i的重要性,N为网络节点总数,aij等于1当且仅当节点i 与j相连,否则为0。
村小在李打油眼里,是李湾唯一的最高学府。带我去报到那天,他当着全校师生郑重宣布,学校“借钱做衫裤——一身是债”的日子一去不复返啦!而且,当场给每位师生发一套服装。我记得很清楚,当时有二百五十三个学生,十九位老师,无论男女、师生,一律的白衬衣蓝长裤。是的确良的,大家都美滋滋地叫真凉快。
k-壳分解法迭代地移除度值低于k的节点,直到所有剩余节点的度值至少为k。根据连接到的剩余节点的数量,为每个步骤移除的节点分配“壳指数”或“k-壳指数”。因此,具有高壳层索引的节点与网络中的剩余节点高度连接,对于维持网络的整体连接性和结构非常重要,对网络的行为和功能具有更大的影响力。
相关研究表明,度中心性指标筛选关键节点时在大规模网络中应用受限;介数中心性无法准确区分不在最短路径上的节点重要性;接近中心性对于大型网络计算时间复杂度较高;特征向量中心性筛选的网络收敛速度较慢,倾向于关注节点在网络中的局部结构;k-壳分解法对同层的节点的重要程度无法区分。因此,本研究采用综合5种节点中心性评估指标的方法来筛选关键节点。计算每个网络的节点的度中心性、介数中心性、接近中心性、特征向量中心性和k-壳分解值,发现4种酵母菌PPI网络的k-壳分解中k值达到上百层,说明蛋白质网络中存在相当多层级结构,并且有一些高度连接的核心节点。因此,在通过不断尝试后认为选取前4种中心性指标降序排列后都处于前2%,且k壳位于内10层的节点为关键节点较为合适,便于研究分析,并将由所有关键节点构成的网络作为关键子网。
1.3 酵母菌蛋白质-蛋白质相互作用网络关键节点对的筛选
为了筛选出关键子网中紧密联系的关键蛋白质对,本文引入了杰卡德相似度指标。杰卡德相似度指标是一种常用于数据挖掘、机器学习和网络分析的相似性度量,用于比较网络中两组数据或2个节点的相似性。在PPI网络中,由于高度连接的蛋白质通常有更多共同的相邻边,因此,杰卡德相似度指标可基于2种蛋白质相互作用的其他蛋白质的数量来测量2种蛋白质之间的相似性。杰卡德相似度指标的计算公式如下:
J(A,B)=|A∩B|/|A∪B|
若a、b 为酵母菌PPI网络中的2个蛋白质,A表示基于与蛋白质a相互作用的其他蛋白质的集合,B表示基于与蛋白质b相互作用的其他蛋白质的集合,∩表示共同基于与a、b 2个蛋白质相互作用的蛋白质数量,∪表示基于与a、b 2个蛋白质相互作用的所有蛋白质数量。通过杰卡德相似度指标计算并分析关键蛋白质之间的相似性,以获得关键的节点对。
2 结果
2.1 蛋白质-蛋白质相互作用网络中关键蛋白质的识别
本文对Gavin、Babu、LCMS和MALDI 4种酵母菌PPI网络进行度中心性、介数中心性、接近中心性、特征向量中心性、k-壳分解法5个指标的计算,获得前4种中心性指标处于前2%和k壳位于内10层的节点,并将此作为关键蛋白质筛选条件。
随后提取网络中满足关键蛋白质筛选条件的节点,并分析酵母菌PPI网络之间共有的关键蛋白质,发现不存在4个PPI网络共有的蛋白质,但3个PPI网络中存在共有的关键蛋白质,见韦恩图(Fig.1)。由Fig.1显示,在Babu、MALDI、LCMS网络中以及在Gavin、MALDI、LCMS网络中各共有1个关键蛋白质;在Babu、Gavin、MALD网络中共有5个关键蛋白质;Gavin、Babu、LCMS未见共有的关键蛋白质。
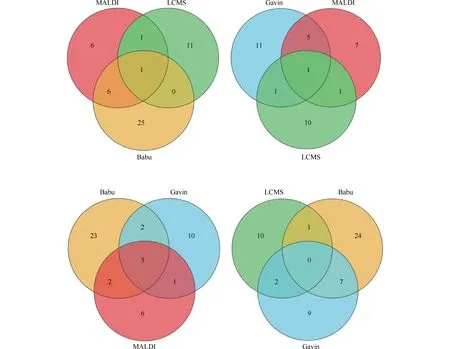
Fig.1 Venn diagram of the distribution of essential proteins shared between the three yeast PPI networks Each color represents a network, and the overlapping parts of the different colors represent essential proteins shared by the different networks
进一步构建关键蛋白质子网,结果正如Fig.2 A-D。分析结果表明,Babu、MALDI、LCMS网络中共有的关键蛋白质为YOR204W;Gavin、MALDI、LCMS网络中共有的关键蛋白质为YER165W;Babu、Gavin、MALDI网络中共有的关键蛋白质则有5个,它们分别是YOR063W、YPL198W、YGL076C、YDR012W和YBR031W。结合关键蛋白质构成的子网分析发现(Fig.2),它们的连接性和中心性较高,均占据着网络的重要位置,是网络中信息流动的关键枢纽。

Fig.2 The subnetworks of essential proteins of the four yeast PPI network Orange, red, and green represent essential proteins that are repeated in different networks
2.2 酵母菌蛋白质-蛋白质相互作用网络中核心子网的识别
对Fig.2中关键蛋白质构成的子网,本文通过计算关键蛋白质之间的杰卡德相似度指标,选取阈值在0.55以上的蛋白质对,获得了拓扑结构相似的关键蛋白质对,这些关键蛋白质对构成酵母菌PPI网络的核心子网。
Fig.3分别是Gavin, Babu, LCMS和MALDI 4种酵母菌PPI网络的核心子网。
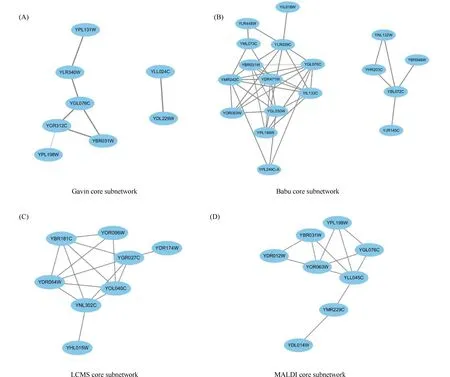
Fig.3 The core subnetworks of the four yeast PPI network The edge between the nodes indicates the Jaccard index above 0.55 between the two proteins
对于Gavin网络,由Fig.3 A显示:YGL076C、YPL131W、YPL198W、 YLR340W、YOR312C和YBR031W构成了一个联系紧密的关键蛋白质组,而YLL024C和YDL229W为另一组,将前一组称为Gavin核心蛋白质组1,简写为Gavin-EPG 1(essential protein group),后一组称为Gavin-EPG 2。
对于Babu网络,由Fig.3 B显示:YBR031W、 YPL198W、YPL249C-A、YLR448W、YGL030W、YIL133C、YLR029C、YGL076C、YDR471W、 YOR063W、YML073C、YIL018W、和YMR242C是一组联系紧密的蛋白质组,YNL132W、YHR203C、YBR048W、YBL072C和YJR145C 为另一组,前一组称为Babu-EPG 1,后一组称为Babu-EPG 2。
对于LCMS网络,由Fig.3 C的结果表明:YDR064W、YGR027C、YBR181C、YOL040C、YNL302C、YOR096W、YDR174W和YHL015W是一组相互作用很强的蛋白质组,称为LCMS-EPG。
对于MALDI网络,由Fig.3 D的结果显示:YGL076W、YPL198W、YLL045C、YOR063W、YBR031W、YDR012W、YMR229C和YDL014W是一组相互作用很强的蛋白质组,称为MALDI-EPG。
这些核心蛋白质组中的蛋白质间有着紧密连接,处在网络中的核心地位,它们共同作用影响着网络的稳定性。
2.3 关键蛋白质和核心蛋白质组的功能分析
通过Fig.2的结果发现,YOR204W、YER165W、YOR063W、YPL198W、YGL076C、YDR012W和YBR031W是不同网络共有的关键蛋白质,其功能见Table 1。
从Table 1可知,Babu、MALDI、LCMS共有的关键蛋白质是依赖ATP的RNA解旋酶,Gavin、MALDI、LCMS共有的关键蛋白质是PolyA的结合蛋白质,Babu、Gavin、MALD网络共有的5个关键蛋白质都是核糖体60S大亚基的组成蛋白质。这些关键蛋白质都参与了蛋白质合成过程中将mRNA转化为多肽链[24]。
通过2.2节分析发现,Gavin和Babu网络各有2个核心蛋白质组Gavin-EPG 1和Gavin-EPG 2,Babu-EPG 1和Babu-EPG 2;LCMS和MALDI网络各有1个核心蛋白质组LCMS-EPG和MALDI-EPG,具体结果见Table 2。
Table 2的结果表明,Gavin-EPG 1和Babu-EPG 1全部由属于大亚基的核糖体蛋白质组成。尽管这些蛋白质都是细胞质核糖体蛋白质,但是不同的核糖体蛋白质在不同的酵母菌PPI网络中形成了不同的核心蛋白质组。Gavin-EPG 2是由细胞质ATP酶和HSP70家族ATP-结合蛋白质构成,2个蛋白质都参与新生肽链的折叠[25]。Babu-EPG 2中除了YNL132W,其他都是属于小亚基的核糖体蛋白质,LCMS-EPG中除了YDR174W,其他也都属于小亚基的核糖体蛋白质。YNL132W是小核糖体亚基生物发生所必需的乙酰转移酶[26],YDR174W是染色质相关高迁移基团((high-mobility group,HMG)家族成员,其对染色质的组装很重要[27]。MALDI-EPG中除了属于大亚基的核糖体蛋白质,还有YMR229C和YDL014W,前者是DNA结合蛋白质,参与18S和5.8S rRNAs的合成[28],后者是组蛋白谷氨酰胺甲基转移酶,参与pre-18S rRNA的加工[29]。

Table 1 Essential proteins and their functions
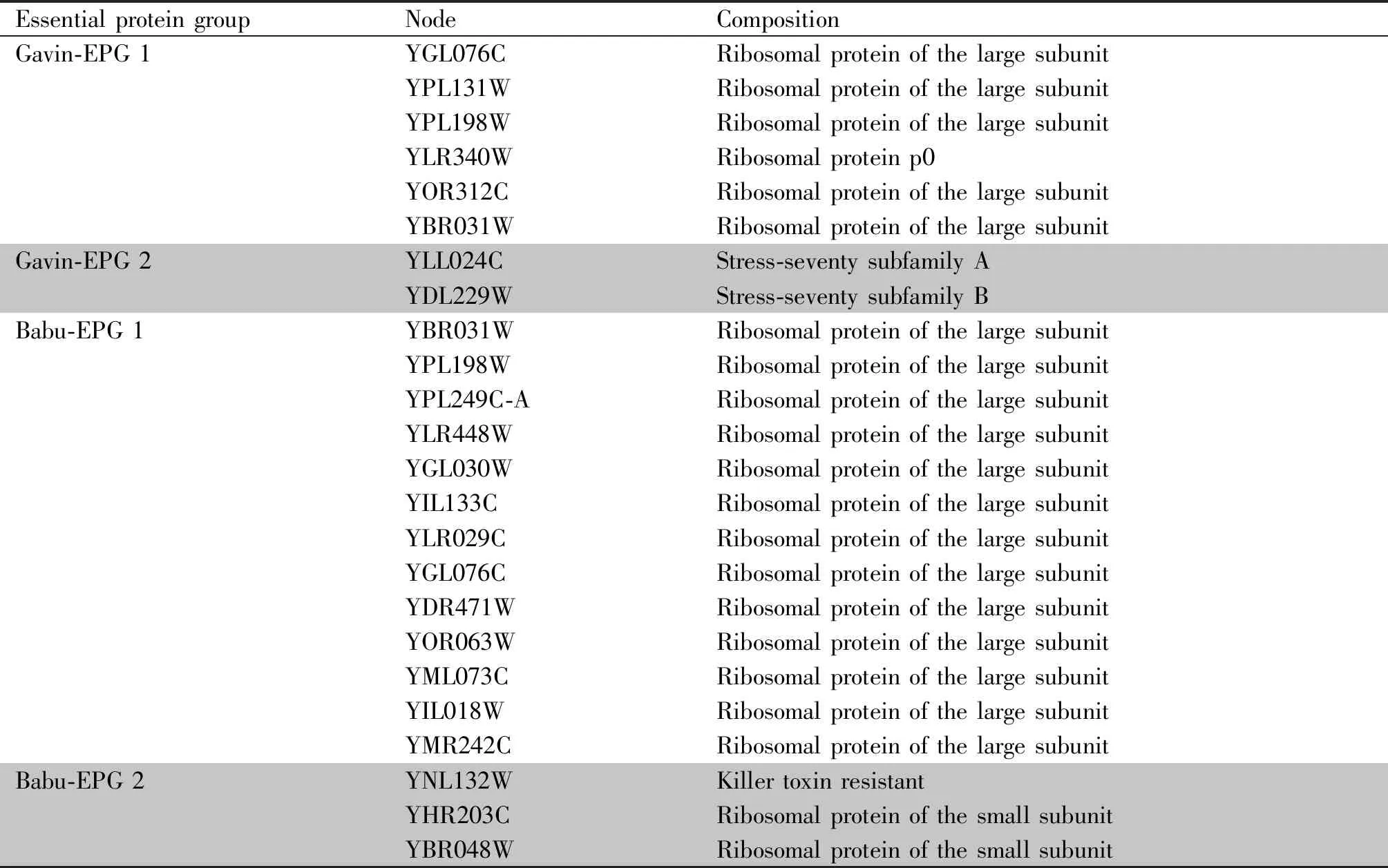
Table 2 The core protein groups
To the next page

Continued Table 2
3 讨论
本文对4个酵母菌PPI网络进行了关键蛋白质和核心蛋白质组的识别,发现不同酵母菌PPI网络中共有的7个关键蛋白质和6个核心蛋白质组,这些关键蛋白质均在将mRNA转化为多肽链的过程中发挥着重要作用。通过采用5种节点重要性排序算法识别关键蛋白质,本文能够准确捕捉到蛋白质在网络中的位置和影响力,成功识别出在维持网络功能和结构稳定性方面起关键作用的蛋白质。面对大规模蛋白质相互作用网络的分析,本文能够高效地筛选出潜在的重要蛋白质。通过杰卡德相似度指标识别核心蛋白质组,本文能够比较蛋白质间的相似性与差异性,捕捉到具有高相似性的蛋白质组成员。然而,本研究忽略了蛋白质的生物学特征和功能信息,仅基于网络拓扑结构来进行分析,可能遗漏了一些在生物学上具有重要功能的蛋白质。
本文揭示了关键蛋白质在细胞代谢和调控中的关键作用,并构建了关键蛋白质对应的核心蛋白质组,为进一步研究这4个酵母菌PPI网络提供了具体的蛋白质组合。这些关键蛋白质和核心蛋白质组为深入研究核糖体上蛋白质相互作用对肽链合成和折叠的影响提供了重要的理论基础。
