不同鲜食品质橄榄果实转录组测序及酚类代谢途径相关调控基因挖掘
2024-04-17谢倩江来贺进刘玲玲丁明月陈清西
谢倩 江来 贺进 刘玲玲 丁明月 陈清西
(福建农林大学园艺学院,福州 350002)
橄榄(Canarium album L.)是我国的特色木本果树,作为重要的药食同源果树之一,涩味感与回甘度是影响其鲜食口感的主要因子[1-2],选育涩味淡、回甘明显是橄榄新品种选育的主要目标。酚类物质类别含量直接影响橄榄涩味程度[3-6],糖[7]、氨基酸[8]等直接影响橄榄回甘程度。橄榄中酚类物质主要包括水解单宁、酚酸、黄酮类等[6,9-13],对橄榄果实品质具有重要作用。因此,挖掘橄榄酚类物质调控关键基因,解析其分子调控机制,对橄榄新品种选育具有重要指导意义。
转录组测序(RNA sequencing, RNA‑seq)技术是高效获取有效参考功能基因及标记位点的重要方式,也是获得转录本定性和定量结果的有用工具,已经成为揭示目的性状潜在分子调控机制的有效技术之一,加速了对未知功能基因的挖掘[14]。转录组是生物体在某种条件下所有转录产物的综合,对于像橄榄这种没有参考基因组的物种进行转录组测序,是连接基因型与表型的重要桥梁。转录因子(transcription factor, TF)通过调控其他基因的表达来影响相应的表型,是信号转导的关键性节点,对于园艺植物次生代谢合成具有十分重要的作用[15-17]。转录组测序技术已被广泛应用于园艺作物品质相关基因挖掘的相关研究中,如刺葡萄抗白腐病关键基因发掘[18]、猕猴桃果肉色泽调控基因挖掘[19]、镉胁迫下梭鱼草叶片苯丙烷代谢途径相关基因挖掘[20]等。
酚类化合物主要通过莽草酸途径(shikimate pathway, SP)、苯丙烷代谢途径(phenylpropanoid metabolic pathway, PMP)与类黄酮代谢途径(flavonoid metabolic pathway, FMP)合成,由糖酵解(embden‑meyerhof‑pamas pathway, EMP)途径生成的磷酸烯醇式丙酮酸(phosphoenolpyruvate, PEP)和磷酸戊糖(phosphoenolpyruvate, PPP)途径生成的赤藓糖‑4‑磷酸(erythrose‑4‑pyosphate, E4P)经SP 途径形成苯丙氨酸,苯丙氨酸在各种酶的催化作用下,经过PMP途径和FMP 途径形成的[21]。为深入研究不同鲜食品质橄榄酚类物质代谢途径的分子调控机制,探究4 个品种(系)在成熟期基因转录水平差异,选取鲜食品质差异显著的‘子阳1 号’和‘东山长穗’2个品种(系),比较分析在成熟过程的转录组差异,并进一步挖掘调控橄榄酚类合成相关转录因子,为深入研究酚类代谢途径参与橄榄鲜食品质差异形成的分子机制奠定理论基础。
1 材料与方法
1.1 材料
‘惠圆’(HP)、‘子阳1 号’(SP)口味苦涩,较不适合鲜食;‘甜榄1 号’(TQ)、‘东山长穗’(DQ)涩味较淡,适合鲜食[2]。本研究以这4 个橄榄品种(系)(C.album)为材料,成熟果采于2020 年11 月中下旬成熟期,不同成熟时期果实采于2020 年8-11月(花后80 d、95 d、160 d)。样本采自福州市橄榄种质资源圃(26°8'6'' N,119°15'58'' E),采后用液氮快速冷冻并于‑80℃冰箱保存备用。
为方便分析将花后80 d、95 d、160 d 分别表示为a、b、c。
1.2 方法
1.2.1 果实总酚测定 总酚提取采用水浴锅浸提[22],测定采用福林酚试剂法[23]进行,标准曲线为Y 浓度=(X吸光值- 0.008 9)/2.424 7(R2= 0.999 8,标准液为没食子酸),试验重复3 次。
1.2.2 RNA 提取与质量检测 RNA 提取选用天根多糖多酚试剂盒,每个样品设置3 个生物学重复,共构建24 个文库。提取的RNA 采用1%琼脂糖凝胶电泳(电压180 V,电泳时间16 min)、Agilent 2100生物分析仪进行RNA 纯度、完整性检测;采用NanoPhotometer 分光光度计进行RNA 纯度(OD260/280和OD260/230)检测;采用Qubit 2.0 荧光计进行RNA浓度测定。
1.2.3 cDNA 文库构建、质检与测序 提取总RNA 后,通过Oligo(dT)磁珠富集mRNA,加入Fragmentation buffer 打断成短片段,以短片段mRNA为模板,合成第一链cDNA;加入缓冲液、dNTPs与DNA polymerase I 合成二链cDNA 后纯化双链cDNA;纯化的双链cDNA 通过末端修复、加A 尾后连接测序接头,AMPure XP beads 进行片段大小选择后进行PCR 富集,完成cDNA 文库构建。Insert size 符合预期后进行文库浓度定量,然后用Illumina HiSeq 平台进行混合样本测序。
1.2.4 序列拼接 测序得到的原始数据经过滤、测序错误率检查、GC 含量分布检查,获得Clean reads后,采用Trinity 组装软件对Clean reads 进行拼接。拼接得到的转录本序列,以Corset(官方网站https://code.google.com/p/corset‑project/)层 次 聚 类 后得到最长Cluster 序列作为Unigene 用于本研究后续分析。
1.2.5 转录本功能注释与编码区预测 Unigene 序列使用BLAST 软件分别在KEGG、NR、SwissProt、GO、KOG、Trembl 数据库中进行比对(E‑value ≤10-5);完成Unigene 氨基酸序列预测后,使用Pfam数据库与HMMER 软件进行序列比对,获得相应Unigene 的功能注释信息。
1.2.6 表达量注释、差异表达基因筛选及功能分析 将得到的序列数标准化为FPKM 值,标准化后的FPKM 值即为该基因的表达量。差异表达基因(differentially expressed genes,DEGs) 选 用TBtools的DESeq2 插件,使用负二项分布模型来鉴定差异表达基因。成熟果DEGs 筛选条件:鲜食品质差的高酚品种(系)(SPc、HPc)与鲜食品质好的低酚品种(系)(TQc、DQc)间的“P < 0.05,|log2FPKM低酚/FPKM高酚|≥1”,定义了4 组差异基因,即SPc vs TQc、SPc vs DQc、HPc vs TQc、HPc vs DQc。其 中FPKM低酚>FPKM高酚,基因上调表达,称为正选择基因,反之下调表达称为负选择基因。使用TBtools(1.1043)对差异基因进行KEGG 通路富集分析,采用Origin Pro 2021 进行结果可视化。
1.2.7 权重基因共表达网络分析 对橄榄不同品种(系)及成熟过程的24 个样本的DEGs 进行加权基因共表达网络分析(weighted gene co‑expression network analysis,WGCNA)。首先对DEGs 进行过滤,设置IQR 筛选,Cutoff =0.5;数据过滤后对样本进行WGCAN 分析,软阈值β 值=16,最小模块基因数50,相似模块合并阈0.25。目标模块基因互作网络可视化采用Maximum clique centrality(MCC)拓扑分析法,以degree 值≥1 筛选与代谢途径相关的关键转录因子。
1.2.8 RT‑qPCR 验 证 选 择12 个 差 异 表 达 基 因Unigene 在2 个品种(系)果实间进行RT‑qPCR,验证转录组数据的可靠性。引物序列、RT‑qPCR 橄榄品种(系)见表1,共18 个样品。RNA 提取同1.2.2,以RNA 为 模 板, 按 TransScript® All‑in‑One First‑Strand cDNA Synthesis SuperMix for qPCR 的说明书配制RT‑qPCR 反应液,42℃孵育15 min,85℃灭活5 s,得到cDNA,‑20℃冰箱保存备用。
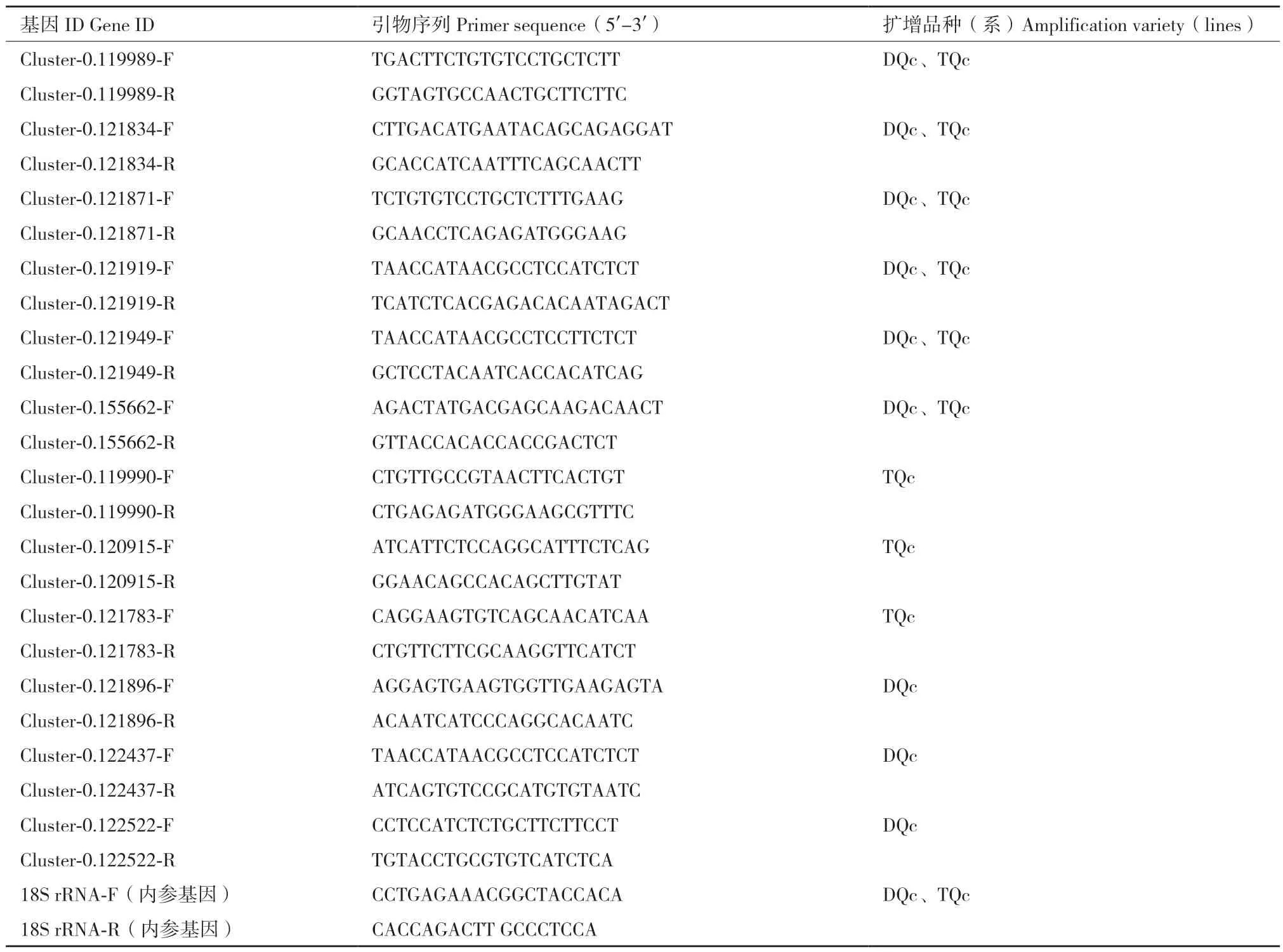
表1 RT-qPCR 引物序列Table 1 Primer sequences for RT-qPCR
根据Real Universal Color PreMix(SYBR Green)操作规程,用LightCycler96 仪进行RT‑qPCR 扩增。以18S rRNA 基因为内参,反应体系:体系总容量20 μL,含10.0 μL 2× PerfectStart® Green qPCR SuperMix(+Dye II),2.0 μL 样品cDNA,上游和下游引物各0.4 μL(20 μmol/L),ddH2O 7.2 μL;反应程序:94℃预变性30 s;94℃变性5 s;60℃退火和延伸30 s,40个循环,4℃保存,每个样品重复3 次,利用溶解曲线检测引物的特异性,按照2-ΔΔCT公式计算基因相对表达量[24]。
1.2.9 数据分析 PCA 分析采用SIMCA14.1 软件分析;层次聚类分析、柱状图、差异代谢物热图、KEGG 气泡图、韦恩图等采用Origin Pro 2021 软件分析制作;代谢路径图采样Adobe Illustrator CC 2017 SP 软件完成。DEGs 加权基因共表达网络构建和分析以R 语言软件中的 WGCNA 包进行,目标模块内基因互作网络可视化使用Cytoscape 3.9.1 软件。
2 结果
2.1 橄榄果实总酚含量差异分析
‘子阳1 号’(SP)与‘东山长穗’(DQ)随着果实的成熟,总酚含量呈上升到趋于平缓的趋势,成熟前期(80 DAF)两品种(系)总酚含量差异不大(P >0.05),后期SP 总酚含量上升趋势快于DQ,成熟时表现显著高于DQ(P<0.05)(图1)。4个品种(系)成熟果总酚含量SP 含量显著高于其他3 个品种(系)(P<0.05);HP 显著高于TQ 与DQ(P<0.05);TQ 与DQ 含量差异不显著。

图1 橄榄果实成熟过程及成熟期总酚含量Fig.1 Total phenol content in Chinese olive fruit ripening process and maturity period
为方便分析,把鲜食品质差的‘惠圆’(HP)、‘子阳1 号’(SP)称为高酚橄榄,鲜食品质好的‘甜榄1 号’(TQ)、‘东山长穗’(DQ)称为低酚橄榄。
2.2 RNA‑Seq序列拼接与功能注释
序列拼接得到296 314 条Unigene,平均长度为1 163 bp,N50 为1 764 bp,超过2/5 的序列(123 611条Unigene)长度超过1 000 bp(表2)。

表2 橄榄转录组序列拼接结果Table 2 Sequence splicing results of Chinese olive transcriptome
215 062 条Unigene(72.58%)在NR 数据库中获得功能注释的基因中,通过NR 数据库比对发现(图2),与橄榄同源性最高的序列主要为甜橙(Citrus sinensis)、克莱门氏小柑橘(Citrus clementina)、蜜柑(Citrus unshiu)、葡萄(Vitis vinifera)。

图2 橄榄Unigene 基于NR 数据库比对物种分布Fig.2 Species distribution of Chinese olive Unigene based on NR database alignment
2.3 转录组数据质量与差异基因筛选
转录组数据根据样本基因FPKM 表达量计算24个样本的相关性,统计结果见图3,重复样本间具有较高的相关性,相关性系数均大于0.8,说明样本转录组数据重复性好。
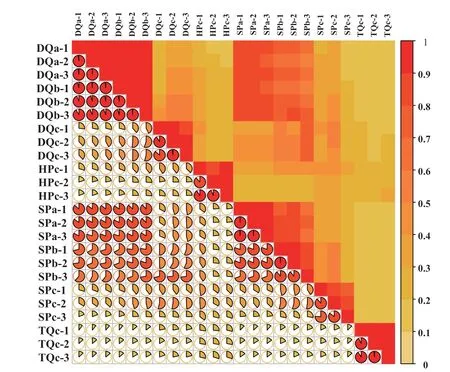
图3 橄榄各样本转录组数据相关性热图Fig.3 Correlation heatmap of transcriptome data of Chinese olive samples
2.4 不同品种(系)橄榄成熟果差异基因分析
根据24 个样品的转录水平,将至少在某一个样品中FPKM > 0.1 的基因定义为表达基因。为筛选鲜食品质差异显著(高酚/低酚)橄榄在转录组水平上的差异,对高酚低酚橄榄成熟果的表达基因进行DEGs 筛选,筛选条件为|log2FC|≥1,且P <0.05,结果如图4‑A 所示,22 631、17 313、19 763、16 958 个DEGs 在SPc 与DQc、SPc 与TQc、HPc 与DQc、HPc 与TQc 间呈现差异表达,其中低酚橄榄相对于高酚橄榄分别有11 360/11 271(SPc vs DQc)、8 036/9 277(SPc vs TQc)、9 802/9 961(HPc vs DQc)和7 778/9 180(HPc vs TQc)个基因显著上调/下调表达。进一步通过维恩图分析,见图4‑B,发现存在1 628 个共有DEGs,并进行KEGG 通路富集分析,富集结果如图4‑C,在“初级代谢”“乙醛酸和二羧酸代谢”“类黄酮生物合成”“过氧化物酶体”“类胡萝卜素生物合成”等通路中显著富集(P<0.05)。

图4 不同品种(系)橄榄差异基因与KEGG 富集分析Fig.4 Differential genes and KEGG enrichment analysis of Chinese olive in different varieties(lines)
2.5 不同品种(系)橄榄成熟过程差异基因分析
基于q 值与差异倍数的双重筛选低酚橄榄DQ与高酚橄榄SP 成熟过程差异表达的基因数量。高酚橄榄SP 在成熟过程19 479、25 854、12 871 个基因分别在SPa 和SPb、SPa 和SPc、SPb 和SPc 中呈现差异表达,取并集得到38 277 个基因在果实成熟阶段差异表达;低酚橄榄‘东山长穗’(DQ)在成熟过程2 946、25 929、22 635 个基因分别在DQa 和DQb、DQa 和DQc、DQb 和DQc 中呈现差异表达,取并集得到33 418 个基因在果实成熟阶段差异表达(图5‑A、B)。

图5 不同品种(系)橄榄成熟过程差异基因与KEGG 富集分析Fig.5 Differential genes and KEGG enrichment analysis of different varieties(lines)of Chinese olive ripening process
对SP 和DQ 成熟过程差异基因分别进行KEGG富集分析,分别63.3%(SP)、64.5%(DQ)的差异基因被比对到KEGG 数据库中。富集结果如图5‑C,两品种(系)在成熟过程,代谢通路在“初级代谢”“蛋白质”“碳水化合物代谢”等通路上显著富集(P<0.05);“氨基酸代谢”途径均富集不显著(P>0.05);酚类物质合成相关代谢通路“苯丙烷类生物合成”“异黄酮生物合成”“类黄酮生物合成”中显著富集(P<0.05)。
2.6 不同品种(系)橄榄成熟过程酚类物质合成途径转录表达分析
橄榄酚类代谢物主要包括酚酸、水解单宁、类黄酮(黄酮、黄酮醇、黄烷醇)化合物,它们合成的前体均是经由莽草酸途径生成,同时成熟果共有差异基因在类黄酮生物合成途径中显著富集(图4‑C)。因此为了进一步探究高酚低酚橄榄成熟过程酚类物质的差异形成分子机制,对莽草酸-水解单宁/苯丙烷-类黄酮生物合成通路差异基因进行了表征。文中基因表达量用FPKM 值表示,差异表达倍数描述为某一基因的表达量在同一发育成熟期的低酚橄榄DQ 与高酚橄榄SP 的比值,相对表达谱描述为不同成熟过程经Log2 转化的差异倍数。
高酚低酚不同品种(系)橄榄果实成熟过程莽草酸-水解单宁生物合成途径结构基因的相对表达图谱(图6)发现,莽草酸途径作为苯丙烷途径的共同上游路径,多数合成途径上的差异基因在不同品种(系)有表现上调也有表现下调,并没有表现绝对的高酚橄榄中高于低酚橄榄,如DPS、SKDH、SK、CS 等。UGTs 是水解单宁生物合成途径的关键酶,表征的UGTs 差异基因多数表现为高酚橄榄显著高于低酚橄榄。
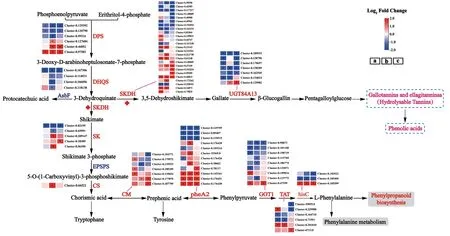
图6 不同品种(系)橄榄成熟过程莽草酸-水解单宁生物合成途径差异基因分析Fig.6 Differential gene analysis of shikimic acid and hydrolyzed tannin biosynthesis pathway in different varieties(lines)of Chinese olive during maturation
高酚低酚不同品种(系)橄榄果实成熟过程苯丙烷-类黄酮生物合成途径结构基因的相对表达图谱(图7)发现,多数合成途径上的差异基因有上调也有下调。PAL 是整个苯丙烷代谢途径的起始酶和限速酶,表征的PAL 差异基因多数表现为高酚橄榄高于低酚橄榄。CHS 是进入类黄酮途径的起始酶和关键酶,也是限速酶,成熟期高酚橄榄的CHS 显著高于低酚橄榄;CHI 是黄酮类化合物生物合成途径上游的关键酶,筛选的多数CHI 差异基因表达量高酚橄榄高于低酚橄榄。F3H、FLS、DFR、ANS、ANR 基因在类黄酮合成阶段的表达有所差异,F3'5'H 在不同品种(系)橄榄的表达水平没有差异;LAR 平在不同品种(系)橄榄的表达水平没有差异,但ANS、ANR 在不同品种(系)表达有高有低,ANS 在低酚橄榄表达量高于高酚橄榄。

图7 不同品种(系)橄榄成熟过程苯丙烷-类黄酮生物合成途径差异基因分析Fig.7 Differential gene analysis in phenylpropanes and flavonoids biosynthesis pathways in different varieties(lines)of Chinese olive during maturation
2.7 酚类代谢基因与转录因子的WGCNA分析
2.7.1 构建差异基因共表达网络 试验中共获得80 981 个DEGs,根据IQR 值过滤50%的基因,选择45 489 个DEGs 进行WGCNA 分析。结果如图8‑A-C,设定软阈值为16,产生19 个共表达模块,除了320 个基因归入M20 模块,代表未获得指定模块外,其余45 169 个均聚类到19 个共表达模块中,一种颜色代表一个模块,模块内基因数量介于145-8 103 之间,权重值介于0.100-0.349;其中模块最大的为M4 模块,包含8 103 个基因,最小的为M12模块,包括145 个基因。
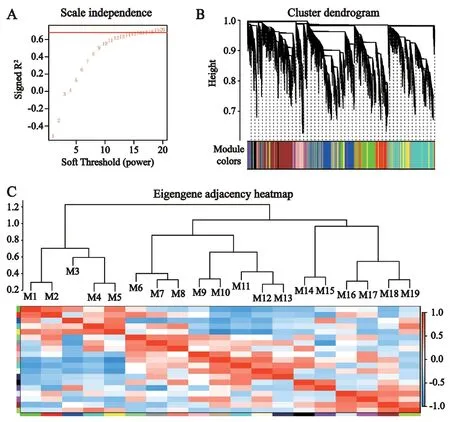
图8 基因共表达网络的可视化Fig.8 Visualization of gene co-expression network
2.7.2 鉴定与酚类化合物相关的模块及转录因子挖掘 为了探究橄榄酚类化合物生物合成的调控机制,结合模块-性状的热图,利用模块内基因的调控关系筛选与模块内莽草酸-水解单宁/苯丙烷-类黄酮代谢途径结构基因共表达的转录因子。根据图6、图7 表征的莽草酸-水解单宁/苯丙烷-类黄酮代谢途径的差异基因,其主要位于M3(20)、M2(19)、M18(17)、M4(13)、M1(8)、M5(4)、M17(3)、M8(3)、M9(1)、M15(1)、M10(1) 和M7(1)模块。结合模块-总酚热图中每个模块与性状的相关性(R2)和P 值,见图9‑A,M3、M4、M5 和M9与总酚的相关性较高,R2均大于0.5,且均达到极显著水平(P<0.01),采用MCC 拓扑分析法,计算这4个模块各节点degree 得分,以degree 值≥1 来筛选关键转录因子,并对这4 个模块关键基因进行网络可视化(图9‑B)。虽然M6 模块与多数酚类化合物具有显著相关性,但该模块内没有莽草酸-水解单宁/苯丙烷-类黄酮代谢路径上的基因。
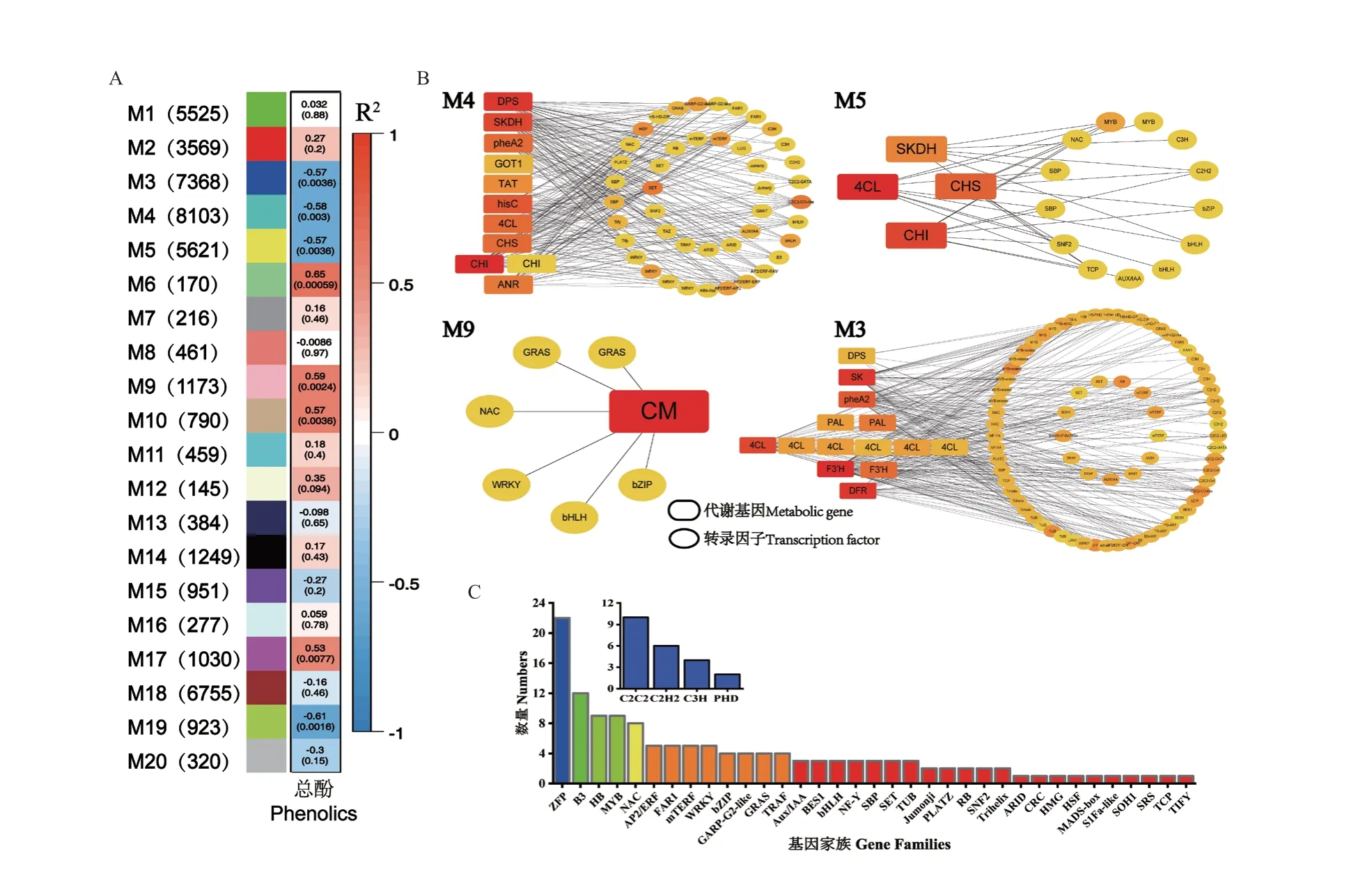
图9 酚类化合物模块鉴定及其相关转录因子挖掘Fig.9 Module identification of phenolic compounds and mining of their associated transcription factors
M3 模块中包含莽草酸途径DPS(1 个)、SK(1个)、pheA2(1 个)基因的Unigene,苯丙烷途径PAL(1 个)、4CL(6 个)基因的Unigene,类黄酮途径F3'H(2 个)基因的Unigene。利用模块内基因的调控关系筛选与上述基因共表达转录因子的Unigene,共筛选到72 个转录因子的Unigene,这些转录因子Unigene 与上述1-9 个基因Unigene 具有一定的连通性,且以B3 类转录因子中的ARF 家族、HB 转录因子、MYB 转录因子为主,其中B3 以ARF家族、HB 以HD‑Zip 家族为主。
M4 模块包含莽草酸途径多个基因的Unigene,DPS(1 个)、SKDH(1 个)、pheA2(1 个)、GOT1(1个)、TAT(1 个)、hisC(1 个);苯丙烷途径4CL(1 个)基因Unigene;类黄酮途径CHS(1 个)、CHI(2 个)、ANR(1 个)基因Unigene。利用模块内基因的调控关系筛选与上述基因共表达的转录因子,筛选到45个转录因子相关的Unigene 与上述基因Unigene 共表达,来自bZIP、bHLH、HB、WRKY 等基因家族。
M5 模块包含莽草酸途径SKDH(1 个)基因的Unigene,苯丙烷途径4CL(1 个)基因的Unigene,类黄酮途径CHS(1 个)、CHI(1 个)基因的Unigene。利用模块内基因的调控关系筛选与上述基因共表达的转录因子,筛选到14 个转录因子的Unigene 与上述基因共表达,来自SBP、MYB、bHLH 等基因家族。
M9 模块包含莽草酸代谢途径上CM(1 个)基因的Unigene。利用模块内基因的调控关系筛选与上述基因共表达的转录因子,筛选到6 个转录因子Unigene 与之共表达,来自GRAS、NAC、WRKY、bHLH、bZIP 基因家族。
综上,共筛选到137 个转录因子Unigene 与莽草酸-水解单宁/苯丙烷-类黄酮生物合成途径的30 个结构基因Unigene 共表达,转录因子Unigene功能注释后来自35 个基因家族,类别统计见图9‑C,其中最多的为锌指蛋白(C2C2、C3H、C2H2、PHD),其次为B3、HB、MYB、NAC 基因家族,其他如bHLH、WRKY、bZIP 等。
2.8 差异表达基因的RT‑qPCR验证
为验证转录组数据的可靠性,结合转录组差异表达基因和WGCNA 分析结果,随机筛选18 个转录组FPKM 值对应的差异基因在不同品种(系)进行RT‑qPCR。结果如图10 所示,RT‑qPCR 与转录组数据中的基因表达水平相关系数为0.822 1(P<0.01),表明转录组分析数据是可靠的。

图10 RNA-Seq 与RT-qPCR 基因表达水平间的相关性Fig.10 Correlation of the gene expression obtained from RNA-Seq and RT-qPCR
3 讨论
3.1 橄榄转录组测序分析
转录组测序为研究橄榄鲜食品质形成及代谢途径与关键基因挖掘提供了丰富的数据基础。本研究共获得了296 314 条Unigene,72.58% Unigene 在数据库中获得功能注释;橄榄没有基因组信息做参考,在NR 数据库中获得功能注释的基因中,同源性最高的序列主要为甜橙(Citrus sinensis)、克莱门氏小柑橘(Citrus clementina),未在公共数据库中得到注释的Unigene,可能是橄榄中特异的编码基因或潜在的长非编码RNA,今后可进行深入探究。
3.2 酚类化合物生物合成
通过RNA‑seq 技术获得的高通量数据可以客观、完整地反映试验样本间的转录表达,许多研究试图通过RNA‑seq 来阐明酚类物质生物合成的分子基础,如通过对不同涩味类型柿子的转录组学和代谢组学分析,确定了参与黄酮类生物合成的关键基因与转录因子[25];通过对山茶花花瓣的阶段性转录组分析,确立了参与黄酮类生物合成的关键基因[26]。本研究4 个品种(系)橄榄成熟果共有DEGs 进行KEGG富集分析发现,在“类黄酮生物合成”途径显著富集,结合影响橄榄鲜食品质的酚类代谢物,包括酚酸、水解单宁、类黄酮(黄酮、黄酮醇、黄烷醇)等,确定与莽草酸-水解单宁/苯丙烷-类黄酮生物合成途径有关的基因。酚类物质的生物合成复杂多样,水解单宁需从莽草酸途径中获得底物[27],类黄酮、酚酸从苯丙烷途径获得底物[28],苯丙烷途径又是经由莽草酸途径合成前体物质[29]。很多编码该途径中酶的结构基因已被鉴定,如SKDH、UGTs、PAL、C4H、4CL、CHS、CHI、F3'H、FLS、F3'5'H等关键基因[30-32]。SKDH 是决定水解单宁生物合成的第一个关键酶,UGTs 负责催化没食子酸糖基化,合成水解单宁的前体物质β-葡聚糖,是水解单宁生物合成途径的关键酶,UGTs 在低酚橄榄表达量低于高酚橄榄,这也导致了高酚橄榄中水解单宁含量较高;PAL 是苯丙烷合成的第一种酶,是整个苯丙烷代谢途径的起始酶和限速酶[33],在酚酸、香豆素、木质素、类黄酮的合成中发挥关键作用[34-35],在鲜食品质差(高酚)的橄榄中多数PAL 基因表达高于鲜食品质好(低酚)的橄榄。类黄酮合成起始于苯丙氨酸,经过苯丙烷类代谢途径催化形成的4‑香豆酰‑CoA 与丙二醛‑CoA 在CHS 和CHI 催化作用下形成黄烷酮,黄烷酮是下游黄酮、黄酮醇、黄烷醇合成之路的同体前体物质[36];CHS 是进入类黄酮途径的起始酶和关键酶,也是限速酶,高酚橄榄CHS 成熟期表达量高于低酚橄榄;CHI 是黄酮类化合物生物合成途径上游的关键酶,对于提高黄酮醇含量有重要作用[21],高酚橄榄CHI 表达量多数高于低酚橄榄。F3H、FLS、DFR、ANS、ANR 基因在类黄酮合成阶段的表达有所差异,黄烷酮在F3H 催化下形成二氢黄酮醇,F3'H 和F3'5'H 催化黄烷醇,其产物是花青素和缩合单宁合成的关键中间体[37],F3'5'H在不同品种(系)橄榄的表达水平没有差异;LAR、ANS 是类黄酮生物合成的下游基因,分别将无色花色素催化成2R,3S‑黄烷‑3‑醇和花青素[38],ANR催化花青素生成2R,3R‑黄烷‑3‑醇[36,39-40],本研究中不同品种(系)橄榄的LAR 表达水平没有差异,但ANS、ANR 在不同品种(系)表达有高有低,ANS 在低酚橄榄表达量高于高酚橄榄,这也导致了高酚橄榄中儿茶素含量较高。
3.3 酚类化合物合成相关转录因子
TF 在植物次生代谢中处于信号转导的关键节点,可通过与基因启动子上特有的顺式作用原件结合,在特定时间和空间激活或者抑制靶基因表达,以实现对靶基因的转录调控[41]。果实酚类物质含量为数量性状[42],多基因假说认为数量性状受多基因控制[43]。许多来自不同基因家族和不同的物种的转录因子被报道参与酚类物质生物合成的转录调控网络,如R2R3‑MYB、NAC、bZIP 和bHLH等[34,44-45]。R2R3‑MYB 是MBW 复合体的核心成员,通过与结构基因启动子区域结合参与调控类黄酮的生物合成[46]。PpMYB17 已经被证实通过激活梨果实中的结构基因PpCHS、PpCHI、PpF3H 和PpFLS来正向调控类黄酮的生物合成[47]。bHLH 可以特异性识别并结合R3 类MYB 特异基序,通过彼此间的相互作用形成二元复合体从而完成对类黄酮合成的调控[48-49]。NAC 调控酚类物质的形成,如MdNAC9与红肉苹果的黄酮醇生物合成密切相关,在苹果愈伤组织中过表达MdNAC9 后黄酮醇含量显著增加[50]。银杏GbbZIP08 和GbbZIP15 能参与银杏花青素生物合成[51];苹果bZIP 类转录因子MdHY5 参与调控苹果花青苷素的合成,并且与MdWRKY41、MdWRKY11、MdMYB 等转录因子互作影响花青苷素的积累合成[52-54]。锌指蛋白转录因子调控酚类物质合成机制研究相对较少[55],在柿果脱涩研究中发现,C2H2 型锌指蛋白在高CO2环境下对柿果实脱涩的调节机制[56]。B3 基因家族主要在调控植物的生长发育、生物与非生物胁迫反应、器官形态建成、开花结实等生物学功能中发挥重要作用[57]。本研究通过WGCNA 挖掘了137 个转录因子Unigene 与莽草酸-水解单宁/苯丙烷-类黄酮生物合成途径的30个结构基因Unigene 共表达,转录因子Unigene 来自35 个基因家族,最多的为锌指蛋白(C2C2、C3H、C2H2、PHD),其次为B3、HB、MYB、NAC 基因家族,其他如bHLH、WRKY、bZIP 等基因家族。
4 结论
4 个品种(系)橄榄成熟果共鉴定到1 628 个DEGs,DEGs 在“类黄酮生物合成”途径显著富集;挖掘到137 个调控酚类物质合成的转录因子Unigene与30 个酚类合成结构基因Unigene 相关,转录因子Unigene 功能注释来自35 个基因家族,最多的为锌指蛋白(C2C2、C3H、C2H2、PHD),其次为B3、HB、MYB、NAC 基因家族。
