环状RNA编码蛋白质在胃肠肿瘤中的研究进展
2024-04-10江杰罗再张昊亮裘正军黄陈
江杰 罗再 张昊亮 裘正军 黄陈
基金项目:国家自然科学基金面上项目(82072662)和上海市级医院临床技能与临床创新三年行动计划(SHDC2020CR4022);第一、二位作者对本文贡献一致
摘要:环状RNA(CircRNA)是一类具有连续、共价闭合、环状结构的单链RNA,生成机制与线性RNA存在较大差异。目前研究已发现部分CircRNA可以通过内部核糖体进入位点、N6-甲基腺苷和滚环翻译等非帽依赖性蛋白翻译机制来编码蛋白质,且编码的蛋白质可进一步通过蛋白诱饵或其他作用机制来调控同源线性蛋白质或下游信号通路,进而发挥生物学功能。已有研究表明CircRNA在各种疾病发生发展中发挥重要作用,特别是参与肿瘤生长增殖、侵袭转移和免疫调节等发生发展过程中。因此,通过阐明编码CircRNA产生的蛋白质在肿瘤发生发展中的表达情况和作用机制,有望为肿瘤诊治提供新的肿瘤标志物和潜在靶点。
关键词:环状RNA;内部核糖体进入位点;蛋白质;蛋白诱饵
中图分类号: R735.2 文献标识码: A 文章编号:1000-503X(2024)01-0072-10
DOI:10.3881/j.issn.1000-503X.15498
Circular RNA-Encoded Proteins in Gastrointestinal Cancer:A Review
JIANG Jie,LUO Zai,ZHANG Haoliang,QIU Zhengjun,HUANG Chen
Department of Gastrointestinal Surgery,Shanghai General Hospital,Shanghai Jiaotong University School of Medicine,Shanghai 200080,China
Corresponding author:HUANG Chen Tel:021-63240090-3121,E-mail:richard-hc@hotmail.com
ABSTRACT:Circular RNAs(CircRNAs)are a class of non-coding RNAs with a covalently closed-loop structure,high stability,and tissue specificity,with the production mechanisms different from linear RNAs.Recent studies have discovered that some CircRNAs can encode proteins via cap-independent translation mechanisms such as internal ribosome entry site,N6-methyladenosine,and rolling loop translation.The encoded proteins regulate homologous linear proteins or downstream signaling pathways via protein bait or other mechanisms,thereby exerting biological functions.Studies have shown that CircRNAs play a role in various diseases,especially in tumor progression,proliferation,invasion,and metastasis and immune regulation.Therefore,by elucidating the expression and roles of proteins encoded by CircRNAs in tumorigenesis and development,this paper is expected to provide new tumor markers and potential targets for tumor diagnosis and treatment.
Key words:circular RNA;internal ribosome entry site;protein;protein bait
Acta Acad Med Sin,2024,46(1):72-81
环状RNA(circular RNA,CircRNA)是一类具有环状结构的共价闭合RNA分子,通常被认为是一种非编码RNA[1-2]。目前关于CircRNA的大部分研究主要聚焦于miRNA海绵和蛋白结合等非编码RNA作用机制,如在最经典的miRNA海绵机制中,CircRNA通过吸附miRNA来抑制miRNA与靶基因的结合,间接调节靶基因表达来发挥作用[3]。随着研究的深入,越来越多的学者发现了在特定情况下可编码蛋白的CircRNA,这为CircRNA的研究提供了新方向。本文将深入探讨CircRNA蛋白编码、与蛋白质交互作用、翻译蛋白质等作用机制以及潜在临床应用。
1 CircRNA的概述
Sanger等[4]于1976年首先发现CircRNA。经典的线性mRNA是由RNA聚合酶Ⅱ转录并剪接去除内含子形成的,CircRNA形成方式则不同,大多是通过反向剪接形成的,即CircRNA外显子上游的3端跨过外显子区域与其下游的5端相拼接,形成5-磷酸二酯鍵闭合的环状结构RNA,小部分CircRNA是因传统剪接中形成的套索结构未降解,而形成闭合的环状结构RNA[5-8]。已有研究表明CircRNA主要在细胞核内产生,随后被运输至不同的作用位点,进而发挥作用,而RNA结合蛋白、参与剪接的蛋白因子和内含子互补元件可以通过互相配对来调节CircRNA的生成速率[7]。含有内含子的CircRNA大多定位在细胞核内,而具有外显子的CircRNA则通过核酸长度依赖的方式运至细胞质内,其中大于1300 nt的CircRNA由人类DExD-box解旋酶39B介导运输[9]。CircRNA降解由核酸内切酶和核酸外切酶介导,尽管反向剪接生成的CircRNA较少,但得益于共价闭合的环状结构,可耐受核酸外切酶,故在时间累积下CircRNA在细胞内有一定的丰度[10-12]。RNA甲基化修饰普遍存在方式是N6-甲基腺苷(N6-methyladenosine,m6A)修饰,m6A修饰的CircRNA可以通过m6A阅读蛋白YTH结构域家族蛋白2和热反应蛋白12被核糖核酸酶P-多药耐药性蛋白复合体降解[13]。除上述降解方式外,在病毒感染的情况下,CircRNA可以由病毒源性的双链RNA激活的核糖核酸酶L降解[14]。CircRNA对于生物体的生长调控、免疫调节和肿瘤发生方面有着重要的作用,调控的作用机制主要包括miRNA海绵机制、蛋白海绵机制和与蛋白互作(如增强、支架、招募作用等)[15]。
随着对CircRNA研究的进展,越来越多的证据表明CircRNA在特定情况下也具有蛋白编码的功能,具有编码功能的CircRNA需要相对应的密码子,类似于其他蛋白编码的mRNA,且序列保守性会比非编码性CircRNA高[16]。与常规线性mRNA的帽依赖性翻译机制不同,目前研究发现的CircRNA存在非帽依赖性的翻译机制,这种非帽依赖性翻译机制也是CircRNA是否能翻译关键之一。
2 CircRNA编码蛋白的机制
mRNA在核内转录后需要进行加工,通常在mRNA的5端处加帽,即5末端7-甲基鸟苷残基帽结构,并于3端被多聚腺苷酸化,生成poly A尾[17]。当mRNA运出细胞核进入胞质进行翻译时,便开启帽依赖性翻译的过程,当帽结合蛋白真核生物翻译起始因子(eukaryotic translation initiation factor,eIF)4E结合至5末端7-甲基鸟苷残基帽结构处,就招募eIF4G和eIF4A组装成eIF4F复合物,然后又和eIF3、eIF1、40S小核糖体亚单位等组合构成43S预启动复合物,从5端非翻译区识别起始密码子,最后与60S大核糖体亚单位结合,开启后续翻译[18]。CircRNA中不存在5末端7-甲基鸟苷残基帽结构,无法以类似mRNA的帽依赖性翻译机制启动翻译,只能以非帽依赖性方式进行翻译,如内部核糖体进入位点(internal ribosome entry site,IRES)、RNA甲基化修饰和滚环翻译介导等的非帽依赖性机制启动翻译[2,19]。
2.1 IRES介导的非帽依赖性翻译起始
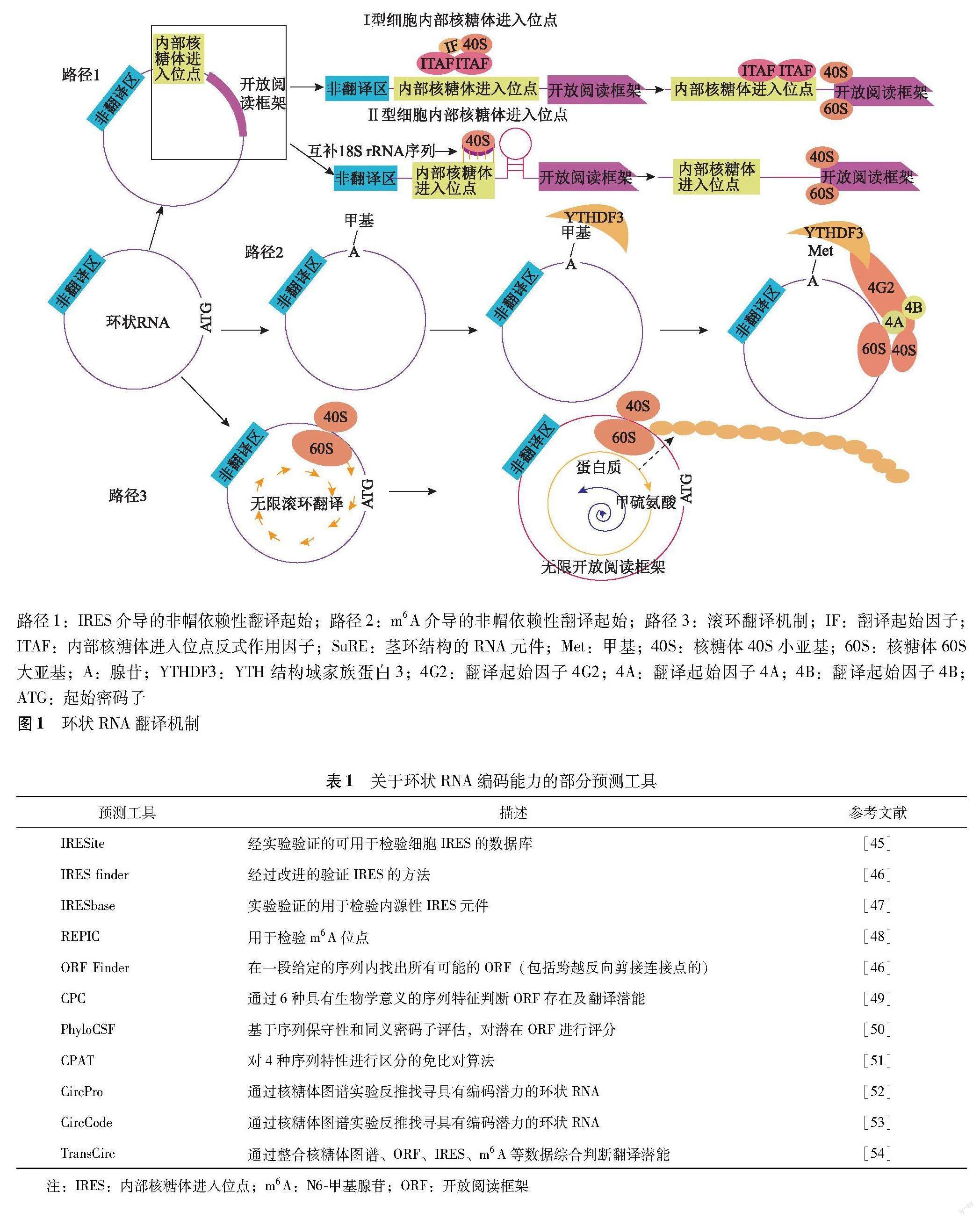
IRES是招募核糖体到mRNA内部区域的一段核酸序列,最早在病毒中发现,IRES介导的翻译有部分还需要翻译起始因子(initiation factor,IF)和IRES反式作用因子(IRES trans-acting factor,ITAF)帮助[20]。通常来说结构越是紧密的IRES需要较少的ITAF和IF进行协助翻译[18]。根据对IF和ITAF的需求,病毒IRES可被分成4组[21]。Ⅰ组病毒IRES通常具有很强的活性,不需要多余的ITAF和IF,仅凭借RNA序列形成假结或茎环结构直接与40S小核糖体亚单位相结合开始翻译,少部分甚至可以从非起始密码子开始翻译[22]。Ⅱ组病毒IRES虽然也可以直接与40S小核糖体相结合,但同时需要几个IF来协调。Ⅲ组和Ⅳ组两组病毒IRES则不能直接与40S小核糖体结合,需要ITAF和IF来招募40S小核糖体亚单位,主要区别是Ⅲ组病毒IRES不需要40S小核糖体识别起始密码子,直接可在IRES位点进行翻译,而Ⅳ组病毒IRES则需要识别起始密码子,才能开始进行翻译[23]。
相较于病毒IRES,细胞内IRES的发现则相对较晚,这是因为细胞IRES介导的翻译通常不如病毒IRES介导的翻译有效,且细胞内IRES介导的翻译还受到多种复杂机制的调控[24-25]。虽然细胞内的IRES发现较晚,但并不少见,任何只要大于50 nt的CircRNA都可能包含1个IRES六聚体存在,由此可进行细胞内IRES介导的翻译[26]。在细胞非应激状态下,IRES位点仅支持低水平的翻译,而在有丝分裂、凋亡、缺氧等应激情况下,帽依赖的细胞翻译机制不能正常运行时,IRES位点则可支持细胞稳定翻译,尽量维持细胞内正常的生理活动[20,25,27]。细胞IRES相较于病毒IRES拥有更少的RNA结构和序列保守性,给细胞IRES的分类带来了一定的难题,细胞IRES大致分为2类:(1)I型IRES是位于RNA上的顺式作用元件,可以与ITAF结合,且与核糖体相互作用促进翻译;(2)Ⅱ型IRES也是位于RNA上的短顺式元件,在距IRES起始位置大约40~60nt左右的位置有1个茎环结构的RNA元件(stem-loop structured RNA element,SuRE)结构,通过核糖体40S亚基中的18S rRNA与IRES相配对来促进翻译[20](图1)。
Ⅰ型IRES通常为一类RNA结合基序,可以募集ITAF,并进一步募集核糖体进行翻译,ITAF大多为RNA结合蛋白,也是在细胞核和细胞质中穿梭的异质核糖核蛋白组,可能与RNA转录加工之间存在干扰调控关系[24,28]。ITAF可以增加IRES和翻译复合物之间的结合力,但具体介导IRES翻译的机制尚不清晰,以下是2个可能的机制:(1)ITAF重塑IRES空间结构,产生更高的亲和力;(2)ITAF可在mRNA和核糖体之间充当桥梁关系,代替IF在mRNA和核糖体之间充当桥梁关系[20,24-25]。已有研究发现关于ITAF作用的9类途径,如伴侣作用、竞争结合、核质易位、启动子依赖性募集、与IF相互作用、与核糖体相作用和作为核糖体固有成分等[29]。也有研究发现,ITAF在细胞内分布的位置和数量是影响IRES活性的重要因素[30]。
Ⅱ型IRES,类似于细菌翻译起始的Shine-Dalgarno序列,帮助招募18S RNA核糖体定位至核酸上[31]。Ⅱ型IRES最大的特征是在距IRES起始位置40~60 nt位置含有SuRE結构,SuRE具有以位置依赖性和结构依赖性方式促进CircRNA非帽依赖性翻译活动的发生,具体机制为当18S rRNA识别到SuRE时,可延缓CircRNA解旋,从而增加IRES上18S rRNA与核糖体18S rRNA互补的机会来促进翻译。SuRE结构为CircRNA特有促进翻译的结构,不存在于线性IRES中[31]。
2.2 m6A介导的非帽依赖性翻译起始
CircRNA含有类似IRES的活性存在的m6A修饰位点共同基序,即RRm6ACH。这些位点主要集中在终止密码子附近和长的内部外显子上,并且在人类和鼠之间高度保守,有研究发现单个m6A位点足以驱动非帽依赖性翻译起始[32]。 在m6A介导的非帽依赖性翻译起始中,YTH结构域家族蛋白3负责读取RNA序列的m6A甲基,读取后招募eIF4G2以及核糖体等,促进翻译进行(图1),甲基转移酶蛋白3/14增强非帽依赖性翻译效率,而去甲基化酶降低非帽依赖性翻译效率。非帽依赖性翻译效率在热休克时上调,这可能是通过YTH结构域家族蛋白2从胞质转移至胞核中阻断了去甲基化酶,提高了m6A水平,从而增强了翻译效率[32]。
2.3 滚环翻译介导的非帽依赖性翻译起始
滚环翻译是指核糖体在CircRNA上无限滚动翻译,从而产生含有大量重复肽段蛋白质的一种翻译机制[33]。Abe等[34]在2013年首次发现病毒CircRNA可通过滚环翻译机制来翻译蛋白质。在2015年,有研究证明真核细胞内CircRNA也可通过滚环翻译机制来翻译蛋白质[35]。不同于IRES和m6A介导的翻译机制,滚环翻译不需要CircRNA内在特殊的启动因子,仅需凭借起始密码子AUG就可以启动翻译[2,19,36-37]。一般来说,可进行滚环翻译的CircRNA的核苷酸数量均为3的倍数[35]。CircRNA没有现成的终止密码子,存在无限开放阅读框架(open reading frame,ORF),但已有研究证明CircRNA滚环翻译可通过程序性-1核糖体移位方式产生终止密码子,最终停止翻译[38-40]。在滚环翻译机制中,CircRNA仅含有起始密码子AUG,且没有终止密码子存在,理论上核糖体可以在无限ORF中进行无限循环翻译,从而产生不同分子量的蛋白质[35](图1)。
3 验证CircRNA编码蛋白的工具和实验
CircRNA通过特定的翻译机制来编码蛋白,在生物体内可能有着十分重要的调控作用。常规对于CircRNA编码能力的验证主要包括生物信息学预测和实验验证。
3.1 基于生物信息学对CircRNA编码能力的预测
CircRNA可以非帽依赖性方式进行翻译,如IRES、m6A和滚环翻译,除此之外,还需有ORF的存在,即从起始密码子至终止密码子的序列存在,才能完成精准翻译。一般来说,可翻译CircRNA的ORF常常是大于100个密码子的序列存在,但进一步研究发现,许多小于100个密码子的短ORF也具有翻译的能力。不同于线性mRNA,在CircRNA中ORF的范围变异性很大,可跨过或不跨过反向剪接连接点,同时,ORF不仅可以有小于一圈的长度存在,还可以有多圈的长度存在。
CircRNA编码能力预测主要是依赖于生物信息学工具对于IRES序列、m6A位点和ORF预测[26,32,41-43]。核糖体图谱常用于对ORF进行定位,在裂解RNA的情况下,依靠核糖体对20~30 nt片段RNA的保护(免于降解),通过鉴定这些片段(又称为核糖体保护片段、核糖体足迹等),配合RNA转录组测序技术可以定位到ORF的准确位置,以此预测CircRNA编码能力[44]。表1总结了对单个或整合CircRNA翻译因素的部分预测工具。
3.2 基于实验对CircRNA编码能力的验证
预测CircRNA的翻译能力后,仍需要实验进一步验证CircRNA编码能力,检验CircRNA与核糖体的结合情况、起始活性以及ORF的翻译是主要的实验验证手段。
路径1:IRES介导的非帽依赖性翻译起始;路径2:m6A介导的非帽依赖性翻译起始;路径3:滚环翻译机制;IF:翻译起始因子;ITAF:内部核糖体进入位点反式作用因子;SuRE:茎环结构的RNA元件;Met:甲基;40S:核糖体40S小亚基;60S:核糖体60S大亚基;A:腺苷;YTHDF3:YTH结构域家族蛋白3;4G2:翻译起始因子4G2;4A:翻译起始因子4A;4B:翻译起始因子4B;ATG:起始密码子
核糖体的验证主要是通过多聚核糖体分析技术,检测结合的核糖体多少来判断CircRNA编码能力,RNA上结合的核糖体越多,在蔗糖密度梯度溶液当中下沉的速率也就越快,RNA被翻译的可能性就越大,收集不同密度层中富集的CircRNA,并进行高通量测序,就可验证CircRNA的编码能力[41-42]。
IRES元件的验证主要是通过测定预测的IRES是否有活性,来判断CircRNA编码能力,常常使用以下两个报告系统:(1)反向切割绿色荧光蛋白片段的单外显子CircRNA报告系统:可以经反向剪接形成完整跨过反向剪接连接点的CircRNA,并编码完整的绿色荧光蛋白,这样可通过检测绿色荧光蛋白的存在来判断IRES是否具有翻译起始活性;(2)完整海肾荧光素酶基因的自剪接报告系统:通过前体RNA体外自剪接形成的CircRNA,并编码完整的绿色荧光蛋白,这样可通过检测转录出来的荧光信号来判断IRES是否具有翻译起始活性,主要用于体外实验[55]。
m6A位点的验证可以通过甲基化RNA免疫沉淀测序直接筛选是否具有m6A修饰位点的存在,来判断CircRNA编码能力[56]。此外,Yang等[32]通过设计针对m6A位点的抗体进行免疫共沉淀实验可以确定m6A位点,从而鉴定含有内源性m6A的CircRNA的存在。
滚环翻译的验证可以通过ORF的序列分析,确定CircRNA是否存在无限ORF,来判断IRES是否具有翻译起始活性[33]。ORF是否能翻译蛋白质也是对CircRNA编码能力的验证方法之一,对于蛋白质的验证而言,质谱分析是最为常用的高通量技术,也是蛋白质组学的金标准[41,43,57]。联合运用质谱分析与RNA转录组测序技术可以有效用于ORF存在的鉴定实验[33,58]。研究者还可根据想要验证的ORF的特异序列设计对应的抗体,进行免疫印迹检测,但应注意当所检测蛋白质浓度过低时,常常会显现出假阴性[59-60]。当不能获取对应ORF抗体时,可以使用CPRIPR-CaS9基因编辑载入插入标记基因(如GFP、Flag),翻译后再利用免疫印迹法或免疫染色检验标记基因表达来验证,滚环翻译机制产生的蛋白质,往往会在Western-blot结果中呈现依据CircRNA翻译的圈数存在一定规律的蛋白条带[39]。除了上述验证实验外,可以通过凝胶电泳和放射自显影检验CircRNA是否能够在体外以35S-甲硫氨酸为原料翻译蛋白质,验证其翻譯能力[41,51]。
4 CircRNA翻译蛋白的潜在作用机制
CircRNA编码蛋白的作用方式分为4类:(1)蛋白诱饵;(2)与线性蛋白作用;(3)非癌性调节;(4)调控信号转导通路[62]。
4.1 蛋白诱饵
CircRNA编码的蛋白与线性编码的蛋白具有同源性,提示两者拥有相似的蛋白作用机制,CircRNA编码蛋白质会与线性同源编码的蛋白质竞争结合功能蛋白,如辅助因子或者效应器,由此蛋白诱饵假说应运而生[43]。CircRNA编码蛋白相对线性同源蛋白短,可能是由于缺少表面屏蔽结构域,易暴露结合位点,从而拥有与线性同源物不一样的活性,发挥更强蛋白诱饵作用[43]。蛋白诱饵假说有以下3个先决条件:(1)诱饵可以通过同源序列与功能蛋白配偶体相结合;(2)功能蛋白至少包含1个结合位点和1个调节位点;(3)诱饵功效取决于配偶体的可及性,即两者的亚细胞定位和相对浓度,因为CircRNA相对含量较少,CircRNA编码蛋白浓度较线性同源蛋白低,所以推测CircRNA编码蛋白具有更强的生物活性[43]。
通过蛋白诱饵机制,CircRNA编码蛋白可以发挥促进或抑制线性同源蛋白的作用。Circ-SHPRH编码蛋白SHPRH-146aa与线性同源蛋白SHPRH具有结构相似性,SHPRH-146aa可以与E3泛素连接酶竞争相互作用来保护SHPRH免受其降解,以此来抑制肿瘤生长[63]。Circ-AKT3由蛋白激酶B第3~7外显子环化而成,编码蛋白激酶B-174aa,蛋白激酶B-174aa作为一种肿瘤抑制因子,与蛋白激酶B竞争性结合磷酸肌醇依赖性蛋白激酶-1,可抑制蛋白激酶B的磷酸化,下调磷脂酰肌醇-3-激酶/蛋白激酶B信号通路,从而抑制肿瘤生长[64]
4.2 与线性蛋白作用
CircRNA编码蛋白可以与线性同源蛋白相作用,发挥生物学作用。CircFGFR1可通过显性负调控的方式来抑制应激条件下的细胞增殖 ,一般情况下成纤维细胞生长因子结合正常FRFR1二聚体胞外端段后,会使胞内段自身磷酸化激酶结构域活化,从而触发下游信号通路,可促进细胞增殖,而CircFGFR1p虽然也定位在细胞膜上,包含成纤维细胞生长因子受体1二聚化和结合结构域,但缺乏激酶结构域,所以当CircFGFR1p与成纤维细胞生长因子受体1相结合时,胞内端仅有1个激酶结构域,无法自身活化,反而发挥显性负调控作用,抑制细胞增殖[31]。CircGprc5a在膀胱肿瘤中高表达并编码CircGprc5a肽发挥作用,G蛋白偶联受体家族C5组成员A是一种在膀胱癌中高度表达CircGprc5a肽的线性同源蛋白,CircGprc5a肽可与G蛋白偶联受体家族C5组成员A结合,具有促进膀胱癌细胞自我更新和转移的功能[65]。
4.3 非癌性调节
指在除癌症之外的生理过程中的调节作用。 CircMbl是由甘露糖结合凝集素第2外显子环化而形成的,通过非翻译区内的IRES介导启动翻译,从而产生CircMbl编码蛋白。CircMbl及其编码蛋白存在于大脑突触体中,可通过叉头框O信号通路调节神经突触功能[62]。Circ-ZNF609及其编码蛋白存在于肌肉组织当中,可特异性控制肌母细胞增殖[66]。
4.4 调控信号转导通路
CircRNA参与调控信号转导通路,影响下游目标,从而调节生物体的生理功能。CircPINT是由长非编码RNA-p53诱导转录本的第2个外显子环化形成,CircPINT编码产生p53诱导转录本-87aa,p53诱导转录本-87aa主要定位在细胞核当中,可通过与聚合酶相关因子复合物结合,使聚合酶相关因子复合物保持在靶基因启动子上发挥调控作用,从而抑制多个癌基因的转录[67]。CircHER2由人类表皮生长因子受体2基因的第3~7个外显子环化形成,在三阴性乳腺癌中显著上调,CircHER2编码蛋白可激活表皮生长因子受体和促进AKT磷酸化来促进肿瘤增殖[68]。Circ-0000437编码人冠蛋白1C-47aa功能肽,人冠蛋白1C-47aa通过与转录因子背景转录相关酸性卷曲蛋白3竞争结合芳香烃受体核转位蛋白,抑制内皮细胞增殖、迁移和分化,从而抑制血管生成和血管内皮生长因子信号传导,具有一定的抗肿瘤作用[69]。
5 CircRNA编码蛋白在肿瘤进展中的作用
CircRNA编码蛋白具有调控细胞生长增殖、侵袭转移、血管生成和免疫调节等功能,CircRNA编码蛋白调控功能失衡是许多疾病演进的基础,已有研究证明CircRNA编码蛋白与肿瘤、心血管等多种疾病相关[3,15,70]。有文献表明CircRNA编码蛋白可以在胃肠肿瘤的发生发展中发挥重要作用,CircRNA编码蛋白不仅可以作为肿瘤标志物用于胃肠肿瘤等的早期筛查和预后预测,也有望作为抗体药物的靶点用于胃肠肿瘤等的治疗[19,62,71-78]。
5.1 CircRNA编码蛋白与胃癌
CircMAPK1可通过IRES位点编码肿瘤抑制因子丝裂原活化蛋白激酶1-109aa,丝裂原活化蛋白激酶1-109aa与丝裂原活化蛋白激酶1竞争性结合,可抑制丝裂原活化蛋白激酶1的磷酸化,从而进一步抑制丝裂原活化蛋白激酶1下游信号通路的活化,发挥抑制胃癌细胞增殖、迁移和侵袭作用。Jiang等[71]发现CircMAPK1的表达水平与胃癌患者预后情况正相关,可作为胃癌患者预后标志物。 CircFNDC3B是由Ⅲ型纤维连接蛋白域蛋白3B基因的外显子5~6环化反剪接而形成的,CircFNDC3B编码一个相对分子质量为25×103的蛋白,可降低E-钙黏蛋白的表达,也可通过形成CircFNDC3B/胰岛素样生长因子II mRNA结合蛋白3/白细胞分化抗原4三元复合物,促进白细胞分化抗原44信使RNA的表达,促进胃癌细胞的增殖和侵袭,与胃癌患者的不良预后相关[72]。CircDIDO1编码蛋白水平同样也与胃癌患者的不良预后相关,如CircDIDO1可以编码一个529aa蛋白,529aa蛋白通过与其线性同源蛋白死亡诱导终结因子1-1a竞争性结合,可抑制多腺苷二磷酸核糖聚合酶活性,影响胃癌患者预后情况[73]。CircAXIN1在胃癌中高表达并编码轴蛋白1-295aa,轴蛋白1-295aa竞争性地与腺瘤性大肠息肉病基因相作用,释放出β-连环蛋白于核内,与启动子的T细胞因子共识位点结合,从而诱导下游基因表达,并激活Wnt信号通路,可以促进胃癌细胞进展,也与胃癌患者的不良預后相关[74]。
5.2 CircRNA编码蛋白与结直肠癌
CircPPP1R12A可编码翻译一种功能蛋白肌球蛋白磷酸酶抗体-73aa,肌球蛋白磷酸酶抗体-73aa可通过激活Hippo-YAP通路,促进结直肠癌细胞增殖、迁移和侵袭作用[75]。CircPLCE1编码的磷脂酶Cε1-411蛋白不仅可抑制结直肠癌细胞增殖和迁移,还可以与热休克蛋白90α/核糖体蛋白S3复合物中的热休克蛋白90α的腺嘌呤核苷三磷酸结构域结合,使核糖体蛋白S3游离出来被泛素依赖性降解,降低核因子-κB核易位,进而抑制了核因子-κB的活性,同样也具有抑制结直肠癌进展的作用[76]。Circ-FNDC3B在结直肠癌中编码一个218aa的蛋白,可抑制锌指转录因子的表达,促进二磷酸果糖激酶1的抗肿瘤作用,抑制结直肠癌进展[62]。CircMAPK14编码的一个175aa的肽链,可通过蛋白诱饵机制竞争性结合人丝裂原活化蛋白激酶6蛋白,从而减少丝裂原活化蛋白激酶14的核易位,并促进叉头框蛋白C1的泛素化降解,同样也具有抑制结直肠癌进展的作用[77]。CircARHGAP35在结直肠癌组织中高度表达,并可以编码一个长达1289aa的蛋白,具有促癌作用[78]。
5.3 CircRNA编码蛋白与其他肿瘤
CircFBXW7的编码蛋白含F-框WD重复域蛋白7-185aa具有抑制胶质母细胞瘤进展的作用,含F-框WD重复域蛋白7-185aa 可以竞争性的结合泛素特异性蛋白酶28,进而避免其线性同源蛋白遭到去泛素化降解,而逃脱的FBXW7a则会诱导c-myc泛素降解,抑制其信号转导并减少癌细胞增殖和转移[79]。此外,CircSHPRH、CircAKT3和CircPINT编码蛋白也在胶质母细胞瘤的进程中起到抑制作用[63-64,67]。CircE-cadherin可以编码出长度为245个氨基酸的C-E-钙黏蛋白,C-E-钙黏蛋白凭借其尾部的14个氨基酸与表皮生长因子受体作用,并促进表皮生长因子受体的磷酸化,从而促进胶质母细胞瘤恶性表型的进展,反而具有促癌作用[80]。CircSMO可編码平滑蛋白-193aa ,平滑蛋白-193aa可以与其线性同源蛋白的N端相结合,促进其胆固醇化,从而促进Hedgehog信号传导,增强胶质母细胞瘤的致癌性,在胶质母细胞瘤中发挥促癌作用[81]。Circβ-catenin编码的β-连环蛋白-370aa可通过蛋白诱饵机制竞争性结合糖原合成酶激酶3β,从而保护β-连环蛋白免受磷酸化和泛素化,而避免降解的β-连环蛋白则会通过介导Wnt/β-连环蛋白途径,促进肝癌的发生发展[82]。CircARHGAP35编码蛋白可通过与细胞核内的反式作用因子Ⅱ-I蛋白相互作用也促进肝癌细胞的进展[78]。CircMRPS35在肝癌中高表达,与顺铂化疗的耐药性高度相关,影响化疗的效果[80]。有研究发现由CircMRPS35编码的CircMRPS35-168aa可以抵消顺铂诱导产生的含半胱氨酸的天冬氨酸蛋白水解酶-3,从而产生耐药性[83]。
6 总结
本综述分别回顾了CircRNA的特性、翻译机制、与CircRNA翻译相关的实验技术、CircRNA编码蛋白的作用机制以及与肿瘤关联的5方面最新进展。随着高通量技术的发展和研究的不断深入,CircRNA编码蛋白为我们打开了人类蛋白质组学的大门,增强了我们对CircRNA在人类肿瘤中重要性的认识,越来越多的证据表明CircRNA可以通过非帽依赖性的方式来翻译蛋白,并在肿瘤等疾病的发生发展中发挥重要作用。尽管CircRNA编码蛋白在肿瘤患者的临床诊断和治疗方面具有巨大的潜力,但下面一系列难题仍未完全厘清:(1)具备翻译潜能的CircRNA特征尚未阐明;(2)CircRNA的其他翻译机制尚待发现;(3)CircRNA编码蛋白的作用机制仍待探索;(4)如何避免无义蛋白干扰,纯化短肽的相关技术尚需完善,待我们进一步研究。
利益冲突 所有作者声明无利益冲突
作者贡献声明 江杰:数据文献搜集阅读并起草文章;罗再:文章选题并修改文章;张昊亮:图表制作并起草文章;裘正军、黄陈:文章设计并修订文章并同意対研究工作诚信负责
参 考 文 献
[1]Mattick JS,Makunin IV.Non-coding RNA[J].Hum Mol Genet,2006,15 Spec No 1:R17-R29.DOI:10.1093/hmg/ddl046.
[2]He L,Man C,Xiang S,et al.Circular RNAs cap-independent translation protein and its roles in carcinomas[J].Mol Cancer,2021,20(1):119.DOI:10.1186/s12943-021-01417-4.
[3]Kristensen LS,Jakobsen T,Hager H,et al.The emerging roles of circRNAs in cancer and oncology[J].Nat Rev Clin Oncol,2022,19(3):188-206.DOI:10.1038/s41571-021-00585-y.
[4]Sanger HL,Klotz G,Riesner D,et al.Viroids are single-stranded covalently closed circular RNA molecules existing as highly base-paired rod-like structures[J].Proc Natl Acad Sci USA,1976,73(11):3852-3856.DOI:10.1073/pnas.73.11.3852.
[5]Lee Y,Rio DC.Mechanisms and regulation of alternative pre-mRNA splicing[J].Annu Rev Biochem,2015,84:291-323.DOI:10.1146/annurev-biochem-060614-034316.
[6]Black DL.Mechanisms of alternative pre-messenger RNA splicing[J].Annu Rev Biochem,2003,72:291-336.DOI:10.1146/annurev.biochem.72.121801.161720.
[7]Chen LL.The expanding regulatory mechanisms and cellular functions of circular RNAs[J].Nat Rev Mol Cell Biol,2020,21(8):475-490.DOI:10.1038/s41580-020-0243-y.
[8]Zhang Y,Zhang XO,Chen T,et al.Circular intronic long noncoding RNAs[J].Mol Cell,2013,51(6):792-806.DOI:10.1016/j.molcel.2013.08.017.
[9]Huang C,Liang D,Tatomer DC,et al.A length-dependent evolutionarily conserved pathway controls nuclear export of circular RNAs[J].Genes Dev,2018,32(9-10):639-644.DOI:10.1101/gad.314856.118.
[10]Enuka Y,Lauriola M,Feldman ME,et al.Circular RNAs are long-lived and display only minimal early alterations in response to a growth factor[J].Nucleic Acids Res,2016,44(3):1370-1383.DOI:10.1093/nar/gkv1367.
[11]Zhang Y,Xue W,Li X,et al.The biogenesis of nascent circular RNAs[J].Cell Rep,2016,15(3):611-624.DOI:10.1016/j.celrep.2016.03.058.
[12]徐昊,方夢蝶,李超,等.新型肿瘤靶标环状RNA的研究进展[J].中国医学科学院学报,2021,43(3):435-444.DOI:10.3881/j.issn.1000-503X.12920.
[13]Lee Y,Choe J,Park OH,et al.Molecular mechanisms driving mRNA degradation by m(6)A modification[J].Trends Genet,2020,36(3):177-188.DOI:10.1016/j.tig.2019.12.007.
[14]Liu CX,Li X,Nan F,et al.Structure and degradation of circular RNAs regulate PKR activation in innate immunity[J].Cell,2019,177(4):865-880.e21.DOI:10.1016/j.cell.2019.03.046.
[15]Wang J,Zhu S,Meng N,et al.ncRNA-encoded peptides or proteins and cancer[J].Mol Ther,2019,27(10):1718-1725.DOI:10.1016/j.ymthe.2019.09.001.
[16]Derrien T,Johnson R,Bussotti G,et al.The GENCODE v7 catalog of human long noncoding RNAs:analysis of their gene structure,evolution,and expression[J].Genome Res,2012,22(9):1775-1789.DOI:10.1101/gr.132159.111.
[17]Das S,Vera M,Gandin V,et al.Intracellular mRNA transport and localized translation[J].Nat Rev Mol Cell Biol,2021,22(7):483-504.DOI:10.1038/s41580-021-00356-8.
[18]Sonenberg N,Hinnebusch AG.Regulation of translation initiation in eukaryotes:mechanisms and biological targets[J].Cell,2009,136(4):731-745.DOI:10.1016/j.cell.2009.01.042.
[19]Meng E,Deng J,Jiang R,et al.CircRNA-Encoded peptides or proteins as new players in digestive system neoplasms[J].Front Oncol,2022,12:944159.DOI:10.3389/fonc.2022.944159.
[20]Yang Y,Wang Z.IRES-mediated cap-independent translation,a path leading to hidden proteome[J].J Mol Cell Biol,2019,11(10):911-919.DOI:10.1093/jmcb/mjz091.
[21]Kieft JS.Viral IRES RNA structures and ribosome interactions[J].Trends Biochem Sci,2008,33(6):274-283.DOI:10.1016/j.tibs.2008.04.007.
[22]Kanamori Y,Nakashima N.A tertiary structure model of the internal ribosome entry site(IRES)for methionine-independent initiation of translation[J].RNA,2001,7(2):266-274.DOI:10.1017/s1355838 201001741.
[23]Pestova TV,Hellen CU,Shatsky IN.Canonical eukaryotic initiation factors determine initiation of translation by internal ribosomal entry[J].Mol Cell Biol,1996,16(12):6859-6869.DOI:10.1128/mcb.16.12.6859.
[24]Komar AA,Hatzoglou M.Internal ribosome entry sites in cellular mRNAs:mystery of their existence[J].J Biol Chem,2005,280(25):23425-23428.DOI:10.1074/jbc.R400041200.
[25]Komar AA,Hatzoglou M.Cellular IRES-mediated translation:the war of ITAFs in pathophysiological states[J].Cell Cycle,2011,10(2):229-240.DOI:10.4161/cc.10.2.14472.
[26]Lei M,Zheng G,Ning Q,et al.Translation and functional roles of circular RNAs in human cancer[J].Mol Cancer,2020,19(1):30.DOI:10.1186/s12943-020-1135-7.
[27]Shatsky IN,Dmitriev SE,Terenin IM,et al.Cap-and IRES-independent scanning mechanism of translation initiation as an alternative to the concept of cellular IRESs[J].Mol Cells,2010,30(4):285-293.DOI:10.1007/s10059-010-0149-1.
[28]Lewis SM,Holcik M.For IRES trans-acting factors,it is all about location[J].Oncogene,2008,27(8):1033-1035.DOI:10.1038/sj.onc.1210777.
[29]Godet AC,David F,Hantelys F,et al.IRES trans-acting factors,key actors of the Stress response[J].Int J Mol Sci,2019,20(4):924.DOI:10.3390/ijms20040924.
[30]Spriggs KA,Bushell M,Mitchell SA,et al.Internal ribosome entry segment-mediated translation during apoptosis:the role of IRES-trans-acting factors[J].Cell Death Differ,2005,12(6):585-591.DOI:10.1038/sj.cdd.4401642.
[31]Chen CK,Cheng R,Demeter J,et al.Structured elements drive extensive circular RNA translation[J].Mol Cell,2021,81(20):4300-4318.e13.DOI:10.1016/j.molcel.2021.07.042.
[32]Yang Y,Fan X,Mao M,et al.Extensive translation of circular RNAs driven by N(6)-methyladenosine[J].Cell Res,2017,27(5):626-641.DOI:10.1038/cr.2017.31.
[33]Liu C,Wu X,Gokulnath P,et al.The functions and mechanisms of translatable circular RNAs[J].J Pharmacol Exp Ther,2023,384(1):52-60.DOI:10.1124/jpet.122.001085.
[34]Abe N,Hiroshima M,Maruyama H,et al.Rolling circle amplification in a prokaryotic translation system using small circular RNA[J].Angew Chem Int Ed Engl,2013,52(27):7004-7008.DOI:10.1002/anie.201302044.
[35]Abe N,Matsumoto K,Nishihara M,et al.Rolling circle translation of circular RNA in living human cells[J].Sci Rep,2015,5:16435.DOI:10.1038/srep16435.
[36]Wawrzyniak O,Zarbska Z·,Kuczyński K,et al.Protein-related circular RNAs in human pathologies[J].Cells,2020,9(8):1841.DOI:10.3390/cells9081841.
[37]Wang Y,Wu C,Du Y,et al.Expanding uncapped translation and emerging function of circular RNA in carcinomas and noncarcinomas[J].Mol Cancer,2022,21(1):13.DOI:10.1186/s12943-021-01484-7.
[38]Perriman R,Ares M Jr.Circular mRNA can direct translation of extremely long repeating-sequence proteins in vivo[J].RNA,1998,4(9):1047-1054.DOI:10.1017/s135583829898061x.
[39]Liu Y,Li Z,Zhang M,et al.Rolling-translated EGFR variants sustain EGFR signaling and promote glioblastoma tumorigenicity[J].Neuro Oncol,2021,23(5):743-756.DOI:10.1093/neuonc/noaa279.
[40]Wen SY,Qadir J,Yang BB.Circular RNA translation:novel protein isoforms and clinical significance[J].Trends Mol Med,2022,28(5):405-420.DOI:10.1016/j.molmed.2022.03.003.
[41]Makarewich CA,Olson EN.Mining for micropeptides[J].Trends Cell Biol,2017,27(9):685-696.DOI:10.1016/j.tcb.2017.04.006.
[42]Sinha T,Panigrahi C,Das D,et al.Circular RNA translation,a path to hidden proteome[J].Wiley Interdiscip Rev RNA,2022,13(1):e1685.DOI:10.1002/wrna.1685.
[43]Zhao H,Zhou Q,Li X.Protein bait hypothesis:circRNA-encoded proteins competitively inhibit cognate functional isoforms[J].Trends Genet,2021,37(7):616-624.DOI:10.1016/j.tig.2021.04.002.
[44]Van Heesch S,Witte F,Schneider-Lunitz V,et al.The translational landscape of the human heart[J].Cell,2019,178(1):242-260.e29.DOI:10.1016/j.cell.2019.05.010.
[45]Mokrejs M,Masek T,Vopálensky V,et al.IRESite--a tool for the examination of viral and cellular internal ribosome entry sites[J].Nucleic Acids Res,2010,38(Database issue):D131-D136.DOI:10.1093/nar/gkp981.
[46]陳新,王鲁泉,黄弋,等.用IRE_FINDER算法在非翻译区搜索人和小鼠的铁反应元件[J].生物化学与生物物理学报,2002,34(6):743-747.
[47]Zhao J,Li Y,Wang C,et al.IRESbase:a comprehensive database of experimentally validated internal ribosome entry sites[J].Genomics Proteomics Bioinformatics,2020,18(2):129-139.DOI:10.1016/j.gpb.2020.03.001.
[48]Liu S,Zhu A,He C,et al.REPIC:a database for exploring the N(6)-methyladenosine methylome[J].Genome Biol,2020,21(1):100.DOI:10.1186/s13059-020-02012-4.
[49]Kong L,Zhang Y,Ye ZQ,et al.CPC:assess the protein-coding potential of transcripts using sequence features and support vector machine[J].Nucleic Acids Res,2007,35(Web Server issue):W345-W349.DOI:10.1093/nar/gkm391.
[50]Lin MF,Jungreis I,Kellis M.PhyloCSF:a comparative genomics method to distinguish protein coding and non-coding regions[J].Bioinformatics,2011,27(13):i275-i282.DOI:10.1093/bioinformatics/btr209.
[51]Wang L,Park HJ,Dasari S,et al.CPAT:coding-potential assessment tool using an alignment-free logistic regression model[J].Nucleic Acids Res,2013,41(6):e74.DOI:10.1093/nar/gkt006.
[52]Meng X,Chen Q,Zhang P,et al.CircPro:an integrated tool for the identification of circRNAs with protein-coding potential[J].Bioinformatics,2017,33(20):3314-3316.DOI:10.1093/bioinformatics/btx446.
[53]Sun P,Li G.CircCode:a powerful tool for identifying circRNA coding ability[J].Front Genet,2019,10:981.DOI:10.3389/fgene.2019.00981.
[54]Huang W,Ling Y,Zhang S,et al.TransCirc:an interactive database for translatable circular RNAs based on multi-omics evidence[J].Nucleic Acids Res,2021,49(D1):D236-D242.DOI:10.1093/nar/gkaa823.
[55]Chen C,Yang Y,Wang Z.Study of circular RNA translation using reporter systems in living cells[J].Methods,2021,196:113-120.DOI:10.1016/j.ymeth.2021.03.011.
[56]Kong S,Tao M,Shen X,et al.Translatable circRNAs and lncRNAs:driving mechanisms and functions of their translation products[J].Cancer Lett,2020,483:59-65.DOI:10.1016/j.canlet.2020.04.006.
[57]Ingolia NT,Ghaemmaghami S,Newman JR,et al.Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling[J].Science,2009,324(5924):218-223.DOI:10.1126/science.1168978.
[58]Slavoff SA,Mitchell AJ,Schwaid AG,et al.Peptidomic discovery of short open reading frame-encoded peptides in human cells[J].Nat Chem Biol,2013,9(1):59-64.DOI:10.1038/nchembio.1120.
[59]Housman G,Ulitsky I.Methods for distinguishing between protein-coding and long noncoding RNAs and the elusive biological purpose of translation of long noncoding RNAs[J].Biochim Biophys Acta,2016,1859(1):31-40.DOI:10.1016/j.bbagrm.2015.07.017.
[60]Kristensen LS,Andersen MS,Stagsted LVW,et al.The biogenesis,biology and characterization of circular RNAs[J].Nat Rev Genet,2019,20(11):675-691.DOI:10.1038/s41576-019-0158-7.
[61]Anderson DM,Anderson KM,Chang CL,et al.A micropeptide encoded by a putative long noncoding RNA regulates muscle performance[J].Cell,2015,160(4):595-606.DOI:10.1016/j.cell.2015.01.009.
[62]Khan FA,Nsengimana B,Khan NH,et al.Chimeric peptides/proteins encoded by circRNA:an update on mechanisms and functions in human cancers[J].Front Oncol,2022,12:781270.DOI:10.3389/fonc.2022.781270.
[63]Zhang M,Huang N,Yang X,et al.A novel protein encoded by the circular form of the SHPRH gene suppresses glioma tumorigenesis[J].Oncogene,2018,37(13):1805-1814.DOI:10.1038/s41388-017-0019-9.
[64]Xia X,Li X,Li F,et al.A novel tumor suppressor protein encoded by circular AKT3 RNA inhibits glioblastoma tumorigenicity by competing with active phosphoinositide-dependent Kinase-1[J].Mol Cancer,2019,18(1):131.DOI:10.1186/s12943-019-1056-5.
[65]Gu C,Zhou N,Wang Z,et al.CircGprc5a promoted bladder oncogenesis and metastasis through Gprc5a-targeting peptide[J].Mol Ther Nucleic Acids,2018,13:633-641.DOI:10.1016/j.omtn.2018.10.008.
[66]Legnini I,Di Timoteo G,Rossi F,et al.Circ-ZNF609 is a circular RNA that can be translated and functions in myogenesis[J].Mol Cell,2017,66(1):22-37.e9.DOI:10.1016/j.molcel.2017.02.017.
[67]Zhang M,Zhao K,Xu X,et al.A peptide encoded by circular form of LINC-PINT suppresses oncogenic transcriptional elongation in glioblastoma[J].Nat Commun,2018,9(1):4475.DOI:10.1038/s41467-018-06862-2.
[68]Li J,Ma M,Yang X,et al.Circular HER2 RNA positive triple negative breast cancer is sensitive to Pertuzumab[J].Mol Cancer,2020,19(1):142.DOI:10.1186/s12943-020-01259-6.
[69]Li F,Cai Y,Deng S,et al.A peptide CORO1C-47aa encoded by the circular noncoding RNA circ-0000437 functions as a negative regulator in endometrium tumor angiogenesis[J].J Biol Chem,2021,297(5):101182.DOI:10.1016/j.jbc.2021.101182.
[70]Wu P,Mo Y,Peng M,et al.Emerging role of tumor-related functional peptides encoded by lncRNA and circRNA[J].Mol Cancer,2020,19(1):22.DOI:10.1186/s12943-020-1147-3.
[71]Jiang T,Xia Y,Lv J,et al.A novel protein encoded by circMAPK1 inhibits progression of gastric cancer by suppressing activation of MAPK signaling[J].Mol Cancer,2021,20(1):66.DOI:10.1186/s12943-021-01358-y.
[72]Hong Y,Qin H,Li Y,et al.FNDC3B circular RNA promotes the migration and invasion of gastric cancer cells via the regulation of E-cadherin and CD44 expression[J].J Cell Physiol,2019,234(11):19895-19910.DOI:10.1002/jcp.28588.
[73]Zhang Y,Jiang J,Zhang J,et al.CircDIDO1 inhibits gastric cancer progression by encoding a novel DIDO1-529aa protein and regulating PRDX2 protein stability[J].Mol Cancer,2021,20(1):101.DOI:10.1186/s12943-021-01390-y.
[74]Peng Y,Xu Y,Zhang X,et al.A novel protein AXIN1-295aa encoded by circAXIN1 activates the Wnt/β-catenin signaling pathway to promote gastric cancer progression[J].Mol Cancer,2021,20(1):158.DOI:10.1186/s12943-021-01457-w.
[75]Zheng X,Chen L,Zhou Y,et al.A novel protein encoded by a circular RNA circPPP1R12A promotes tumor pathogenesis and metastasis of colon cancer via Hippo-YAP signaling[J].Mol Cancer,2019,18(1):47.DOI:10.1186/s12943-019-1010-6.
[76]Liang ZX,Liu HS,Xiong L,et al.A novel NF-κB regulator encoded by circPLCE1 inhibits colorectal carcinoma progression by promoting RPS3 ubiquitin-dependent degradation[J].Mol Cancer,2021,20(1):103.DOI:10.1186/s12943-021-01404-9.
[77]Wang L,Zhou J,Zhang C,et al.A novel tumour suppressor protein encoded by circMAPK14 inhibits progression and metastasis of colorectal cancer by competitively binding to MKK6[J].Clin Transl Med,2021,11(10):e613.DOI:10.1002/ctm2.613.
[78]Li Y,Chen B,Zhao J,et al.HNRNPL circularizes ARHGAP35 to produce an oncogenic protein[J].Adv Sci(Weinh),2021,8(13):2001701.DOI:10.1002/advs.202001701.
[79]Yang Y,Gao X,Zhang M,et al.Novel role of FBXW7 circular RNA in repressing glioma tumorigenesis[J].J Natl Cancer Inst,2018,110(3):304-315.DOI:10.1093/jnci/djx166.
[80]Gao X,Xia X,Li F,et al.Circular RNA-encoded oncogenic E-cadherin variant promotes glioblastoma tumorigenicity through activation of EGFR-STAT3 signalling[J].Nat Cell Biol,2021,23(3):278-291.DOI:10.1038/s41556-021-00639-4.
[81]Wu X,Xiao S,Zhang M,et al.A novel protein encoded by circular SMO RNA is essential for Hedgehog signaling activation and glioblastoma tumorigenicity[J].Genome Biol,2021,22(1):33.DOI:10.1186/s13059-020-02250-6.
[82]Liang WC,Wong CW,Liang PP,et al.Translation of the circular RNA circβ-catenin promotes liver cancer cell growth through activation of the Wnt pathway[J].Genome Biol,2019,20(1):84.DOI:10.1186/s13059-019-1685-4.
[83]Jie M,Wu Y,Gao M,et al.CircMRPS35 suppresses gastric cancer progression via recruiting KAT7 to govern histone modification[J].Mol Cancer,2020,19(1):56.DOI:10.1186/s12943-020-01160-2.
(收稿日期:2023-01-17)
