利用CRISPR/Cas9系统创制水稻品种GW2基因的突变体
2024-04-08颜静宛陈子强周淑芬王锋
颜静宛 陈子强 周淑芬 王锋
摘要:培育具有育种价值的GW2基因编辑的水稻优异新品种在水稻育种中具有重要意义,利用CRISPR/Cas9基因编辑技术,以生产上广泛推广应用的13份水稻品种为材料,对粒质量基因(GW2)进行定向性状改良,通过农杆菌转化创制出一批无T-DNA元件的水稻非转基因GW2突变纯合株系。结果表明:13份T0代水稻转基因中,有28.0%~59.1%植株的GW2基因发生了突变,纯合突变株数量占总突变株数量的35.0%,双等位突变株数量占总突变株数量的14.2%,杂合突变株数量占总突变株数量的50.8%。此外,不同水稻品种发生的突变类型也略有不同。对13份T2代非转基因水稻GW2突变纯合株进行千粒质量性状的考种分析。与对应的野生型亲本品种相比,纯合突变水稻植株的千粒质量显著提高10.81%~58.22%。本研究结果极大地丰富了GW2的突变类型,为不同水稻品种的高产稳产创造了重要的种质资源,同时也为利用基因编辑提高水稻产量提供了有价值的育种信息。
关键词:水稻;CRISPR/Cas9;基因编辑;粒质量;GW2基因;突变
中图分类号:Q344+.14;S511.01 文献标志码:A
文章编号:1002-1302(2024)03-0073-06
水稻(Oryza sativa L.)是世界上种植最广泛的作物之一,是全球超过35亿人的主食,尤其是在亚洲。据预测,到2050年,水稻產量必须增加约42%,才能跟上全球日益增长的粮食需求。粒质量是影响水稻产量潜力的重要农艺性状之一,一直是育种家的主导性状[1]。研究发现,水稻千粒质量每提高1 g,可增加水稻产量400 kg/hm2[2]。因此,增加粒质量对于水稻新品种的进一步高产稳产越来越重要[3]。因此,迫切需要我们运用更精准的技术和方法来创制高产优质的水稻新品种。
近年来,国内外学者纷纷克隆出控制水稻粒型的基因,如GW2[4]、GW5[5]、TGW6[6]、GW8[7]等,其中水稻粒宽GW2基因已被Song等定位在第2号染色体,这是一个主要调控水稻粒宽和粒质量的主效基因,GW2编码的环型E3泛素连接酶通过将其底物靶向到蛋白酶体来调节蛋白水解,从而负向调节细胞的分裂[4]。大粒水稻中氨基酸残基的缺失使得GW2无法结合底物,无法调控泛素介导的蛋白质降解,从而增加谷壳细胞数量,使小穗壳更大、更宽,加快籽粒灌浆速度,增加水稻籽粒的宽度、质量和产量。
基因编辑技术是近几年来快速发展的热门新型技术,该技术通过核酸酶对目标靶基因序列进行精准编辑,经过编辑后的目标序列可产生不同形式的突变类型,如碱基的缺失、插入或替换等形式的突变,这些变异形式都具有可遗传性[8-10]。相比传统的ZFNs、TALENs技术,基因编辑技术具有更精准改良作物的重要农艺性状,可大大缩短培育新品种的时间,有利于基因功能研究和精确分子育种。在基因编辑技术中,CRISPR/Cas9系统由于载体易于构建、编辑效率高等优点,成为当前主流的基因编辑系统[11]。目前,CRISPR/Cas9系统已经被广泛应用于作物上,可以创造出更多、性状更优良的新品种,也可以为解决全球粮食安全问题提供更广阔的前景[12-16]。
本研究主要对生产推广中应用广泛的13份水稻品种进行基因编辑,利用构建的CRISPR/Cas9系统,对粒质量基因(GW2)进行定向改良,通过基因编辑创制出一批优异的非转基因水稻新品种,以期为进一步开展品种设计和育种价值奠定基础。
1 材料与方法
1.1 试验材料
供试受体材料是目前生产上应用广泛的13份水稻品种,分别是:三系保持系50B;恢复系3550R、736R、5466R、615R、华占;常规籼稻品种福香9号、福泰8522、TN05、粤抗1522、福香98;常规粳稻品种台粳9号、李粳2号。试验于2021年5月至2022年12月在福建省农业科学院寿山基地试验田内进行,按照常规方法进行正常田间种植及管理。
1.2 主要试剂
Golden DNA聚合酶,购自北京天根生物科技有限公司;植物Cas9/gRNA质粒构建试剂盒,购自北京唯尚立德生物科技有限公司;引物合成和DNA测序由福州尚亚生物技术有限公司完成;组织培养试剂,购自Sigma公司。
1.3 载体和菌株
供试菌株为大肠杆菌DH5α和农杆菌菌株EHA105,均购自北京博迈德生物有限公司。
1.4 利用CRISPR/Cas9技术构建基因敲除载体
利用植物Cas9/gRNA质粒构建试剂盒(VK005-01)设计引物,试剂盒步骤如下:oligo二聚体的形成;oligo二聚体插入到载体中;转化;阳性克隆的鉴定测序。构建水稻粒质量相关性状的水稻基因编辑载体。
1.5 水稻遗传转化
以13个水稻供试转化受体的成熟胚经诱导后产生的愈伤组织作为转化受体,水稻愈伤组织的诱导、继代及与农杆菌的共培养,抗性愈伤组织的筛选、分化及生根等相关培养基和具体试验步骤参照笔者所在实验室的常用操作方法[17]进行。
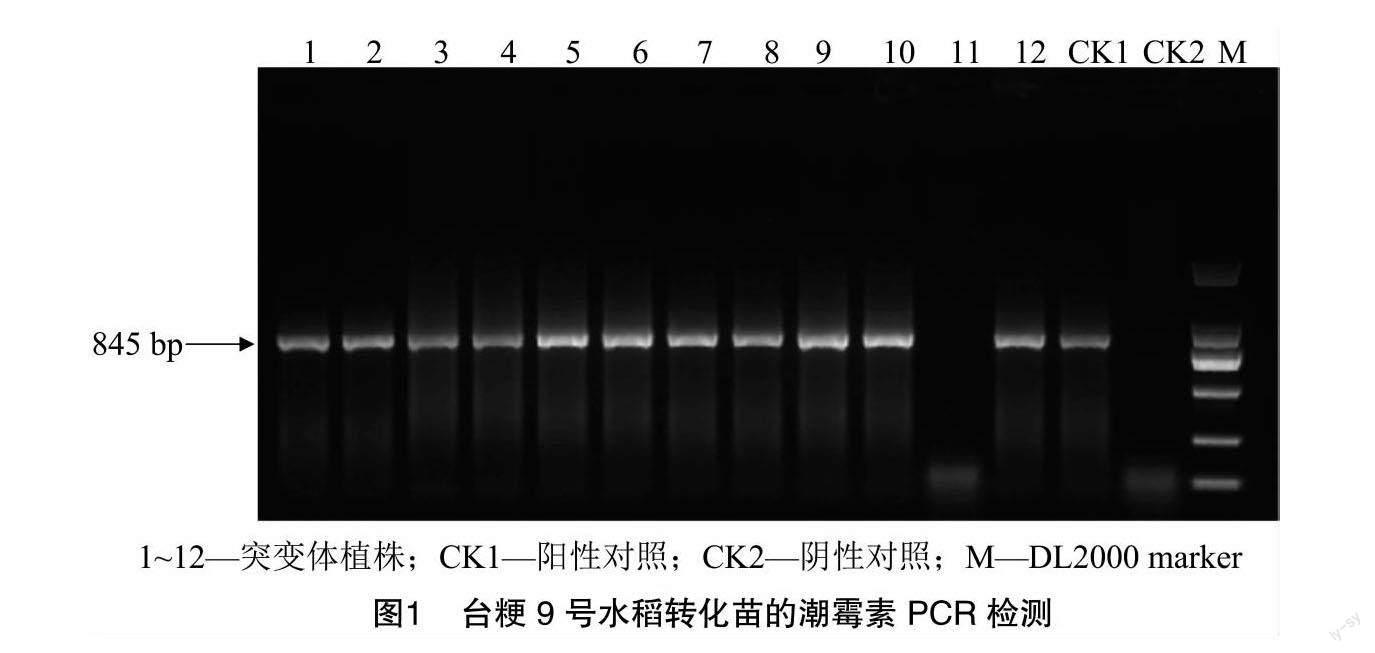
1.6 T0代基因编辑转化体植株的PCR检测
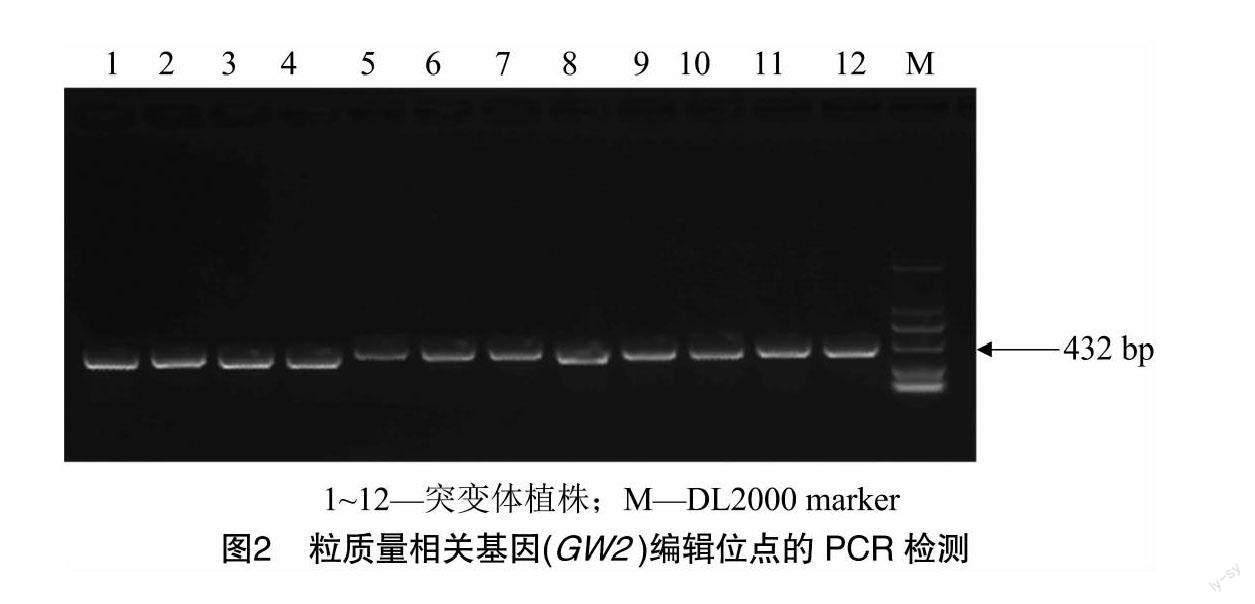
采用十六烷基三甲基溴化铵(CTAB)法提取水稻幼嫩叶片的基因组DNA[18]。设计潮霉素磷酸转移酶基因(Hpt)的PCR引物,引物编号及序列如下:hyg1,5′-TACACAGCCATCGGTCCAGA-3′;hyg2,5′-TAGGAGGGCGTGGATATGTC-3′。以提取的T0代各单株的基因组DNA为模板,通过PCR扩增潮霉素磷酸转移酶基因(Hpt)鉴定转基因植株。PCR扩增程序:94 ℃ 3 min;94 ℃ 45 s,59 ℃ 45 s,72 ℃ 1 min,35个循环;72 ℃,10 min。PCR产物大小为845 bp。同时,设计粒质量的CRISPR鉴定引物:gw2-1F,5′-GGTGAGGGTTTCATCTGCGG-3′;gw2-1R,5′-CCTACTCTCGCAACAACAAG-3′。PCR扩增程序:94 ℃ 3 min;94 ℃ 45 s,59 ℃ 45 s,72 ℃1 min,35个循环;72 ℃,10 min。PCR产物大小为432 bp。
1.7 系统分析T0代基因编辑转化体的序列信息
对13份水稻T0代转化苗进行突变位点的测序,找出双等位突变、杂合突变和纯合突变的克隆,并对突变株的突变类型进行统计分析。
1.8 非转基因水稻GW2突变体的鉴定及其千粒质量性状的考种分析
将PCR测序结果为突变类型的T0代转基因单株经过自交結实后,得到的即为T1代种子。以T1代各单株的基因组DNA为模版,通过引物hyg1/hyg2进行PCR扩增Hpt基因,无目的条带的单株即为无T-DNA元件的GW2突变体。
采用随机区组设计种植无T-DNA元件的T1代纯合GW2突变株系及其对应的亲本对照品种,每个株系种植5行,每行种植7株。株行距为20 cm×20 cm,单本栽插。按照大田常规方法进行田间管理。收获成熟种子烘干后,从中随机数取饱满、无病种子1 000粒,用YTS-5D水稻数字化考种机(武汉谷丰光电科技有限公司)称质量(g),3个重复。数据使用GraphPad Prism软件计算平均值、标准误,采用单因素方差分析进行比较。
2 结果与分析
2.1 T0代转基因水稻植株的PCR检测
用植物Cas9/gRNA质粒构建试剂盒(VK005-01)设计引物,根据试剂盒说明书按步骤操作,进行转化,挑3~5个白色菌落摇菌后进行测序,测序结果均正确。将载体命名为CRISPR/Cas9-GW2。
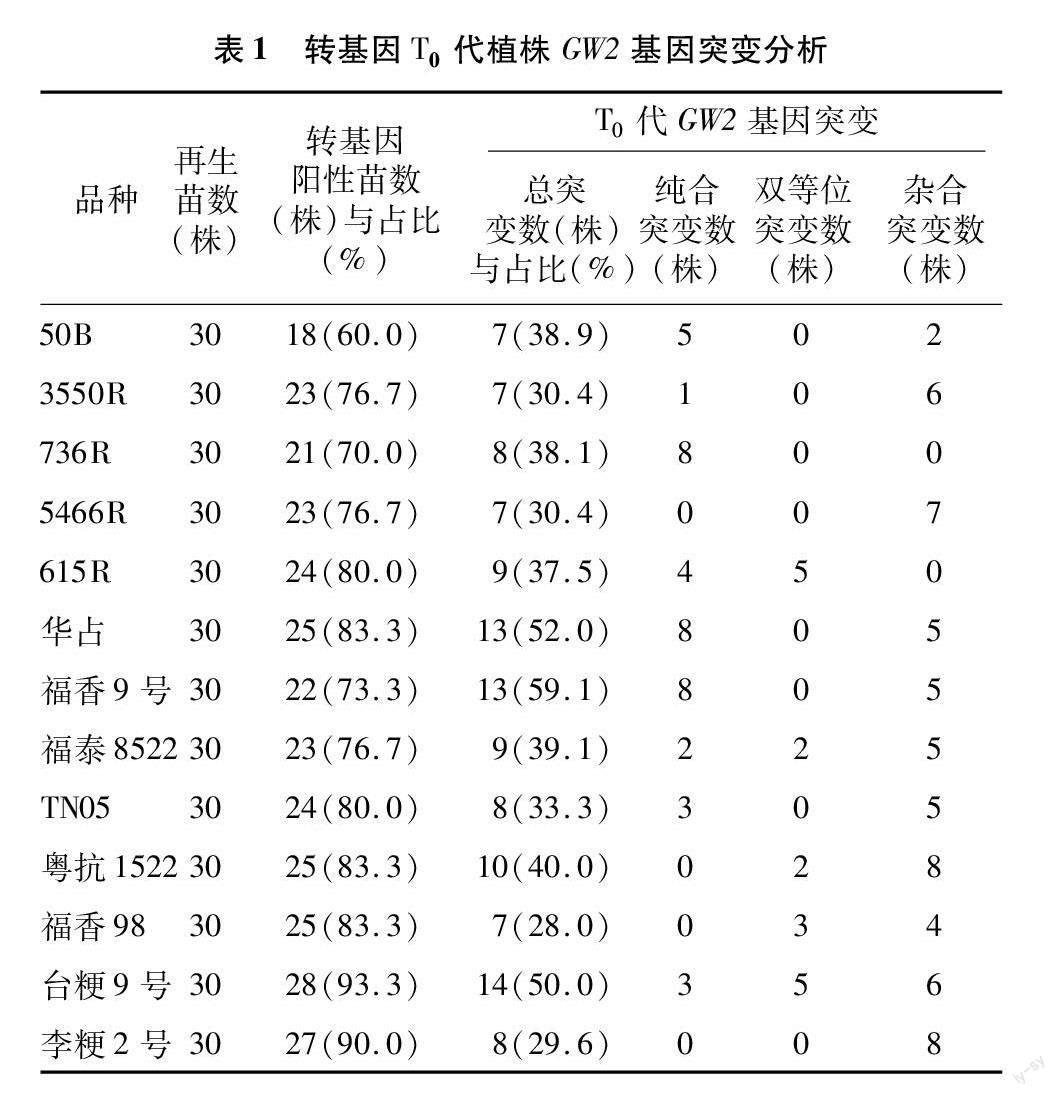
用编辑载体CRISPR/Cas9-GW2转化13个水稻受体材料,每个转化受体各转化30个克隆,分别提取这些克隆水稻转化植株的基因组DNA,用PCR扩增潮霉素基因。从转基因植株中扩增出目的条带,说明目的序列已经整合到水稻基因组中。在13个水稻T0代转化材料中,转基因阳性植株检测结果显示,转化阳性率为60.0%~93.3%(表1)。其中,台粳9号水稻转基因苗中有28个克隆能扩增出845 bp大小的DNA目标条带,而阴性对照则不能扩增出目的基因大小的条带,转化阳性率为93.3%。PCR检测结果如图1所示。
2.2 T0代转基因水稻GW2基因突变类型分析
为了解T0代转基因阳性植株GW2基因的突变情况,用gw2-1F、gw2-1R特异引物PCR扩增上述转基因阳性植株的GW2基因靶位点序列(图2),并对PCR产物进行测序鉴定,分析结果见表1。由统计结果可知,在13份T0代水稻转基因中,有28.0%~59.1%植株的GW2基因发生了突变。纯合突变株占总突变株数的35.0%,双等位突变株占总突变株数的14.2%,杂合突变株占总突变株数的50.8%。不同品种发生的突变类型也略有不同,8株736R突变株只产生1种纯合突变类型;8株李粳2号突变株也只产生1种杂合突变类型;14株台粳9号突变株中却有3株纯合突变株、5株双等位突变株、6株杂合突变株。综上,利用CRISPR/Cas9-GW2质粒是可以有效编辑不同水稻品种的GW2基因。
2.3 非转基因水稻GW2突变体的鉴定及其千粒质量性状的考种分析
将PCR测序结果为突变类型的T0代水稻转基因种子种植,自交结实后得到T1代种子,对其DNA基因组进行潮霉素基因PCR检测,扩增得到没有潮霉素基因的单株,确定为不含T-DNA元件的非转基因GW2突变体,图3列出了台粳9号T1代水稻转化苗的潮霉素PCR检测的结果,进一步种植,自交结实得到T2代种子,对13份非转基因水稻GW2突变纯合株进行千粒质量性状的考种分析。T2代种子的千粒质量统计分析结果见表2,与对应的野生型品种相比,所分析的纯合突变植株都显著提高了水稻的千粒质量,增幅为10.81%~58.22%。其中常规籼稻品种福香9号、 福泰8522、粤抗1522的纯合突变株的千粒质量都比亲本增幅超过50%。
3 讨论与结论
近年来,随着分子生物学的不断发展,基因编辑技术特别是CRISPR/Cas9系统的不断改进,使基因编辑成为一种简单、高效、易使用的多功能靶向遗传操作工具。用CRISPR/Cas9系统来改良作物的性状包括产量、品质及生物和非生物胁迫抗性,有望加速作物改良和提高可持续农业发展的潜力[19]。如在水稻中同时敲除3个与粒质量相关的基因(GW2、GW5和TGW6)会促使籽粒质量聚合,从而大大增加了粒质量[20]。吴明基等通过CRISPR/Cas9编辑Badh2基因来改良优质粳稻品种的香味性状[21]。尹丽颖等利用CRISPR/Cas9技术来创制出高效抗除草剂水稻[22]。Zhang等通过CRISPR/Cas9敲除Waxy,产生了直链淀粉含量低的大米,从而改善了大米的食用和烹饪质量[23]。张元野等利用CRISPR/Cas9基因编辑技术成功获得恢复红种皮表型的纯合株系[24]。本研究利用CRISPR/Cas9基因编辑技术,以生产上推广应用广泛的13份水稻品种为材料,对粒质量基因(GW2)进行定向性状改良,一般有28.0%~59.1%植株的GW2基因发生了突变,能非常有效地改良不同水稻品种的粒质量性状,说明利用CRISPR/Cas9系统是可以快速编辑不同背景不同地域的水稻品种的GW2基因,这也极大地突破和提高了水稻常规育种的进程。但可能由于靶基因、靶位点、脱靶效应等因素的影响,本研究所用的CRISPR/Cas9载体的编辑效率还不是非常高。随着基因编辑器的不断开发,Zeng等开发出一套植物高效广靶向胞嘧啶碱基编辑器PhieCBEs,平均编辑效率提高了3~8倍,展现出稳定的编辑能力[25]。随后,Tan等在2022年又开发了一个强大的高效腺嘌呤碱基编辑器PhieABEs,可以接近100%的编辑效率,在编辑窗口中有高比例的纯合碱基取代[26]。 Wei等也开发出高效的腺嘌呤碱基编辑rABE8e,在水稻中表现出约100%的编辑效率和较高的纯合替代率[27]。这些高效基因编辑器的开发,可实现多种形式精准的基因修饰,满足不同的基因编辑需求,从而为我们今后进一步研究基因功能和分子育种奠定了基础。
水稻产量是水稻重要的农艺性状,籽粒大小是决定水稻产量高低的重要因素之一,它不仅是粮食产量的组成部分,而且是决定市场价值的品质性状。本研究利用CRISPR/Cas9基因编辑技术对不同水稻背景下的GW2位点进行敲除,增产效果明显,达到了预期的目的。发现不同水稻背景的突变类型略有不同,可发生单碱基或多碱基的缺失或插入,所产生不同的突变类型其千粒质量的增产各不相同,即便不同水稻品种产生相同位点的突变类型,其千粒质量的增产也各不相同。在GW2基因同一个位点发生腺嘌呤单碱基的缺失(-A),恢复系品种3550R的千粒质量增加10.81%,华占的千粒质量增加17.42%;常规籼稻福泰8522的千粒质量增加26.51%,福香9号的千粒质量增加38.63%;而常规粳稻台粳9号的千粒质量增加30.07%。而在GW2基因同一位点发生胞嘧啶单碱基的插入(+C),保持系50B的千粒质量增加36.32%,常规籼稻粤抗1522的千粒质量则增加58.22%。由此推测,这些不同背景的水稻品种突变后导致氨基酸移码突变,从而使转录提前终止,编码的蛋白跨膜螺旋结构域发生了改变,从而导致GW2无法结合底物,代谢途径可能存在显著差异,因此当GW2突变后,不同品种的GW2突变体加快谷粒灌浆的速度产生了极显著差异。GW2基因是一种遗传决定因子,是今后选育良种的理想遗传资源,可用于定向改良更多生产上主推的水稻品种。此外,需要一个全面综合的研究策略,通过多重基因组编辑系统构建更多有利的遗传决定因素,以便有目的地快速培育高产优质的水稻品种,以应对和保障未来的粮食安全。
参考文献:
[1]Niu Y N,Chen T X,Wang C C,et al. Identification and allele mining of new candidate genes underlying rice grain weight and grain shape by genome-wide association study[J]. BMC Genomics,2021,22(1):602-615.
[2]符福鸿,王 丰,黄文剑,等. 杂交水稻谷粒性状遗传分析[J]. 作物学报,1994,20 (1):39-45.
[3]王松凤,李 辉,刘 喜,等. 水稻粒形相关性状及千粒重QTL的稳定性分析[J]. 南京农业大学学报,2008,31(3):1-7.
[4]Song X J,Huang W,Shi M,et al. A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase[J]. Nat Genet,2007,39(5):623-630.
[5]Weng J F,Gu S H,Wan X Y,et al. Isolation and initial characterization of GW5,a major QTL associated with rice grain width and weight[J]. Cell Research,2008,18(12):1199-1209.
[6]Ishimaru K,Hirotsu N,Madoka Y,et al. Loss of function of the IAA-glucose hydrolase gene TGW6 enhances rice grain weight and increases yield[J]. Nat Genet,2013,45(6):707-711.
[7]Wang S K,Wu K,Yuan Q B,et al. Control of grain size,shape and quality by OsSPL16 in rice[J]. Nat Genet,2012,44(8):950-954.
[8]Shan Q W,Wang Y P,Li J,et al. Targeted genome modification of crop plants using a CRISPR-Cas system[J]. Nat Biotechnol,2013,31(8):686-688.
[9]Li J F,Norville J E,Aach J,et al. Multiplex and homologous recombination-mediated genome editing in Arabidopsis and Nicotiana benthamiana using guide RNA and Cas9[J]. Nat Biotechnol,2013,31(8):688-691.
[10]Xie K B,Yang Y N. RNA-guided genome editing in plants using a CRISPR-Cas system[J]. Molecular Plant,2013,6(6):1975-1983.
[11]劉 妮,陆 沁,刘 娟,等. CRISPR/Cas系统最新研究进展[J]. 生物技术通报,2017,33(2):53-58.
[12]Wu M J,Liu H Q,Lin Y,et al. In-frame and frame-shift editing of the Ehd1 gene to develop japonica rice with prolonged basic vegetative growth periods[J]. Front Plant Sci,2020,11:307-320.
[13]Shan Q W,Wang Y P,Li J,et al. Genome editing in rice and wheat using the CRISPR/Cas system[J]. Nature Protocols,2014,9(10):2395-2410.
[14]Lu Y M,Ye X,Guo R M,et al. Genome-wide targeted mutagenesis in rice using the CRISPR/Cas9 system[J]. Mol Plant,2017,10(9):1242-1245.
[15]Zhang S J,Zhang R Z,Gao J,et al. Highly efficient and heritable targeted mutagenesis in wheat via the Agrobacterium tumefaciens-mediated CRISPR/Cas9 system[J]. Int J Mol Sci,2019,20(17):4257-4275.
[16]Zhang Y L,Ma X L,Xie X R,et al. CRISPR/Cas9-based genome editing in plants[J]. Prog Mol Biol Transl Sci,2017,149:133-150.
[17]苏 军,胡昌泉,翟红利,等. 农杆菌介导籼稻明恢86高效稳定转化体系的建立[J]. 福建农业学报,2003,18(4):209-213.
[18]Murray M G,Thompson W F. Rapid isolation of high molecular weight plant DNA[J]. Nucleic Acids Research,1980,8(19):4321-4325.
[19]Chen K L,Wang Y P,Zhang R,et al. CRISPR/Cas genome editing and precision plant breeding in agriculture[J]. Annual Review of Plant Biology,2019,70(1):667-697.
[20]Xu R F,Yang Y C,Qin R Y,et al. Rapid improvement of grain weight via highly efficient CRISPR/Cas9-mediated multiplex genome editing in rice[J]. Genet Genomics,2016,43(8):529-532.
[21]吳明基,林 艳,刘华清,等. CRISPR/Cas9编辑Badh2基因改良优质粳稻品种香味性状[J]. 福建农业学报,2020,35(5):465-473.
[22]尹丽颖,张元野,李荣田,等. 利用CRISPR/Cas9 技术创制高效抗除草剂水稻[J]. 中国水稻科学,2022,36(5):459-466.
[23]Zhang J S,Zhang H,Botella J R,et al. Generation of new glutinous rice by CRISPR/Cas9-targeted mutagenesis of the Waxy gene in elite rice varieties[J]. J Integr Plant Biol,2018,60(5):369-375.
[24]张元野,尹丽颖,李荣田,等. 利用CRISPR/Cas9技术创制Rc基因恢复红稻[J]. 中国水稻科学,2022,36(6):572-578.
[25]Zeng D C,Liu T L,Tan J T,et al. PhieCBEs:plant high-efficiency cytidine base editors with expanded target range[J]. Molecular Plant,2020,13(12):1666-1669.
[26]Tan J T,Zeng D C,Zhao Y C,et al. PhieABEs:a PAM-less/free high-efficiency adenine base editor toolbox with wide target scope in plants[J]. Plant Biotechnol J,2022,20(5):934-943.
[27]Wei C,Wang C,Jia M,et al. Efficient generation of homozygous substitutions in rice in one generation utilizing an rABE8e base editor[J]. J Integr Plant Biol,2021,63(9):1595-1599.
