鹅源副鸡禽杆菌全基因组重测序及比较基因组学分析
2024-04-02苏文楠刘佳琪钟嘉诚陈济铛朱婉君张溢珊张济培
苏文楠,刘佳琪,钟嘉诚,陈济铛,朱婉君,张溢珊,张济培,*
(1.佛山科学技术学院,佛山 528225;2.佛山天牧生物科技有限公司,佛山 528225)
副鸡禽杆菌(Avibacteriumparagallinarum,Apg)属于巴氏杆菌科禽杆菌属,是一种无鞭毛、无芽胞的革兰阴性菌,可引起鸡传染性鼻炎。该菌不属于泛嗜性细菌,在以往文献报道中,除了鸡以外,甚少有其它动物感染Apg的报道,即使可以从其他动物上分离到Apg,也无法用本体动物复制出相同的症状和病理变化[1]。但1981年Reece等[2]报道了日本地区的鹌鹑群体出现疑似传染性鼻炎病例,通过对病原体分离鉴定,证实为Apg感染所致,之后该病也出现在印度尼西亚,成为当地鹌鹑群体中的一种常见病,从鹌鹑分离的菌株重新接种回本体动物,可以复制出同样的临床症状和病理变化[3-5],说明Apg已开始出现跨禽种传播的趋势。2020年初,钟嘉诚等[6]从广东地区发生传染性鼻炎的种鹅中分离出了一株Apg,经过动物回归试验证实了该菌株对鹅具有致病性。目前关于Apg引起鹅传染性鼻炎的报道不多,鲜有对于鹅源Apg的研究报道,对于鹅源Apg的来源以及与鸡源Apg基因组之间的异同仍不清楚。因此本研究对3株鹅源Apg和4株鸡源Apg菌株进行全基因测序及基因组分析,通过对基因组进行基于核心基因SNPs的同源性分析来了解其系统进化关系,并根据宿主源和致病性的差异对菌株归类,利用同源基因分析比较不同宿主源Apg之间的基因差异,以探讨Apg对不同宿主的感染机制。
1 材料与方法
1.1 菌株
由本实验室分离保存的3株鹅源Apg(G-AP1、G-AP2、G-AP3)及4株鸡源Apg(C-AP1、C-AP2、C-AP3、C-AP4),菌株的具体信息见文献[7](表1)。
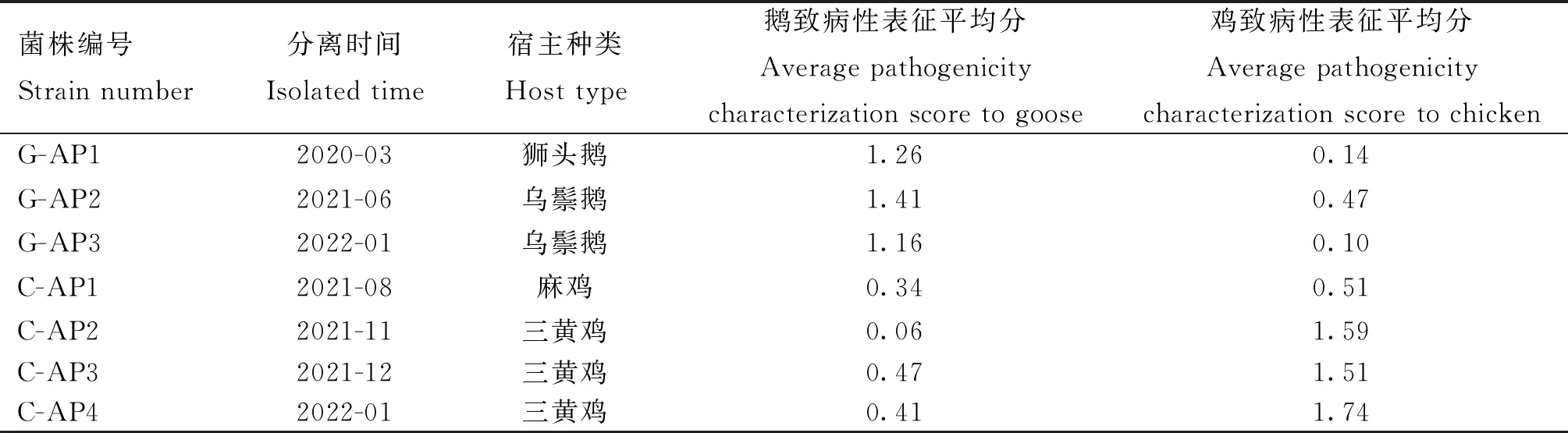
表1 菌株信息
1.2 主要试剂及分析软件
主要试剂:Ezup柱式细菌基因组DNA抽提试剂盒,生工生物工程(上海)有限公司。
分析软件:测序数据质量评估软件FastQC(version 0.11.9),测序数据过滤软件Trimmomatic(version 0.39),基因组序列拼接软件Soapdenovo2(version 2.40),基因组注释软件prokka(version 1.14.6),圈图绘制软件CGView,序列比对相关软件MEGA7、BLAST+(version 2.13.0)、BRIG(version 0.95),SNP分析软件kSNP3(version 3.1.2),基因同源性分析软件OrthoFinder(version 2.3.12),画树工具FastTree。
1.3 菌株基因组DNA的提取
参照Ezup柱式细菌基因组DNA抽提试剂盒说明书对7株菌株进行细菌基因组的提取,干冰冻存送至生工生物工程(上海)股份有限公司完成测序工作。
1.4 基因组重测序与组装
7株Apg菌株的全基因组重测序工作由生工生物工程(上海)股份有限公司完成,采用二代测序平台Illumina PE150 进行全基因组测序。将测序的原始数据(raw data)通过FastQC软件作序列数据的质量评估[4],利用Trimmomatic软件进行过滤处理获得有效数据(clean data);将有效数据利用Soapdenovo2软件[8]进行denovo组装后获得高质量的Contig片段,并进行序列补洞与修正,最终获得基因组扫描图。
1.5 一般生物信息学分析
采用Prokka软件[9]对基因组扫描图进行基因元件(编码基因、tRNA、rRNA等)预测,Prokka软件根据COG等数据库对编码蛋白进行功能预测与注释;基因岛通过IslandViewer 4在线预测,以副鸡禽杆菌ESV-135(登录号为NZ_CP050 316.1)作参照基因组;噬菌体序列由PHASTER[10]在线预测;利用VFDB数据库(Virulence Factor Database)作毒力基因的预测。使用CGView软件绘制基因组圈图,并将基因岛、前噬菌体信息整合到圈图中。
1.6 基于核心基因SNPs的系统发育分析
运用kSNP3软件[11],以Apg菌株ESV-135基因组作参考基因组,分别与本试验的7株菌株基因和NCBI已公布的42个Apg基因组进行比对,获得所有菌株的SNPs(单核苷酸多态性位点),根据核心基因SNPs,通过ML法(最大似然法)进行系统发育分析。
1.7 同源基因分析
1.7.1 7株菌株基因组的同源性分析 提取7株菌株基因组的蛋白质序列文件,运用OrthoFinder软件[12]分析各个基因组之间共有和特有的基因。
1.7.2 鹅源菌株与鸡源菌株基因组的同源性分析 为了解该地区的鹅源菌株群体与鸡源菌株群体之间的同源基因分布情况,将本试验的3株鹅源菌株设为A组,4株鸡源菌株设为B组,分别对二组进行同源基因分析,随后将鹅源菌株共有基因与鸡源菌株共有基因进行聚类分析,从而获到二者的特有基因。
1.7.3 Apg对鹅致病基因的筛查 7株Apg对鹅的致病性表征评分分别为G-AP1(1.26)、G-AP2(1.41)、G-AP3(1.16)、C-AP1(0.34)、C-AP2(0.06)、C-AP3(0.47)、C-AP4(0.41),C-AP2株对鹅的致病力最弱,且在鹅机体内停留的时间最短,而鹅源Apg对鹅的致病性最强,另外3株鸡源Apg次之[7]。为探究是否存在有助于Apg对鹅致病的相关基因,分别将低毒力的C-AP2株基因组设为A组,高毒力的鹅源菌株(G-AP1、G-AP2、G-AP3)和中等毒力的鸡源菌株(C-AP1、C-AP3、C-AP4)的基因组集合设为B组,通过对A组和B组进行同源性分析比较,获得B组特有基因。
1.7.4 Apg对鸡致病基因的筛查 从Apg对鸡的致病性表征表征评分分别为G-AP1(0.14)、G-AP2(0.47)、G-AP3(0.1)、C-AP1(0.51)、C-AP2(1.59)、C-AP3(1.51)、C-AP4(1.74),所有鹅源菌株和鸡源菌株G-AP1对鸡致病性弱,感染鸡没有明显的临床表征[7],为了寻找有助于Apg对鸡致病的相关基因,将低毒力鹅源菌株(G-AP1、G-AP2、G-AP3)和中等毒力鸡源菌株C-AP1基因组集合设为A组,3株高致病性鸡源菌株(C-AP2、C-AP3、C-AP4)的基因组集合设为B组,通过对A组与B组进行同源性分析比较,获得B组特有基因。
2 结 果
2.1 菌株基因组分析
2.1.1 基因组分析 7株Apg全基因组原始测序结果经数据过滤校正后,通过组装处理,得到7个大小为2.567 339~2.637 621 Mb的基因组片段,GC含量介于40.79%~41.02% 之间,分别预测到2 481~2 624个蛋白编码基因(protein coding sequences,CDSs)、tRNA 53~55个、rRNA 5~9个;利用IslandViewer4对菌株进行基因岛预测,分别预测到9~14个基因岛;通过PHASTER在线预测,可见菌株带有5~7个前噬菌体序列(详见OSID “开放科学内容与数据”)(表2)。鹅源菌株与鸡源菌株的全基因组相似,结果无显著差异。
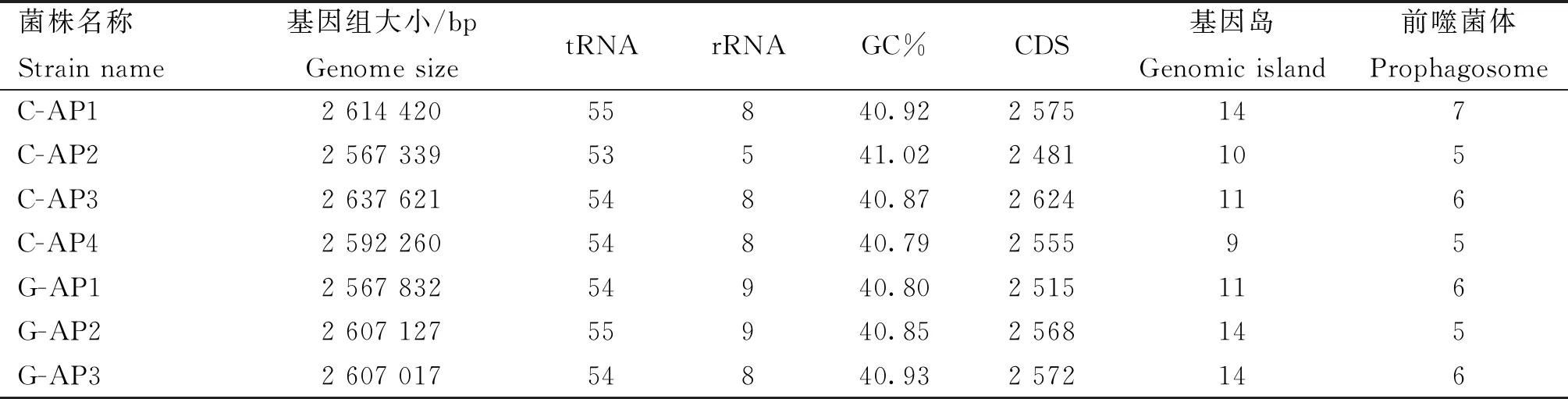
表2 菌株基因组信息
2.1.2 功能聚类分析 COG、KEGG和GO注释分类统计图都分别包含了7株Apg各种基因功能的平均基因数和对应的标准差,从图1可见,基因功能主要聚集在细胞的新陈代谢、细胞的合成、膜转运、信号转导、DNA复制及修复、转录、翻译;根据各基因功能的基因数标准差,可知7株Apg各种基因功能对应的基因数相差甚少。
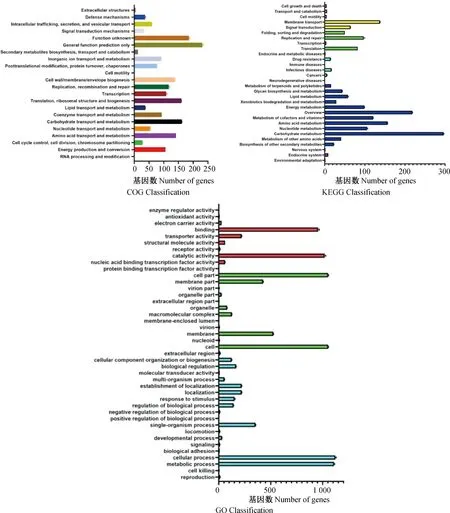
图1 COG、KEGG和GO基因功能聚类分析Fig.1 Functional cluster analysis of COG, KEGG and GO genes
2.1.3 毒力基因分析 利用细菌毒力因子数据库(VFDB)在线分析,3株鹅源菌株和4株鸡源菌株均预测出75个毒力相关基因,且所有菌株携带相同的毒力基因谱,其中占比最大的是内毒素类的毒力基因,详情见表3。
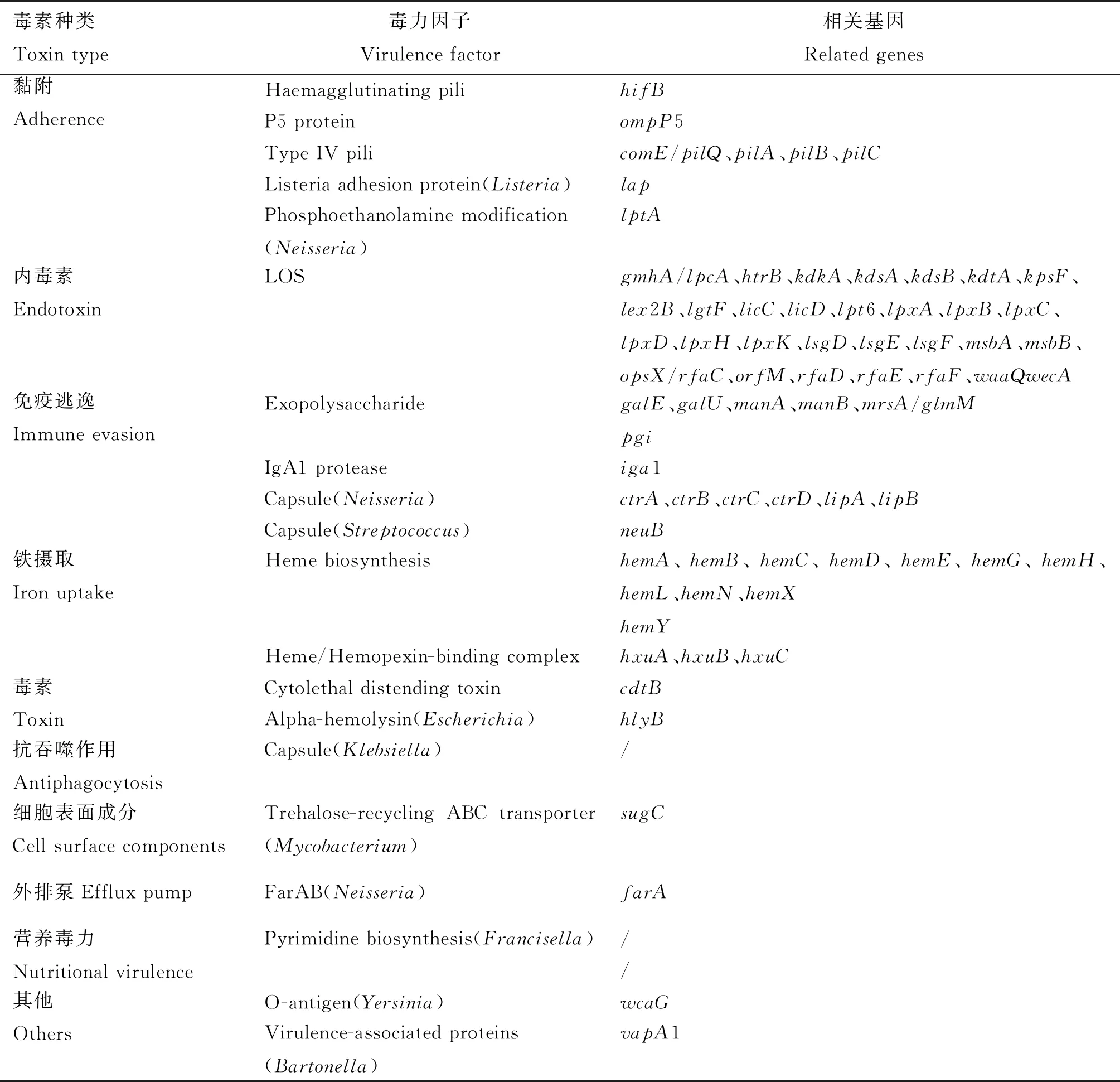
表3 毒力基因统计分析
2.2 基于核心基因SNPs的系统发育分析
根据核心基因SNPs构建进化树,发现Apg的进化树分布情况与菌株的分离地区有密切关系,各地区菌株基本会聚集在邻近的分支上,国内除了Hp8、A2S-2-1和Z1S-2-1株外,其他菌株均聚集在相邻分支上,包括本试验的7株菌株。在进化树中,3株鹅源菌株和鸡源菌株C-AP3以及p4chr1株位于同一小分支上,另外3株鸡源菌株分布在其它国内菌株分支上(图2)。
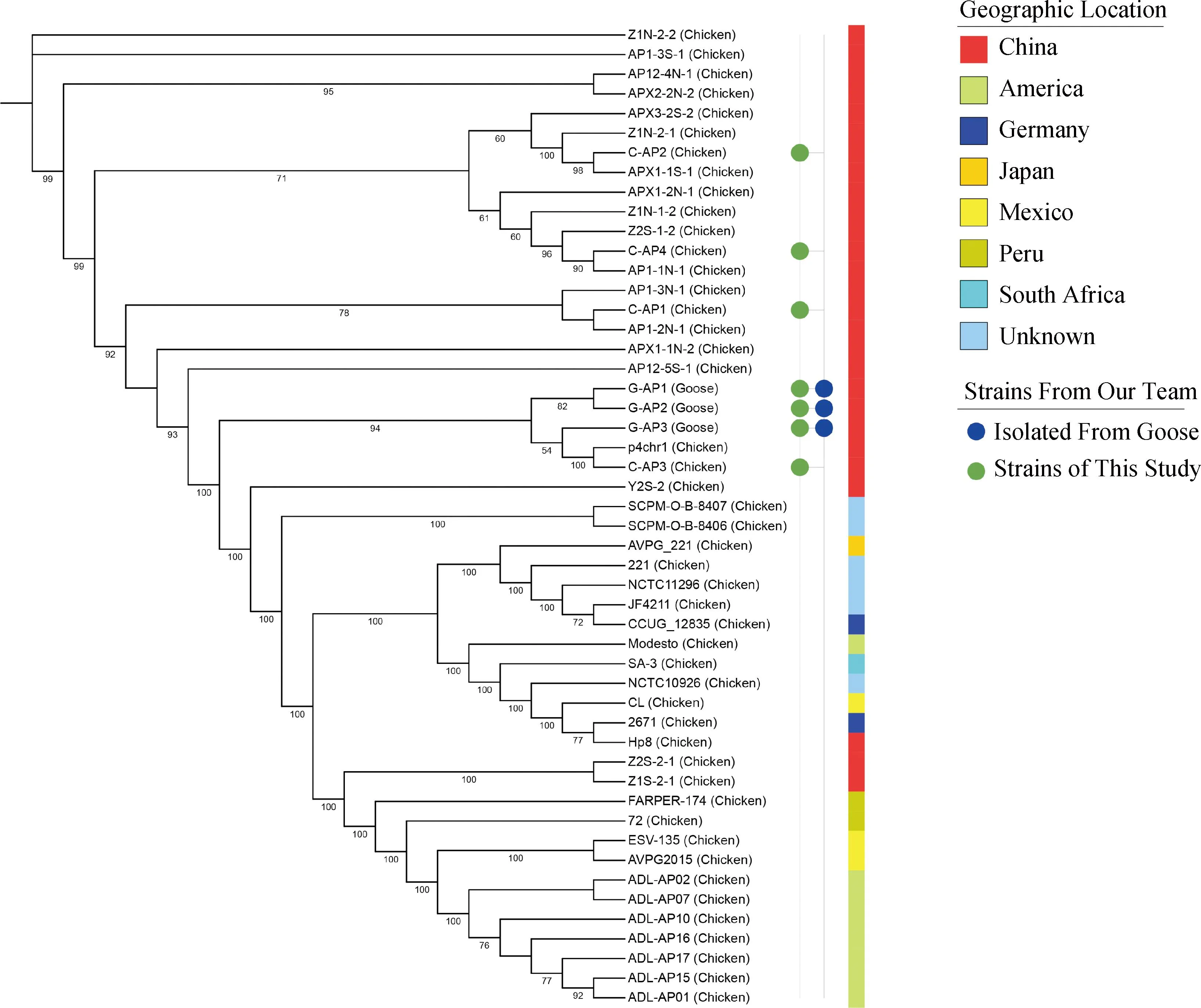
图2 核心基因SNPs进化树Fig.2 Phylogenetic tree of core SNPs
2.3 直系同源基因分析
2.3.1 7 株菌株同源性分析 为了解Apg菌株整体的同源基因分布情况,基于所有基因组的蛋白质序列,利用OrthoFinder软件分析各基因组间的共有基因和特有基因。结果显示,7 个基因组共计17 200条蛋白质序列,其中17 131个基因之间至少有两个基因存在同源性,还有69个基因与其他基因没有同源性;17 131个基因共计分出的2 241个共有基因家族,菌株普遍携带2 ~ 3个特有基因,而C-AP2株有14个特有基因、C-AP3株有44个特有基因;根据同源单拷贝基因构建的系统进化树,可见G-AP1株和G-AP2株独立于其它5株菌株(图3)。
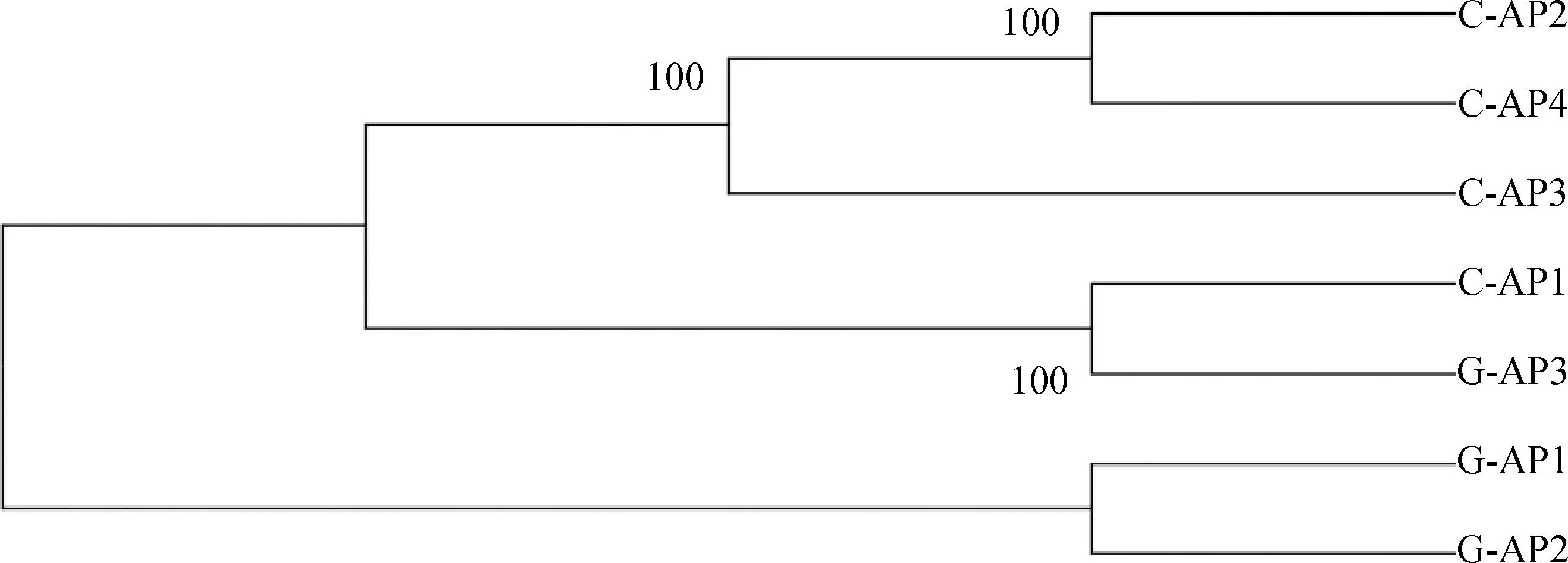
图3 基于同源单拷贝基因构建的进化树Fig.3 Phylogenetic tree of homologous single copy genes
2.3.2 鹅源菌株与鸡源菌株的同源性分析 通过对鹅源菌株共有基因与鸡源菌株共有基因作比较,发现鹅源和鸡源菌株共有4 711个基因,其中共有基因4 598个分为2 225个基因家族类群。另有113个基因属于特有基因,其中鹅源菌株特有基因有76个,鸡源菌株特有基因有37个。研究表明鹅源菌株与鸡源菌株在基因水平上存在差异。
2.3.3 鸡源Apg对鹅适应性基因的筛查 对A、B二组的基因组进行同源性分析,经排除与A组(C-AP2)基因组同源的基因,剩余79个特有基因存在于B组(G-AP1、G-AP2、G-AP3、C-AP1、C-AP3、C-AP4),通过对79个特有基因的功能注释可见,仅有22个基因编码已知蛋白,包括ATP酶RavA、冷休克样蛋白CspD、50S核糖体蛋白L3谷氨酰胺甲基转移酶、双官能团(IPC转移酶和DIPP合成酶)、转运蛋白家族EamA、胆碱/乙醇胺激酶、腺苷酸环化酶、Ⅰ型限制性内切酶EcoR124II R蛋白、跨膜蛋白A、EcoKⅠ限制性修饰系统蛋白HsdS、抗阻遏物AntA/AntB、海藻酸盐生物合成蛋白、岩藻糖转移酶、DNA结合蛋白H-NS、转肽酶D、脂多糖核心生物合成蛋白、碳水化合物二酸调节剂、唾液酸转移酶PMO188、DoxX、乙酰转移酶OatA、半乳糖、肠杆菌素EntS。其余57个蛋白均为假定蛋白;22个已知蛋白中,只有脂多糖核心生物合成蛋白与致病性有较大关联,其他蛋白与菌株致病性无直接联系;由于存在大量假定蛋白,暂时无法判断这79个基因中是否存在影响Apg对鹅的适应性基因,需要作进一步研究。
2.3.4 鹅源Apg对鸡适应性基因的筛查 对鹅源菌株G-AP1、G-AP2、G-AP3和鸡源菌株C-AP1共计9 831个基因进行同源基因分析,其中2 327个基因家族类群存在于所有样本中,将2 327个基因家族类群记为A组。对鸡源菌株C-AP2、C-AP3、C-AP4共计7 369个基因进行同源基因分析,其中2 278个基因家族类群存在于所有样本中,将2 278个基因家族类群记为B组。对A、B二组进行同源性分析,筛选出39个B组特有基因,其中10个为有明确蛋白功能注释的基因,包括灰分蛋白家族、β-自转运域蛋白、Ogr/Delta样锌指、转座酶DDE结构域蛋白、P2噬菌体J蛋白、噬菌体CP4-57调节蛋白(AlpA)、螺旋-转角-螺旋结构域蛋白、质粒稳定系统蛋白、DNA结合转录抑制因子PuuR、噬菌体CI抑制因子螺旋-转角-螺旋结构域蛋白,29个为假定基因;10个功能明确的基因中,除了β-结构域自转运蛋白编码基因与革兰阴性菌毒力有较直接关系外,其它基因仍有待进一步研究才能证实是否存在介导Apg对鸡适应的作用(表4)。

表4 已知功能蛋白
3 讨 论
细菌基因组是细菌全部遗传信息的集合,它在本质上决定了细菌的外观与功能,由于其基因数目庞大,且复杂多样,导致研究难度较高。随着全基因测序的发明以及比较基因组学方法的建立,让细菌基因组的研究变得便捷且科学。本试验3株鹅源Apg菌株基因组的整体参数与鸡源Apg菌株的基因组相比差异不大[13]。借助COG、KEGG和GO数据库对菌株基因组作注释和聚类,可见所有鹅源菌株的基因功能主要聚集在细胞的新陈代谢、细胞的合成、膜转运、信号转导、DNA复制及修复、转录、翻译,与鸡源菌株分布情况相似。
目前VFDB数据库仍未公布Apg的毒力因子信息,但由于大部分细菌的毒力因子为种属特异的,不同物种间的比对会面临假阳性高的风险,因此本试验以亲缘关系较为接近的流感嗜血杆菌数据库作替代,结果显示7株Apg均预测出75个相同的毒力基因,未找到菌株间毒力基因的差异,但可以肯定的是,仍有部分未知的Apg毒力基因在Apg感染宿主过程中起关键作用。
基因同源性分析试验中,通过对鹅源菌株共有基因与鸡源菌株共有基因作比较,结果显示鹅源菌株与鸡源菌株有2 225个共有基因,得到鹅源菌株特有基因76个,鸡源菌株特有基因37个,说明鹅源菌株和鸡源菌株存在基因水平上的差异。为了进一步了解这些基因差异是否会导致菌株的致病性差异以及是否会改变菌株对宿主的嗜性,作者根据菌株对不同宿主的致病性强弱进行物种的适应性筛查。Apg对鸡适应性基因筛查结果显示,共聚类出79个基因,其中编码的蛋白大部分为假定蛋白,另外在22个已知蛋白中,目前较明确与致病性相关的基因为编码脂多糖核心合成蛋白的基因,其它基因与细菌的黏附、抗宿主免疫系统、毒力因子和摄铁系统没有直接关联[14-16];此外,由于假定蛋白的数量众多,影响Apg对鹅适应性的基因也可能存在于此,暂时无法下明确结论。在Apg对鸡适应性基因的筛查结果中,共聚类得到39个基因,其中10个编码已知蛋白,29个编码假定蛋白,在已知蛋白里,除β域自转运蛋白具有明确的致病性外[17],其他蛋白与菌株的致病性没有直接关系,另外29个假定蛋白编码基因也可能是影响Apg感染鸡的关键因素。
SNP是指基因组中由单个碱基突变引起的DNA序列多态性,是遗传变异的重要依据[18-21],为研究物种起源、进化,本次利用SNP序列构建了包含50株Apg的进化树,从进化树中可以发现,Apg菌株的SNP变化基本是以地区为单位,同一国家的菌株会分布在同一或相邻的分支上,本试验的3株鹅源菌株与鸡源菌株都分布在我国新近分离株的大分支上,且鹅源菌株和鸡源C-AP3株聚集在p4chr1株的小分支上,从菌株对鹅的致病性试验可见,这4株菌对鹅的适应性要强于另外3株鸡源菌株,由此推测,Apg对鹅机体产生适应性的原因不一定是物种适应性基因的拥有,也可能是由于菌株基因发生突变,改变了Apg的黏附能力、毒力或抗吞噬力等,从而达到实施感染致病的目的。
4 结 论
通过比较基因组学分析,发现与鹅源副鸡禽杆菌(Apg)宿主适应性有关的基因为118个,对鹅适应性基因的筛查共聚类出79个基因,其中编码已知蛋白的基因有22个,其余编码假定蛋白;对鸡适应性基因的筛查聚类出39个基因,其中10个编码已知蛋白,29个编码假定基因为进一步阐释Apg跨物种感染的分子生物学机制奠定了基础。
