基于SSR 分子标记吉安地区的水稻亲缘关系鉴定分析
2024-03-25张天舒王莉莉贺卫东孙惠敏
郑 卓,张天舒,李 华,王莉莉,吴 杨,贺卫东,孙惠敏
(1.井冈山大学生命科学学院,江西,吉安 343009;2.吉安市青原区农业农村局,江西,吉安 343009;3.井冈山大学医学部,江西,吉安 343009)
水稻是最重要的粮食作物之一,全球近50%以上的人口以之为主食,其地位在亚洲尤其突出[1]。目前我国稻米总产量位居世界第一位,作为世界上最大的水稻生产与消费国,我国稻米单产量处于世界领先水平。农业种质资源是保障国家粮食安全、推动现代种业发展的物质基础,是维护生物多样性、建设生态文明的重要保障[2]。伴随着人民生活水平的逐步提升,水稻高产已不再是育种所追求的唯一目标,在达到高产的同时,稻种品质也成为了育种工作者选育的重要指标以及我国水稻业能否实现可持续发展的关键问题之一[3]。
早期由于缺乏对水稻资源完善、成熟且有效的鉴定方法,在我国出现水稻同种异名和同名异种的混乱现象非常普遍,极大地阻碍了水稻资源的充分利用。长久以来,大部分水稻的品种鉴定方法还停留在观察种子或植株的表观特征,这种方式极易受主观因素的影响和环境因素的干扰,准确性不高。DNA 分子标记技术现已广泛应用于种质鉴定、亲缘关系鉴别以及分子指纹图谱的构建等研究中[4]。其中,SSR 分子标记具有扩增条件稳定、在基因组中分布广泛、多态性好、操作简单且不易受环境影响等优点,能够有效地鉴别品种之间的亲缘关系。目前,SSR 分子标记技术已成为种质资源鉴定、作物遗传多样性分析等领域的最常见的遗传标记之一[5]。
例如,种子老化是导致遗传完整性丧失的关键因素,Z Huiying 等以东乡野生稻、湘谷、9194和日本野稻种子为材料,利用44 个SSR 标记对不同萌发梯度的种质资源进行遗传完整性分析,其研究结果为确定水稻资源的繁殖更新种群大小和发芽率临界值提供了重要依据[6]。而为了分析高原粳稻主栽品种的遗传状况,寻找与农艺性状相关联的分子标记,为水稻新品种选育提供参考,世荣等在2 种海拔条件下调查81 份高原粳稻主栽品种的农艺性状,并利用48 个SSR 标记对供试品种进行多态性扫描和群体遗传结构分析,最终检测出的7 个标记可以为高原粳稻杂交配组及分子标记辅助育种提供理论依据[7]。范家萌等以杂交水稻丰两优3305 和常规稻润稻118 为试验材料,结合品种指纹数据和PCR 产物片段大小之间的差异,筛选出适用于该品种纯度鉴定的SSR 分子标记,其研究结果可为高效检测水稻种子纯度提供技术参考[8]。为了解12 份水稻恢复系之间的遗传亲缘关系,从而为水稻恢复系选育和杂交组合选配提供分子依据,王衍坤等利用40 对SSR 引物进行遗传多样性研究,构建该12 份恢复系的指纹图谱,并以标记数据为基础计算遗传距离和生成聚类图[9],结果明确了相互之间的亲缘关系,从分子层面为恢复系选育和亲本组配提供了较好的遗传信息。
综上所述,由于SSR 分子标记自身独特的优势,如不受水稻发育阶段及环境的限制,且多态性水平高,有利于增强标记的效果等,使得在品种鉴定中SSR 标记发挥了巨大作用[10]。而本研究选取吉安市种植面积广泛、具有代表意义的8 种水稻样品,意在明确其相互之间的亲缘关系,为当地水稻种质资源的鉴定及分子标记辅助育种提供理论指导和依据。
1 材料与方法
1.1 实验材料
实验所用水稻材料为本地常用的水稻育种材料,由吉安市农科所提供,供试水稻材料为早-1、迟-2、井冈山迟A03、井冈山早A05、赣迟A04、赣早A02、吉安早A07 和吉安迟A09,分别编号为1、2、3、4、5、6、7、8。在这些水稻材料水稻材料中,早-1、井冈山早A05、赣早A02、吉安早A07 为早熟水稻品种,迟-2、井冈山迟A03、赣迟A04、吉安迟A09 为晚熟水稻品种。
1.2 实验方法
1.2.1 水稻基因组总DNA 的提取
根据欧阳昊婷等对于SSR 标记分析中的DNA提取现状的总结[11],本研究采用CTAB 法进行水稻基因组DNA 的提取,具体步骤如下:
剪取6 ~8 cm 水稻叶片,加入800 μL1.5×CTAB,充分研磨;将匀浆转入2 mL 的离心管,放入水浴锅前上下颠倒摇匀,60℃水浴30 min 后冷却至室温。
加入700 μL 氯仿,上下颠倒试管摇匀,10000 rpm 离心5 min;取上清液转入1.5 mL 离心管,10000 rpm离心5 min;将上清液转入新的1.5 mL离心管(如果上清取得很好,此步可省略)。
向提取的上清液中加入700 μL 无水乙醇,上下颠倒摇匀,-20℃放置20 min(可放置过夜);后经离心机12000 rpm 离心10 min,弃上清液,沉淀即为水稻基因组DNA;用75%酒精洗涤沉淀,风干DNA;最后加入50 ~100 μL 双蒸水,4℃保存备用。
1.2.2 PCR 扩增
利用均匀分布在水稻12 条染色体上的SSR 引物对8 组稻株的基因组DNA 进行PCR 扩增,SSR引物的核苷酸序列见表1;参考丁晓华等[12]的方法,选择20 μL 的PCR 扩增体系,其中含18 μL mix for PAGE 缓冲液(含buffer,Taq Enzyme,dNTP,Mg2+),上游引物0.4 μL,下游引物0.4 μL,0.8 μL模板DNA,余量为ddH2O;
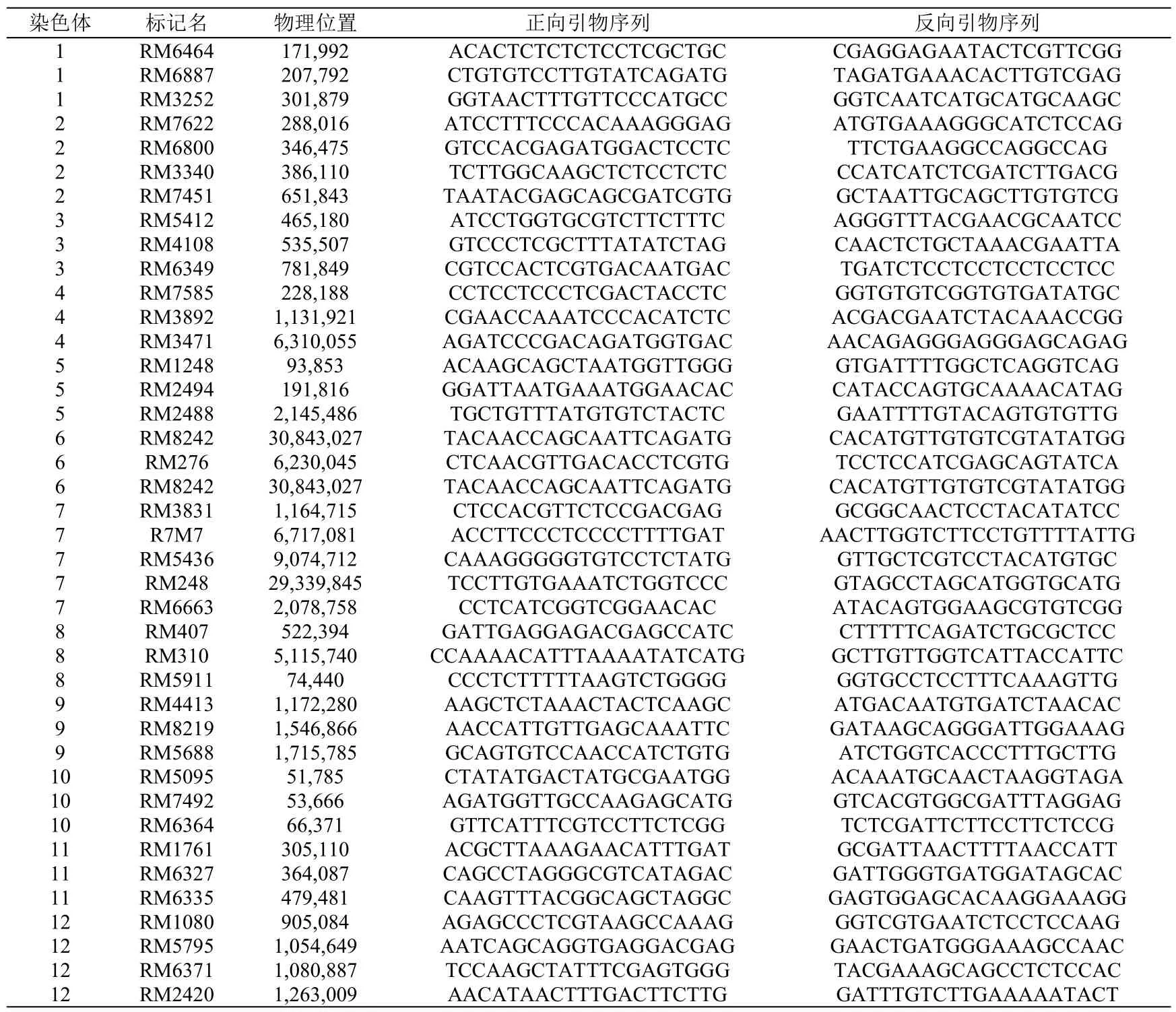
表1 本实验采用的SSR 引物序列信息Table 1 Sequence information of SSR primers used in this experiment
PCR 反应程序为:95℃预变性5 min;95℃变性15 s,55℃退火15 s,72℃延伸1 min,35 个循环;72℃延伸10 min。最后置于冷藏柜4℃保存[13]。
1.2.3 琼脂糖凝胶电泳
取50×TAE 缓冲液10 mL 以及490 mL 蒸馏水,配置1×TAE 缓冲液;称取1 g 琼脂糖加入100 mL 的TAE 缓冲液中,加热至琼脂糖完全熔解,静置3 ~5min,按照1:10000 加入10 μL 核酸染料于琼脂糖中[14];将琼脂糖凝胶液体倒入制胶槽,插入梳子,静置40 min;待凝胶凝固后,拔出梳子,将琼脂糖凝胶和制胶槽一并放入电泳槽内,将1×TAE 电泳缓冲液倒入电泳槽没过凝胶;向凝胶孔内分别加入DNA marker 4 μL 把8 种稻株的DNA 分别点样;连接正负极,打开电源,于120 V恒定电压下电泳30 ~40 min;停止电泳,将琼脂糖凝胶用成像仪观察并拍照。
1.2.4 数据处理
观察成像结果,根据每组标记扩增的条带数据,将PCR 扩增后依次出现的条带类型以数字的方式表现出来,依据带型出现的顺序、大小记录并标号。根据SSR 标记扩增检测的结果,通过NTsys pc2.01 软件,利用ntedit 直接录入数据,第1 行第1 个数字一般填1,代表要处理的数据为数字矩阵;第1 行第2 个数字填行数,第3 个数字填列数;第1 行第4 个数字,如果有缺失值填0,没有缺失值填1。然后用similarity 程序组中的qualitative data 计算相似系数矩阵;之后以上一步的结果作为输入文件,利用clustering 程序组进行计算,聚类分析法选用UPGMA。
将计算出的聚类树矩阵和距离矩阵进行比较,进行Mantel 检测,计算协表距离矩阵及相关性系数,以表示该聚类树对原始距离矩阵的代表程度。最后将上步方法得到的tree file 文件输入Graphics程序组中的tree plot 程序中计算,得到各水稻品种的SSR 聚类结果图,以此判断它们之间的相似度及亲缘关系[15]。
2 结果与分析
2.1 观察并记录
通过40 对SSR 引物对各水稻样品基因组DNA 的PCR 扩增,并对琼脂糖凝胶电泳后的这样带型统计如图1,同一引物对不同的水稻样品扩增的结果中,用数字“0”“1”分别标记带型。

图1 SSR 标记引物RM248 在8 种水稻样品中的PCR 扩增带型图谱Fig.1 PCR amplification band patterns of SSR marker primers RM248 in 8 rice samples
2.2 聚类分析
根据SSR 标记扩增检测的结果,通过NTsys pc2.01 软件[16-17],利用ntedit 直接录入数据,然后用similarity 程序组中的qualitative data 计算形似系数矩阵;之后以上一步的结果作为输入文件,利用clustering 程序组中的SHAN 进行计算,聚类分析法选用UPGMA,ties 选用FⅠND;最后将上步方法得到的tree file 文件输入到Graphics 程序组中的tree plot 程序中计算,得到聚类图2。

图2 待测水稻样品的UPGMA 聚类图Fig.2 UPGMA clustering diagram of rice samples to be tested
图中所示的1-8 号分别对应实验材料中所记录的8 种稻株。经分析可知,在遗传相似系数0.65处,待测的8 份水稻样品聚为两大类群,其中5 号‘赣迟A04’单独为一类,其他样品稻种聚为一类。在遗传相似系数0.80 处,可划分为三大类群:第一类囊括了6 种样品,表明这6 个品种亲缘关系较近,其中6 号‘赣早A02’与7 号‘吉安早A07’亲缘关系最近,其次是3 号‘井冈山迟A03’与4 号‘井冈山早A05’,而6 号、7 号同时与8 号‘吉安迟A09’保持有较近的亲缘关系;第二类只含有2 号‘迟-2’样品株;第三类则仅有5 号稻株‘赣迟A04’,说明其与其他7 份水稻样品的亲缘关系为最远。
3 讨论
本研究利用均匀分布于水稻基因组12 对染色体上的40 对SSR 分子标记对8 个水稻样品的亲缘关系进行了鉴定,总体PCR 扩增结果与琼脂糖凝胶电泳结果较为成功,有值得借鉴的经验,同时也有待于探讨的不足之处。
首先在优点方面,SSR 标记技术由于揭示的多态性水平较高,因此十分适合水稻品种遗传多样性的研究分析,遗传育种家也可以此方法来进一步判断不同稻种间的遗传距离和亲缘关系,而不是只依靠表型来选择亲本,因此便大大降低了在育种时选配亲本的盲目性,有效提升了选育新品种的工作效率[18]。同时,在实际的育种工作当中,大多学者认为亲本选配的重要原则之一就是扩大亲本的多样性水平和遗传距离,因为亲本间的基础遗传差距越大,后代杂种优势可能表现得越明显,所以更容易选出性状优于双亲的新品种[19]。
其次本次研究也存在一些不足之处,例如PCR 扩增时的退火温度就有待于改进。PCR 反应的退火温度取决于引物的碱基组成、长度以及其浓度,除此之外其还受目的基因所在序列长度的影响。由于退火温度能够直接影响产物生成量和反应的特异性,因此实验中通常选择较高的退火温度,以此来提高PCR 反应的特异性,减少引物与模板之间的非特异性结合,但扩增产物量可能会有所降低[20]。
此外,琼脂糖凝胶电泳和聚丙烯酰胺凝胶电泳作为常用的分离纯化和鉴定核酸的方法,二者之间存在差异。琼脂糖凝胶电泳所分离的DNA 片段范围较大,不同浓度的琼脂糖凝胶可分离长度从50 bp 至2000 bp 的DNA 片段。如今,由于操作简便、快速,一般实验室多用琼脂糖凝胶电泳进行DNA 的电泳分离。相较于琼脂糖凝胶电泳,聚丙烯酰胺凝胶电泳对分离小片段DNA 的效果最好,其分辨力极高,用于测序时可分离相差仅1bp的DNA 片段[21-22],因此其观察效果也比琼脂糖凝胶电泳要好的多,基本不会出现类似本研究中存在多态性但却无法观察其多态性的情况。
目前上述研究方法在水稻的遗传育种中发挥着重要的作用,今后研究中,通过进一步发掘新技术,利用最新手段对水稻等作物进行相关的遗传研究,将对我国乃至世界粮食问题的解决会产生重要的影响[23]。
