盐胁迫下大豆抗感品种根系微生物群落结构分析
2024-02-20陈昕宇张真张早立宋圆圆曾任森卢龙
陈昕宇, 张真, 张早立, 宋圆圆, 曾任森, 卢龙
(福建农林大学农学院,作物遗传育种与综合利用教育部重点实验室,福建 福州 350002)
土地盐渍化已成为当今农业不可忽视的一大难题[1]。盐渍化在造成盐胁迫的同时又会引发渗透胁迫与氧化损伤,进而影响作物种子萌发、作物生长及器官发育,在严重情况下会导致作物萎蔫,甚至死亡[2]。中国当前土地盐渍化扩大严重,由此造成耕地面积不断减少,是制约大豆生产的重要原因之一。大豆是中国重要的粮食和油料作物,其种子是重要的植物蛋白与油脂来源,也是中国工业和农业生产的重要原材料。但大豆是一种中等盐敏感作物,盐胁迫会降低大豆的发芽率与出苗率,干扰节瘤,减少大豆生物量累积,最终严重影响大豆产量[3-5]。
自然界中植物的根部聚集着特定种类的微生物,被称为根系微生物组[6-8]。根系微生物根据其受根系影响和调控的程度被分为根际微生物、根表微生物及根内微生物[9]。根系微生物通过改进土壤养分的有效性、与植物的互作、拮抗病原菌、诱导植物抗性等方式,促进植物生长,增强植物抗逆性,对植物生长产生影响[10-11]。根系微生物的变化既能反应土壤性质的变化,也能在一定程度上反应植物生长的变化[12-13]。当植物受到外界胁迫时,其根系微生物也会发生相应改变,部分微生物甚至参与了植物抵御胁迫的过程。例如,在植物遭遇盐胁迫时,根系微生物会分泌生长素等植物激素类物质帮助植物抵御盐胁迫[14]。在干旱或洪涝胁迫下,如根瘤菌等根系微生物会分泌胞外多糖(EPSs)帮助植物抵御胁迫[15]。
随着微生物组学及高通量测序技术的兴起与发展,拟南芥[16]、水稻[17]、玉米[18]等植物的根系微生物结构特征得到研究。已有研究表明,植物根系微生物的结构受其所处环境,植物的基因型及其生长状态等因素决定[19],而在特定的土壤环境中,根系微生物的结构受到植物根系分泌物的调控[20]。
近年来,已有学者对盐害条件下多种植物的根系微生物进行研究[21]。但多集中于群落结构分析及农艺性状改良应用方面[22-23],对盐胁迫下不同基因型大豆根系微生物群落结构变化及耐盐大豆根系微生物的筛选研究较少。本研究通过16S扩增子测序技术研究盐敏感和耐盐2个大豆品种在对照条件和盐胁迫条件下的根系微生物群落结构差异,揭示盐胁迫条件下大豆根系微生物群落结构的变化,从中筛选可能影响大豆耐盐性的有益菌株,进而深入了解根系微生物介导的大豆耐盐机制。
1 材料与方法
1.1 试验材料及方法
供试大豆品种分别为盐敏感的科丰1(KF)和耐盐的南农1183-2(NN)。依照处理方式不同将所有样本分为4组:KF_R_W(科丰1对照处理)、KF_R_N(科丰1盐处理)、NN_R_W(南农1183-2对照处理)、NN_R_N(南农1183-2盐处理)。
将大豆种子播于内径7 cm,高8 cm的塑料花盆中,所有花盆置于托盘中,待大豆出苗后,每3 d于托盘内用1 L Hoagland营养液浇灌1次。每盆大豆定苗4株,待第1片复叶长出时,取生长情况相同的大豆按品种与处理方式不同分为4组。试验组每3 d浇灌1次1 L 120 mmol·L-1NaCl的Hoagland营养液。处理7 d后,挖出大豆的完整根系,取样参照胡雅丽等[24]的方法,先用无菌水洗掉粘在大豆根上的松散的土壤颗粒,接着放入装有25 mL PBS缓冲液的50 mL管中,180 r·min-1摇晃清洗3次,然后用无菌滤纸吸干根表面水分,最后将样本放入-80 ℃中保存。
1.2 DNA提取、16S扩增子文库构建和测序
采用MoBio的土壤微生物DNA强力提取试剂盒(PowerSoil® DNA Isolation Kit)对大豆根系样品完成基因组DNA抽提后,利用质量分数1%琼脂糖凝胶电泳检测抽提基因组DNA后,采用简并PCR引物799F:AACMGGATTAGATACCCKG以及1492R:ACGTCATCCCACCTTCC,对16SRNA基因的V5~V7区进行PCR扩增,扩增完成后对PCR产物使用质量分数1%琼脂糖凝胶电泳检测扩增目的条带大小,并用Agencourt AMPure XP核酸纯化试剂盒纯化,再进行Miseq文库构建,最后进行Miseq上机测序。
1.3 数据分析与制图
对测序得到的Pair-End(PE)双端序列数据,使用Trimmomatic (v0.36)、Pear (v0.9.6)、Flash (v1.20)、Vsearch (v2.7.1)软件进行质控与拼接处理,采用uchime方法及自比对(denovo)方法去除嵌合体以及不合要求的短序列,最终得到优质序列。使用Qiime(version 1.8.0)软件对所有序列进行OTU(operational taxonomic units,可操作分类单元)聚类(相似水平97%)分析,并进行注释,最后计算各样本的Alpha多样性指数和Beta多样性指数;采用PICRUSt2软件进行功能预测分析。采用SPSS 26对数据进行差异性分析;采用R语言工具及graph8进行统计与作图;通过MAGE11软件选择Test UPGMA Tree算法构建系统树状图;使用R及其igraph包,psych包对共现性网络进行可视化操作。
2 结果与分析
2.1 不同样本根系微生物群落结构
2.1.1 OTU及多样性分析 Shannon-wiener是反映样本中微生物多样性的指数,利用各样本的测序量在不同测序深度时的微生物多样性指数构建曲线,以此反映各样本在不同测序数量时的微生物多样性。测序结果表明,所有样本的Shannon-wiener曲线最终都趋于平坦(图1),且覆盖率都在99%以上(表1),表明测序结果达到要求,可以反映样本的真实情况。各组的Chao指数与物种数没有明显差异,KF_R_W的Shannon-wiener指数与Simpson指数低于KF_R_N,而NN_R_W的Shannon-wiener指数与Simpson指数显著高于NN_R_N(p<0.05),这表明在盐胁迫条件下大豆的根系微生物多样性均发生改变。
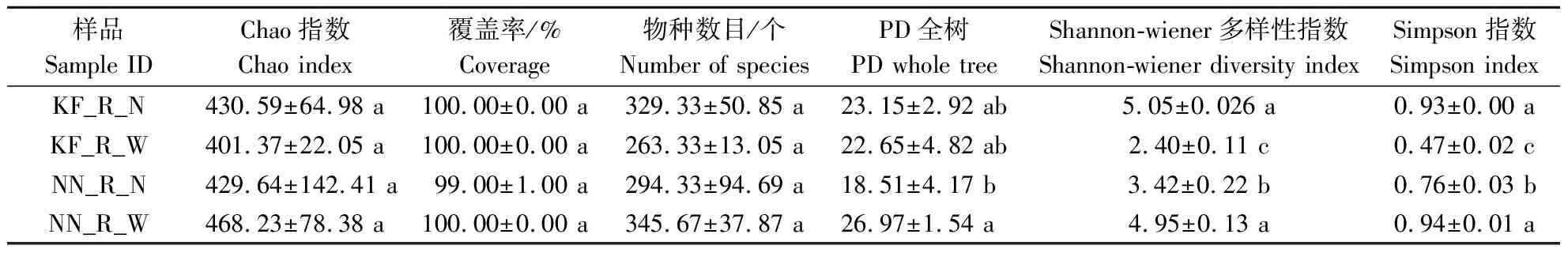
表1 4组大豆根系样本的Alpha多样性指数Table 1 Alpha diversity index of root-associated samples from four groups of soybeans
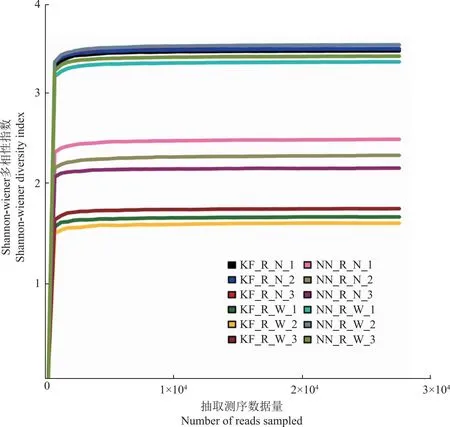
图1 4组大豆根系样本Shannon-wiener曲线Fig.1 Shannon-Wiener curves of microorganism in root-associated samples from four groups of soybeans
由各组根系微生物的OTU(操作分类单元,operational taxonomic units)所构建的Venn图可知(图2),KF_W、NN_W、KF_N、NN_N各处理共有820个OTU。所有处理共有的OTU数目为209个,这可能代表了KF与NN 2种大豆根系中存在稳定的菌种。KF_W、NN_W、KF_N、NN_N 4种处理方式特有的OTU个数依次为31、95、63、105。无论是对照条件下还是盐胁迫条件下,NN的OTU数大于KF。因此,在盐胁迫处理下,两种大豆的根系微生物群落菌属均发生改变,不但存在相似部分也衍生出特有部分,群落结构发生变化。
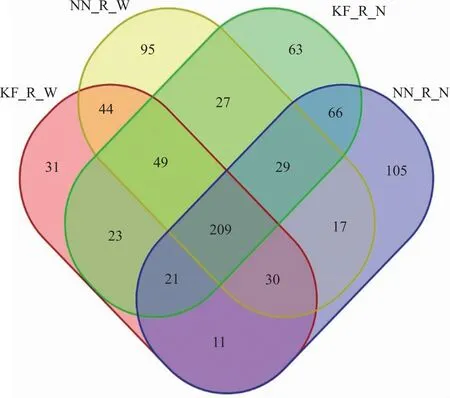
图2 4组大豆根系微生物样本OUT数Venn图Fig.2 Venn diagram based on OUT number of root microbiota samples from four groups of soybeans
2.1.2 不同样本微生物群落差异性分析 通过基于unifrac及bray-curtis的主坐标(principal coordinates analysis PCoA)分析4组大豆根系样本的微生物多样性(图3)。图3显示,在第一主坐标轴上2种大豆的根系微生物组按照处理环境被分为了2簇,表明环境差异导致了大豆根系微生物的组成不同,是本研究造成微生物差异的主要来源,证明盐胁迫处理是影响2种大豆根系微生物群落结构组成的重要因素。
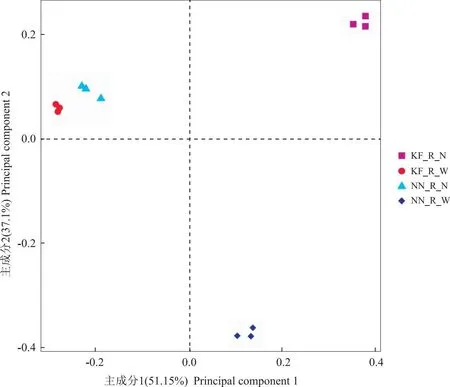
PCoA横纵轴上所标注的比例即该主坐标对样品矩阵数据差异的贡献度。4组样本的根系微生物组在PCoA的第一轴上(主成分1)按处理方式分开,第二轴上(主成分2)按基因型分开,组间差异显著(p<0.001)。The values displayed on the horizontal and vertical axis of principal coordinate analysis (PCoA) represent the extent to which each principal coordinate contributes to the dissimilarity observed in the sample matrix data. The root microbiomes of the four groups were distinguished by treatment along the first axis (principal component 1) and by genotype along the second axis (principal component 2). These differences were found to be statistically significant among the groups (p<0.001).
利用树枝结构描述和比较多个样本间的相似性和差异关系,即根据beta多样性距离矩阵进行层次聚类(hierarchical clustering)分析[25],使用非加权组平均法UPGMA(unweighted pair group method with arithmetic mean)算法构建树状结构,得到4组大豆的根系微生物群落结构树状关系如图4所示,同一处理条件下的不同基因型大豆各自处于不同的分支,表明基因型也是影响大豆根系微生物群落结构组成的重要因素之一。
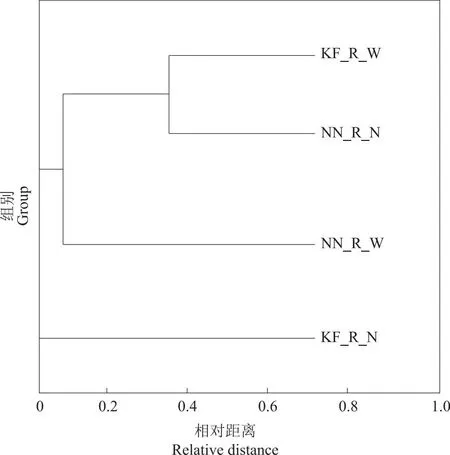
图4 4组大豆根系微生物样本Hcluster分析结果Fig.4 Hierarchical clustering analysis (Hcluster) of root microbiota from four groups of soybeans
2.2 盐胁迫对大豆根系细菌群落结构门水平的影响
4组大豆根系样本中的微生物在门水平上的相对丰度如图5所示,门一级的优势菌门为变形菌门Proteobacteria,变形菌门无论在对照还是盐胁迫下,在2种大豆的根系中所占比例均超过90%,高于其他任何菌门。KF_R_N相较于KF_R_W根系中的放线菌门Actinobacteriota与拟杆菌门Bacteroidota所占比例升高,分别由0.347%上升至6.810%,0.072%上升至1.043%。而NN_R_N变形菌门由97.890%上升至98.870%,其余菌门丰度均未超过0.500%。
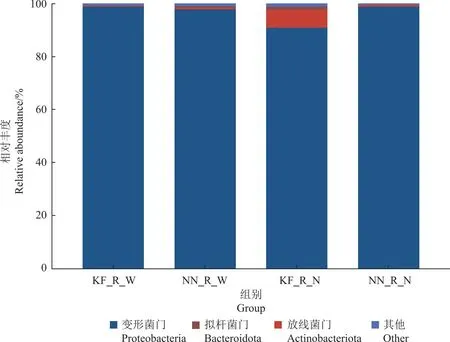
图5 4组大豆根系微生物样本在门水平上的相对丰度Fig.5 The relative abundance of root microbiota in samples from four groups of soybeans at phylum level
2.3 盐胁迫对大豆根系细菌群落结构属水平的影响
对4组大豆根系样本中的微生物在属水平上的相对丰度如图6所示。在属水平上, KF_R_W的根系微生物中,排名前3的优势菌属为:艾德昂菌属Ideonella(72.45%),根瘤菌属Allorhizobium-Neorhizobium-Pararhizobium-Rhizobium(4.94%),马赛菌属Massilia(2.43%)。艾德昂菌属占据绝对优势,所占比例超过70.00%, 其余菌属比例均未超过10%;在盐胁迫条件下,排名前3的优势菌属为:根瘤菌属(22.39%),假黄单胞菌属Pseudoxanthomonas(17.04%),艾德昂菌属(12.32%)。艾德昂菌属比例显著下降,由72.45%下降至12.32%,根瘤菌属与假黄单胞菌属比例显著上升,分别从4.94%上升至22.39%,0.22%上升至17.04%。NN_R_W根系微生物各个优势菌属比例相对均匀,排名前3的优势菌属依次为:艾德昂菌属(13.82%),马赛菌属Massilia(11.81%),嗜甲基菌属Methylophilus(11.58%);在盐胁迫条件下,排名前3的优势菌属为:艾德昂菌属(45.40%),假单胞菌属Pseudomonas(25.78%),根瘤菌属(6.83%),假单胞菌属占比上升,由对照条件下时的2.15%提高至25.78%,成为NN_R_N的优势菌属。
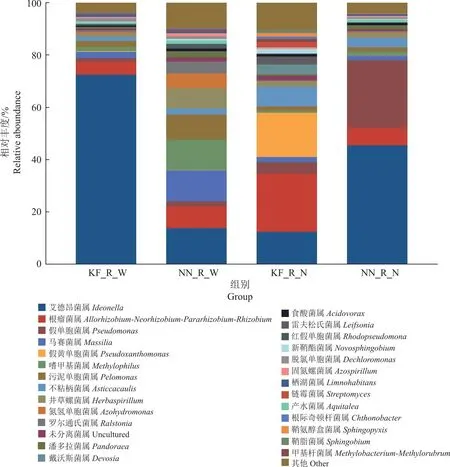
图6 4组大豆根系微生物样本在属水平上的相对丰度Fig.6 The relative abundance of root microbiota in samples from four groups of soybeans at genus level
2.4 Co-occurrence network共现性网络分析
在对微生物群落的研究中,Co-occurrence network共现性网络常被用以表示群落中微生物的互作关系[26]。以所有样本中绝对丰度前20的属水平结果进行相互关联性分析并构建共显性网络结构,结果如图7所示。所有大豆根系样本中参与构建网络的微生物绝对丰度前20的菌属均为变形菌门Proteobacteria或放线菌门Actinobacteriota,且变形菌门无论在丰度与菌属的种类上均占据绝对优势地位。艾德昂菌属尽管丰度占比最大,但多与其他菌属负相关,而作为植物促生根系菌(PGPB)的根瘤菌属及假单胞菌属与多数菌属呈正相关。

图中各点代表微生物的属,点的大小代表微生物属丰度的大小,线的粗细代表相关性大小;点的颜色代表所属门,线为红色表示呈正相关,蓝色表示呈负相关关系。In the given diagram, each data point corresponds to a specific genus of microorganism. The size of the data point is indicative of the relative abundance of the genus, while the thickness of the connecting lines represents the strength of correlation between the genera. Additionally, the color of the data points corresponds to the category to which they belong. Notably, a red line signifies a positive correlation, whereas a blue line signifies a negative correlation.
2.5 菌群相关功能预测
通过PICRUSt (phylogenetic investigation of communities by reconstruction of unobserved states)对2种大豆正常环境下与盐胁迫条件下根系微生物群落的代谢功能进行预测,结果如图8所示。在对照条件下,大豆根系微生物中,生物降解代谢(xenobiotics biodegradation and metabolism),辅助因子及维生素代谢(metabolism of cofactors and vitamins),碳水化合物代谢(carbohydrate metabolism),氨基酸代谢(amino acid metabolism)丰度最高,表明大豆根系微生物代谢状态活跃。相对于对照条件,NN_R_N根系微生物在生物降解代谢、脂类代谢(lipid metabolism)以及氨基酸代谢方面丰度高于KF_R_N,尤其在色氨酸代谢方面,NN_R_N高于KF_R_N(图9),而色氨酸的一条关键代谢途径则是合成吲哚乙酸(IAA)。这表明耐盐大豆根系微生物可能通过生物降解以及脂类、氨基酸代谢参与调节并提高大豆对盐胁迫的耐受性。
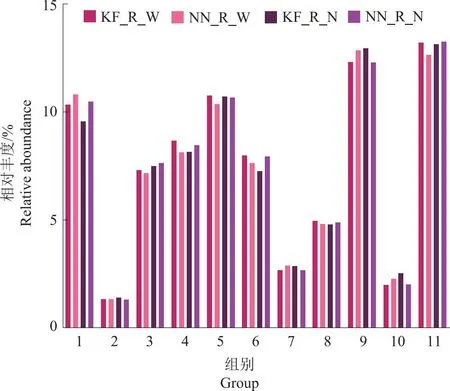
1: 生物降解和代谢; 2: 核苷酸代谢; 3: 萜类和多酮类代谢; 4: 其它氨基酸代谢; 5: 辅助因子和维生素代谢; 6: 脂类代谢; 7: 聚糖生物合成和代谢;8: 能量代谢; 9: 碳水化合物代谢; 10: 其它次生代谢产物的生物合成; 11: 氨基酸代谢。1: Xenobiotics biodegradation and metabolism; 2: Nucleotide metabolism; 3: Metabolism of terpenoids and polyketides; 4: Metabolism of other amino acids; 5: Metabolism of cofactors and vitamins; 6: Lipid metabolism; 7: Glycan biosynthesis and metabolism; 8: Energy metabolism; 9: Carbohydrate metabolism; 10: Biosynthesis of other secondary metabolites;11: Amino acid metabolism.
图9 4组大豆根系微生物的色氨酸代谢通路差异Fig.9 Differences in the tryptophan metabolism pathway among four groups of soybean root microbiota
3 结论与讨论
基于16S高通量测序技术盐胁迫下大豆根系微生物多样性的研究结果表明,基因型及环境均导致大豆根系微生物群落结构发生变化。同样在小麦,冬枣,烟草等植物的研究中均发现基因型与环境是改变植物根系微生物的重要驱动因素[27-28]。基于样本OTU建立的Venn图结果显示,在对照与盐胁迫条件下,2种大豆的根系微生物群落均发生改变。对样本的主坐标分析表明了盐胁迫是影响根系微生物群落的主要因素,而对样本的Hcluster分析则表明,基因型也是影响大豆根系微生物的因素之一。反映出根系微生物受环境与基因型影响的复杂性。
根系微生物对作物生长、发育、抗病,抵御非生物胁迫等方面有着积极作用,根系微生物的改变对于作物的生长发育有着很大影响[29-30]。本研究对盐胁迫下大豆根系微生物物种分析发现,NN根系微生物群落中假单胞菌属的丰度占比明显提高,所占比例高达25%。假单胞菌属是一种根系促生菌,可通过分泌吲哚乙酸(IAA)类似物促进植物生长与根部发育,同样被视作一种可以缓解植物盐胁迫的根系促生菌[31]。由日本学者分离的恶臭假单胞菌(Pseudomonasputida)KT440能够改善盐胁迫下大豆与玉米的生长情况[32]。此外,已有学者在大豆根系微生物中成功分离出4株假单胞菌属菌株,4株菌株均表现出对高盐极强的耐受性,且其中一株菌株改善了盐碱土壤中大豆的生长情况[33]。目前已有研究证明了假单胞菌属是一种有大豆特异亲和性的根系促生菌[34]。
通过对根系微生物的代谢分析进行功能预测表明,盐处理样品中,NN大豆的根系微生物在生物降解及代谢,脂类代谢以及氨基酸代谢方面丰度高于KF,说明NN的根系微生物可能通过上述几种代谢途径参与调控大豆耐盐。在色氨酸代谢方面,NN代谢丰度高于KF,而色氨酸是合成吲哚乙酸的前体物质,这与假单胞菌属可以分泌生长素类似物的功能恰好吻合。近年来有相关报道表明,作物根系微生物产生的生长素可以缓解盐胁迫对作物生长的影响,增强作物耐盐性[35-36],这可能是NN相对于KF有着更强耐盐性的原因[37]。基于本文研究结果,推测在盐胁迫条件下,耐盐大豆会招募耐盐根系促生微生物如假单胞菌属等定殖根部,利用其分泌的生长素类似物增强自身对盐胁迫的耐受性,本文主要研究盐敏感大豆与耐盐大豆在盐胁迫条件下根系微生物的变化以及功能预测,而假单胞菌是否在大豆耐盐胁迫中起到重要作用有待于进一步研究。因此,后续将对根系微生物中假单胞菌属进行分离与功能验证,并对大豆根系生长素含量变化等方面进行深入研究。
综上所述,盐胁迫与基因型是影响大豆根系微生物群落的重要因素。相较于盐敏感大豆科丰1,盐胁迫下耐盐大豆南农1183-2中假单胞菌属丰度明显上升。该菌作为一种有高盐耐受性,且有大豆特异亲和性的根系际促生菌,或可成为改善大豆耐盐性的可利用资源。
