掌叶大黄(Rheum palmatum L.)WRKY基因家族鉴定与分析
2024-02-05吴圳张明英闫锋李依民高静颜永刚张岗
吴圳 张明英 闫锋 李依民 高静 颜永刚张岗
(1. 陕西中医药大学药学院 陕西省秦岭中草药应用开发工程技术研究中心,西安 712046;2. 陕西中医药大学省部共建特色秦药资源研究开发国家重点实验室[培育],咸阳 712083;3. 陕西中医药大学陕西省中医药管理局“秦药”研发重点实验室,西安 712046)
大黄,别名将军、黄良等,是我国传统大宗中药材,其功效为泻下攻积、清热泻火、凉血解毒、逐瘀通经、利湿退黄[1],其3个基源分别为蓼科植物掌叶大黄(Rheum palmatum L.)、唐古特大黄(R.tanguticum Maxim.ex Bal)和药用大黄(R. officinale Baill.),入药部位为干燥根及根茎。掌叶大黄应用历史悠久,主要分布于甘肃东部及东南部、青海东部、四川西部及西北,多生于林缘、灌丛、草地[2]。大黄的主要生物活性成分是蒽醌类成分,研究发现其有抗肿瘤,抗炎,保护心血管等药理作用[3]。
转录因子是可以与基因顺式作用元件发生特异性相互作用,并激活或抑制基因转录的一种DNA结合蛋白。植物在高度可变的环境中,转录因子通过调控下游靶基因表达直接或间接地参与调节植物对环境的生理适应[4]。目前植物中发现了相当多的转录因子家族,如MYB、bZIP、bHLH、WRKY。其中很多家族,如MYB、bHLH和WRKY,被证实参与了植物生长发育和次生代谢的转录调控[5]。WRKY是植物中最大的转录因子家族之一。WRKY结构由两部分组成:N端包含一个保守的WRKYGQK基序,C端包含一个60氨基酸长锌指基序,二者对于WRKY转录因子与靶基因启动子的结合至关重要[6]。WRKY蛋白按其WRKY domain的数目及锌指基因的种类可划分为三类[7]。I族成员有两个WRKY结构域和C2H2型锌指。II族成员只有一个WRKY结构域和一个C2H2锌指基序,根据其WRKY结构域可进一步分为5个亚组(IIa、IIb、Ic、IId和IIe)。III族成员有一个WRKY结构域和一个C2HC型锌指。
第一个WRKY基因SPF1于1994年在甘薯中被克隆[8],自此之后WRKY家族被报道广泛参与植物对非生物胁迫和激素信号转导。如SbWRKY30参与了高粱对干旱胁迫的响应[9],过表达马鞭草VbWRKY32基因转基因植株耐寒性显著增强[10]。在南非醉茄中鉴定的一个WsWRKY1,能通过调节烷基内酯的积累而参与防御反应[11]。WRKY基因家族还通过激素信号分子广泛参与了植物种子萌发和生长发育。研究表明,AtWRKY44和AtWRKY75在拟南芥中调节根毛的发育[12]。生长素诱导AtWRKY23的表达从而调节植物根系的正常生长和类黄酮的局部合成[13]。WRKY57作为MeJA诱导叶片衰老的抑制因子,参与了MeJA和生长素介导的信号通路[14]。敲除了AtWRKY2的拟南芥突变体在种子萌发和萌发后早期生长期间对ABA的反应更敏感[15]。
在药用植物中,WRKY转录因子参与次生代谢产物的生物合成调控。CjWRKY1的异位表达可显著增加小檗碱生物合成相关基因的表达水平[16]。PgWRKY4X与角鲨烯环氧化酶启动子中的W-box结合上调人参皂苷生物合成基因[17]。红豆杉TcWRKY8/20/26/47调控紫杉醇的生物合成[18]。过表达IiWRKY34显著提高菘蓝中6种木脂素类化合物的含量[19]。鉴于WRKY在药用植物次生代谢调控中的潜在作用,其有可能作为中药材质量控制的关键因子。本研究以掌叶大黄为对象,利用全长转录组测序技术结合RNA-seq挖掘WRKY转录因子家族基因及其差异表达特征,为后续深入研究其在掌叶大黄生长发育及蒽醌类成分代谢调控中的作用机制提供科学支撑。
1 材料与方法
1.1 材料
1.1.1 仪器 NanoDropTM2000分光光度计(美国Thermo-fisher公司); PacBio Seque III测序仪(美国PacBio公司); StepOnePlusTMReal-Time PCR(qPCR)仪(美国Applied Biosystems公司); K5800自动检测超微量分光光度计(凯奥公司)。
1.1.2 试剂 茉莉酸甲酯(methyl jasmonate, MeJA);Trizol试剂盒(Life technologies公司); SMARTer PCR cDNA Synthesis Kit(美国Clontech公司);RN38-EASYspin Plus植物RNA提取试剂盒(北京艾德莱公司); PrimeScriptTMRT Master Mix反转录试剂盒; TB Green® Premix ExTaqTMII(TliRNaseH Plus)(TaKaRa公司)。
1.1.3 植物材料 2022年8月在甘肃省陇南市甘肃中医药大学和政药用植物园分别采集掌叶大黄(R.palmatum L.)一年生植株和成熟种子,经陕西中医药大学胡本祥教授鉴定。
选择外观一致且饱满的种子点播于黑色塑料花盆中(直径9 cm、高12.5 cm), 每盆点播4颗种子,每盆用200 g泥炭土(klasmann)。培养温度为23±2℃,在9 000 Lx光强下光周期为16 h/8 h。第一次浇水至盆底有水排出即可,每隔3 d补水50 mL。
1月后,选取长势一致、生命旺盛的掌叶大黄幼苗3株,将其根、叶等量混合用于全长转录组测序分析。同时,对1月龄幼苗叶片喷施200 μmol/L MeJA为处理组、喷施溶剂为模拟对照组,以0 h为空白对照,所有样品重复3次,分别于处理后3、6、12和24 h取样,液氮速冻后于-80℃冰箱保存。
取200 μmol/L MeJA处理12 h即MeJA-12 h、Mock-12 h 叶片样本,3次生物学重复,送广州基迪奥生物科技有限公司进行 RNA-seq 分析;取一年生植株3株,根、根茎、叶3个部位各等量混合后进行RNA-Seq分析。
1.2 方法
1.2.1 全长转录组测序及WRKY鉴定 利用Trizol试剂盒提取总RNA,采用NanoDropTM 2000分光光度计检测RNA样品的浓度,检测合格后的总RNA使用SMARTer PCR cDNA Synthesis Kit及PCR扩增合成全长cDNA并进行SMRT bell文库构建。上机测序采用PacBio Seque III,所得到的原始数据用SMRT Link V8.0.0进行分析。先提取高质量的环形一致性序列(circular consensus sequencing, CCS),聚类FLNC reads,得到完整的isoform。通过BlastX(http://www.ncbi.nlm.nih.gov/BLAST/)将isoform比对到蛋白数据库SwissProt(http://www.expasy.ch/sprot)、Nr(http://www.ncbi.nlm.nih.gov)和KEGG(http://www.genome.jp/kegg)进行功能注释。
使用转录因子数据库plant TFdb(http://planttfdb.gao-lab.org/index.php)在蛋白序列中找到有已知转录因子的motif,进行转录因子家族归类。挑选WRKY转录因子家族基因,再次进行BlastX比对,候选基因包含起始密码子和终止密码子的完整开放阅读框(open reading frame, ORF)利用ORFfinder(https://www.ncbi.nlm.nih.gov/orffinder/)在线网站进行预测,使用标准密码子翻译表将其中最长的ORF翻译成全长蛋白序列。用ExPASy(https://prosite.expasy.org/prosite.html)分析基因编码蛋白质的结构域,同时用CDD(https://www.ncbi.nlm.nih.gov/cdd)进行验证。
1.2.2 蛋白序列分析 采用ExPASy ProtParam(http://cn.expasy.org/tools)和SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)在线网站进行蛋白一级、二级结构理化性质分析。亚细胞定位使用WoLF PSORT(http://www.genscript.com/psort/wolf_psort.html)预测。
根据WRKY保守结构域来分类RpWRKYs,该结构域在Jalview软件中进行可视化分析。使用MEGA 11软件中邻接法(neighbor-joining, NJ)进行RpWRKYs系统进化树构建分析,Bootstrap为1 000次,再利用itol在线网站进行发育树修饰和标注。保守氨基酸基序的分析使用在线保守基序预测网站MEME(https://meme-suite.org/meme/tools/streme)完成。利用TBtools可视化分析RpWRKYs蛋白保守基序。
1.2.3 基因组织表达分析 基于RNA-Seq转录组测序数据,筛选出RpWRKYs在掌叶大黄根、根茎和叶中的表达数据,以FPKM值表示丰度,经log2标准化后运用TBtools绘制RpWRKYs在不同组织中的相对表达量热图,分析其在不同组织部位的表达模式。
1.2.4 候选基因qPCR验证 参照RN38-EASYspin Plus植物RNA提取试剂盒提取掌叶大黄各样品总RNA,1.0%琼脂糖进行凝胶电泳检测完整性,经K5800自动检测超微量分光光度计检测RNA浓度和纯度合格之后用PrimeScriptTMRT Master Mix反转录试剂盒合成cDNA第一链,-20℃保存备用。
利用实时荧光定量PCR检测RpWRKYs在大黄不同样品中基因表达,以β-actin为内参基因。使用TB Green®Premix ExTaqTMII(TliRNaseH Plus)进行qPCR。20 μL反应体系:2×TB Green®Premix ExTaqTMII(TliRNaseH Plus)10 μL、forward/reverse primer各0.4 μL、cDNA模板1 μL、50×ROX Reference Dye 0.2 μL、ddH2O 7 μL。反应程序:预变性95℃ 30 s;变性95℃ 5 s,退火60℃ 30 s,延伸60℃ 34 s,40个循环,反应结束后于95℃ 15 s,60℃ 1 min,95℃ 15 s条件下绘制熔解曲线。包括不加模板的对照在内,所有qPCR反应技术重复和实验重复各3次,应用2-ΔΔCt法计算相对表达量。
1.2.5 蒽醌合成关键酶基因与RpWRKYs蛋白相互作用分析 课题组前期根据二代转录组数据NR注释结果筛选得到蒽醌类生物合成途径的关键酶基因,并绘制蒽醌类生物合成代谢途径[20]。蒽醌母核的生物合成主要涉及莽草酸(shikimic acid)和聚酮(polyketide)途径,由于中间产物共用,甲基赤藓糖醇(methylerythritol 4-phosphate, MEP)、甲羟戊酸(mevalonic acid, MVA)途径也参与其中(图1)。使用string 11.5(http://string-db.org/)数据库预测RpWRKYs蛋白与蒽醌类物质合成关键酶基因的相互作用网络,选定模式植物拟南芥为物种参数,去除不成簇和单个节点的蛋白得到蛋白互作网络图。
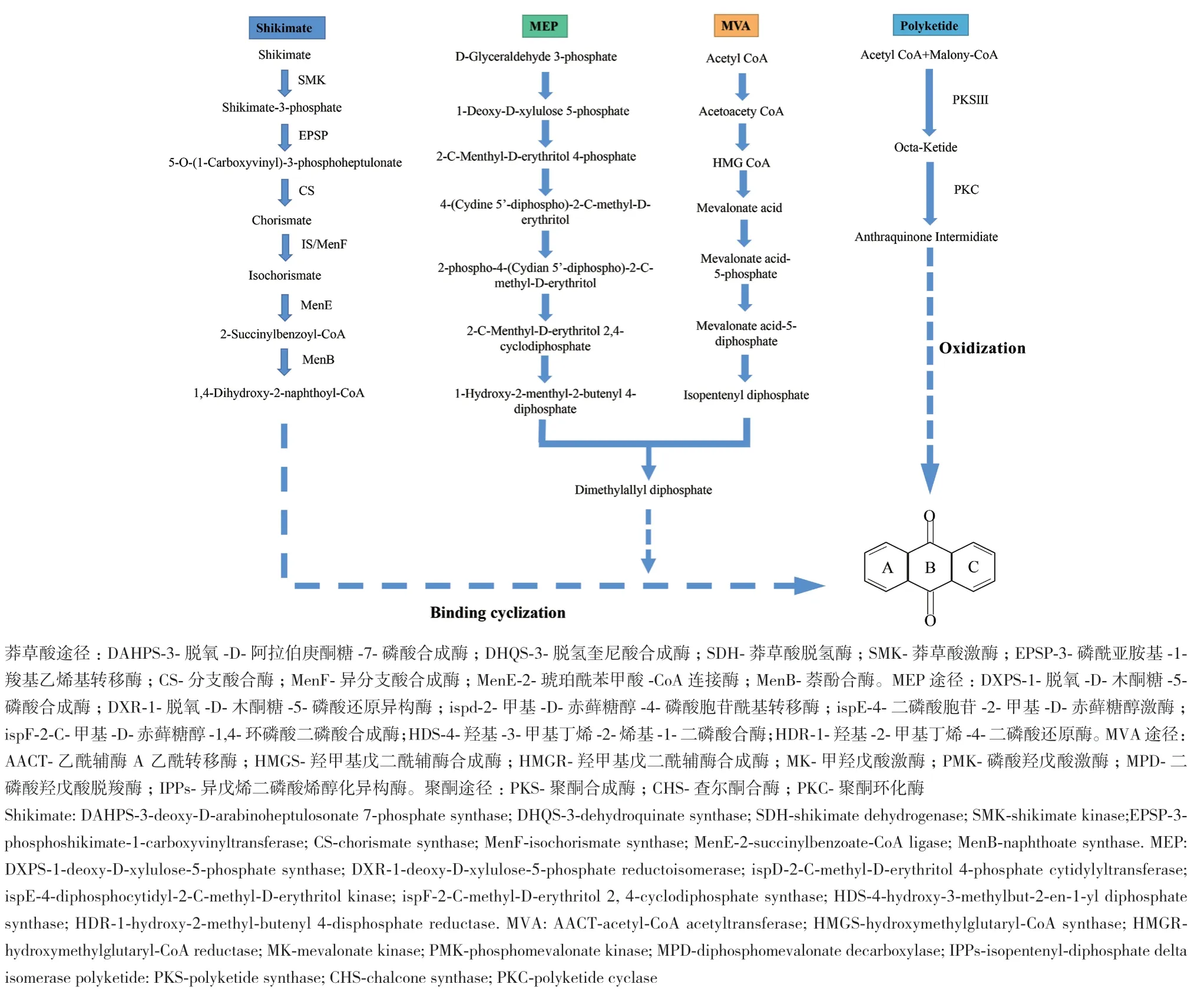
图1 大黄蒽醌类生物合成途径Fig. 1 Anthraquinone biosynthesis pathway in R. officinale Baill
2 结果
2.1 RpWRKYs序列分析
掌叶大黄全长转录组中有106条Isoform编码WRKY蛋白,其中,包含完整ORF的Isoform有62个。整理ORF差异位点并合并重复,最终获得53个全长RpWRKYs,编号RpWRKY1-RpWRKY53(表1)。序列最长的是RpWRKY1,由748个氨基酸编码,最短的是RpWRKY53,由192个氨基酸编码。
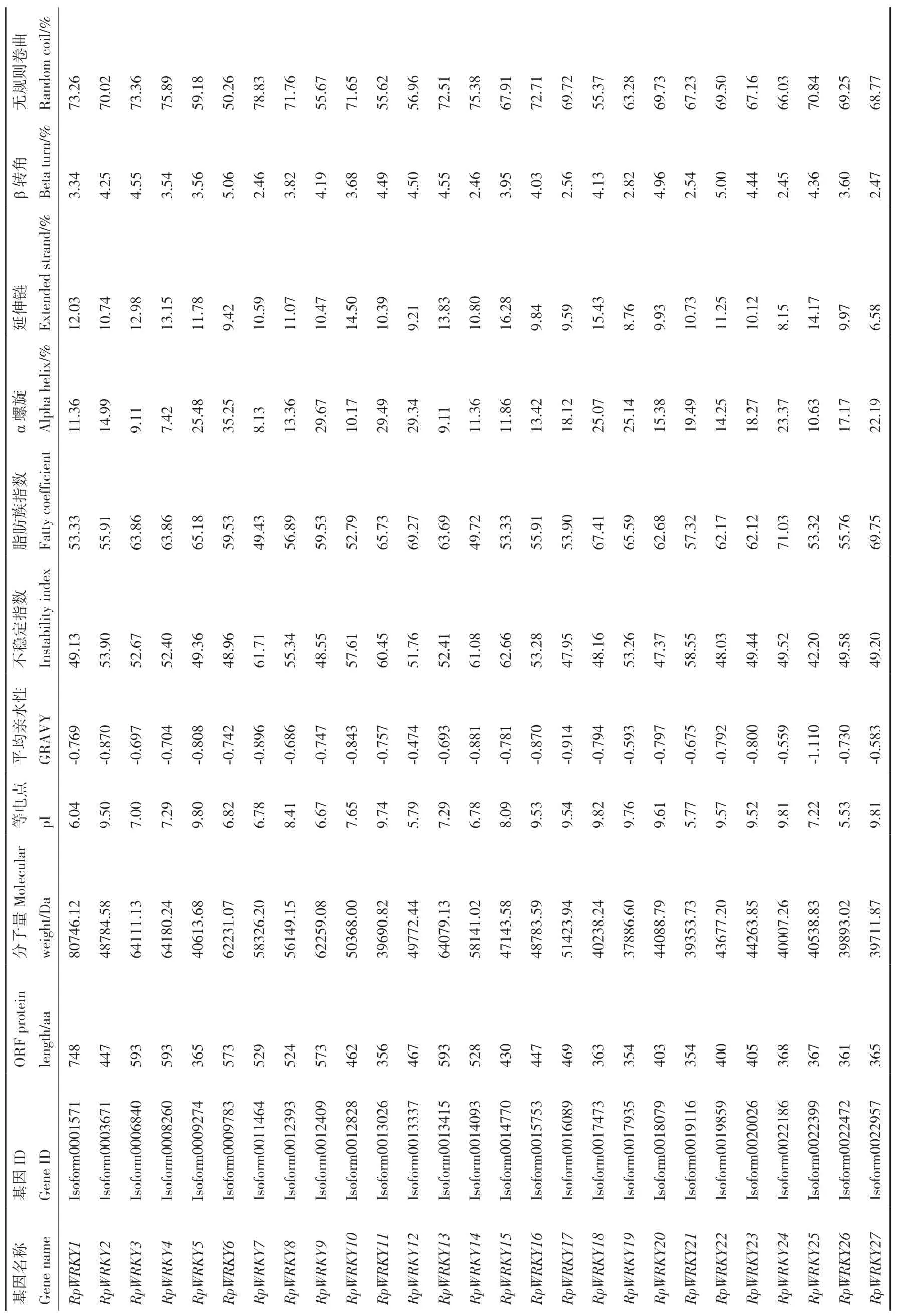
表1 掌叶大黄RpWRKY 转录因子的基本信息Table 1 Basic information of RpWRKY transcription factors in R. palmatum
蛋白质一级结构分析发现,基因编码蛋白的相对分子量在21.184(RpWRKY53)-80.746(RpWRKY1)kD之间,理论等电点介于5.40-9.92,包括37个碱性蛋白,16个酸性蛋白。所有RpWRKYs蛋白的平均亲水性数值是负值,说明其均为亲水性蛋白;RpWRKY44/53的不稳定系数<40,推测其为稳定蛋白,其余51个为不稳定蛋白;脂肪系数为41.61-73.92,说明RpWRKY转录因子基因编码蛋白的热稳定性较好。
RpWRKYs家族蛋白均具有α螺旋、延伸链、β转角和无规卷曲,所占比例较大的是α螺旋和无规卷曲(所占比平均值分别为20.59%、64.56%),β-折叠和延伸链占比较小(所占比平均值分别为11.25%、3.57%),散布于整个蛋白中。亚细胞定位结果显示,53个RpWRKYs蛋白均定位于细胞核。
2.2 系统进化分析
利用MEGA11构建掌叶大黄与拟南芥WRKY转录因子家族系统进化树。图2结果表明,53个RpWRKYs蛋白分为I、II、III 3个组,与拟南芥WRKY分类结果一致。I组的RpWRKY蛋白有16个,占30.18%,II组又分为II-a、II-b、II-c、II-d、II-e 5个亚组,含有RpWRKY家族成员31个,占58.49%,II-e(1个)和II-b(3个)两亚组RpWRKY基因分布较少;其次是III组,含有6个RpWRKYs基因,占11.32%。
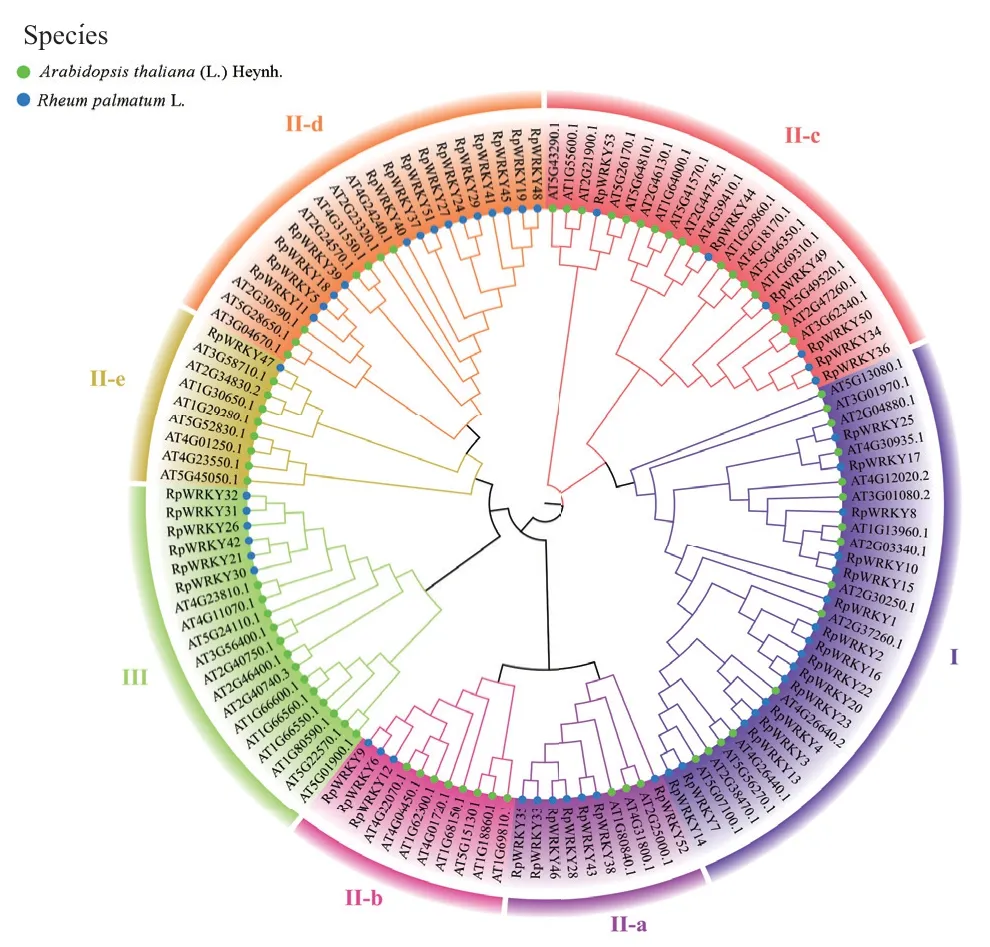
图2 掌叶大黄和拟南芥中WRKYs成员的进化树Fig. 2 Evolutionary tree of WRKYs members in R. palmatum and Arabidopsis thaliana
2.3 保守基序分析
通过MEME在线工具对RpWRKYs分析,结果(图3-A-B)表明,同组的RpWRKYs包含的保守元件数量和种类一致,而组间存在一定的差异性。53个RpWRKYs蛋白包含8个Motif保守基序,最短基序有29个氨基酸残基,最长的有50个氨基酸残基。由图3-C可知Motif1和Motif3为RpWRKYs蛋白的WRKYGQK七肽保守序列,Motif2和Motif5属于锌指结构基序,RpWRKYs蛋白基序分布与基因分类结果一致。

图3 RpWRKYs的进化树(A)、保守基序(B)和保守基序特征(C)Fig. 3 Evolution tree(A), conserved motifs(B)and conserved motif signatures(C)of RpWRKYs
2.4 基因组织表达和响应MeJA的表达分析
利用不同组织及MeJA组的RNA-seq数据分析掌叶大黄53个RpWRKYs基因的表达谱。结果显示(图4-A),同一基因在根、根茎、叶中表达差异较大,仅在根、根茎和叶中高表达的基因分别有21、11、10个。7个基因(RpWRKY5/12/25/28/30/42/47)在根和根茎中表达量均较高,叶中表达量最低。在MeJA处理下(图4-B),53个RpWRKYs基因呈现差异表达,其中RpWRKY6/7/9/14/33/35/38/43/46/47被MeJA显著诱导,RpWRKY5/18被显著抑制。
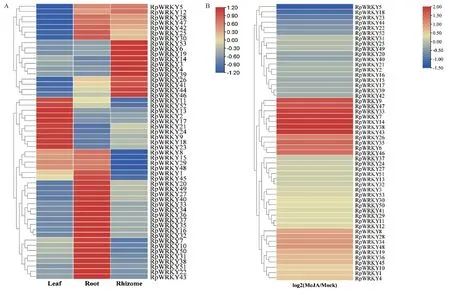
图4 掌叶大黄不同组织中RpWRKYs的表达模式(A)和MeJA处理下的响应(B)Fig. 4 Expression patterns of RpWRKYs in different tissues of R. palmatum(A)and in response to MeJA treatment(B)
2.5 RpWRKYs表达模式分析
qPCR分析检测RpWRKY5/6/9/33响应MeJA处理的表达结果见图5。以0 h(CK)为空白对照,结果显示4个基因于MeJA处理12 h的表达量与转录组测序数据基本一致。RpWRKY5在MeJA激素处理下4个时间段表达量均下降,且在12 h下调最明显,为Mock的0.09倍。RpWRKY6/9/33在12 h上调表达,分别为Mock的58.19、2.67、10.66倍,且RpWRKY6于12 h达峰值。
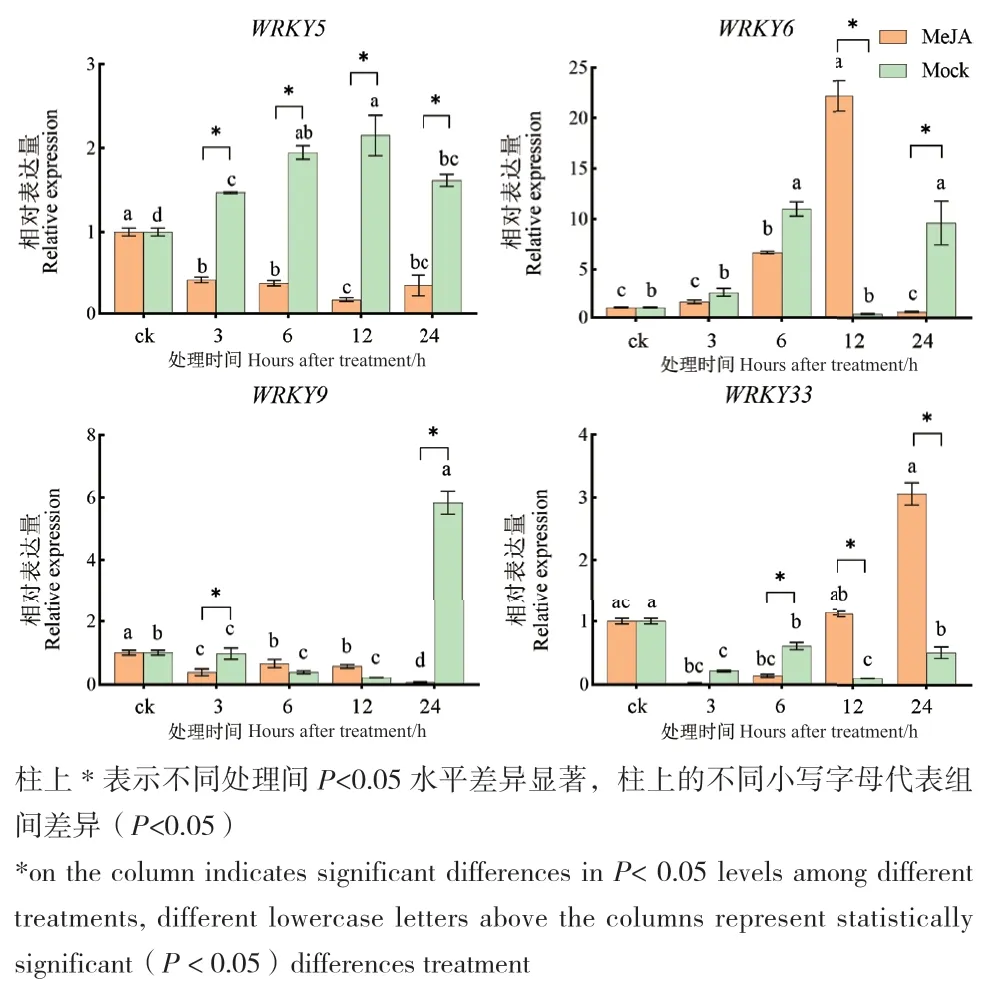
图5 RpWRKY5/6/9/33在MeJA处理下的表达模式Fig. 5 Expression pattern of RpWRKY5/6/9/33 in response to MeJA treatment
2.6 RpWRKYs蛋白的相互作用分析
使用STRING 11.5在线软件构建53个RpWRKY蛋白与蒽醌类生物合成关键酶蛋白的蛋白质-蛋白质相互作用网络(图6),模式植物设置为拟南芥,去除不成簇和单个节点的蛋白。结果显示,共有19个RpWRKY蛋白相互作用,其中AtWRKY40(RpWRKY28/33/35/38/43/46)属于网络中的中心节点,其余6个AtWRKY均与AtWRKY40有相互作用。5个RpWRKYs蛋白(RpWRKY2/16/20/22/23)通过CHS与其他蒽醌合成途径关键酶基因连接成簇。
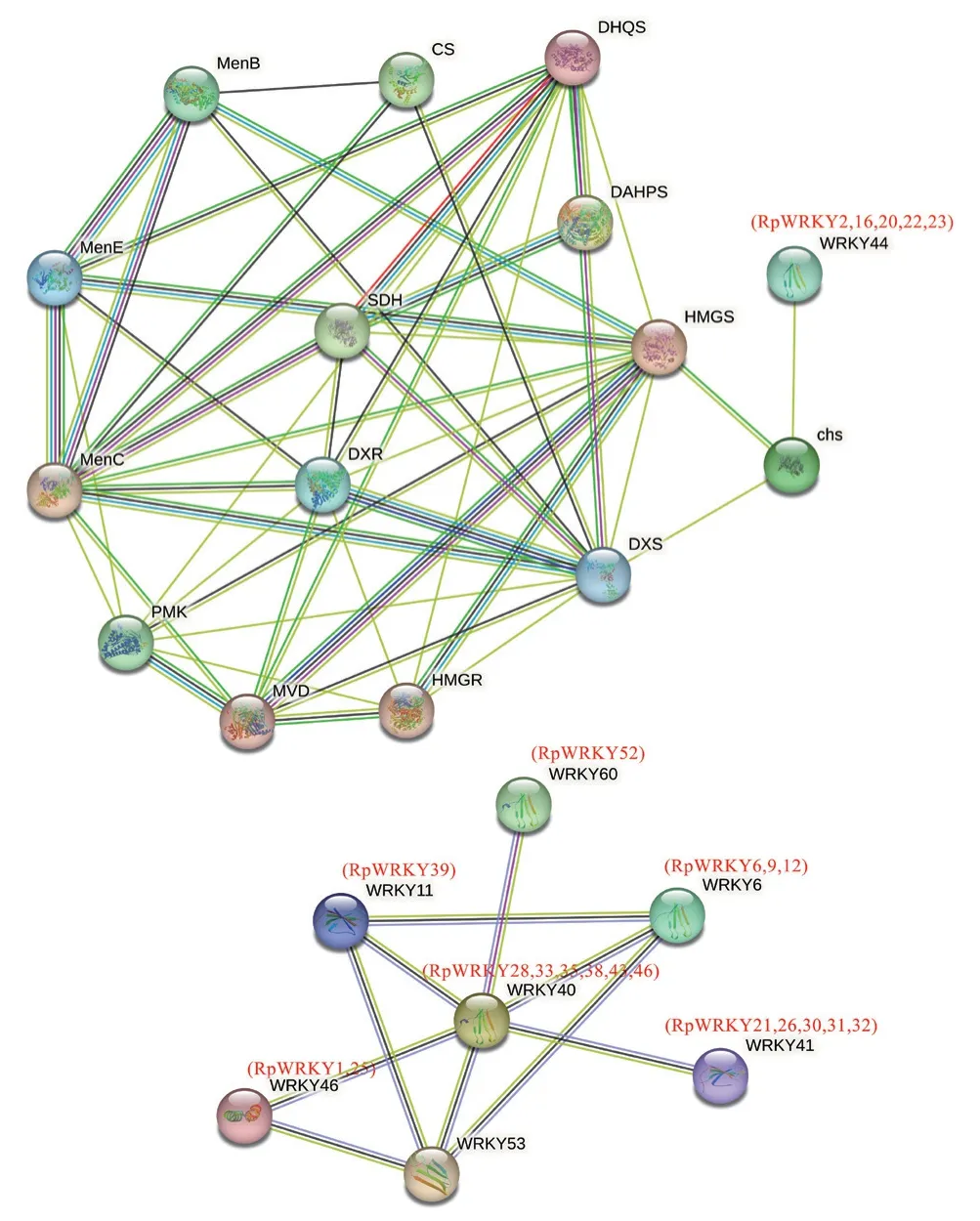
图6 RpWRKY蛋白质-蛋白质相互作用网络Fig. 6 Protein interactions of protein RpWRKYs
3 讨论
WRKY基因家族是调节植物生长和次生代谢的最大转录因子家族之一,广泛参与植物对生物、非生物和激素胁迫的响应,以及植物生长发育过程[21]。WRKY家族已经在模式植物拟南芥[22]、经济作物大豆[23]、马铃薯[24]和许多药用植物如人参、丹参、板蓝根[25]中进行了研究。本研究利用第三代测序技术分析了掌叶大黄的全长转录组,从掌叶大黄中鉴定到53个RpWRKYs全长基因,和黄连(41)[26]、马蓝(65)[27]及杜仲(51)[28]等药用植物中WRKY基因数量差距不大。但相对于大豆、拟南芥和马铃薯基因组的188、72、79个基因[29],掌叶大黄RpWRKYs基因数量较少,说明在药用植物中WRKY基因家族的数量相较于模式植物和经济作物来说发现的较少,随着未来掌叶大黄基因组测序的深入研究,将会鉴定出更多未发现的RpWRKYs基因,进一步丰富大黄WRKY基因家族。
系统发育分析显示RpWRKYs基因被分为3大组(I、II和III),与拟南芥WRKY基因家族[21]分组一致。其中,II-e(1个)和II-b(3个)组RpWRKYs分布较少。研究发现II-e组AT1G30650.1(WRKY14)基因为拟南芥热形态发生的抑制因子[30],RpWRKY47与其聚为一个分支,说明RpWRKY47可能在掌叶大黄温度胁迫机制中起重要作用。
基因结构决定基因功能,对RpWRKYs的结构进行预测发现其均为热稳定性良好的亲水性蛋白,亚细胞定位预测RpWRKYs均定位在细胞核中,说明RpWRKYs的转录调控过程主要在细胞核内进行。相同或相似的基序是维系蛋白质结构域进而发挥生物学功能的重要前提。对53个RpWRKYs蛋白的保守基序分析发现,组内Motif分布基本一致,组间存在差异且Motif在不同组的分布符合WRKY家族分类的特征。Motif1和Motif2存在于所有RpWRKYs序列中,说明这两个基序高度保守,为WRKY的特征基序[31]。
RpWRKYs在掌叶大黄中有着明显的组织表达特异性,仅在根中高表达的有21个,高表达基因数目明显多于根茎(11)和叶(10),RpWRKY5/12/25/28/30/42/47等7个基因在根和根茎中表达量均较高,可能参与了掌叶大黄根及根茎的生长发育过程。相较于RpWRKYs主要在根及根茎中有较高表达,马蓝中高表达WRKY则主要分布于根茎及叶片中,说明WRKY在不同物种不同部位中的表达差异较大。Li等[32]检测发现PgWRKYs表达趋势与桔梗皂苷含量变化一致,推测PgWRKYs可能调节桔梗皂苷的代谢。本课题组后续也会结合掌叶大黄全生育期活性成分含量变化及其与RpWRKYs表达量的关联分析,进一步明确可能参与大黄蒽醌类成分合成的关键WRKY基因。
MeJA参与了植物生长发育和胁迫反应等多个生理过程,同时能够调节植物的次生代谢[33]。为研究中药活性成分生物合成途径,通常在可控培养环境下,以MeJA等激素处理药用植物,通过多组学分析,挖掘关键调控基因。课题组前期研究发现,外源MeJA处理可显著诱导掌叶大黄蒽醌类成分积累,RNA-seq揭示MeJA信号通路、次生代谢通路、抗病防御通路均得到显著富集,而我们关注的RpWRKYs基因家族成员呈现差异表达。10个RpWRKYs基因显著诱导,只有RpWRKY5和RpWRKY18被MeJA抑制。掌叶大黄不同组织表达模式及其对MeJA的响应表明,这些基因可能在掌叶大黄的生长发育和次级代谢中发挥重要作用。本研究中RpWRKYs在MeJA处理下显示出不同的响应模式,RpWRKY6/9/33受MeJA显著诱导,推测可能参与掌叶大黄对外界逆境的防御反应。
蛋白质-蛋白质互作网络,有助于调节生物信号传递和相关基因表达。研究表明AtWRKY6和AtWRKY42相互作用,参与拟南芥对低Pi胁迫的响应[34]。对于RpWRKY蛋白互作网络预测分析表明,AtWRKY40(RpWRKY28/33/35/38/43/46)属于网络中的中心节点,可能在掌叶大黄生长发育以及生物和非生物胁迫等应答过程中发挥重要作用。CHS基因是聚酮代谢途径的关键基因,在蒽醌类物质合成中扮演着重要的角色,如决明CHS-L9参与蒽醌类物质的生物合成[35]。蛋白互作预测AtWRKY44(RpWRKY2/16/20/22/23)与CHS关系密切,推测RpWRKY2/16/20/22/23可能参与了掌叶大黄蒽醌类物质的生物合成过程。综上所述,本研究为进一步研究WRKY转录因子在大黄次生代谢调控及药材品质形成过程中作用机制提供依据。
4 结论
本研究鉴定了掌叶大黄53个WRKY基因,并对其进行理化性质、遗传进化和表达模式分析,探究了不同RpWRKY对MeJA的响应模式以及RpWRKY的组织表达特异性。研究结果显示WRKY家族基因在掌叶大黄根及根茎中表达量较高,RpWRKY在不同时间MeJA处理下有着不同的响应模式。蛋白互作结果表明有5个RpWRKY基因可能与大黄中蒽醌类物质的合成有关。