基于大球盖菇基因组的SSR分子标记开发及指纹数据库应用
2024-02-05李雪松孙达锋刘绍雄张俊波岳万松李建英
李雪松,孙达锋,刘绍雄,张俊波,岳万松,李建英,华 蓉,*
(1.中华全国供销合作总社昆明食用菌研究所,云南 昆明 650221; 2.云南省食用菌产业发展研究院,云南 昆明, 650221)
大球盖菇(StrophariarugosoannulataFarl. ex Murrill,S.rugosoannulata),别名皱环球盖菇、酒红色球盖菇等,商品名为赤松茸,属于担子菌门(Basidomycota) 层菌纲(Hymenomycetes)伞菌目(Agaricales)球盖菇科(Strophariaceae)球盖菇属(Stropharia)[1]。大球盖菇原产于美国,引进国内后在我国的西南地区得到广泛种植[2]。大球盖菇的栽培条件简单,模式多样,原料广泛,可生料栽培,发展前景广阔[3]。大球盖菇是一种营养丰富、口味鲜美、品质优良的食用菌。同时,在抗疲劳[4]、抗氧化[5-7]、抗肿瘤[8]和免疫调节[9]、降血糖血脂[10]、抑菌[11]等方面具有一定的药用价值。据中国食用菌协会统计,2018年和2019年我国皱环球盖菇产量比上一年分别增长了96.92%和149.99%,2019 年产量超过了14万t[12]。
随着大球盖菇栽培面积逐渐扩大,优异栽培品种短缺、品种混乱、种性退化等菌种问题尤为突出[13],阻碍了大球盖菇产业的健康发展。当前大量学者在大球盖菇的生物学特性、农艺性状、规范化栽培等方面开展了一系列研究,在分子水平开展大球盖菇的遗传多样性、分子标记、育种等方面的研究较少。分子标记是一种遗传标记,是以个体间遗传物质内核苷酸序列变异为基础的,可以直接反映出DNA水平的遗传多态性[14],其中简单重复序列标记就是应用较为广泛的标记之一。
简单重复序列(simple sequence repeat, SSR)又被称为短串联重复序列(simple tandm reprat,STR)或微卫星标记(microsatellita),是由2~6个核苷酸的串联重复片段组成,分布于真核生物基因组中。SSR 分子标记具有分布广泛、位点特异、重复性高等优点[15-17],因此,在食用菌的相关研究中被应用于种质资源鉴定、遗传差异及多样性、分子标记辅助育种等方面[18-21]。其中,在羊肚菌、灵芝、黑木耳、金针菇、双孢蘑菇、蛹虫草、暗褐网柄牛肝菌等食药用菌的研究中均有报道[22-28]。近年来,随着越来越多的大型真菌基因组的完成及公布,使得基于基因组开发物种特异的SSR引物成为可能。但目前尚未见基于基因组序列开发大球盖菇SSR标记与应用的相关研究报道。
本研究收集了13个大球盖菇菌株,基于已公布的大球盖菇基因组数据开发了SSR分子标记,并利用开发的SSR分子标记对13个大球盖菇菌株进行鉴别,构建了指纹编码,明确13个大球盖菇菌株间的遗传差异;同时,基于本研究开发的SSR标记为大球盖菇的种质资源鉴定、品种选育、分子标记辅助育种等研究提供理论基础。
1 材料与方法
1.1 材料
13份测试菌株收集自我国的多个地区,其中DQG-1、DQG-2、DQG-4、DQG-5和ZJJQG001来自云南昆明,DQG-3和DQG-8来自云南曲靖,DQG-9来自云南普洱,DQG-6和DQG-7来自江苏,DQG-10、DQG-11、DQG-12分别来自湖北、山东、四川。目前所有菌株保存于中华供销合作总社昆明食用菌研究所菌种与资源研究中心。参考卯晓岚[29]的《中国大型真菌》,13份测试菌株均为大球盖菇菌。测试菌株中除了ZJJQG001子实体菌盖颜色为金黄色,其余菌株子实体菌盖颜色均为酒红色。
此外,从GenBank数据库中下载球盖菇属(Stropharia)与香菇(Lentinulaedodes)的ITS序列,用于分子鉴定及系统发育分析,其中大球盖菇的ITS登录号为FJ810166.1、KC176328.1、MH860190.1,吉林球盖菇(S.jilinensis)的ITS登录号为JF961347.1,齿环球盖菇(S.coronilla)的ITS登录号为MH856747.1,浅赭色球盖菇(S.hornemannii)的ITS登录号为MT955153.1,木生球盖菇(S.lignicola)的ITS登录号为MW492530.1,铜绿球盖菇(S.aeruginosa)的ITS登录号为MW492534.1,哈迪球盖菇(S.hardii)的ITS登录号为MW821365.1,香菇的ITS登录号为KT693242.1。从NCBI数据库下载大球盖菇的基因组序列(GCA_003314255.1)[30]。
1.2 方法
1.2.1 菌丝体培养
取不同菌株的菌丝接种于马铃薯葡萄糖琼脂平板培养基(PDA)上,25 ℃避光静置培养20 d,收集菌丝,-80 ℃保存。
1.2.2 基因组DNA提取
取保存的菌丝,使用DNA提取试剂盒(TSP101-200)提取DNA。DNA纯度和浓度用0.8%琼脂糖凝胶和NanoDrope 2000检测,-20 ℃保存备用。
1.2.3 ITS 扩增与检测
使用ITS 通用引物ITS4、ITS5进行PCR扩增。25 μL扩增体系包括:金牌Mix(green,擎科 TSE101)20 μL、0.5 μmol·L-1上下游引物、50 ng 基因组模板DNA。PCR 扩增程序为:94 ℃ 2 min;94 ℃ 15 s、60 ℃ 30 s、72 ℃ 60 s,30个循环;72 ℃ 10 min。PCR完成后,加6×上样缓冲液5 μL ,混匀,1.0%琼脂糖凝胶电泳检测。检测合格后,进行测序。
1.2.4 序列比较与系统发育分析
以球盖菇属(Stropharia)物种的ITS序列作为参照,对13个菌株是否为大球盖菇进行分子鉴定。首先,使用Clustal W 1.8进行多重比对,对结果进行人工修正。然后,使用MEGA 7软件进行系统发育分析树构建,以Kimura 2-parameter模型计算遗传距离,比对结果中空位或缺失碱基覆盖率小于95%的位置进行删除。以香菇(Lentinulaedodes)作为复合外群,用NJ 法(neighbor-joining method)构建分子进化树,设置1 000次自展值进行检验。
1.2.5 基于基因组序列设计和筛选大球盖菇的特异性微卫星引物
使用软件Krait检测并设计 SSR 引物,筛选出适合扩增的位点不重复的100对引物,进行合成。SSR-PCR扩增反应体系(20 μL):2×TSINGKE Master Mix(blue) 16.45 μL,10 μmol·L-1Primer F(加接头)0.15 μL,10 μmol·L-1Primer R 1.2 μL,10 μmol·L-1Tag (荧光)1.2 μL,模板DNA 1 μL。反应条件:98 ℃预变性2 min;98 ℃变性10 s,60 ℃退火10 s,72 ℃延伸5 min,共35次循环。将扩增好的PCR产物进行琼脂糖凝胶电泳。根据鉴定结果来确定模板浓度,加水稀释到合适浓度,进行毛细管电泳。
1.2.6 SSR引物的多样性分析
获取毛细管电泳的下机数据后,使用Gene mapper4.1软件进行分析,去除不合格数据,最终建立原始数据矩阵。使用Popgen32软件计算SSR位点的各项遗传多样性指标,包括观测等位基因(Na)、有效等位基因(Ne)、香农指数(I)、多态性信息指数(PIC)、观测杂合度(Ho)、期望杂合度(He)等。同时,使用populations软件构建UPGMA树进行遗传结构分析,自展值设置为1 000。
2 结果与分析
2.1 大球盖菇的ITS鉴定与系统发育分析
将获得的原始测序数据进行质控,经过质控后的数据使用BLAST软件将其与GenBank 数据库进行比对。结果表明,13个菌株的ITS与数据库大球盖菇的相似度在99%以上。同时,从系统发育树可以看出球盖菇属的物种与香菇分开为两个大的进化枝;在球盖菇属的进化分枝中,13个菌株与大球盖菇聚为一个小枝,且支持率为100%。结合形态学的鉴定结果,可以认定13个收集菌株均为球盖菇属的大球盖菇(图1)。
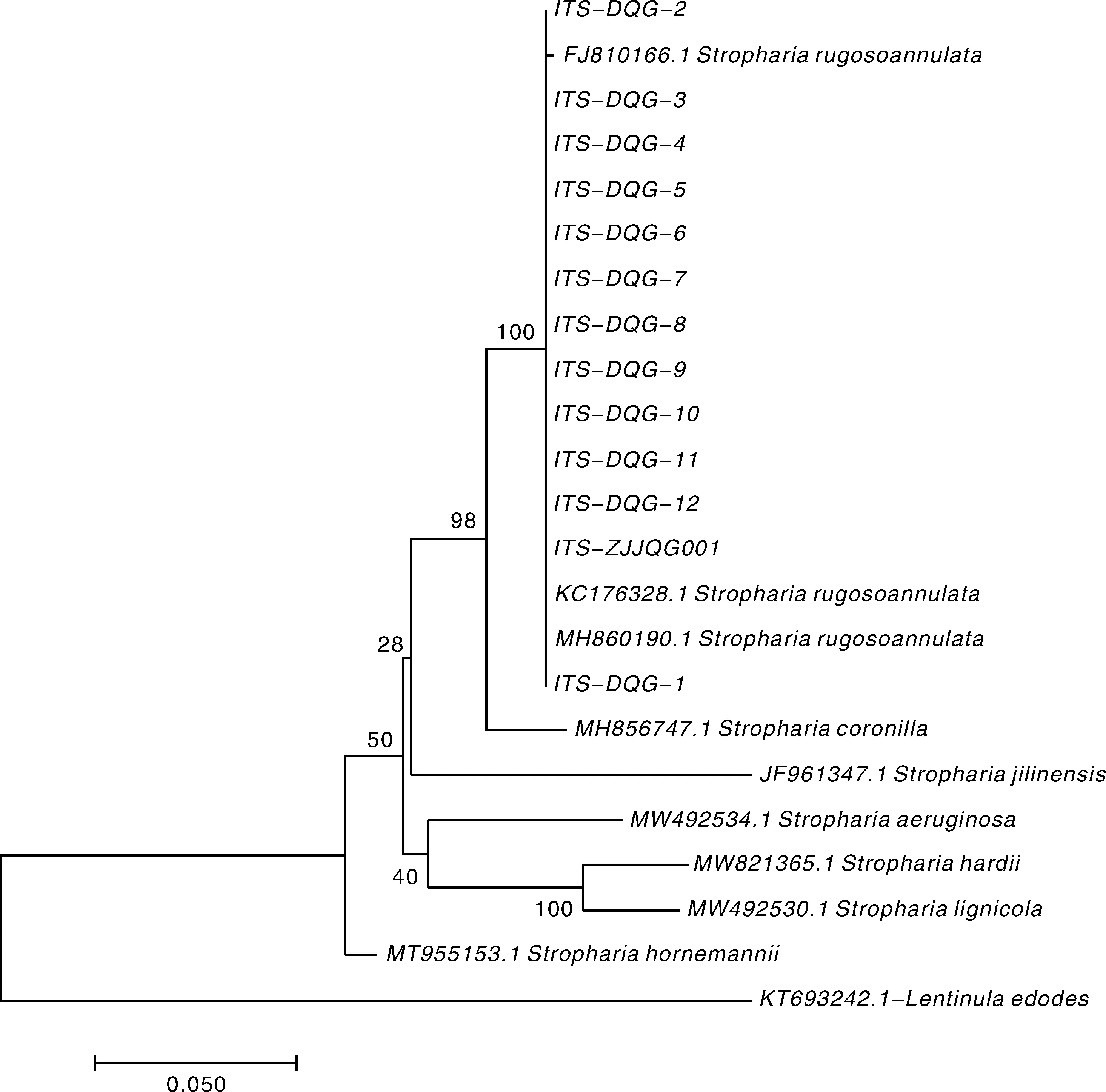
图1 基于ITS的13个大球盖菇菌株的NJ系统发育树Fig.1 NJ phylogenetic tree of 13 S. rugosoannulata strain based on ITS
2.2 大球盖菇全基因组中SSR分布特点
共检索到2 124个SSR,有6种SSR类型,其中主要以三核苷酸(P3)类型为主。单碱基至六碱基SSR分别为372、320、704、418、153和157个,其中三碱基重复类型的SSR数目最多,共计704,占总数的33.15%。单碱基重复占总SSR的17.51%,主要以AT类型为主,有346个,CG类型有26个;其次,二碱基重复占总SSR的15.07%,主要类型有AC/GT、AG/CT、AT、GC、GA/TC、TA、CG和CA/TG,分别为156,31,39,3,27,33,4和27个;四碱基的重复类型也较多,占总数的19.68%;五、六碱基重复类型较多,但总和只占全部SSR数量的14.59%(图2)。
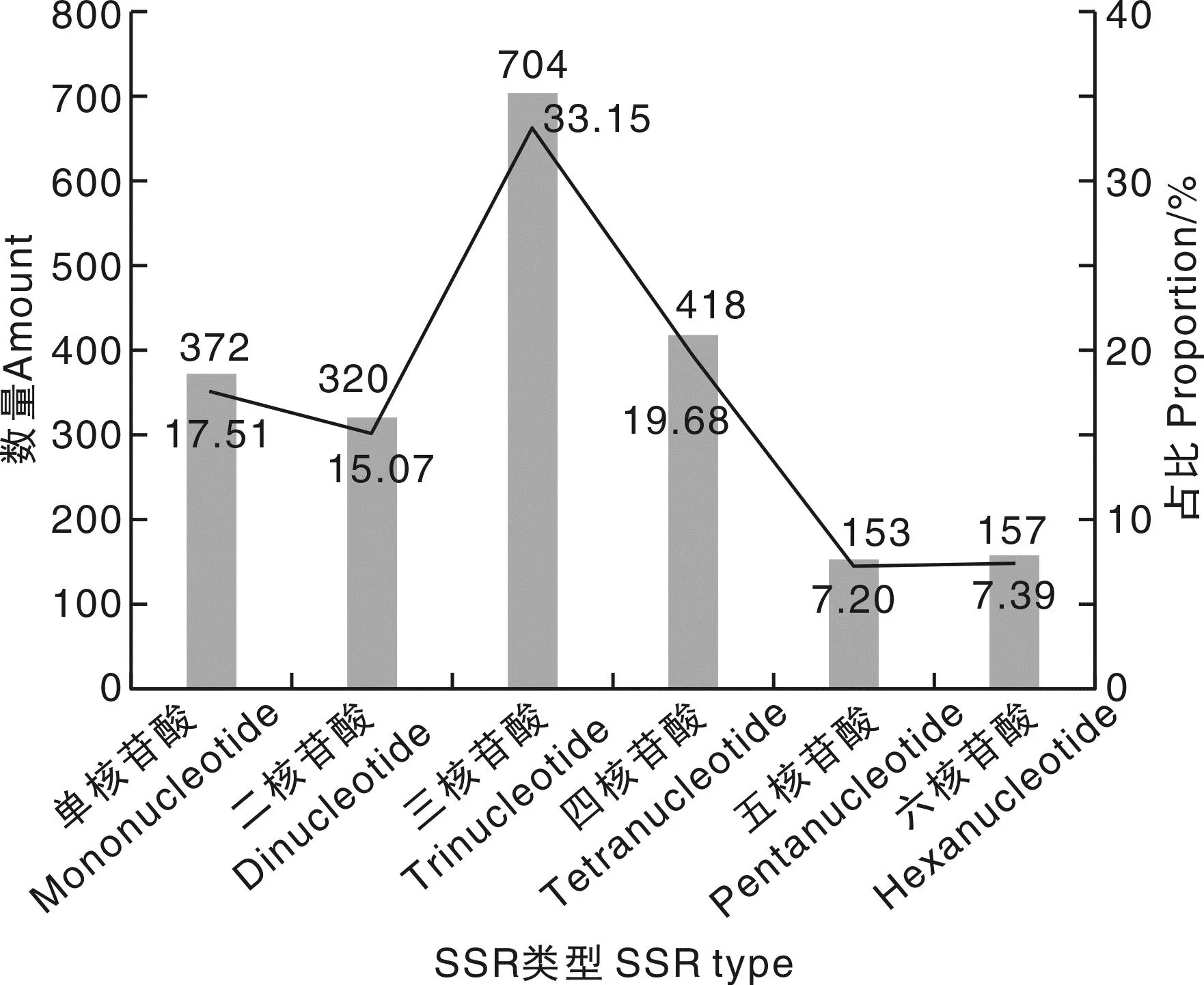
图2 大球盖菇基因组中SSR主要重复基元的分布Fig.2 Distribution of SSR repeat motifs of S. rugosoannulata
2.3 大球盖菇SSR引物筛选与遗传多样性
根据大球盖菇的基因组SSR的位点统计与引物设计结果,从中选择符合要求的100对SSR进行合成和实验筛选。通过扩增实验和毛细管电泳检测100对SSR引物的多态性与稳定性,最终筛选出16对多态性高、重复性好、特异性好的引物,分别为DQGSSR006、DQGSSR007、DQGSSR020、DQGSSR029、DQGSSR031、DQGSSR034、DQGSSR035、DQGSSR037、DQGSSR039、DQGSSR052、DQGSSR063、DQGSSR065、DQGSSR077、DQGSSR081、DQGSSR088和DQGSSR097。16对SSR引物的信息见表1。
筛选获得的16对SSR引物的扩增条带最少为2条,最多为5条。毛细管电泳检测获得的片段条带清晰、无杂峰,有助于后续的大球盖菇的遗传聚类和指纹编码等分析。16对SSR引物。其中,DQGSSR020、DQGSSR031、DQGSSR077的多态性较高、特异性好、无杂峰、荧光亮度高、区分度好,扩增出的片段与预期的基本一致(表2)。

表2 16对SSR引物的扩增信息统计
16对SSR引物在13个大球盖菇菌株中共检测出42个等位基因位点(,频率最高的等位基因为原始型)(表3)。其中,最小等位基因数目为2,最大等位基因数目为5,平均每个位点等位基因数目为2.63。有效等位基因总数为34.72,数值变化范围为1.17(DQGSSR006)~3.80(DQGSSR020),平均每个位点有效等位基因数目为2.17。其中,等位基因在群体中分布得越均匀,Ne越接近实际检测到的等位基因数。香农指数的数值范围为0.27(DQGSSR006)~1.36(DQGGSSR020),平均值0.79。多态信息含量为0.13(DQGSSR006)~0.69(DQGSSR020),平均值0.41,其中14对引物具有较高的多态信息(PIC>0.25)。
将13个大球盖菇菌株的16对SSR引物进行扩增,使用populations软件对扩增产物进行分析,基于Nei的遗传距离构建13个大球盖菇菌株的UPGMA聚类树。图3显示出13个样品大致分成了3个大的进化枝,DQG-1、DQG-4、DQG-5、DQG-8、DQG-9、DQG-10、DQG-11、DQG-12为一个大的进化枝,DQG-2、DQG-3、DQG-6、DQG-7又为一个大的进化分枝,子实体为金黄色的ZJJQG001单独为一个分枝。UPGMA树的聚类结果与菌盖颜色的表型呈现一致,即除ZJJQG001外,其余的大球盖菇的菌盖均体现为酒红色。
2.4 大球盖菇指纹编码构建
用DQGSSR006、DQGSSR007、DQGSSR020、DQGSSR029、DQGSSR031、DQGSSR034、DQGSSR035、DQGSSR037、DQGSSR039、DQGSSR052、DQGSSR063、DQGSSR065、DQGSSR077、DQGSSR081、DQGSSR088和DQGSSR097这16对引物进行大球盖菇DNA 指纹编码数据库的构建。DNA指纹数据库编码的构建方法参照陈世军等[31]和陈昌文等[32]的赋值原则,根据16对引物扩增出的片段大小的顺序对13个大球盖菇菌株进行编码(表4),编码串联后的数字组合即为该大球盖菇菌株的DNA 指纹数据库编码。基于这16对SSR标记对每个大球盖菇菌株的扩增片段组合进行编码,获得每个大球盖菇菌株的36位数分子指纹数据库编码(表5)。
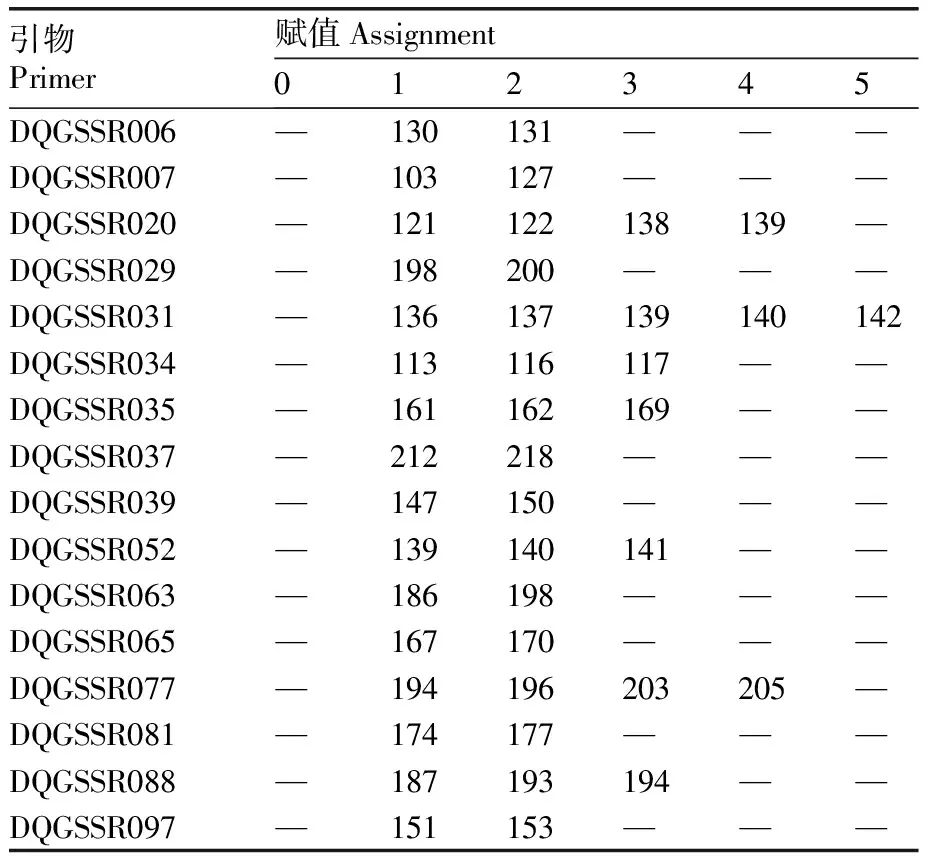
表4 扩增片段与赋值
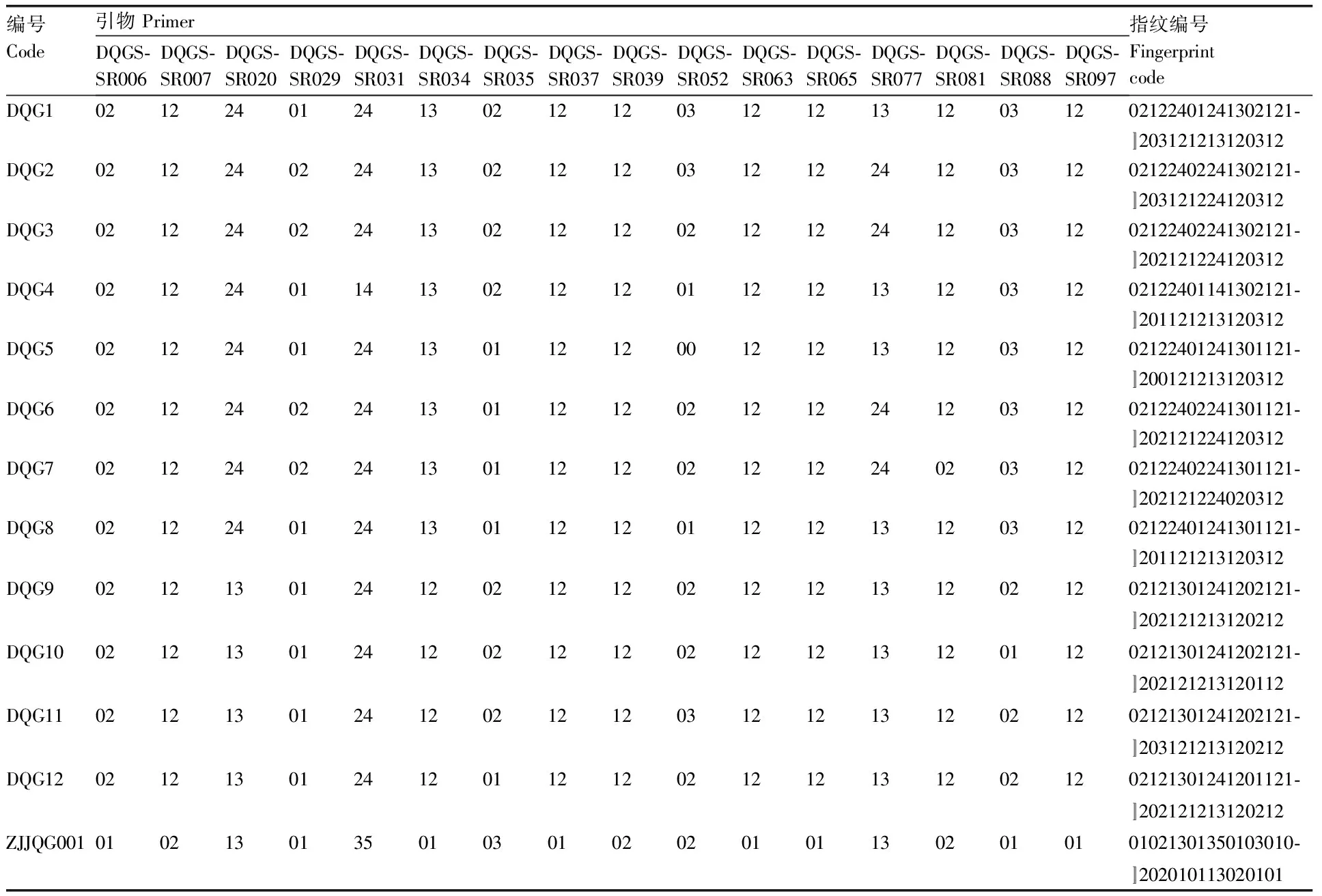
表5 大球盖菇资源指纹数据库编码
3 讨论
随着食用菌产业的不断发展,越来越多的菌种将流通于市场,菌种真实性鉴定也将广泛推广[33]。目前,菌株的鉴定主要是通过对菌丝体的形态特征、拮抗试验、子实体的形态特征、生理生化指标等来完成,但以上鉴定方法存在易受环境因素、人为误差等不确定因素的影响。随着分子生物学技术的发展,一些分子标记技术和方法已经应用于菌株鉴定的相关研究中,使得不同品种的鉴定变得便捷。
大球盖菇是具有食药用价值、栽培稳定、产量高的食用菌之一,具有一定的发展前景及优势。而如何对不同的大球盖菇菌株进行准确鉴定区分,就是大球盖菇产业发展中急需解决的问题。SSR分子标记有分布广泛、位点特异、重复性高等优点[15-17],在多个食用菌品种的相关研究中均有报道,如:真姬菇[20]、金针菇[19,22]、香菇[21]、黑木耳[23]、灵芝[24]、双孢蘑菇[26]等。在羊肚菌[25]、暗褐网柄牛肝菌[28]等食用菌中已有将SSR分子标记应用于品种鉴定的报道。
本研究中,基于已公布的大球盖菇基因组数据开发了SSR分子标记。首先开展了大球盖菇基因组SSR分布的分析,共检索到2 124个SSR,总数多于云芝[18],与花脸香蘑[34]接近;重复类型主要以三核苷酸类型为主,占总数的33.15%;五、六碱基重复类型较多,但总和只占全部SSR数量的14.59%。而花脸香蘑[34]主要以单碱基类型为主,占总数的67.70%。这说明不同大型真菌的SSR总数、重复类型、重复数目等都存在较大的差异。
通过设计100对引物进行验证,筛选出16对多态性高、特异性好的SSR引物。最近,有学者在花脸香蘑SSR标记[34]的开发中就使用了花脸香蘑的基因组进行SSR引物的设计及验证,结果表明,开发出的SSR标记可以较好地区分不同花脸香蘑菌株,可以精确地进行菌株之间的区分。
筛选出的16对SSR引物的Shannon信息指数平均值为0.79,低于暗褐网柄牛肝菌[28],但高于工厂化主栽的双孢蘑菇品种[35],说明大球盖菇SSR引物的多态性介于两者之间。而期望杂合度和观察杂合度高于青海黄色类群羊肚菌[25]。陈绪涛等[12]参考NYT1730—2009《食用菌菌种真实性鉴定 ISSR法》中28个ISSR引物开展了大球盖菇菌株遗传多样性分析,结果表明,使用的ISSR引物可以进行不同类群的划分,但无法对每一个菌株进行精准鉴定,而且结果不易统计,存在统计误差。基于16对大球盖菇SSR引物构建的13个大球盖菇菌株的UPGMA聚类树显示,13个大球盖菇菌株大致分成了3个大的类群,金黄色的大球盖菇(ZJJQG001)独立为一类。其他的12个红色大球盖菇的聚类与地理来源不符,说明栽培使用的大球盖菇菌株较为混乱,12个红色大球盖菇菌株原始的来源可能是源于3个不同的菌株。
使用本研究开发的16对SSR分子标记对13个大球盖菇菌株构建指纹编码库,获得13个大球盖菇菌株的32位数分子指纹编码。16对大球盖菇SSR引物组合虽然不能对实验使用的13个大球盖菇菌株进行精准区分,但可区分大部分的大球盖菇菌株,结果为后续大球盖菇种质资源鉴定、品种选育、分子标记辅助育种等提供了基础。
