脂肪酸代谢途径影响聚酮化合物生物合成的机制及应用研究进展
2024-01-26曹丽朱梓榕夏梓源金多夏立秋
曹丽 朱梓榕 夏梓源 金多 夏立秋
摘 要:聚酮化合物(PKs)作为一大类次级代谢产物,有着重要的生物活性和潜在的应用价值。链霉菌具有合成多种聚酮化合物的潜力,但野生型菌株合成聚酮化合物的产量难以满足工业化生产的需求。贮藏脂质的降解能为聚酮化合物生物合成提供大量的酰基CoA前体,因此,控制好脂肪酸与聚酮化合物生物合成通量,有利于促进目标聚酮化合物的合成。本文综述了强化脂肪酸β-氧化途径提高聚酮化合物产量的研究进展,为利用β-氧化途径促进聚酮化合物生物合成提供了新的研究策略。
关键词:聚酮化合物;脂肪酸代谢;β-氧化;生物合成;链霉菌
中图分类号:Q936 文献标志码:A DOI:10.3969/j.issn.1007-7146.2023.01.001
Research Progress in the Mechanism and Application of Fatty Acid Metabolism Pathway Affecting the Biosynthesis of Polyketides
CAO Li, ZHU Zirong, XIA Ziyuan, JIN Duo, XIA Liqiu*
(Hunan Provincial Key Laboratory for Microbial Molecular Biology, State Key Laboratory of Developmental Biology of Freshwater Fish, College of Life Science, Hunan Normal University, Changsha 410081, China)
Abstract: As a large class of secondary metabolites, PKs have important biological activity and economic value. Streptomyces has the potential to synthesize a variety of PKs, whereas the yield of PKs synthesized by wild-type strains is difficult to meet the needs of industrial production. The degradation of storage lipids can provide a large number of acyl-CoA precursors for the biosynthesis of PKs. Therefore, controlling the balance of the biosynthetic flux of fatty acids and PKs is conducive to improving the production of the target PKs. This paper briefly reviews on improving the production of PKs by strengthening the fatty acids β-oxidation pathway, and prospects prospective research ideas on improving the biosynthesis of PKs by engineering the β-oxidation pathway.
Key words: polyketides; fatty acid metabolism; β-oxidation; biosynthesis; Streptomyces
(Acta Laser Biology Sinica, 2023, 32(1): 001-007)
聚酮化合物(polyketides,PKs)是一大类结构复杂且生物活性多样的次级代谢产物。细菌、放线菌、真菌和植物均是聚酮化合物的生产者,其中,放线菌科链霉菌属(Streptomyces)更适合或兼容高GC含量的含聚酮合成酶(polyketide synthases,PKSs)的异源生物合成基因簇,且链霉菌胞内代谢物及中间产物极其丰富,可为聚酮化合物的生物合成提供大量的前体,因此,链霉菌在聚酮化合物生产中极具优势[1-2]。聚酮化合物通过多功能聚酮合成酶催化合成,其合成過程与脂肪酸合成酶(fatty acid synthase,FAS)催化的脂肪酸生物合成类似,均是通过酰基辅酶A(acyl-CoA)扩展单元的脱羧缩合反应进行的生物合成[3]。根据聚酮合成酶的结构及其合成机制,聚酮化合物可被分成3类。I型聚酮化合物,聚酮合成酶是以模块形式存在的,每个模块均含有非重复使用的催化结构域[4]。I型聚酮化合物每个模块均包含酰基转移酶(acyltransferase,AT)、酰基载体蛋白(acyl carrier protein,ACP)和酮合成酶(ketosynthase,KS)结构域。AT将特定的酰基CoA构建块加载到ACP上,KS催化上游模块的中间体与ACP之间的碳碳键形成,最后,由位于聚酮合成酶碳末端的硫酯酶(thioesterase,TE)结构域完成水解或环化[5]。除此之外,Ⅰ型聚酮化合物还可能含有将β-酮基修饰为羟基、双键或单键的酮还原酶(ketoreductase,KR)、脱水酶(dehydratase,DH)或烯醇还原酶(enoylreductase,ER)的结构域[1]。II型聚酮化合物,聚酮合成酶只使用一套重复的蛋白质催化聚酮碳链的缩合反应,其主要由酮合成酶α(ketosynthase α,KSα)、酮合成酶β(ketosynthase β,KSβ)和酰基载体蛋白3种蛋白质组成,产生不同的芳香族代谢物[6]。Ⅲ型聚酮化合物,聚酮合成酶是一个同源二聚体,聚酮化合物生物合成的所有催化反应均在一个活性位点完成[4]。Ⅲ型聚酮合成酶通常催化不同酰基辅酶A起始单元与丙二酰辅酶A(malonyl-CoA)的迭代延伸形成查尔酮和二苯乙烯产物[7]。
聚酮化合物由于具有抗肿瘤、免疫抑制剂、抗菌以及驱虫杀虫等活性,在医药和农业领域得到了广泛应用。例如,红霉素(erythromycin)具有抗细菌活性[8],雷帕霉素(rapamycin)具有免疫抑制活性[9],多杀菌素(spinosad)和阿维菌素(avermectin)具有驱虫和杀虫活性[10-11]。野生型链霉菌合成聚酮化合物的产量远不能达到工业化生产规模的需求,增强链霉菌合成聚酮化合物的能力一直是研究的热点。目前,代谢工程是提高次级代谢产物产量行之有效的方法之一,比如,在聚酮化合物生物合成过程中,采用重组限速酶的过表达、合成基因簇的异源表达和强化前体供应技术等。
磷酸激酶(polyphosphate kinase,PPK)的基因ppk被阻断,使链霉菌产抗生素的能力增强。研究表明,ppk基因被阻断的突变株中发生的是贮藏脂质的降解,而不是己糖分解代谢[12]。另外,通过对两种具有相同的脂质和次级代谢产物生物合成途径的模式链霉菌进行分析,结果表明,天蓝色链霉菌(Streptomyces coelicolor)A3(2)是放线菌紫红素(actinorhodin,Act)的高效生产者,其三酰甘油(triacylglycerols,TAGs)含量约为114 nmol/mg,比变铅链霉菌(Streptomyces lividan)少50%左右。TAGs的降解产生脂肪酸和甘油,脂肪酸部分通过β-氧化降解,产生乙酰辅酶A(acetyl-CoA),增加Act生物合成所需前体的供给,促进Act的生物合成。因此,TAGs的积累导致Act生物合成所需前体的减少,从而减少了Act的产量[13]。链霉菌中脂质含量与抗生素活性之间的负相关是一般规则,而个别例外可能是由一些与乙酰辅酶A产生或消耗相关的各种途径中的一些遗传差异造成的[14]。链霉菌不仅能够产生结构独特的聚酮化合物,还能够在膜脂中产生高比例的支链脂肪酸(branched chain fatty acids,BCFA),通过工程策略将酰基辅酶A前体通量从PKs重新定向到BCFA,能够获得高产的BCFA生产菌株[15]。同理,通过强化脂肪酸β-氧化途径,也能将更多的酰基辅酶A前体引流向聚酮化合物的生物合成[16]。这些研究结果表明,贮藏脂质代谢和聚酮化合物生物合成之间具有一定的联系,平衡好脂质代谢与聚酮化合物生物合成通量有利于促进目标产物的合成。本文根据脂肪酸β-氧化途径工程的研究进展,综述脂肪酸合成乙酰辅酶A代谢途径与聚酮化合物生物合成之间的联系,强化脂肪酸β-氧化途径能改善聚酮化合物生物合成,为利用β-氧化途径提高链霉菌产聚酮化合物能力的研究提供新的思路。
1 脂肪酸代谢与聚酮化合物合成
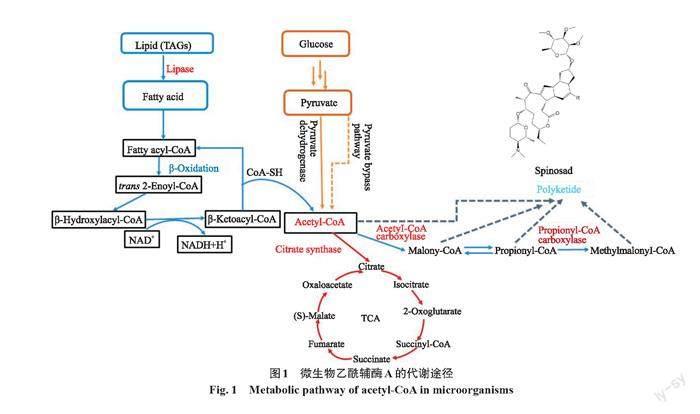
聚酮化合物的生物合成过程与脂肪酸的生物合成过程类似,即通过酰基辅酶A活化的底物之间的重复脱羧缩合而合成[3]。聚酮化合物则是一大类结构多样的次级代謝产物,其生物合成较复杂,由聚酮合成酶以“流水线”方式组装生产,每个延伸步骤均需要多个酶结构域催化,由此增加两个碳单元[17-18]。在聚酮化合物的生物合成过程中,起始单元主要是单酰辅酶A,如乙酰辅酶A、丙酰辅酶A(propionyl-CoA)。延伸单元主要是双酰辅酶A,如丙二酰辅酶A、甲基丙二酰辅酶A(methylmalonyl-CoA)。如图1所示,乙酰辅酶A主要来源于脂肪酸的β-氧化、糖酵解产生的丙酮酸氧化脱羧、支链氨基酸的分解代谢。乙酰辅酶A的流向除了进入三羧酸循环(tricarboxylic acid cycle,TCA)为微生物的生长发育提供能量和营养物质外,还能在乙酰辅酶A羧化酶(acetyl-CoA carboxylase,ACC)催化下生成丙二酰辅酶A,作为前体物质进入脂肪酸的生物合成过程[19-20]。乙酰辅酶A作为脂肪酸和聚酮化合物生物合成过程中的共同前体,两者存在一定的竞争关系,它不仅是中心碳代谢的一种中间产物,同时,在决定细胞分解代谢和合成代谢之间的平衡上至关重要[21]。
乙酰辅酶A作为大多数聚酮化合物生物合成的前体,可以通过β-氧化途径使脂肪酸降解来增加细胞内浓度。脂肪酸首先是在脂酰辅酶A合成酶(fatty acyl-CoA synthetase,ACS)的催化下,生成脂肪酰辅酶A。此后,脂肪酰辅酶A进入β-氧化途径,在一系列酶的作用下,在α碳原子和β碳原子之间断裂,β碳原子被氧化成羧基,生成含有2个碳原子的乙酰辅酶A和较原来少2个碳原子的脂肪酸[22]。因此,脂肪酸的多周期循环的β-氧化途径产生的乙酰辅酶A可能是聚酮化合物生物合成的重要组成部分。催化脂肪酸β-氧化途径第一步的极长链酰基辅酶A脱氢酶(very long-chain acyl-CoA dehydrogenase,vLCAD)的氧化还原敏感调节蛋白PirA的失活,使β-氧化途径遭到破坏,导致聚酮化合物生物合成前体单体供应不平衡,从而抑制聚酮化合物的合成[23]。因此,控制好脂肪酸代谢通量与聚酮化合物生物合成通量之间的平衡,有利于提高目标聚酮化合物的产量。在外源脂肪酸的存在下,细菌会对自身脂肪酸生物合成途径做出下调反应[24],减少脂肪酸生物合成与聚酮化合物生物合成竞争相应的前体物质,有利于聚酮化合物产量提高。
培养基中加入植物油的优化策略能够提高聚酮化合物的产量,但植物油的添加会影响脂肪酸的代谢和前体供应[25]。在阿维链霉菌(Streptomyces avermitilis)的发酵培养基中添加豆油,强化脂肪酸的代谢途径,不仅可以减缓细胞内脂肪酸生物合成与阿维菌素生物合成竞争共同的前体,而且可使更多的脂肪酸进入β-氧化途径生成乙酰辅酶A,提供更多阿维菌素合成前体[26]。在刺糖多孢菌(Saccharopolyspora spinosa)多杀菌素生物合成过程中,当外源脂肪酸存在时,脂肪酸生物合成基因转录水平下降,而多杀菌素生物合成和β-氧化相关基因转录水平上调,且利用长链酰基辅酶A合成细胞膜脂,短链酰基辅酶A如乙酰辅酶A和丙二酰辅酶A被用作多杀菌素合成前体,这解释了为什么外源脂肪酸添加能提高聚酮化合物的产量[27]。另外,增强以初级代谢产物为代表的底物供应也能提高聚酮化合物的产量,初级代谢提供的前体通常是通过碳底物(如脂肪酸、单糖和蛋白质)的分解代谢形成的[28]。
2 强化脂肪酸β-氧化途径促进聚酮化合物生物合成
如何提高链霉菌产聚酮化合物的能力,一直是菌株改良、代谢工程和发酵工程的重点研究内容。以往的研究大多集中在调控因子水平上,但这种操作很大程度上依赖基因的基因工程或者仅限于在生物合成基因簇上操作,而面对链霉菌次生代谢复杂的调控网络和高拷贝的基因簇,这给操作带来一定的挑战以及仅获得低产量的聚酮化合物的问题,效果并不理想[29-30]。链霉菌在发酵过程中经历了从初级代谢到次级代谢的转变,所有次级代谢都是以初级代谢的产物为底物进行的,了解代谢是如何转变的有利于最大限度生产聚酮化合物等目标次级代谢产物[31]。在初级代谢过程中,细胞消耗外部营养呈指数生长,当外部营养物被消耗完时,细胞停止生长进入稳定期并开始产生次级代谢产物,如聚酮化合物[32]。链霉菌不仅能够产生具有生物活性的聚酮化合物,还能在初级代谢过程中积累大量的TAGs。TAGs作为一种中性脂质化合物,最高可由65%(占细胞干重)的脂肪酸组成,这代表了链霉菌所描述的最高脂质含量[33-34]。脂肪酸除了是TAGs的主要组分外,还能合成细胞膜脂(磷脂和糖脂),用以维持细胞的正常生长。链霉菌体内TAGs的有效水解能够产生大量的游离脂肪酸,游离脂肪酸经β-氧化途径进行降解。因此,链霉菌体内大量积累TAGs,会导致脂肪酸降解的减少[35]。链霉菌初级代谢和次级代谢过程具有密切的联系,初级代谢向次级代谢转变的触发因子以及初级代谢产物如何过渡到稳定期用作次级代谢产物合成底物的机制尚不清楚。近年来,在初级代谢到次级代谢的代谢转变方面的研究获得突破性进展。Wang等[36]首次在代谢水平上发现链霉菌胞内TAGs在衔接初级代谢和聚酮化合物合成过程中起着关键作用。在生长阶段,链霉菌利用外源营养生长的同時积累内源TAGs,当菌株停止生长时,TAGs开始降解,为聚酮化合物生物合成提供必要的前体和还原力。研究发现,增强嗜油白色链霉菌ZD11(Streptomyces albus ZD11)TAGs和脂肪酸的降解,有利于为菌体生长和盐霉素(salinomycin)的生物合成提供丰富的碳前体[37]。通过转录组学分析发现,脂肪酸生物合成基因在TAGs合成时上调,在Act合成和TAGs降解时下调,而β-氧化相关基因则相反[36]。据此推测β-氧化相关基因是TAGs降解的关键控制点。
启动子工程是提高天然产物产量或发现新的天然产物的有效策略。组成型启动子持续调节基因的表达,不能在时间上协调目标产物的生物合成和宿主其他生理代谢之间的平衡,给宿主带来了代谢负担。因此,利用组成型启动子工程改善宿主的生产能力难以达到理想的效果,而诱导型启动子能在时间和强度两个方面微调基因的表达水平,更适合用于代谢工程提高代谢物的产量[38-39]。为了合理地控制TAGs动态降解,对脂肪酸降解途径进行优化,将编码ACS的基因sco6196置于cumate诱导启动子控制下,选择性控制TAGs降解的时间和强度,从而使更多碳源流向Act的生物合成[36]。该方法使Act、杰多霉素B(jadomycin B)、土霉素(oxytetracycline)和阿维菌素B1a 4种不同模块的聚酮化合物的产量均显著提高。其中,阿维菌素B1a产量从6.2 g/L提高到9.3 g/L,产量增加50%,表明该方法具有广泛的应用性[36]。聚酮化合物的生物合成基因簇可以通过携带β-氧化基因来调节β-氧化途径,使链霉菌能够有效合成目标聚酮化合物并经济地利用环境中的营养[40]。该研究再次证明了初级代谢和次级代谢的耦合机制,为促进链霉菌聚酮化合物的合成制定策略提供了理论依据。将天蓝色链霉菌中参与β-氧化途径的fadD和fadE基因重组到刺糖多孢菌中,重组菌株产多杀菌素能力显著增强[25]。
无独有偶,美国Almer等[41]报道了真菌系统——解脂耶氏酵母菌(Yarrowia lipolytica)利用β-氧化相关策略改善真菌系统中多种不同的聚酮化合物生产。通过柠檬酸途径、丙酮酸旁路途径和丙酮酸脱氢酶复合物途径探索发现,丙酮酸旁路途径和丙酮酸脱氢酶复合物途径均可以提高聚酮化合物三油酸内酯(triacetic acid lactone,TAL)的产量,且丙酮酸旁路途径使TAL达到了前所未有的效价,但其只能在复杂的培养基中实现,而不能在规定的培养基中实现,这限制了其在工业上的实际应用。另外,通过过表达PEX10证明了β-氧化修饰同样可以提高TAL的产量[42]。PEX10编码过氧化物酶体生物发生因子,过氧化物酶体含多种氧化酶和触酶,其主要功能是催化脂肪酸的β-氧化,将长链脂肪酸分解为短链脂肪酸,从而增强脂肪酸β-氧化途径。这些结果表明,脂肪酸β-氧化途径是提高聚酮化合物产量的有效改良靶点。
3 强化脂肪酸β-氧化途径提高聚酮化合物产量的优势
为了提高聚酮化合物的产量,人们采用了过表达其合成途径中的限速酶、在发酵培养基中添加前体物质以及生物合成基因或合成基因簇异源表达等策略。聚酮化合物生物合成基因簇异源表达虽然已有很多成功的案例,但异源表达偶尔获得较低的产量甚至不能产生目标化合物。在链霉菌中,聚酮化合物的异源生产可能需要添加前体和辅助因子以调节基因簇的转录和催化蛋白的酶活性[43]。此外,异源生产通常需要构建工业底盘菌株,以消除异源宿主次级代谢产物同目标产物的竞争,这是一项繁杂的研究工作[10]。过表达合成途径中的限速酶,虽然在一定程度上提高了聚酮化合物产量,但通常仅操纵单一限速酶所达到的效果有限,为了获得更高产量的聚酮化合物,无疑要求对聚酮化合物合成途径中的多个限速酶同时进行基因修饰[44]。例如,在过表达了PEX10的基础上进一步过表达耶氏酵母菌内源性乙酰辅酶A羧化酶ACC1,使TAL、柚皮苷(naringenin)、白藜芦醇(resveratrol)以及双去甲氧基姜黄素(bisdemethoxycurcumin)的产量进一步增加,其中柚皮苷的发酵产量高达898 mg/L[41]。
脂肪酸降解代谢及其相关酶在提供聚酮化合物生物合成的前体物质方面起着重要的作用。一方面,通过脂肪酸β-氧化途径的强化,加强体内前体供应来增加聚酮化合物的生物合成。另一方面,脂肪酸通过多轮β-氧化,产生高水平的还原力和腺苷三磷酸(adenosine triphosphate,ATP)[36]。有研究表明,高水平的还原力和ATP控制着TCA,对TCA循环过程中α-酮戊二酸脱氢酶(α-ketoglutarate dehydrogenase,KGDH)、异柠檬酸脱氢酶(isocitrate dehydrogenase,IDH)和柠檬酸合成酶(citrate synthase,CS)的活性具有一定的抑制作用[45-46]。因此,增强脂肪酸β-氧化能够削弱三羧酸循环,减少其与聚酮化合物生物合成竞争共同前体物质,使更多碳源流向聚酮化合物生物合成途径,促进聚酮化合物的生物合成。通过合理控制脂肪酸β-氧化相关基因表达,使多杀菌素[25]、阿维菌素B1a、杰多霉素[36]以及三油酸内酯[47]、柚皮苷和白藜芦醇[41]等多种聚酮化合物类抗生素的产量得到了提高,这表明该方法具有广谱适用性。
4 总结与展望
聚酮化合物是一大类结构复杂多样的次生代谢产物,其中许多聚酮化合物具有重要的生物活性。虽然聚酮化合物结构复杂多样,但都是由简单的小分子酰基辅酶A分子作为起始单元和延伸单元进行连续的脱羧缩合完成生物合成的,如乙酰辅酶A和丙二酰辅酶A。链霉菌发酵是产生次级代谢产物的有效途径,在发酵过程中发生了从初级代谢到次级代谢的剧烈转变,所有次级代谢产物都是建立在初级代谢的底物基础上,因此,利用代谢工程策略重新分配碳流量走向,可引导更多的底物从初级代谢到次级代谢,有利于高效合成次生代谢产物。乙酰辅酶A既是聚酮化合物生物合成的主要前体物质,也是脂类物质生物合成的底物,两者在生物合成过程中共同竞争乙酰辅酶A,而脂类降解产生的脂肪酸经β-氧化途径,又可以产生大量的乙酰辅酶A。因此,通过对脂肪酸β-氧化途径进行合理工程设计优化和基因编辑,合理调控细胞内代谢物相关途径,将更多碳源引向聚酮化合物生物合成,能有效提高聚酮化合物的产量。
脂肪酸主要储存在中性脂质化合物TAGs中,TAGs具有极强的疏水性,是极好的储备材料,可以大量积累而不影响细胞的渗透压。TAGs的生物合成途径已被广泛研究,但其分解涉及到的遗传因子研究较少[48]。脂肪酸β-氧化途径在所有生物体中高度保守,链霉菌中存在许多潜在的β-氧化相关基因的同源物,其在β-氧化过程中的作用不明确,这是企图通过脂肪酸β-氧化工程提高聚酮化合物产量所面临的一个挑战。转录组学、蛋白质组学、代谢组学等多组学技术的运用,有望解决这一难题。之前通过β-氧化途径提高聚酮化合物产量的策略主要集中在直接参与β-氧化途径的相关酶水平上,例如,催化脂肪酸活化生成脂酰辅酶A的脂酰辅酶A合成酶、催化脂酰基辅酶A脱氢的脂酰基辅酶A脱氢酶以及烯酰辅酶A水合酶。脂肪酶(lipase)能促进TAGs高效、稳定水解。然而,通过对脂肪酶进行遗传操作来增强TAGs水解,使更多的脂肪酸进入β-氧化途径,从而为聚酮化合物生物合成提供更多的前体物质的研究尚未见报道。因此,脂肪酶将会是今后促进聚酮化合物生物合成的代谢工程的重要研究对象。
在放线菌进化中,大多数代谢途径已经被平衡了,以至于没有一种酶能完整控制通过该途径的通量。聚酮化合物生物合成通常受级联调控,比如受多效调控因子或全局调控因子的影响[49-50]。因此,单基因遗传修饰虽有可能提高聚酮化合物的产量,但细胞代谢的复杂性会造成细胞内酶水平的失衡,导致中间产物的过量积累,有可能对细胞造成毒害作用,从而限制菌体生长发育和代谢产物的合成[51]。多途径组合工程策略将是改善聚酮化合物生产的主要途径。利用定量蛋白质组、转录组和代谢组学等多组学技术实时监测基因转录、蛋白表达水平和代谢物浓度是有必要的。有了上述研究基础,可以利用合适的启动子控制基因的表达水平,在提高聚酮化合物的产量的同时减少代谢紊乱。在今后的研究中,我们将利用脂肪酸代謝与聚酮化合物合成的关系,通过合成生物学手段,结合调控β-氧化途径的全局调控因子的修饰表达,来协调二者的代谢通量,同时,可以结合CRISPRi技术通过抑制TCA、脂肪酸合成关键基因的表达,减少乙酰辅酶A流向TCA、脂肪酸合成,使更多的前体物质流向聚酮化合物的生物合成,促进目标聚酮化合物生物合成,以满足生产聚酮化合物工业化的需求。利用启动子工程结合代谢组学、转录组学或蛋白质组学等组学研究方法,筛选在初级代谢过程中活性较弱而在次级代谢过程中活性较强的内源性生理时序启动子,合理调控脂肪和聚酮化合物代谢通量,强化代谢流方向,有利于目标产物聚酮化合物的生物合成。
参考文献(References):
[1] TAN G Y, LIU T G. Rational synthetic pathway refactoring of natural products biosynthesis in actinobacteria[J]. Metabolic Engineering, 2017, 39: 228-236.
[2] DEVINE R, MCDONALD H P, QIN Z, et al. Re-wiring the regulation of the formicamycin biosynthetic gene cluster to enable the development of promising antibacterial compounds[J]. Cell Chemical Biology, 2021, 28(4): 515-523.
[3] WLODEK A, KENDREW S G, COATES N J, et al. Diversity oriented biosynthesis via accelerated evolution of modular gene clusters[J]. Nature Communication, 2017, 8(1): 1206.
[4] RISDIAN C, MOZEF T, WINK J, et al. Biosynthesis of polyketides in Streptomyces[J]. Microorganisms, 2019, 7(5): 124.
[5] DUTTA S, WHICHER J R, HANSEN D A, et al. Structure of a modular polyketide synthase[J]. Nature, 2014, 510(7506): 512-517.
[6] QIAN Z, BRUHN T, DAGOSTINO P M, et al. Discovery of the streptoketides by direct cloning and rapid heterologous expression of a cryptic PKS II gene cluster from Streptomyces sp.Tu 6314 [J]. Journal of Organic Chemistry, 2020, 85(2): 664-673.
[7] SAKAMOTO S, MORITA Y, YUSAKUL G, et al. Molecular cloning and characterization of type III polyketide synthase from Plumbago zeylanica[J]. Journal of Asian Natural Products Research, 2021, 23(5): 478-490.
[8] FALLAH F, ZARGAR M, YOUSEFI M, et al. Synthesis of the erythromycin-conjugated nanodendrimer and its antibacterial activity[J]. European Journal of Pharmaceutical Sciences, 2018, 123: 321-326.
[9] ZHANG X, CHEN W, GAO Q, et al. Rapamycin directly activates lysosomal mucolipin TRP channels independent of mTOR[J]. PLoS Biology, 2019, 17(5): e3000252.
[10] SONG C, LUAN J, CUI Q, et al. Enhanced heterologous spinosad production from a 79 kb synthetic multi-operon assembly[J]. ACS Synthetic Biology, 2019, 8(1): 137-147.
[11] HUANG J, CHEN A L, ZHANG H, et al. Gene replacement for the generation of designed novel avermectin derivatives with enhanced acaricidal and nematicidal activities[J]. Applied and Environmental Microbiology, 2015, 81(16): 5326-5334.
[12] LE MARECHAL P, DECOTTIGNIES P, MARCHAND C H, et al. Comparative proteomic analysis of Streptomyces lividans wild-type and ppk mutant strains reveals the importance of storage lipids for antibiotic biosynthesis[J]. Applied and Environmental Microbiology, 2013, 79(19): 5907-5917.
[13] ESNAULT C, DULERMO T, SMIRNOV A, et al. Strong antibiotic production is correlated with highly active oxidative metabolism in Streptomyces coelicolor M145[J]. Scientific Reports, 2017, 7(1): 200.
[14] DAVID M, LEJEUNE C, ABREU S, et al. Negative correlation between lipid content and antibiotic activity in Streptomyces: general rule and exceptions[J]. Antibiotics (Basel), 2020, 9(6): 280.
[15] YI J S, YOO H W, KIM E J, et al. Engineering Streptomyces coelicolor for production of monomethyl branched chain fatty acids[J]. Journal of Biotechnology, 2020, 307: 69-76.
[16] HAO Y, YOU Y, CHEN Z, et al. Avermectin B1a production in Streptomyces avermitilis is enhanced by engineering aveC and precursor supply genes[J]. Applied Microbiology and Biotechnology, 2022, 106(56): 2191-2205.
[17] FABIEN C, FRANOISE G, GUILLAUME T, et al. Carbon-flux distribution within Streptomyces coelicolor metabolism: a comparison between the actinorhodin-producing strain M145 and its non-producing derivative M1146[J]. PLoS One, 2013, 8(12): e84151.
[18] MASCHIO L, PARNELL A E, LEES N R, et al. Cloning, expression, and purification of intact polyketide synthase modules[J]. Methods in Enzymology, 2019, 617(63): 63-82.
[19] WU P, CHEN K, LI B, et al. Polyketide starter and extender units serve as regulatory ligands to coordinate the biosynthesis of antibiotics in actinomycetes[J]. Molecular Biosystems, 2021, 12(5): e0229821.
[20] YE J, DEBOSE-BOYD R A. Regulation of cholesterol and fatty acid synthesis[J]. Cold Spring Harbor Perspectives Biology, 2011, 3(7): a004754.
[21] PIETROCOLA F, GALLUZZI L, BRAVO-SAN PEDRO J M, et al. Acetyl coenzyme A: a central metabolite and second messenger[J]. Cell Metabolism, 2015, 21(6): 805-821.
[22] BANCHIO C, GRAMAJO H. A stationary-phase acyl-coenzyme A synthetase of Streptomyces coelicolor A3(2) is necessary for the normal onset of antibiotic production[J]. Applied and Environmental Microbiology, 2002, 68(9): 4240-4246.
[23] TALA A, DAMIANO F, GALLO G, et al. Pirin: a novel redox-sensitive modulator of primary and secondary metabolism in Streptomyces[J]. Metabolic Engineering, 2018, 48: 254-268.
[24] PARSONS J B, MATTHEW W F, CHITRA S, et al. Metabolic basis for the differential susceptibility of Gram-positive pathogens to fatty acid synthesis inhibitors[J]. Proceedings of the National Academy of Sciences, 2011, 108(37): 15378-15383.
[25] HUANG Y, ZHANG X, ZHAO C, et al. Improvement of spinosad production upon utilization of oils and manipulation of beta-oxidation in a high-producing Saccharopolyspora spinosa strain[J]. Journal Molecular Microbiology and Biotechnology, 2018, 28(2): 53-64.
[26] 曹鵬, 胡栋, 张君,等. 基于比较代谢组学的理性优化方法提高阿维菌素产量[J]. 微生物学报, 2017, 57(2): 281-292.
CAO Peng, HU Dong, ZHANG Jun, et al. Enhanced avermectin production by rational feeding strategies based on comparative metabolomics[J]. Acta Microbiologica Sinica, 2017, 57(2): 281-292.
[27] JHA A K, POKHREL A R, CHAUDHARY A K, et al. Metabolic engineering of rational screened Saccharopolyspora spinosa for the enhancement of spinosyns A and D production[J]. Molecules and Cells, 2014, 37(10): 727-733.
[28] TANAKA Y, IZAWA M, HIRAGA Y, et al. Metabolic perturbation to enhance polyketide and nonribosomal peptide antibiotic production using triclosan and ribosome-targeting drugs[J]. Applied Microbiology and Biotechnology, 2017, 101(11): 4417-4431.
[29] VANWEZEL G P, MCDOWALL K J. The regulation of the secondary metabolism of Streptomyces: new links and experimental advances[J]. Natural Product Reports, 2011, 28(7): 1311-1333.
[30] MENZELLA H G, REID R, CARNEY J R, et al. Combinatorial polyketide biosynthesis by de novo design and rearrangement of modular polyketide synthase genes[J]. Nature Biotechnology, 2005, 23(9): 1171-1176.
[31] GAMBOA-SUASNAVART R A, VALDEZ-CRUZ N A, GAYTAN-ORTEGA G, et al. The metabolic switch can be activated in a recombinant strain of Streptomyces lividans by a low oxygen transfer rate in shake flasks[J]. Microbial Cell Factories, 2018, 17(1): 189.
[32] LIU G, CHATER K F, CHANDRA G, et al. Molecular regulation of antibiotic biosynthesis in Streptomyces[J]. Microbiology and Molecular Biology Reviews, 2013, 77(1): 112-43.
[33] RODRIGUEZ E, NAVONE L, CASATI P, et al. Impact of malic enzymes on antibiotic and triacylglycerol production in Streptomyces coelicolor[J]. Applied and Environmental Microbiology, 2012, 78(13): 4571-4579.
[34] ROTTIG A, STRITTMATTER C S, SCHAUER J, et al. Role of wax ester synthase/acyl coenzyme A: diacylglycerol acyltransferase in oleaginous Streptomyces sp. Strain G25[J]. Applied and Environmental Microbiology, 2016, 82(19): 5969-5981.
[35] ZHANG J, LIANG Q, XU Z, et al. The inhibition of antibiotic production in Streptomyces coelicolor over-expressing the TetR regulator SCO3201 is correlated with changes in the lipidome of the strain[J]. Frontiers in Microbiology, 2020, 11: 1399.
[36] WANG W, LI S, LI Z, et al. Harnessing the intracellular triacylglycerols for titer improvement of polyketides in Streptomyces[J]. Nature Biotechnology, 2020, 38(1): 76-83.
[37] LI H, WEI J, DONG J, et al. Enhanced triacylglycerol metabolism contributes to efficient oil utilization and high-level production of salinomycin in Streptomyces albus ZD11[J]. Applied and Environmental Microbiology, 2020, 86(16): e00763-20.
[38] LI S, WANG J, XIANG W, et al. An autoregulated fine-tuning strategy for titer improvement of secondary metabolites using native promoters in Streptomyces[J]. ACS Synthetic Biology, 2018, 7(2): 522-530.
[39] ZHOU Q, NING S, LUO Y, et al. Coordinated regulation for nature products discovery and overproduction in Streptomyces[J]. Synthetic and Systems Biotechnology, 2020, 5(2): 49-58.
[40] WEI J, CHEN B, DONG J, et al. Salinomycin biosynthesis reversely regulates the beta-oxidation pathway in Streptomyces albus by carrying a 3-hydroxyacyl-CoA dehydrogenase gene in its biosynthetic gene cluster[J]. Microbial Biotechnology, 2022, 15(12): 2890-2904.
[41] ALMER C M, MILLER K K, NGUYEN A, et al. Engineering 4-coumaroyl-CoA derived polyketide production in Yarrowia lipolytica through a beta-oxidation mediated strategy[J]. Metabolic Engineering, 2020, 57: 174-181.
[42] MARKHAM K A, PALMER C M, CHWATKO M, et al. Rewiring Yarrowia lipolytica toward triacetic acid lactone for materials generation[J]. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(9): 2096-2101.
[43] HUANG J, YU Z, LI M H, et al. High level of spinosad production in the heterologous host Saccharopolyspora erythraea[J]. Applied and Environmental Microbiology, 2016, 82(18): 5603-5611.
[44] JIANG H, WANG Y Y, RAN X X, et al. Improvement of natamycin production by engineering of phosphopantetheinyl transferases in Streptomyces chattanoogensis L10[J]. Applied and Environmental Microbiology, 2013, 79(11): 3346-3354.
[45] KUMAR P, DUBEY K K. Modulation of fatty acid metabolism and tricarboxylic acid cycle to enhance the lipstatin production through medium engineering in Streptomyces toxytricini[J]. Bioresource Technology, 2016, 213: 64-68.
[46] VEMURI G N, EITEMAN M A, MCEWEN J E, et al. Increasing NADH oxidation reduces overflow metabolism in Saccharomyces cerevisiae[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(7): 2402-2407.
[47] LIU H, MARSAFARI M, WANG F, et al. Engineering acetyl-CoA metabolic shortcut for eco-friendly production of polyketides triacetic acid lactone in Yarrowia lipolytica[J]. Metabolic Engineering, 2019, 56: 60-68.
[48] MENENDEZ-BRAVO S, PAGANINI J, AVIGNONE-ROSSA C, et al. Identification of FadAB complexes involved in fatty acid beta-oxidation in Streptomyces coelicolor and construction of a triacylglycerol overproducing strain[J]. Frontiers in Microbiology, 2017, 8: 1428.
[49] HE H, YUAN S, HU J, et al. Effect of the TetR family transcriptional regulator Sp1418 on the global metabolic network of Saccharopolyspora pogona[J]. Microbial Cell Factories, 2020, 19(1): 27.
[50] YANG R, LIU X, WEN Y, et al. The PhoP transcription factor negatively regulates avermectin biosynthesis in Streptomyces avermitilis[J]. Applied Microbiology and Biotechnology, 2015, 99(24): 10547-10557.
[51] LIU C L, HAO R B, BAI Z H, et al. Engineering and manipulation of a mevalonate pathway in Escherichia coli for isoprene production[J]. Applied Microbiology and Biotechnology, 2019, 103(1): 239-250.
收稿日期:2022-09-30;修回日期:2022-10-24。
基金項目:国家自然科学基金项目(31770106)。
作者简介:曹丽,博士研究生。
* 通信作者:夏立秋,教授,主要从事微生物次生代谢产物合成生物学的研究。E-mail: xialq@hunnu.edu.cn。
