计算机模拟筛选食用藻蛋白源PAR2 抑制肽
2024-01-09高立芳曾新安金可沆范土贵彭名军曾巧辉
高立芳,曾新安,金可沆,范土贵,彭名军,曾巧辉*
(1.佛山科学技术学院食品科学与工程学院,广东省食品智能制造重点实验室,广东佛山 528225)(2.广州市食品检验所,广东广州 511400)
蛋白质是人体所必须的重要的营养物质之一。通常,蛋白质进入人体后,经过胃肠道消化酶的降解才能被机体吸收,其在人体不是仅以氨基酸形式被吸收,更多是以肽的形式被吸收[1]。小分子生物肽的分子量小、结构简单,且可快速透过小肠黏膜被吸收,相较于游离氨基酸,吸收耗能低、转运速度快、载体不易饱和[2]。因此,食品蛋白质源的小分子活性肽的吸收、转化和利用具有高效性[3]。海洋生物中存在丰富的生物活性天然产物可以用于开发治疗多种人类疾病[4]。近年来,人们越发关注从鱼类、藻类、甲壳类动物和海绵中分离和鉴定具有广泛生物活性的蛋白质和多肽。
藻类蛋白是人类重要的海洋蛋白质来源,且其经酶解后能释放出具有生物活性的小分子肽。研究表明,大豆、牛奶、带鱼等食物的酶解产物具有降血脂、抗炎症、降血糖、抗抑郁等多种生物活性[5-7]。蛋白酶激活受体(PARs)是G 蛋白偶联受体家族的一员,与人体内许多的生理反应相关[8],且参与诱发多种疾病的发生,如各种炎症、癌症、冠心病和糖尿病等[9-11]。通常情况下,PARs 的N 末端序列被丝氨酸蛋白酶水解切割而发生不可逆的激活,暴露含有系锁配体的新的N 末端,新的N 末端作为系锁配体可与PARs 进行分子内结合,从而实现自发激活,进而引发细胞级偶联反应以响应炎症信号和其它刺激反应[12]。PARs 包含有四种亚型,分别为PAR1、PAR2、PAR3、PAR4,其中蛋白酶激活受体2(PAR2)比较特殊,且不能被凝血酶激活[13],而PAR2 主要激活剂包括胰蛋白酶、类胰蛋白酶和弹性蛋白酶,其中胰蛋白酶是最有效的PAR-2 激活剂[14]。
我国是藻类生产消费大国,常见的藻类品种有海带(Laminariajaponica)、紫菜(Porphyra)、裙带菜(UndariapinnatifidaSuringar)、江蓠(Gracilaria)、螺旋藻(Spirulina)以及微藻杜氏藻(Microalgae Dunaliella)等[15],其富含优质蛋白。目前,基于计算机的分子模拟技术高速发展,能一定程度上减少传统科研实验存在的耗时、费力、缺乏靶向性等问题。传统的生物活性肽主要是从蛋白质酶解产物的分离纯化和活性鉴定开展研究,其不仅存在上述局限性,还有可能导致微量但活性高的生物活性肽的损失。
本研究利用生物数据库和基于计算机模拟的多肽-蛋白分子对接技术,理论上从食用藻类蛋白质中筛选具有蛋白酶激活受体2(PAR2)抑制作用的小分子多肽;以对接分数和配体与受体相互作用力作为评估藻类蛋白质源多肽对PAR2 的抑制潜力的依据,为藻类蛋白的开发利用以及PAR2 抑制剂发掘研究提供参考。
1 材料与方法
1.1 试验材料
在NCBI 数据库(https://www.ncbi.nlm.nih.gov/)[16]中输入“Edible algae”,结合蛋白质晶体数据库(PDB),检索出所有种类食用藻类蛋白,并下载各种蛋白质的氨基酸序列。从PDB 数据库获取蛋白酶激活受体2(PAR2)的晶体结构(PDB:5NDD,5NDZ),利用PyMol 软件分别将受体和配体8TZ(C13H15FN2O,结构见图1a),8UN(C30H27BrN4O3,结构见图1b)分离,并保存为PDB 文件。8TZ 作为蛋白酶激活受体2的拮抗剂,结合在受体外表面附近一个完全闭塞的口袋中,其主要与PAR2 氨基酸序列上的Asp228、Tyr82和His135 形成氢键,从而稳定受体的无活性状态以防止激动剂的活化变构。抑制剂8UN 具有高度的亲脂性,主要通过疏水作用与受体结合在蛋白螺旋表面偏远的变构位点上,从而限制激动剂结合后受体激活所需的螺旋间构象重排。

图1 抑制剂8TZ、8UN 的分子结构和蛋白酶激活受体2 与两个抑制剂配体对接的2D 结构Fig.1 Molecular structure of the inhibitors 8TZ and 8UN and 2D structure of the protease-activated receptor 2 docked to two inhibitor ligands
1.2 BIOPEP-UWM 分析
根据Yao 等[5]采用的方法,将从NCBI 数据库获得食用藻中常见的11 种蛋白质序列导入BIOPEP-UWM数据库中,预测被胃蛋白酶、胰蛋白酶和胰凝乳蛋白酶这三种肠胃酶的作用情况,模拟人体胃肠道消化的过程。将模拟人体胃肠消化所得的理论肽进一步分析获得潜在生物活性肽,并且对其进行更深一层的分析。
人体对螺旋藻蛋白的消化能力可以通过理论水解度TDH 来进行表示。
式中:
TDH——蛋白质水解过程中被裂解的肽键数与给定蛋白质的总肽键数之比,%;
D——蛋白质中肽键总数;
d——氢的数目[17]。
1.3 食用藻蛋白质源多肽的活性分析
使用Peptide Ranker 服务器评估螺旋藻蛋白质酶解产物的生物学活性,并对小分子肽进行综合评分筛选。在相应数据库中输入寡肽的氨基缩写序列可得到介于0~1 的评分,评分越高说明该寡肽的生物活性越高。在本研究中,当预测的肽段分数超过0.50 时,则可以认为该肽段具有潜在生物活性。
1.4 食用藻类蛋白和计算机模拟水解后释放的肽段的理化性质筛选
目前,ToxinPred 程序已广泛运用于多肽研究的分子对接虚拟筛选中[18],其预测功能可以有效避免有毒小分子肽进入人体内从而对身体健康造成危害。水溶性是影响生物活性肽吸收、发挥活性作用的主要因素,本研究采用Innovagen(http://www.innovagen.com/)和ToxinPred 网站对高活性肽进行预测,挑选潜在生物活性高、无毒且水溶性好的寡肽片段进行下一步对接研究。
1.5 分子对接
HPEPDOCK 是一种开展蛋白-多肽对接模拟研究的在线工具[16]。在HPEPDOCK 数据库中可将受体与筛选出的多肽进行对接,选择分离出来的配体作为对接参照;将分子对接评分≤-100 的寡肽视为高亲和、分子对接评分≤-150 的寡肽为超高亲和。
同时,用F 值来评价蛋白质抑制蛋白酶激活受体2(PAR2)的能力。
式中:
F——出现频率;
N——蛋白链内高亲和寡肽的数量;
L——蛋白链的长度。
1.6 分子对接结果的可视化
利用PyMol 软件对受体和寡肽进行处理,构建对接复合物的三维空间结构以及分子间作用力。通过LigPlus(版本号:V.2.2)分析高亲和力寡肽与PAR2受体结合的疏水残基和氢键作用[17]。
1.7 数据分析
所有数据均采用Excel 软件进行分析。
2 结果与讨论
2.1 计算机模拟酶解食用藻蛋白的结果分析
根据蛋白质在食用藻中含量高、活性强、研究广泛的原则,从NCBI 数据库中筛选食用藻来源的代表性蛋白质,利用BIOPEP-UWM 数据库模拟胃肠道消化酶(胰凝乳蛋白酶、胰蛋白酶和胃蛋白酶)水解上述筛选的蛋白质。结果如表1 所示。

表1 食用藻类蛋白质情况Table 1 Introduction of edible algae protein
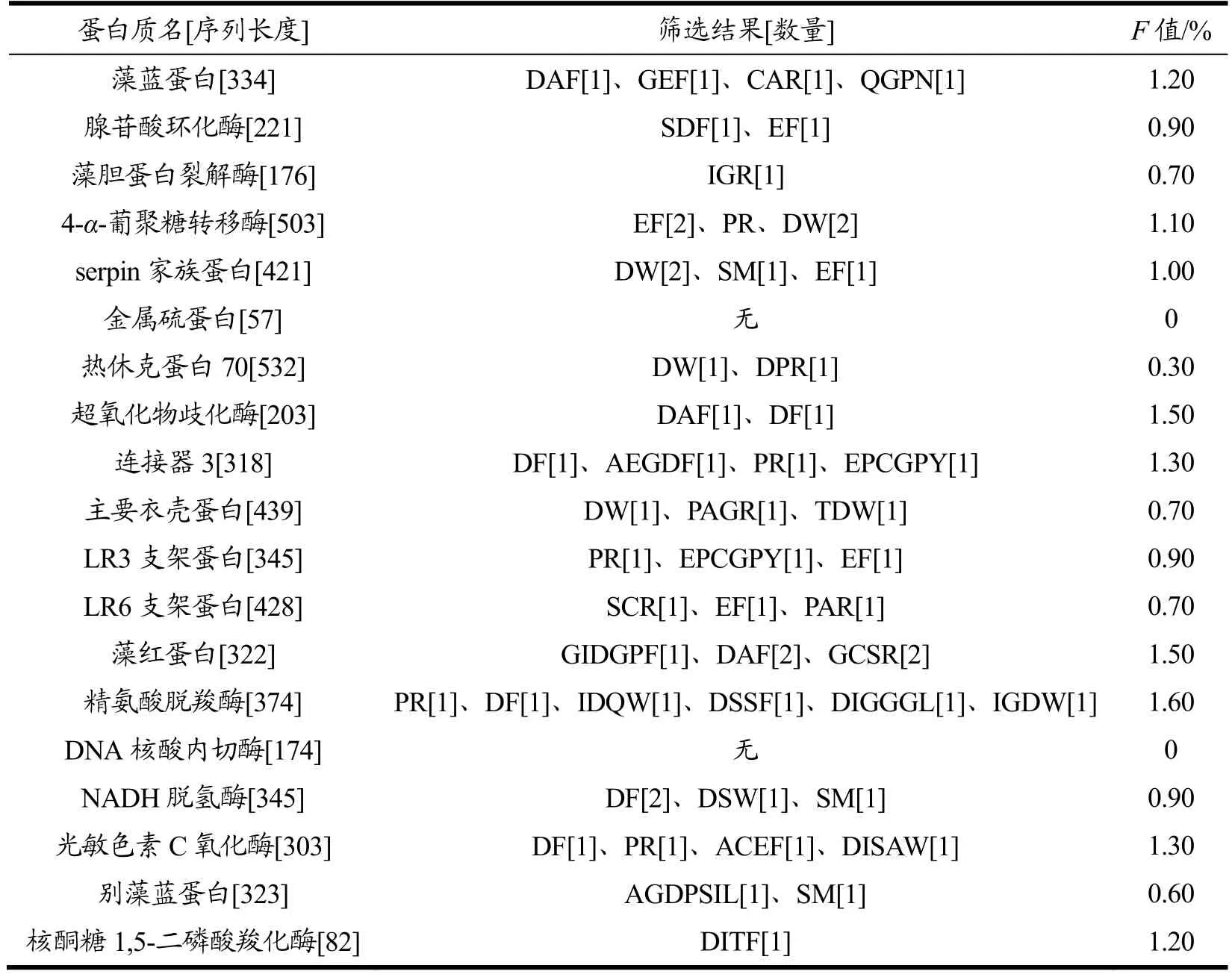
表2 藻类蛋白结合蛋白酶激活受体2 的寡肽序列和频率Table 2 Oligopeptide sequence and frequency of algal protein binding PAR 2
挑选螺旋藻、小球藻、紫菜、江蓠以及裙带菜等五种市场上常见的高营养价值的食用藻类,根据每种酶均有其固定的切割位点这一特性,采用NCBI 和BIOPEP-UWM 数据库处理、模拟酶水解可得到,水解度TDH 基本上大于30%,最高不超过50%;水解后的产物主要是氨基酸和寡肽,其中,NADH 脱氢酶和4-α-葡聚糖转移酶的水解度较高,分别达到了49.13%和43.63%,另外,水解度最低的则是裙带菜的核酮糖1,5-二磷酸羧化酶只有28.24%;水解后发现产生多肽数量最多的是4-α-葡聚糖转移酶可达到121,其次是热休克蛋白70 为118;serpin 家族蛋白产生高活性肽数量最多为46,4-α-葡聚糖转移酶和别藻蓝蛋白均为41。
生物信息学(Bioinformatics)是综合运用生物学、信息科学的各种知识和工具,对复杂的生物数据进行获取、处理、存储、分发、分析和解释,从而得到我们能够理解和接受的各种知识的一种技术。生物信息学工具在近年来发展飞速,在活性肽发掘研究中有着广泛应用[19]。步营等在研究抗SARS-CoV-2 海洋活性肽时运用了PeptideRanker 和ToxinPred 等生物信息学工具[18]。当前,各种数据库与多肽理化性质预测工具如在线工具Innovagen 软件以及预测活性肽的吸收(Adsorptin)、分布(Distribution)、代谢(Metabolism)、排泄(Excretion)和毒性(Toxicity)的AdmetSAR 软件等都在活性肽的模拟筛选发挥着重要作用[18-20]。对比传统的生物活性肽研究方法,利用生物信息学技术可以节省获得活性肽构效关系的成本,实现了天然活性肽的快速高通量筛选。同时,生物质谱中的液相色谱-飞行时间串联质谱以及四级杆线性离子阱质谱被逐渐应用于生物活性肽的结构分析,并辅以质谱数据解析软件ProteinPilot,缩短了活性肽结构表征的周期[21]。
2.2 食用藻源蛋白酶解活性预测结果
使用Peptide Ranker 工具来分析通过水解得到的寡肽,并筛选出具有潜在活性的寡肽,从而开展后续更深入的研究。筛选出的活性潜力较高、水溶性好的寡肽如表3 所示。由表3 可知,蛋白质serpin 家族蛋白(46)来源的且具有理论活性的寡肽数量是最多,其次是4-α-葡聚糖转移酶(41)。活性预测得分位于前三的寡肽分别是DF、DW 和GIDGPF 分子量分别为280.29、319.33 和604.74,肽段的生物活性并没有随分子量的增大而增大,其它肽段没有展现出肽段的生物活性随分子量的增大而降低,所以肽段的分子量与生物活性没有直接的相关性。可以看到预测得分较高的寡肽多集中在含有2~3 个氨基酸序列小分子肽之间。水溶性好的多肽都含有极性氨基酸。

表3 模拟水解获得的高活性寡肽及有效抑制蛋白酶激活受体2 活性的小分子寡肽Table 3 Highly active oligopeptides obtained by simulated hydrolysis and small molecule oligopeptides that effectively inhibit protease-activated receptor 2 activity
蛋白质和多肽是食用藻类中重要的生物营养物质,具有多种保健功效和生物活性,如促进外源营养成分的消化和吸收、抗光老化、抗肿瘤、抗氧化等[22]。大部分寡肽被肽酶再次水解为二肽和三肽,由PepT1转运至细胞内,而后被细胞内肽酶水解为游离氨基酸,进而通过细胞途径和载体转运系统完整吸收进入体循环。多数被吸收的小分子肽都被血管内皮组织肽酶和可溶性血浆肽酶分解为氨基酸[1]。
目前,影响口服肽生物利用度的两个重要因素是肽对胃肠道酶降解的抵抗及其通过肠细胞定向运输的效率。肠道中二肽和三肽的主要转运途径是PepT1 介导的转运系统,四肽和五肽则主要通过细胞旁紧密连接途径转运[23]。一些报道发现,某些具有2~8 个氨基酸残基的肽可抑抑制DPP-IV,其活性受序列长短、疏水性、氨基酸组成等各种因素影响[24]。因此,现有研究往往选用肽链长度在2~8 之间的寡肽开展蛋白酶激活受体2(PAR2)分子对接研究。
使用在线工具Innovagen和ToxinPred程序来分别对筛选出来的具有生物化学潜力的寡肽进行水溶性预测以及预测所选肽段的毒性、测定分子量等其他理化数据,具体结果如表3 所展示。结果发现所选用的寡肽均无毒无害,这很有可能和构成肽段的氨基酸的种类以及其结构相关。采用ToxinPred 对所选寡肽进行毒性预测,发现所挑选的肽段均无毒,可进一步用作食品药品方面的研究。另外,肽段的亲水性的负值越大代表其亲水能力越强;正值越大则代表其疏水能力越强[26]。
2.3 分子对接结果分析
近年来,分子对接(MolecularDocking)技术作为探究关蛋白-配体的结合状态、食品功能性成分作用位点、农兽药作用机理的一种新兴技术,因能克服科学实验存在的局限性,在食品领域的应用日益广泛。Auwal等使用Schrödinger软件对石头鱼蛋白的水解产物抑制肽与血管紧张素转化酶(Angiotensin Converting Enzyme,ACE)进行分子对接,从分子水平上解释了这些抑制肽的ACE 抑制作用。步营借助Discovery Studio 软件筛选抗SARS-CoV-2 海洋活性肽,并分析了分子间的相互作用[18]。该技术利用计算机模拟程序把配体小分子放在受体活性位点处,按照几何互补和能量互补的原则,通过打分函数筛选出配体与受体间的最佳结合模式[27]。
HPEPDOCK(http://huanglab.phys.hust.edu.cn/hpepdock/)是一款模拟蛋白-多肽对接的在线工具。Weng 等[28]测试了14 种常见对接软件,包括:三个蛋白-蛋白对接软件(ZDOCK、FRODOCK and HawkDock),三个蛋白-小分子对接软件(GOLD、Surflex-Dock and AutoDock Vina),以及八个蛋白-多肽对接软件(GalaxyPepDock、MDockPeP、HPEPDOCK、CABS-dock、pepATTRACT、DINC、AutoDock CrankPep (ADCP)、HADDOCK peptide docking)。发现在全局对接中,HPEPDOCK 的表现优于其他的对接软件,因此,本研究选择HPEPDOCK 软件进行分子对接。基于该软件,本研究最终筛选出四个分子对接评分≤-150 的寡肽(PAGR、PAR、IDQW、DISAW),且将这四个寡肽认为是的具有较高蛋白酶激活受体2抑制潜力超高亲和肽。
将经过胃肠道消化模拟水解酶切后且通过生物信息学工具筛选的寡肽与蛋白酶激活受体2(PAR2)进行分子对接。对接得分<-100 被认为是具有亲和能力的寡肽,用F值来评价食源性藻类蛋白质对蛋白酶激活受体2(PAR2)的潜在抑制能力。结果表2 所示,发现小球藻的精氨酸脱羧酶蛋白的F值最高为1.60%,其次是超氧化物歧化酶和藻红蛋白,均为1.50%。F值越高,说明这三种食用藻蛋白的蛋白酶激活受体2(PAR2)的潜在抑制能力优于本文研究的其他食用藻蛋白,酶解后更容易获得高亲和力的寡肽,这三种蛋白广泛存在于常见食用藻类中,相对含量较高,它们是筛选蛋白酶激活受体2 抑制肽的主要优势。
2.3.1 分别以抑制剂8TZ 和8UN 为参照的对接结果
在HPEPDOCK 软件中分别以抑制剂8TZ 和8UN为参照,经过胃肠道消化模拟和水解酶切后且通过生物信息学工具筛选的寡肽与蛋白酶激活受体2(PAR2)进行分子对接,分子对接结果如表5 所示。

表5 分子对接结果Table 5 Results of molecular docking
本研究通过LigPlus 软件分析研究螺旋藻蛋白源寡肽与PAR2 的结合能力,以及参与相互作用的疏水残基和氢键和以抑制剂8TZ 为参照的对接结果发现,分子对接分数最高的寡肽是来自精氨酸脱羧酶的PAGR(-165.80),且多肽主要与其PAR2 氨基酸序列上的His227、Leu151 形成氢键相互作用,以及与Ile327、Tyr82、HIS135、Leu78、Tyr134、Cys226、Ile152、Lys131、Thr334、Trp127、Phe155、Ser124、Phe155、Asn158、Asn336、Phe128、Asp228 形成疏水作用。另外,PAGR 与抑制剂8TZ 有8 个相同结合位点(Ile327、His227、Asp228、His135、Leu78、Tyr82、Phe155、Cys226),其中有5 个是相同的疏水残基位点(Ile327、His135、Leu78、Phe155、Cys226);PAR与抑制剂8TZ 形成8 个相同的疏水作用位点(Tyr82、His227、Leu78、Phe155、Phe327、Cys226、Lys131、Tyr134)和一个相同的氢键(Asp228),综合认为PAR成为PAR2 抑制肽的潜力较高。
以抑制剂8UN 为参照的对接结果发现,对接分数最高的寡肽是来自蛋白质光敏色素C 氧化酶的DISAW(-154.48),且多肽主要与其与PAR2 氨基酸序列上的Ala157、Cys161、Leu123、Phe154、Tyr210、Trp199、Leu203、Ile202、Leu196、Leu200 形成疏水相互作用,没有形成氢键作用。而IDQW 与抑制剂8UN 形成由8 个疏水氨基酸残基(Cys161、Asn158、Ala157、Leu123、Leu203、Trp199、Phe154、Ala120)参与的疏水相互作用。因此,综合认为寡肽DISAW相较IDQW 对PAR2 的抑制潜力无明显差异。
2.4 分子对接结果的可视化
根据以上的分子对接结果,将分子对接评分≤-150 的寡肽视为超高亲和,使用PyMol 软件对受体和寡肽进行处理,构建对接复合物的三维空间结构以及分子间作用力。LigPlus 软件分析超高亲和力寡肽与PAR2 受体结合的疏水残基和氢键作用,分析结果如表6、图2~5 所示。
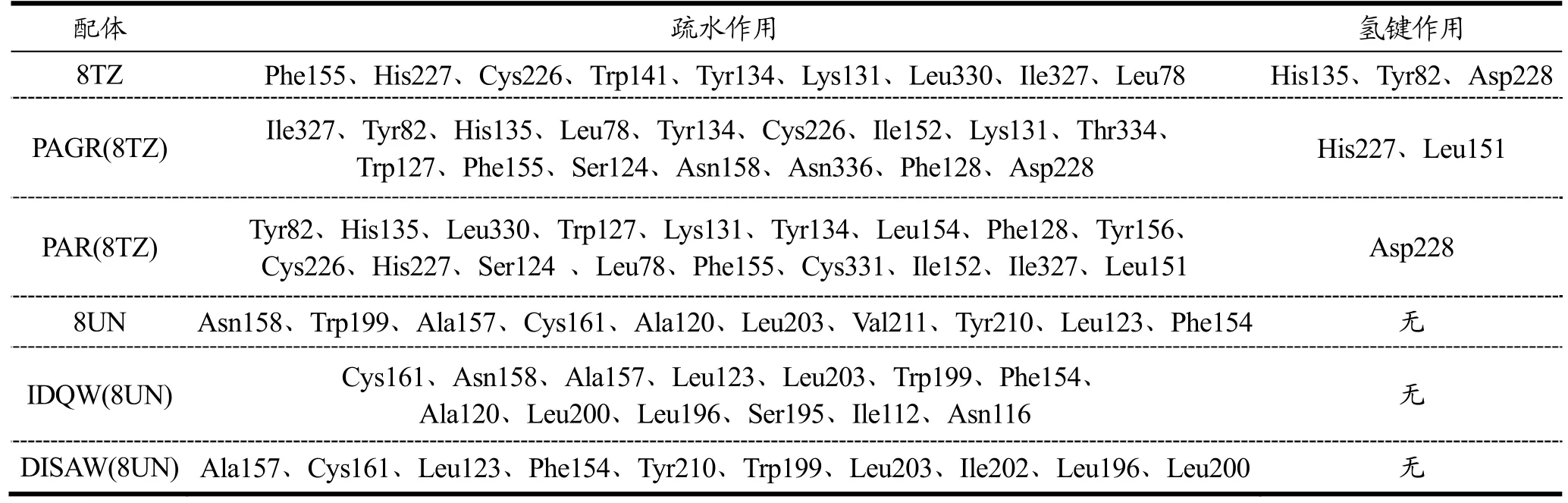
表6 超高亲和寡肽与蛋白酶激活受体的相互作用力Table 6 Interactions between ultrahigh affinity oligopeptides and protease activated receptors

图2 蛋白酶激活受体2(PAR2)与超高亲和肽PAGR 结合的图像Fig.2 Image of PAR2 binding to the ultra-high affinity peptide with sequence of PAGR
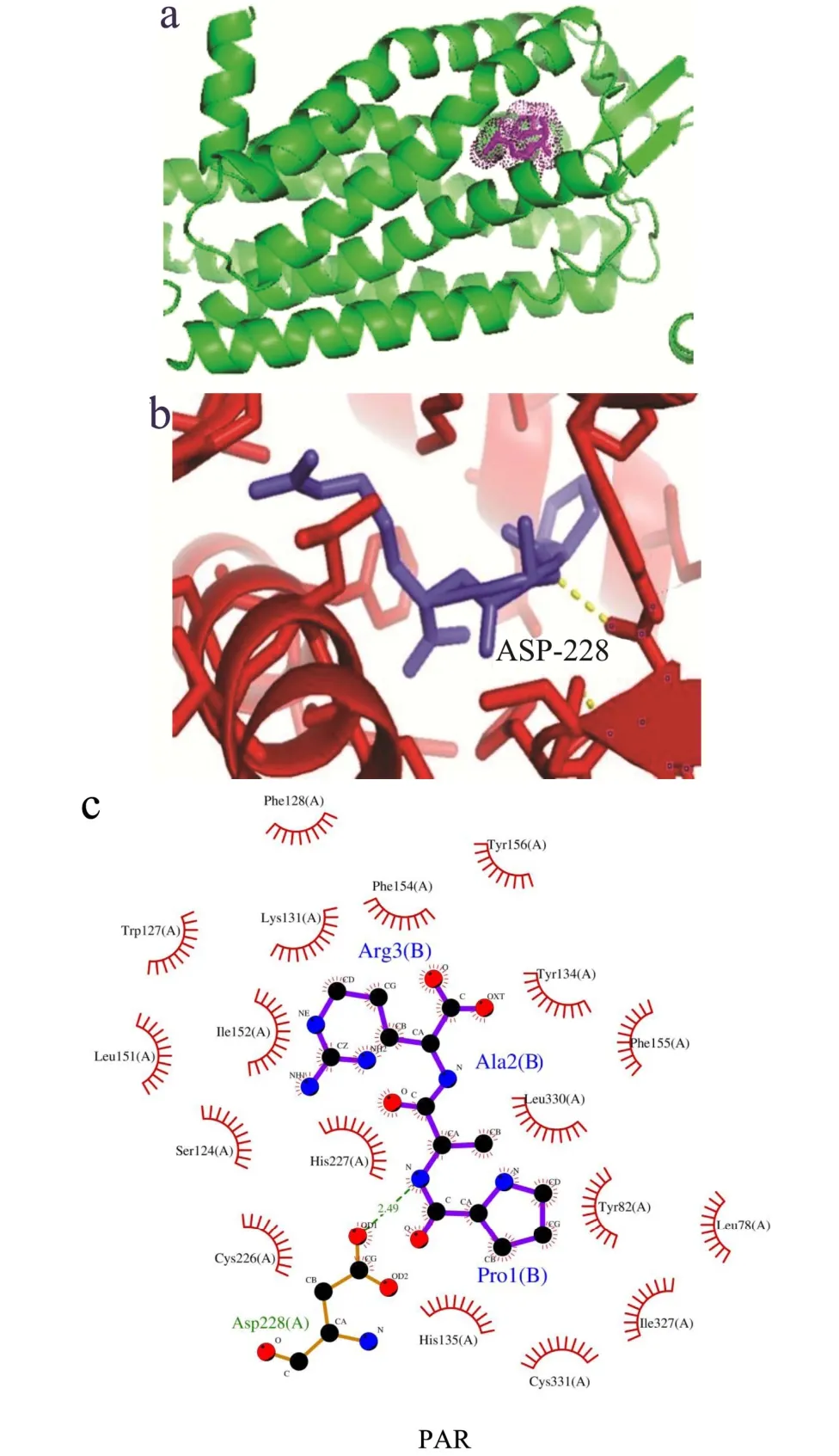
图3 蛋白酶激活受体2(PAR2)与超高亲和肽PAR 结合的图像Fig.3 Image of PAR2 binding to the ultra-high affinity peptide with sequence of PAR
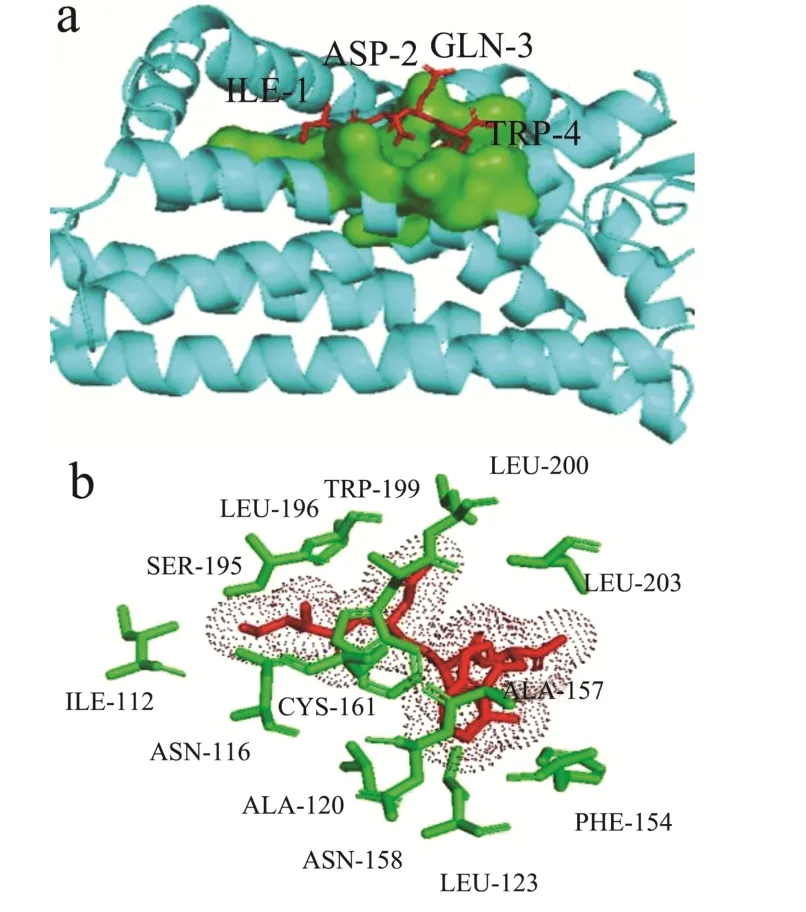
图4 蛋白酶激活受体2(PAR2)与超高亲和肽IDQW 结合的图像Fig.4 Image of PAR2 binding to the ultra-high affinity peptide with sequence of IDQW
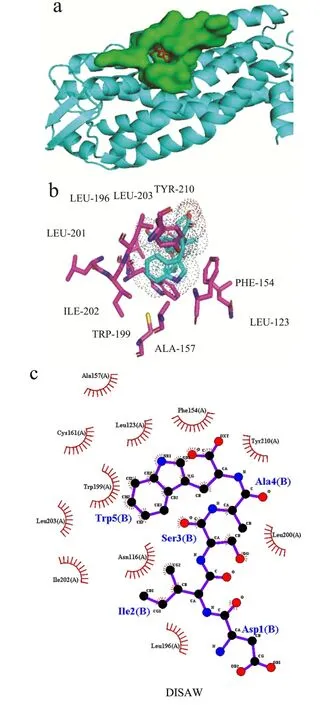
图5 蛋白酶激活受体2(PAR2)与超高亲和肽DISAW 结合的图像Fig.5 Image of PAR2 binding to the ultra-high affinity peptide with sequence of DISAW
分子对接是依据配体和受体之间的“钥匙与锁”的识别关系来模拟小分子配体和大分子受体之间的相互作用力,其从分子层面来解释PAR2 与PAGR、PAR、IDQW 以及DISAW 的作用机制。计算机分子模拟技术作为强大的科学研究工具,可研究分子间相互作用,指导新产品的研发。利用分子对接技术可以清晰地了解活性肽和受体蛋白的对接位点及二者之间存在的相互作用力,从而分辨出肽段的生物活性和有效性[29]。PAR2 具有规范的跨膜螺旋结构,目前已有研究表明PAR2 拮抗剂可以有效通过抑制受体激活和信号传导所需的结构重排、阻断激活配体进入结合位点两种方式发挥作用[8]。
3 结论
食用藻类被认为是绿色健康的食品,其蛋白质含量高于10%,其中螺旋藻蛋白更是符合人体氨基酸比例需求的优质蛋白,且蛋白质含量超过藻干粉质量的65%以上。但我国当下藻类产业链的附加价值不高,综合经济效益逐年下降。基于目前活性多肽的研究路线基本上遵循蛋白质提取→酶解条件优化→活性多肽分离纯化→活性测定→结构鉴定研究途径,不仅耗时费力,而且在反复分离纯化过程中活性肽含量损失严重。因此,生物信息学协同实际实验手段开发活性生物肽的方法正成为当前的研究热点。
本研究主要利用生物数据库以及基于计算机模拟的多肽-蛋白分子对接技术,模拟酶解食用藻类来源的蛋白质制备小分子活性肽,预测分析寡肽片段的生物活性,并开展其水溶性和毒性等理化性质评估,最后开展小分子寡肽与蛋白酶激活受体2(PAR2)晶体结构的分子对接研究,从而筛选与蛋白酶激活受体2 结合能力较高的食用藻蛋白质源寡肽,理论上分析小分子寡肽抑制蛋白酶激活受体2 的潜在活性和作用机制。结果显示,食用藻蛋白源寡肽PAGR、PAR、IDQW、DISAW 与蛋白酶激活受体2 具有较高的结合潜力。研究成果为藻类蛋白的开发利用以及蛋白酶激活受体2(PAR2)抑制剂的发掘提供参考依据,也为未来的体内和体外实验研究提供了理论指导。
