蒜头果ISSR-PCR反应体系建立及验证
2024-01-01张鹏远黄久妍童海珍胡博余潇雷小铃王娟


















摘要:【目的】建立稳定的蒜头果ISSR-PCR反应体系,筛选出适用于蒜头果的多态性引物,为后续蒜头果遗传多样性研究及种质资源保护提供参考依据。【方法】采用单因素试验与L2(5)正交试验相结合的方法,对影响ISSR-PCR反应体系扩增效果的4个主要因素(模板DNA用量、引物浓度、dNTPs浓度、Tag DNA聚合酶)及反应循环数进行优化, 确定最佳反应体系和条件。在此基础上筛选出适用于蒜头果的多态性引物,探究其最佳退火温度,并采集云南省境 内3个野生居群10个蒜头果样品对优化的反应体系进行验证。【结果】蒜头果ISSR-PCR最佳反应体系(20.0uL):模板 DNA 30 ng,ISSR引物0.3 umol/L,dNTPs 0.250 mmol/L,Tag DNA聚合酶1.00 U,ddH,O补足至20.0 uL。ISSR最佳反 应程序为30个循环。利用优化后的反应体系筛选出了8条引物(UBC825、UBC826、UBC827、UBC836、UBC844、UBC861、UBC834和UBC851)的最佳退火温度,分别为50.2、56.5、50.2、56.5、50.2、50.2、50.2和54.0°C,部分引物(如UBC827、UBC836、UBC861等)实测最佳退火温度与软件预测退火温度存在明显差异。采用优化后的ISSR-PCR反应体系及引物对10个蒜头果样品进行ISSR-PCR分子标记检测,结果显示建立的ISSR-PCR反应体系稳定可靠,不同采样点的蒜头果在遗传上相对保守,且10个蒜头果样品的遗传分化系数(G)为0.74,说明其74%的遗传多样性存在于居群间,且基因流(N)为0.18,远小于1.00,说明发生遗传漂变的概率大,易发生遗传多样性降低及居群分化。【结论】通过优化蒜头果ISSR-PCR反应体系,建立可用于蒜头果ISSR-PCR分子标记扩增的稳定反应体系,可用于蒜头果种 质资源保护与利用研究工作。
关键词:蒜头果;ISSR-PCR;体系优化;验证
中图分类号:S792.99
文献标志码:A
文章编号:2095-1191(2024)03-0834-12
Establishment and verification of ISSR–PCR reaction system for Malania oleiferaZHANG Peng-yuan1, HUANG Jiu-yan1, TONG Hai-zhen1, HU Bo1, YU Xiao2,LEI Xiao-ling2, WANG Juan2*
(1College of Life Sciences, Southwest Forestry University, Kunming, Yunnan 650224, China; 2Eco-development Aca-demy, Southwest Forestry University, Kunming, Yunnan 650224, China)
Abstract:[Objective]The purpose of the study was to establish a stable ISSR-PCR reaction system for Malania oleifera and to screen appropriate polymorphic primers for M. oleifera, so as to provide a reference basis for the subsequent study on the genetic diversity of M. oleifera and the conservation of germplasm resources. 【Method]Single factor test combined with L2s (54) orthogonal test was applied to optimize the four main factors (template DNA amount, primer con- centration, dNTPs concentration, Tag DNA polymerase) and the number of reaction cycles affecting the amplification ef- fect of the ISSR-PCR reaction system and to determine the optimal reaction conditions. On this basis, the polymorphic primers suitable for M. oleifera were screened, the optimal annealing temperature was explored, and the optimized reac- tion system was verified by collecting ten M. oleifera samples from three wild populations in Yunnan. 【Result】The opti- mal ISSR-PCR reaction system for M. oleifera (in 20 uL reaction system):30 ng of DNA, 0.3 umol/L of primer, 0.250 mmol/L of dNTPs, 1.00 U of Tag DNA polymerase, and ddH2O replenished to 20.0 uL. The optimal reaction program for ISSR was 30 cycles. The optimal annealing temperature of eight primers (UBC825, UBC826, UBC827, UBC836,UBC844, UBC861, UBC834 and UBC851) was screened out using the optimized reaction system at 50.2, 56.5, 50.2, 56.5, 50.2, 50.2, 50.2 and 54.0 ℃, respectively. The measured optimal annealing temperatures of some primers (such as UBC827, UBC836, UBC861) differed greatly from the predicted annealing temperatures of the software. Ten M. oleifera samples were tested for ISSR-PCR molecular markers using the optimized ISSR-PCR reaction system and primers. The re- sults showed that the established ISSR-PCR reaction system was stable and reliable, and the samples of M. oleifera from different sampling sites were relatively conserved genetically. Additionally, the coefficient of genetic differentiation (Gsa) of the ten M. oleifera samples was 0.74, indicating that 74% of the genetic diversity occurred among the populations, and the gene flow (Nm) was 0.18, which was much less than 1.00, indicating that there was a high probability of genetic drift, which made it prone to the decrease of genetic diversity and the differentiation of the populations. 【Conclusion】By optimizing the ISSR-PCR reaction system of M. oleifera, a stable reaction system for M. oleifera ISSR-PCR molecular markers has been established, which can be used in the research work of M. oleifera germplasm resources conservation and utilization.
Key words: Malania oleifera; ISSR-PCR; system optimization; verification
Foundation items: Yunnan Major Science and Technology Project (202002AA100007) ; Yunnan Biology Highquality Engineering Project (503190106) ; \"Yunling Industrial Technology Leading Talents\" Project of Yunnan Ten Thousand Talents Plan(2018-212)
0 引言
【研究意义】蒜头果(Malania oleifera)是我国特 有的铁青树科(Olacaceae)蒜头果属(Malania)常绿 阔叶树种(梁月芳等,2003;黄晓露等,2022a),也是 国家珍贵用材树种、国家Ⅱ级重点保护植物及云南 省极小种群重点拯救和保护对象(王菖莉等,2019)。 蒜头果是开发利用神经酸的重要潜在资源植物 (Wang et al.,2021),具有极高的开发利用价值,但现 有野生蒜头果分布范围极为狭窄,其自然分布范围 仅限于我国云南省的富宁、广南及广西的百色右江 区、巴马、凤山等地(梁月芳,2001;杨玉玲等,2020), 且蒜头果种群数量小,开发利用难度大。根据2019 年统计数据,我国仅存蒜头果植株总数约22630株 (李洪果等,2019),在现有蒜头果种群数量及生境条 件下,有效地开展蒜头果种质资源保护与良种选育 工作是开发与利用蒜头果种质资源的先决条件。分 子标记技术可在DNA水平上检测物种的遗传多样 性,且具有不受环境因素影响的特点,近年来被广泛 应用于植物品种鉴定(孙淑霞等,2011)、遗传改良 (熊梅,2021)、分子辅助选择育种(郭莹等,2023)及 遗传多样性分析(刘倬宇,2023)等研究中。开发建立稳定的蒜头果ISSR-PCR分子标记体系并进行验 证,从遗传角度探究造成蒜头果濒危的内在原因并 制定相应保护策略对蒜头果种质资源保护与利用具 有重要意义。【前人研究进展】近年来,蒜头果的研究 工作主要集中在形态学及生理生化分析(杨玉玲等, 2020;Wang et al.,2021;黄晓露等,2022b)、微生物资源研究(王毅等,2018;王一凡等,2019;Pan et al., 2021,2022)、神经酸检测、制备及功能研究等领域 (周琴芬等,2016;赖福兵,2018;王四海等,2018;付 一笑等,2020;谷颖卓等,2020)。但蒜头果种群的扩繁工作始终存在人工栽培成活率低的问题(吕仕洪 等,2009,2016),要实现蒜头果种质资源的可持续 开发与利用尚需开展更加全面、深入的研究。近年 来,蒜头果全基因组序列(Xu et al.,2019)、叶绿体基因组序列(Liu et al.,2019)及线粒体基因组序列(Luo et al.,2020)均相继被测定,基因组序列也已完成染色体水平的组装(Yang et al.,2023),为开展蒜 头果遗传多样性及系统发育分析、种质资源保护与 利用等打下良好的前期基础。在蒜头果分子标记研 究方面,仅赖家业等(2006)对蒜头果DNA提取方法及随机扩增多态性DNA标记(RAPD)反应体系进行 优化研究。除此之外,尚未发现其他分子标记反应 体系优化与应用的相关报道。简单序列重复(ISSR)是一种基于PCR技术的分子标记方法,其具有操作 简单、快速及高效的优点(王建波,2002;刘小英等, 2021),被广泛应用于咖啡(闫林,2019)、香蕉(卢家 仕,2020)、白鹤芋(郭甜馨等,2022)、铁线莲(王鑫 等,2022)、野生毛花猕猴桃(王培育等,2023)及紫云 英(张梦等,2023)等不同植物的遗传多样性研究中。ISSR分子标记PCR反应体系不同组分浓度、扩增程 序等因素对扩增效果影响显著。现有研究结果也表明,不同物种的最佳ISSR-PCR反应体系差异显著,如任风鸣等(2014)对影响金钱草ISSR-PCR扩增效果的模板DNA量、引物浓度、dNTP浓度等因素进行优化,研究发现各因素影响程度排序为引物gt;TaqDNA聚合酶gt;模板 DNAgt;Mg2+gt;dNTPs,在峨眉金线 莲(谢孔平等,2022)中也得到了类似的结果。覃创 羿等(2023)研究发现,影响夜来香ISSR-PCR扩增效果的因素排序为模板DNAgt;dNTPsgt;Mg2tgt;Tag DNA 聚合酶gt;引物。因此,针对不同物种建立ISSR-PCR 反应体系是开展遗传多样性研究的关键步骤。为确保所构建反应体系的准确性及可靠性,在建立分子 标记体系时,常会使用少量样品从条带质量、多态性 及鉴定效果等方面对所构建的ISSR-PCR反应体系 进行验证及效果评估。如宁琳等(2017)采用8条 ISSR引物及9份不同种质番石榴样品对建立的 ISSR-PCR反应体系进行验证,并从条带多态性及重 复性方面进行评估;吴望蕊等(2021)利用7条引物 对8个不同产地的朝鲜淫羊藿DNA进行ISSR-PCR 扩增,并对获得的分子标记条带进行质量分析;郭晓 亮等(2022)以建立的ISSR-PCR反应体系成功鉴定 了7个不同来源重齿毛当归的真伪,表明该方法同 样适用于物种的真伪鉴定。【本研究切入点】目前鲜 见有关蒜头果ISSR-PCR反应体系优化的相关研究报道,亦未发现采用ISSR分子标记对蒜头果遗传多 样性开展研究的报道。【拟解决的关键问题】采用单 因素试验和正交试验相结合的方法,对蒜头果ISSR-PCR反应体系中的模板DNA用量、Taq DNA聚合酶用量、dNTPs浓度等多个影响ISSR-PCR扩增效果的因素进行分析,建立与优化蒜头果ISSR-PCR反应体 系并对该体系进行初步验证,为蒜头果的遗传多样 性研究及种质资源保护与利用提供科学参考。
1材料与方法
1.1试验材料
用于蒜头果ISSR-PCR反应体系建立的供试蒜 头果样品采自西南林业大学蒜头果种植圃,反应体 系验证的蒜头果样品分别采集自云南省富宁县者桑 乡(ZSWY)、富宁县板仑乡(MDCWH)及文山州广南县董堡乡(DBX)(图1)。主要试剂:植物基因组DNA提取试剂盒(离心柱型,DP305)购自天根生 化科技(北京)有限公司;EasyTaq DNA Polymerase (AS111-11)、琼脂糖(GS201)及其他试剂均购自北 京全式金生物技术股份有限公司。主要仪器设备:DS-11+微量紫外可见分光度计(美国DeNoVIX公 司)、ABI Veriti PCR仪(美国ABI公司)、NuGenius核 酸成像仪(英国Syngene公司)和超纯水仪(德国ELGA公司)。
1.2试验方法
1.2.1基因组DNA提取及检测
以植物基因组DNA提取试剂盒提取蒜头果叶片基因组DNA,分别采用1.2%琼脂糖凝胶电泳和DS-11+微量紫外可见分光度计检测DNA质量和浓度,仅条带明亮清晰且OD260280介于1.8~1.9的样品可用于后续研究。
1.2.2 ISSR反应体系设计及优化
参考田艳伶等(2015)的研究方案设计单因素试验及正交试验,所 用引物均选自加拿大哥伦比亚大学(University of British Columbia,UBC)公布的UBC系列引物。基 于实验室现有成熟PCR反应体系20uL:1×EasyTaq Buffer,DNA模板50 ng,UBC引物0.2 umol/L,0.200 mmol/L dNTPs,Taq DNA聚合酶1.00 U。扩增程序: 94℃预变性5min;94℃30s,56℃30s,72℃1min, 进行30个循环,72℃10min,4℃保存。根据前期 预试验设计不同模板DNA浓度、引物浓度、dNTPs浓度、Taq DNA聚合酶量及不同循环数,以UBC860引物对上述5个因素开展单因素试验(表1),初步筛 选出适合开展ISSR分子标记的反应体系各组分范围。
根据单因素试验结果,初步确定正交试验L2s (54)的因素和水平,如表2所示。为了保证结果的稳 定性和可重复性,每个试验组设3个重复,依据ISSR一PCR的扩增效率、扩增特异性、条带清晰度、背景质 量等扩增质量因素计分。计分范围1~25分,分数越 高,表示扩增效果越好。经3次重复试验后分别对 每个扩增条件进行计分并取平均值。对所得评分采 用直观量化分析评价法、极差法及方差分析法进行 分析,综合评估出最佳的ISSR-PCR反应体系。
1.2.3引物退火温度预测及实际最佳退火温度确定
采用Tm Calculator软件(https://www.genscript. com.cn/tools/oligo-primer-calculation)对初步筛选的8条UBC引物(表3)进行最佳退火温度预测。以优化后的蒜头果ISSR-PCR反应体系对各引物在8个 退火温度(50.2、50.8、52.0、54.0、56.5、58.5、59.7和60.3℃)下的扩增效果进行检测,确定各引物的最佳 退火温度。
1.2.4优化后的蒜头果ISSR-PCR反应体系验证
为验证所构建及优化的蒜头果ISSR-PCR反应体系,采用优化后的ISSR-PCR反应体系对3个不同地 理居群共10个样品进行扩增并获得0,1矩阵,以 PopGen 32分析计算Nei's基因多样性指数(Nei's gene diversity,H’)、Shannon’s信息多样性指数 (Shannon's information index,I)、多态性百分率(Percentage of polymorphic bands,PPB)、Nei's基因 分化系数(Coefficient of gene differentiation,Gg)和 基因流(Estimate of gene flow from Ga,Nm)等指标,以NTSYS-pc 2.10e计算遗传相似性系数,并以非加权组平均法(UPGMA)构建系统发育进化树。
2结果与分析
2.1蒜头果DNA提取质量检测结果
提取的蒜头果叶片基因组DNA用1.2%琼脂糖 凝胶电泳进行检测,结果如图2所示。所提取的蒜 头果叶片DNA样品条带清晰且无拖尾、弥散等现象,DNA样品OD260280介于1.8~1.9,表明提取的蒜头果叶片DNA质量较好,可用于后续试验。
2.2蒜头果ISSR-PCR反应体系的单因素试验
2.2.1模板DNA用量对蒜头果ISSR-PCR扩增效果的影响
如图3所示,从数量、清晰度、明亮度及 背景来看,6个模板DNA用量扩增获得的条带均无明显差异,表明在蒜头果ISSR-PCR反应体系中,模板DNA用量对扩增反应的影响相对较小。出于节约DNA用量的目的,在20uL反应体系中,选择10~50 ng DNA模板用量进行正交试验。
2.2.2dNTPs浓度对蒜头果ISSR-PCR扩增效果的影响
不同dNTPs浓度对ISSR-PCR扩增效果的影响如图4所示。低浓度dNTPs(0.075~0.100mmol/L)下PCR扩增条带数量较少且清晰度较低,中等浓度范围(0.150~0.300mmol/L)内随dNTPs浓度越高,条带数量和清晰度越高。当dNTPs浓度增加至0.350mmol/L后,条带数量开始减少,清晰度也变差。综合考虑,后续研究选取0.200~0.400mmol/L中的5个 浓度开展正交试验。
2.2.3引物浓度对蒜头果ISSR-PCR扩增效果的影响
由图5可知,不同浓度引物对ISSR-PCR的影响 呈浓度越高,条带数量越多,条带越清晰的趋势。综 合评估条带数量、清晰度等因素后,选取0.3~0.7 umol/L中的5个浓度梯度开展正交试验。
2.2.4 Taq DNA聚合酶用量对蒜头果ISSR-PCR扩增效果的影响
如图6可知,当Taq DNA聚合酶用量为0.10~0.25U时,扩增获得的条带数量少且不清晰。随着TagDNA聚合酶用量的增加,扩增条带数量及亮度达到峰值,但同时抹带情况也显得较为突 出。当Taq DNA聚合酶用量达1.50U后,扩增条带情况差异不明显。综合考虑各因素,选取0.25~2.00U 范围中的5个TaqDNA聚合酶用量进行后续正交试验。
2.2.5反应循环数对ISSR-PCR扩增效果的影响
PCR反应循环数对PCR扩增特异性及扩增效率均明显影响。分别对20、25、30和35个循环进行检测,结果如图7所示。循环数仅为20~25个循环时由于扩增产物太少,不能有效获得扩增条带。随着循环数增加,30个循环时扩增条带最多、最清晰,35个循环时条带数量明显减少。根据上述试验结果,后续正交试验反应循环数均为30个循环。
2.3蒜头果ISSR-PCR反应体系正交试验的直观量化分析结果
采用L23(5)正交试验设计对25个正交组合进行ISSR-PCR及扩增产物电泳检测,结果如图8所示。尽管选取的各因素和水平在单因素试验中的结果多数在可接受范围内,但不同组合的正交试验组合扩增效果存在明显差异。A2B1C2D3、A2B2C3D4和A5B5C4D3组合扩增效果较好,而A1B1C1D1、A2B4C5D1和A3B2C4D1组合扩增效果较差。采用直观法对正交试验结果进行打分并计算出极差。极差反映了各因素对反应体系的影响,R值越大,影响就越显著。由表4可知,在蒜头果ISSR-PCR反应体系中,各因素对扩增效果的影响程度排序为:TaqDNA聚合酶用量gt;dNTPsgt;DNAgt;引物,最大k值对应的水平即为该因素在ISSR-PCR反应体系中的最佳浓度水平,得分最高的组合为A2B1C2D3。
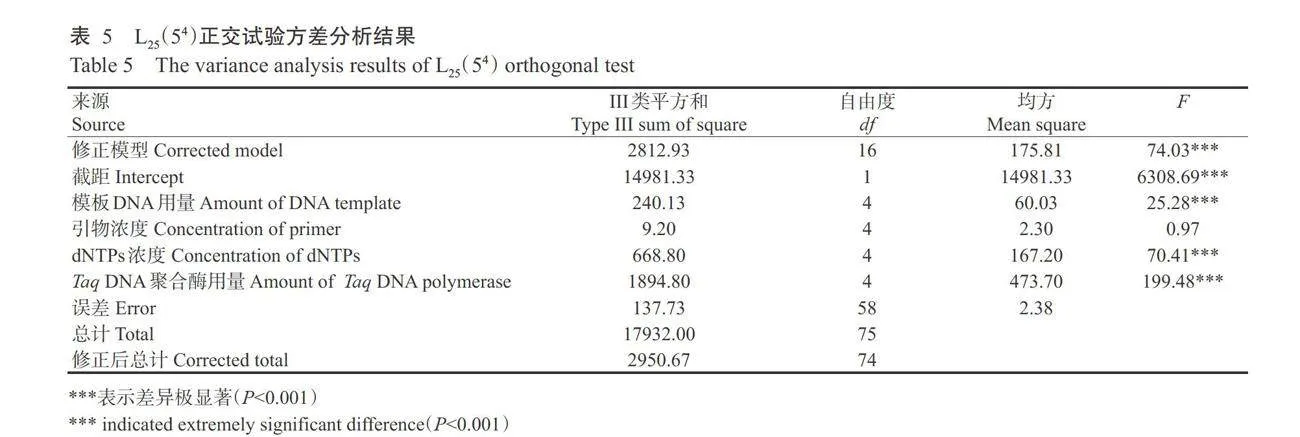
2.4蒜头果ISSR-PCR反应体系正交试验的方差分析及多重比较结果
采用方差分析法和Duncan's多重比对法对正交试验结果进行分析,结果(表5)显示,除引物浓度因素影响不显著(Pgt;0.05,下同)外,其他各因素对蒜头果ISSR-PCR反应影响均为极显著(Plt;0.001)。从F值可知,各因素对扩增效果的影响程度排序为Taq DNA聚合酶用量gt;dNTPs 浓度gt;模板DNA用量gt;引物浓度,与上述极差分析结果(表4)一致。以 Duncan's 法对不同组合进行多重比较可知,A2B1C2D3、A2B2C3D4 和 A5B5C4D3结果最好且无显著性差异。综合考虑直观量化分析、方差分析及琼脂糖凝胶电泳结果,后续研究选取A2B1C2D3组合作为优化后的蒜头果ISSR-PCR反应体系,即在20μL PCR反应体系中加入30ng DNA模板,0.3 umol/L引物,0.250 mmol/L dNTPs,1.00 U TagDNA聚合酶。
2.5退火温度对ISSR-PCR扩增效果的影响
利用优化后的蒜头果ISSR-PCR反应体系对初步筛选出的8条ISSR-PCR分子标记引物进行最佳退火温度检测,结果显示,不同退火温度对ISSR-PCR扩增效果存在明显影响,如UBC827引物扩增结果如图9所示。此外,部分引物实测最佳退火温度与软件预测退火温度存在明显差异,如 UBC861引物预测退火温度与实测最佳退火温度差值为8.9 °℃(表 6)。所测引物的最佳退火温度范围为50.2~56.5°℃。
2.6优化后的蒜头果ISSR-PCR反应体系验证结果
利用优化后的ISSR-PCR反应体系,以UBC827引物对野外采集的3个野生居群共10株蒜头果叶片样品进行ISSR-PCR检测,结果如图10所示。从10个样品中扩增获得清晰、明亮且背景干净的条带,扩增效果理想。
以筛选的5条引物(UBC825、UBC826、UBC827、UBC834、UBC844)对3个野生居群的10个样品进行ISSR-PCR验证,将获得的ISSR-PCR条带转化为0,1矩阵后计算遗传相似性系数和遗传多样性相关指标,并构建系统发育进化树。由遗传多样性相关指标(表7)可知,以5条引物验证10个蒜头 果样品的遗传多样性时,Ga为0.74,表明其74%的遗传多样性存在于居群间,且Nm为0.18,远小于1.00, 说明发生遗传漂变的概率大,易发生遗传多样性降 低及居群分化。基于遗传相似性系数(表8)进行聚 类分析,结果(图11)显示,在遗传相似性系数为0.93 时,10个蒜头果样品被分为两大簇,其中,板仑乡样 品与董堡乡样品亲缘关系较近被划分为一个大簇, 者桑乡样品被划分为另一个大簇;在遗传相似性系 数为0.94时,10个蒜头果样品可被清晰的分为三大 簇,表明建立的蒜头果ISSR-PCR体系可较好地区分 不同自然分布点的蒜头果样品。
3讨论
随着蒜头果应用价值的日益凸显,有效开展蒜头果的种质资源保护与遗传多样性研究已成为当前蒜头果研究工作的重要方向(李洪果等,2019)。建立蒜头果分子标记PCR反应体系将为这些研究工作的开展打下良好的前期基础。ISSR分子标记技术因兼具SSR、RAPD、RFLP及AFLP等分子标记的优点而被广泛应用于植物遗传多样性评价、种质资源鉴定、功能基因挖掘及定位等领域(朱岩芳等,2010)。在ISSR-PCR反应体系中,DNA聚合酶、引物、模板DNA等组分均会影响其反应稳定性和扩增效果,且不同物种的最佳反应体系存在明显差异(赵丽华和李强,2021)。因此,需针对特定物种建立ISSR-PCR最佳反应体系。本研究采用单因素试验与正交试验相结合的方法,确定了各因素对蒜头果ISSR-PCR反应体系扩增效果的影响程度排序为:Taq DNA聚合酶用量gt;dNTPs浓度gt;模板DNA用量gt;引物浓度,与罗湘等(2021)的龙珠果ISSR-PCR反应体系组分影响程度排序基本一致,但与赵春梅等(2012)的甜瓜 ISSR-PCR反应体系组分影响程度排序完全相反。可见,建立各物种特异的ISSR-PCR反应体系对开展其种质资源保护和遗传多样性研究具有重要的意义。
对于常规PCR扩增,常以软件预测或经验公式计算引物退火温度。本研究对筛选出的引物开展退火温度梯度试验,结果显示实测最佳退火温度与软件预测退火温度存在较大差异,如UBC861预测退火温度与实测最佳退火温度相差8.9 °C,且部分引物退火温度间仅相差2.0 C即可明显影响ISSR-PCR扩增条带的数量及清晰度,表明对所用ISSR-PCR引物进行退火温度梯度试验,并根据试验结果确定引物的最佳退火温度,是开展ISSR-PCR分子标记研究不可忽视的关键环节。
本研究采用优化的蒜头果ISSR-PCR反应体系可获得清晰、稳定的ISSR扩增条带,根据有无条带构建0,1矩阵,再计算遗传相关指数,结果发现,与同为珍稀濒危木本植物的毛柄小勾儿茶(B.wibomi)(许凤华等,2006)、白木香(A.sinensis)(何梦玲等,2022)相比,所采集的3个野生居群10个蒜头果样品遗传多样性低且发生遗传漂变可能性大。此外,地理位置相近的云南富宁县板仑乡样品(MDCWH)与云南文山州广南县董河堡乡样品(DBX)样品的遗传距离也较近。但由于本研究所用蒜头果样品和引物数量均较少,后续应加大样本和引物数量,利用建立的ISSR-PCR反应体系对蒜头果种质开展深入的遗传多样性分析。
4结论
通过优化蒜头果ISSR-PCR反应体系,建立可用于蒜头果ISSR-PCR分子标记扩增的稳定反应体系,可用于蒜头果种质资源保护与利用研究工作。
参考文献(References):
付一笑,梁永洁,谷颖卓,张玉红.2020.正交设计优化蒜头果 种子中神经酸提取纯化工艺[J].植物学研究,9(4):380-386. [Fu Y X,Liang Y J,Gu Y Z,Zhang Y H. 2020. Opti-mization of the extraction and purification of nervonic acid from seeds of Malania oleifera using orthogonal design [J]. Botanical Research,9(4):380-386.] doi: 10.12677/br.2020.94047.
谷颖卓,付一笑,张玉红.2020.蒜头果中神经酸的高效液相 色谱法检测[J].植物学研究,9(4):307-313.[GuYZ,Fu Y X, Zhang Y H. 2020. Determination of nervonic acid
in Malania oleifera by high performance liquid chromatography(HPLC) [J]. Botanical Research,9(4):307-313.] doi:10.12677/br.2020.94037.
郭甜馨,Muhammad MA,颜喜玺,李雪,简丽观,彭金彬,陈歆妍,赵锦河,彭俊贤,陈发兴.2022.基于ISSR分子标
记的白鹤芋遗传多样性分析[J].福建农业学报,37(5): 632-640. [Guo T X, Ali M M, Yan X X, Li X, Jian L G, Peng J B, Chen X Y,Zhao J H,Peng J X, Chen F X. 2022. Genetic diversity analysis based on ISSR molecular mar- kers in Spathiphyllum[J]. Fujian Journal of Agricultural Science, 37(5) :632-640.] doi: 10.19303/j.issn.1008-0384.
2022.005.011.
郭晓亮,段媛媛,郭杰,游景茂,王澳炎,朱建波.2022.重齿毛当归ISSR-PCR反应体系的建立与优化[J].分子植物育种,20(8):2682-2688.[Guo XL,Duan YY,Guo J,YouJ M, Wang A Y,Zhu J B. 2022. Establishment and optimiza- tion of ISSR-PCR reaction system of Angelica pubescence
Maxim[J]. Molecular Plant Breeding,20(8): 2682-2688.]doi:10.13271/j.mpb.020.002682.
郭莹,化青春,虎梦霞,王勇,袁俊秀,杨芳萍.2023.分子标记 辅助选择在作物育种中的应用及展望[J].寒旱农业科学,2(9):785-790.[Guo Y,HuaQC,Hu MX,Wang Y, Yuan J X, Yang F P. 2023. Application and prospect of marker-assisted selection in crop breeding[J]. Journal of Cold-Arid Agricultural Science , 2 (9) : 785-790. ] doi : 10.
3969/j.issn.2097-2172.2023.09.001.
何梦玲,林丹,李嘉惠,欧晓华,刘晓莹,高晓霞,马文哲,张宏 意,严寒静.2022.白木香种质资源ISSR标记的遗传多样性分析[J].中草药,53(11):3441-3447.[He ML,Lin D,Li J H,Ou X H,Liu X Y,Gao X X,Ma W Z,Zhang HY, Yan H J. 2022. Evaluation of genetic diversity of Aquilaria sinensis germplasm resources using ISSR markers [J]. Chinese Traditional and Herbal Drugs, 53 (11) : 34413447.] doi:10.7501/j.issn.0253-2670.2022.11.021.
黄晓露,李宝财,骆相华,苏方贵,谢太福,梁文汇.2022a.蒜 头果种子形态特征及脂肪酸组成分析[J].森林与环境学报,42(6):663-672.[Huang XL,Li B C,Luo XH,Su F G, Xie T F, Liang W H. 2022a. Variation analysis of seed morphological characteristics and fatty acid composition of Malania oleifera individuals [J]. Journal of Forest and Environment, 42 (6): 663-672.] doi: 10.13324/j.cnki.jfcf.2022.06.014.
黄晓露,杨日升,骆相华,苏方贵,李宝财,杨卓颖,梁文汇.2022b.广西4个蒜头果家系幼苗光合生理特征分析[J].
广西林业科学,51(4):482-488.[HuangXL,Yang RS, Luo X H, Su F G,Li B C,Yang Z Y, Liang W H. 2022b.
Analysis on photosynthetic physiological characteristics of seedlings of four Malania Oleifera families in Guangxi [J]. Guangxi Forestry Science, 51 (4) :482-488.] doi: 10.
19692/j.issn.1006-1126.20220405.
赖福兵.2018.蒜头果油中高纯神经酸的制备研究[D].南宁:广西大学.[Lai F B.2018.Study on the preparation of nervonic acid in Malania Oleifera's oil [D]. Nanning:
Guangxi University.] doi: 10.7666/d.Y3435329.
赖家业,石海明,刘凯,刘宝,罗荡平,冯家勋,陈放.2006.珍 稀植物蒜头果基因组DNA的提取及RAPD反应体系的 优化[C]//2006年全国博士生学术论坛——中国生物多样性分论坛论文集:304-307.[LaiJY,Shi H M,Liu K, Liu B, Luo D P, Feng J X, Chen F. 2006. Genetic DNA extraction and optimization of RAPD analytic condition of Malania oleifera [C]//National Doctoral Academic Forum- Proceedings of the China Biodiversity Sub-Forum : 304-307.]
李洪果,李武志,邓硕坤,潘启龙,蔡道雄,安宁.2019.蒜头果种质资源及其研究利用现状[J].林业科技通讯,(7):32- 35. [Li H G, Li W Z,Deng S K,Pan Q L, Cai D X,An N.
2019. Research and utilization of Malania oleifera germplasm resources [J]. Forest Science and Technology, (7): 32-35.] doi:10.13456/j.cnki.lykt.2019.01.08.0007.
梁月芳,吴曙光,黎向东.2003.蒜头果的濒危原因研究[J].广西植物,23(5):404-407.[Liang Y F,Wu S G,Li X D.
2003. Study on the endangered causes for Malania Oleifera [J]. Guihaia, 23 (5) : 404-407.] doi: 10.3969/j. issn.1000-3142.2003.05.004.
梁月芳.2001.蒜头果的濒危原因研究及挽救对策[D].南宁:广西大学.[Liang Y F.2001.Study on the endangering causes and the saving strategies for Malania oleifera [D].
Nanning:Guangxi University.] doi: 10.7666/d.Y373774.
刘小英,黄飞龙,刘国强,张游南.2021.枇杷ISSR分子标记的研究进展[J].中国农学通报,37(21):106-110.[LiuX
Y,Huang F L,Liu G Q,Zhang Y N. 2021. ISSR molecular markers in loquat: Research progress [J]. Chinese Agricultural Science Bulletin, 37 (21) : 106-110.] doi: 10.11924/j.issn.1000-6850.casb2020-0644.
刘倬宇,赵永秀,宁琳懿睿,张凌硕,孙瑞芬,刘锦川,牛素清.
2023.基于ISSR标记分析豌豆农家品种遗传多样性[J].
分子植物育种,21(20):6760-6766.[LiuZY,ZhaoY X, Ning L Y R,Zhang L S,Sun R F, Liu J C, Niu S Q. 2023. Genetic diversity analysis of pea germplasm based on ISSR markers[J]. Molecular Plant Breeding,21 (20) :6760-6766.] doi:10.13271/j.mpb.021.006760.
卢家仕,韦绍龙,黄素梅,韦弟,李朝生,龙盛风,覃柳燕.
2020.基于ISSR分子标记的广西香蕉种质资源遗传多样性分析[J].南方农业学报,51(4):775-780.[LuJS,Wei S L,Huang S M,Wei D,Li C S,Long S F,Qin L Y. 2020. Genetic diversity analysis of Guangxi banana germplasm resources based on ISSR molecular marker technology[J].
Journal of Southern Agriculture, 51 (4) : 775-780.] doi: 10.3969/j.issn.2095-1191.2020.04.006.
罗湘,朱玉静,彭滢枫,杨丹,谭勇,赵立春.2021.西番莲科龙 珠果ISSR-PCR体系的建立和优化[J].分子植物育种,19(5): 1598-1605. [Luo X,Zhu Y J,Peng Y F, Yang D, Tan Y, Zhao L C. 2021. Establishment and optimization of ISSR-PCR system for Passiflora foetida of Passionaceae [J]. Molecular Plant Breeding, 19(5): 1598-1605.] doi: 10.
13271/j.mpb.019.001598.
吕仕洪,李先琨,陆树华,向悟生,覃家科,王晓英.2009.桂西 南岩溶地区珍稀濒危树种育苗与造林初报[J].广西植物,29(2):222-226.[Lü S H,Li XK,Lu S H,Xiang W S, Qin J K, Wang X Y. 2009. Preliminary study on seedling and afforestation of rare and endangered trees in karst region of southwest Guangxi[J]. Guihaia,29(2):222-226.]
doi:10.3969/j.issn.1000-3142.2009.02.017.
吕仕洪,韦春强,黄甫昭,何运林,曾丹娟,李先琨,邓振海.
2016.珍稀树种蒜头果种实性状及其在石漠化山区的适 应性[J].生态学杂志,35(1):57-62.[Lü S H,Wei C Q, Huang F Z,He Y L, Zeng D J, Li X K, Deng Z H. 2016. Fruit and seed traits and adaptability to rocky desertifica- tion mountain of rare tree species Malania oleifera[J]. Chi- nese Journal of Ecology, 35 (1) : 57-62.] doi: 10.13292/j.1000-4890.201601.008.
宁琳,陈豪军,黄显雅,潘祖建,严霖,甘卫堂,唐玉娟.2017.番石榴最适ISSR-PCR反应体系的建立与验证[J].中国 南方果树,46(4):57-59.[Ning L,Chen HJ,Huang XY, Pan Z J, Yan L, Gan W T, Tang Y J. 2017. Establishment and verification of optimized ISSR-PCR reaction system for Psidium guajava [J]. South China Fruits, 46 (4) : 57- 59.] doi:10.13938/j.issn.1007-1431.20160449.
任风鸣,胡开治,刘燕琴,焦雁翔,刘杰,罗敏,全健.2014.传 统中药金钱草ISSR-PCR反应体系的正交优化研究[J].中国中药杂志,39(12):2233-2238.[Ren F M,Hu KZ, Liu Y Q, Jiao Y X, Liu J, Luo M, Quan J. 2014. Optimiza- tion for ISSR-PCR system of traditional Chinese medicine Lysimachia christinae by orthogonal design[J]. China Jour- nal of Chinese Materia Medica, 39 (12) : 2233-2238.] doi:
10.4268/cjcmm20141217.
孙淑霞,李靖,陈栋,谢红江,涂美艳,江国良.2011.ISSR分子标记技术在桃品种鉴定中的应用[J].中国农学通报,27(4): 173-177. [Sun S X, Li J, Chen D, Xie H J, Tu M Y, Jiang G L. 2011. Molecular identification of peach germplasm by ISSR markers [J]. Chinese Agricultural Science Bulletin, 27 (4):173-177.] doi: 10.11924/j.issn.1000-6850.2010-2097.
覃创羿,王晓南,鲁宗翰,卢万义,韦杰.2023.夜来香ISSR-PCR反应体系的建立及引物筛选[J/OL].分子植物育种.https:/kns.cnki.net/kems/detail//46.1068.s.20230215.
1711.012.html.[Qin C Y,Wang X N,Lu Z H,Lu W Y,W J. 2023. Establishment and optimization of ISSR-PCR reaction system on Telosma cordata[J/OL]. Molecular Plant
Breeding. https://kns. cnki. net/kems/detail//461068.s.20230215.1711.012.html.]
田艳伶,李志辉,杨模华,张斌.2015.钩栗ISSR-PCR反应体系的建立与优化[J].中南林业科技大学学报,35(2):32- 37. [Tian Y L, Li Z H, Yang M H, Zhang B. 2015. Estab- lishment and optimization of Castanopsis tibetana Hance ISSR-PCR reaction system[J]. Journal of Central South University of Forestry and Technology,35(2):32-37.] doi:
10.14067/j.cnki.1673-923x.2015.02.007.
王菖莉,陈福,徐德兵,杨华斌,贾代顺.2019.广南县发展蒜头果的意义、优势及建议[J].现代园艺,(7):42-44.[Wang C L, Chen F, Xu D B, Yang H B, Jia D S. 2019. Signifi- cance, advantages and suggestions for the development of Malania oleifera in Guangnan County [J]. Contemporary Horticulture, (7): 42-44.] doi: 10.3969/j.issn.1006-4958.
2019.07.019.
王建波.2002.ISSR分子标记及其在植物遗传学研究中的应用[J].遗传,24(5):613-616.[WangJB.2002.ISSR mar- kers and their applications in plant genetics [J]. Hereditas (Beijing),24(5):613-616.] doi: 10.3321/j.issn:0253-9772.2002.05.022.
王培育,叶炜,冯新,赖瑞联,颜沛沛,陈义挺.2023.三十份野 生毛花猕猴桃种质资源的ISSR遗传多样性分析[J].热带农业科学,43(6):29-33.[Wang P Y,Ye W,Feng X, Lai R L, Yan P P, Chen Y T. 2023. ISSR genetic diversity analysis of 30 wild Actinidia eriantha Benth. germplasm resources [J]. Chinese Journal of Tropical Agriculture, 43(6):29-33.] doi:10.12008/j.issn.1009-2196.2023.06.006.
王四海,施蕊,陈剑,杨宇明,王娟.2018.蒜头果油对小鼠的 急性毒性试验研究[J].西部林业科学,47(5):94-98.[Wang S H, Shi R, Chen J, Yang Y M, Wang J. 2018. Acute toxicity study of Malania oleifera oil to mice [J].Journal of West China Forestry Science, 47 (5) : 94-98.]doi: 10.16473/j.cnki.xblykx1972.2018.05.016.
王鑫,郑妍,刘志杰,张存石,刘冬云.2022.铁线莲属植物 ISSR分子标记体系优化及指纹图谱的构建[J].林业与 生态科学,37(1):71-79.[Wang X,Zheng Y,Liu Z J, Zhang C S, Liu D Y. 2022. Optimization of Clematis ISSR molecular marker system and fingerprint construction [J].
Forestry and Ecological Sciences, 37 (1) : 71-79.] doi: 10.13320/j.cnki.hjfor.2022.0010.
王一凡,粟桂娇,李伟光,赖芳,于唱,黄秋容,孙泽宇.2019.
蒜头果内生真菌发酵产微生物油脂的脂肪酸分析[J].食 品科技,44(6):8-12.[Wang YF,Li GJ,Li W G,Lai F, Yu C,Huang Q R,Sun Z Y. 2019. Fatty acids composition analysis of microbial oils produced by Malania oleifera endophytic fungi[J]. Food Science and Technology,44(6): 8-12. ] doi:10.13684/j.cnki.spkj.2019.06.002.
王毅,王娟,王四海,李云琴,原晓龙,陈中华.2018.蒜头果的 内生真菌多样性分析[J].基因组学与应用生物学,37
(9):3859-3866. [Wang Y,Wang J, Wang S H, Li Y Q, Yuan X L, Chen Z H. 2018. Diversity of endophytic fungi of Malania oleifera Chun et Lee [J]. Genomics and Applied Biology, 37 (9) : 3859-3866.] doi: 10.13417/j. gab.037.003859.
吴望蕊,王英哲,国坤,容路生,孟芳芳,孙云龙,肖井雷,姜大 成.2023.朝鲜淫羊藿ISSR-PCR反应体系优化及引物筛选[J].分子植物育种,21(7):2318-2324.[Wu W R,Wang Y Z,Guo K,Rong L S,Meng F F, Sun Y L, Xiao J L, Jiang D C. 2023. Optimization of ISSR-PCR reaction sys- tem and primer selection of Epimedium koreanum Nakai [J]. Molecular Plant Breeding, 21 (7) : 2318-2324. ] doi: 10.
13271/j.mpb.021.002318.
谢孔平,杨适,毛冀,鲁松.2022.峨眉金线莲ISSR反应体系的建立与优化[J].四川林业科技,43(4):76-82.[Xie KP, Yang S, Mao J, Lu S. 2022. Establishment and optimization of ISSR-PCR reaction system of Anoectochilus emeiensis[J]. Journal of Sichuan Forestry Science and Tech-nology,43(4):76-82.] doi:10.12172/202110110001.
熊梅.2021.甘蓝型油菜Y522R等核心骨干亲本根肿病抗性的遗传改良[D].武汉:华中农业大学.[XiongM.2021.
Genetic improvement of the clubroot resistance of the core parents Y522R et al of Brassica napuys L. [D]. Wuhan: Huazhong Agricultural University.]
许凤华,康明,黄宏文,江明喜.2006.濒危植物毛柄小勾儿茶 片断化居群的遗传多样性[J].植物生态学报,30(1): 157-164. [Xu F H, Kang M, Huang H W, Jiang M X.
2006. Genetic diversity in fragmented populations of Berchemiella wilsonii var. pubipetiolata, an endangered plant endemic to eastern China [J]. Journal of Plant Ecology,30(1): 157-164. ] doi:10.17521/cjpe.2006.0022.
闫林,黄丽芳,王晓阳,孙燕,林兴军,董云萍,龙宇宙.2019.
咖啡种质资源遗传多样性的ISSR分析[J].南方农业学报,50(3):491-499.[Yan L,HuangLF,Wang XY,Sun Y, Lin X J,Dong Y P, Long Y Z. 2019. Genetic diversity of coffee gemeplasms by ISSR analysis[J]. Journal of Sou- thern Agriculture , 50 (3) : 491-499. ] doi : 10.3969/j.issn.2095-1191.2019.03.08.
杨玉玲,王序英,魏波,赵大伟.2020.蒜头果种子外观质量分 级标准初步研究[J].广西林业科学,49(4):524-530.[Yang Y L, Wang X Y, Wei B, Zhao D W. 2020. Preliminary study on grading standard of appearance quality of Malania oleifera seeds[J]. Guangxi Forestry Science, 49
(4):524-530.] doi:10.3969/j.issn.1006-1126.2020.04.008.
张梦,史鹏飞,刘春增,李本银,丁丽,张琳,郑春风,张成兰, 何春梅,施海帆.2023.基于ISSR分子标记的紫云英种质资源遗传多样性及结构分析[J].草地学报,31(1):50-60. [Zhang M,Shi P F,Liu C Z,Li B Y,Ding L,Zhang L, Zheng C F, Zhang C L, He C M, Shi H F. 2023. Genetic diversity and structure analysis of germplasm resources based on ISSR markers in Astragalus sinicus L.[J]. Acta Agrestia Sinica, 31 (1) : 50-60.] doi: 10.11733/j.issn. 1007-0435.2023.01.006.
赵春梅,金荣荣,刘英,汪磊.2012.甜瓜ISSR-PCR反应体系的正交优化研究[J].中国农学通报,28(4):146-150.
[Zhao C M, Jin R R, Liu Y, Wang L. 2012. Optimization for ISSR-PCR system of Cucumis melo L. using orthogonal design [J]. Chinese Agricultural Science Bulletin, 28(4):146-150.] doi: 10.3969/j.issn.1000-6850.2012.04.027
赵丽华,李强.2021.杜鹃属ISSR-PCR反应体系优化[J].现 代园艺,44(13):14-15.[Zhao LH,LiQ.2021.Optimiza- tion of ISSR-PCR reaction system for Rhododendron plants [J]. Contemporary Horticulture, 44 (13) : 14-15.] doi: 10.
3969/j.issn.1006-4958.2021.13.006.
周琴芬,李杰,谈满良,栾连军.2016.响应面实验优化蒜头果 油中神经酸甘油酯皂化工艺[J].中国油脂,41(8):45- 48. [Zhou Q F, Li J, Tan M L, Luan L J. 2016. Optimiza- tion of saponification of nervonic acid glyceride from Malania oleifera oil by response surface methodology[J].
China Oil , 41 (8) : 45-48.] doi: 10.3969/j.issn.1003-7969.2016.08.011
朱岩芳,祝水金,李永平,马文广,郑昀晔,胡晋.2010.ISSR分子标记技术在植物种质资源研究中的应用[J].种子,29(2):55-59. [Zhu Y F,Zhu S J,Li Y P,Ma W G,Zheng Y Y, Hu J. 2010. Application of ISSR molecular marker in plant germplasm research [J]. Seed, 29 (2) : 55-59.] doi:
10.3969/j.issn.1001-4705.2010.02.014.
Liu S S, Hu Y H, Maghuly F, Porth I M, Mao J F. 2019. The complete chloroplast genome sequence annotation for Malania oleifera, a critically endangered and important bioresource tree[J]. Conservation Genetics Resources,11:271-274. doi:10.1007/s12686-018-1005-4.
Luo H, Xu J, Jiao S Q, Zhang R G, Mao J F. 2020. The complete mitochondrial genome of an endangered tree: Malania oleifera [J]. Mitochondrial DNA Part B, 5 (4) : 3829-
3830. doi:10.1080/23802359.2020.1841583.
Pan Y,Lei X,Wang P,Yang G,Gu Y,Wang M,Wang J. 2021. First report of leaf spot and stem canker on Malania oleifera caused by Neofusicoccum parvum in China[J]. Journal of Plant Pathology, 104: 413. doi: 10.1007/s42161-021-00978-Z
Pan Y,Wang P,Chen W,Yang G C,Gu Y J,Wang J W,Wang
J. 2022. First report of leaf spot on Malania oleifera caused by Botryosphaeria fabicerciana in China[J]. Plant Disease,106(6):1759. doi:10.1094/PDIS-09-21-2038-PDN.
Wang S H, Chen J, Yang W, Hua M, Ma Y P. 2021. Fruiting character variability in wild individuals of Malania oleifera, a highly valued endemic species [J]. Scientific Re-ports, 11 (1) : 23605-23613. doi: 10.1038/s41598-021-03080-7.
Xu C Q,Liu H,Zhou S S,Zhang D X,Zhao W,Wang S,Chen
F,Sun Y Q, Nie S, Jia K H,Jiao S Q,Zhang R G, Yun Q
Z, Guan W, Wang X, Gao Q, Bennetzen J L,Maghuly F, Porth I, Van de Peer Y, Wang X R, Ma Y, Mao J F. 2019. Genome sequence of Malania oleifera, a tree with great value for nervonic acid production[J]. GigaScience,8(2):1-14. doi:10.1093/gigascience/giy164.
Yang T Q,Zhang R G,Tian X L,Yao G,Shen Y T,Wang S H, Mao J F, Li G Y, Liu A Z, Sun W B, Ma Y P. 2023. The chromosome-level genome assembly and genes involved in biosynthesis of nervonic acid of Malania oleifera [J].
Scientific Data, 10(1) : 298-305. doi: 10.1038/s41597-023-02218-8.
(责任编辑陈燕)
